陆地棉GhCSN6A基因克隆及低磷胁迫下的表达
卿桂霞,耿翡翡,梁颖颖,周俊江,张富厚,孟超敏*
(1 河南科技大学 农学院,河南洛阳 471000;2 洛阳市作物遗传改良与种质创新重点实验室,河南洛阳 471000)
COP9信号复合体(constitutively photomorphogennic signalosome,CSN)是一种进化上保守的多亚基蛋白复合体,参与动植物的许多生理过程,包括蛋白质降解、DNA损伤反应和信号转导[1]。主要在泛素蛋白酶体通路(ubiquitin proteasome system,UPS)中发挥着调节作用[2],UPS是蛋白质进行高效特异性降解的主要途径,其过程为目标蛋白首先被泛素三酶级联反应,然后对靶蛋白进行泛素化修饰,最后由26S蛋白酶体催化将其降解[3-4]。在高等真核生物中典型的CSN蛋白由8个不同亚基构成,它们被命名为CSN1-CSN8,通常包含2个保守结构域,分别是MPN和PCI[5]。其中CSN5和CSN6包含MPN结构域,该结构域是由3条α螺旋和9条β线缠绕成β片层组成,具有识别信号因子,激活下游活动,调控生物学等主要功能[6-7]。COP9信号复合体亚基已从多种生物中克隆并分析,而且发现CSN在植物抵御逆境中具有重要作用。通过研究部分功能丧失突变体VTC1-1的CSN5B双突变拟南芥,发现CSN5B通过26S蛋白酶体途径降解GDP-甘露糖焦磷酸化酶(VTC1),使植物抵抗非生物胁迫的关键因子抗坏血酸(AsA)含量升高,从而增强植物对环境胁迫的耐受性[8];利用基因芯片技术筛选出缺铁条件下的基因,并对水稻CSN6基因进行缺铁胁迫表达模式分析,发现水稻CSN6基因能够应答缺铁胁迫[9]。
棉花是中国当今最主要发展的经济作物之一,其种植产业在整个世界上始终处于领先地位,创造的经济效益在中国国民人均生产要素总值中具有重要意义[10-11]。但是由于棉花吸收利用的磷素在土壤中主要以磷酸根的形式存在,土壤中的磷酸根极其容易被金属阳离子和矿物质吸附固定或转化成有机磷而不能成为有效磷,因此土壤有效磷的供应情况和棉花吸收磷素的能力是使棉花免遭受低磷胁迫危害,降低低磷胁迫给棉花生长、纤维品质和产量造成影响的关键因素[12-13]。在低磷环境下,植物经过长期的进化过程中会形成一系列的应答机制来调节其生理生化反应来应对低磷胁迫[14-16],从而减少缺磷对自身的伤害。因此,可以通过克隆棉花应答低磷胁迫过程中的关键基因,解析其胁迫响应及作用机制,从而来提高棉花对磷的利用效率。
本研究在前期利用基因芯片技术筛选出适磷与低磷胁迫下的高差异表达序列ES816317和陆地棉基因组数据库的基础上,以磷高效棉花品种陆地棉‘新陆早19’(Gossypiumhirsutum‘Xinluzao 19’)为材料进行基因克隆,并对该基因在不同组织及根系低磷胁迫下的相对表达水平进行分析,为深入研究棉花GhCSN6A的生物学功能奠定基础,以期为棉花磷高效基因工程育种提供基因资源。
1 材料和方法
1.1 试验材料及处理
选取自交留种中籽粒饱满的‘新陆早19’种子用 70%酒精杀菌 30 min,冲洗干净后泡种 24 h 至棉花露白,分别播种于沙土中和河南科技大学农学院试验田。沙土中的棉花在28 ℃恒温培养箱培养至四叶期左右用以植株总RNA的提取。在试验田棉花花铃期取根、茎、叶、花等4个组织迅速放入液氮中备用。
将棉花种子播种于含干净湿润细沙的塑料盆内,28 ℃恒温培养箱(光照14 h、黑暗10 h)培养至棉苗三叶期,选择长势良好且一致的幼苗移至水培盆中,用 1/2 Hoagland 营养液培养1周后分为2个处理水平,即适磷处理(SP,1.0 mmol/L)和低磷处理(LP,0.01 mmol/L),磷源采用KH2PO4,为保证K+的浓度一致,以1.0 mmol/L为标准,低磷营养液中以KCl补齐K+,其他营养成分含有2 mmol/L Ca(NO3)2·4H2O、2.5 mmol/L KNO3、0.5 mmol/L NH4NO3、1.4 mmol/L MgSO4·7H2O、1 μmol/L ZnSO4·7H2O、1 μmol/L MnSO4·H2O、0.1 μmol/L CuSO4·5H2O、0.01 mmol/L H3BO3、0.005 μmol/L (NH4)6Mo7O24·4H2O、0.1 mmol/L EDTA-FeNa[17]。分别处理0、4、12、24和72 h后,选取3株生长一致的棉花植株,并混合取其根部组织,然后迅速置于液氮中,-80 ℃保存备用。每日调节营养液 pH,使其保持在 6.5 左右,每3 d更换1次营养液。
1.2 试验方法
1.2.1 低磷胁迫差异表达序列延伸利用基因芯片技术筛选得到‘新陆早19’在低磷和适磷2种不同处理下差异表达序列ES816317,以ES816317为探针在NCBI网站的EST数据库中检索其相似序列,并利用DNASTAR的Seqman将所得到的相似序列(覆盖率>50%,相似度>85%)进行拼接得到其重叠群,后续以所得拼接序列为探针继续检索与拼接,直至没有新的相似序列出现,其所得才为结果序列,利用ORFfinder在线平台查找开放阅读框,并对目的基因进行克隆与分析。
1.2.2 陆地棉基因组DNA的提取取液氮速冻‘新陆早19’完整植株,迅速将其研磨成粉末转移至离心管,然后利用CTAB法提取陆地棉‘新陆早19’的基因组DNA,并以1%的琼脂糖凝胶对其进行检测,最后-20 ℃保存备用。
1.2.3 总RNA提取及cDNA合成按照天根RNAprep Pure多糖多酚植物总RNA提取试剂盒说明书提取棉株总RNA,然后利用超微量分光光度计测定RNA浓度,并用1%琼脂糖凝胶电泳检测其完整性。若RNA的质量符合要求,则根据HiScript®Ⅱ1st Strand cDNA Synthesis Kit cDNA第一链合成试剂盒说明书将所提取的总RNA反转录合成cDNA第一链,用于后续实验。
1.2.4 基因克隆与测序根据拼接所得到的GhCSN6A基因序列信息,利用软件Primer 5.0设计其ORF上、下游引物(表1),并分别以陆地棉‘新陆早19’的DNA及cDNA为扩增模板,进行目的基因的扩增,再以1%琼脂糖凝胶电泳进行PCR扩增检测。将目的条带的PCR产物与TOPO-T载体进行连接,转化、菌液PCR检测,然后目标条带的菌液送至上海生工生物有限公司测序,并保留一些菌液。
1.2.5 生物信息学分析采用NCBI数据库中的blastn和blastp分析GhCSN6A基因的核苷酸序列及编码氨基酸序列的同源性,并使用DNAMAN对GhCSN6A与同源蛋白序列多重比对,使用在线工具ProtParam预测GhCSN6A蛋白的各种理化性质[18];利用SOPMA对其二级结构进行预测。利用SWISS-MODEL和CD-Search分别对GhCSN6A蛋白进行三维结构同源建模和功能结构域的分析。使用SingalP 3.0和TMHMM 2.0分别预测其信号肽和跨膜螺旋结构域;其磷化位点和亚细胞定位分别由NetPhos3.1 Server和Plant-mPLoc进行预测。利用MEGA 5.0的邻接法(neighbor-joining method)构建系统发育进化树。
1.2.6GhCSN6A表达的qRT-PCR基于GhCSN6A基因全长序列及保守域的分析,使用 Primer5.0设计荧光定量 PCR 引物(表1),GhActin作为内参基因。反应体系按照SYBR®Green Premix Pro Taq HS qPCR Kit试剂盒说明配制,使用荧光定量PCR仪型号为CFX96,采用2-ΔΔCt算法计算目的基因相对表达量,利用Origin 9.0对数据进行统计分析并作图。
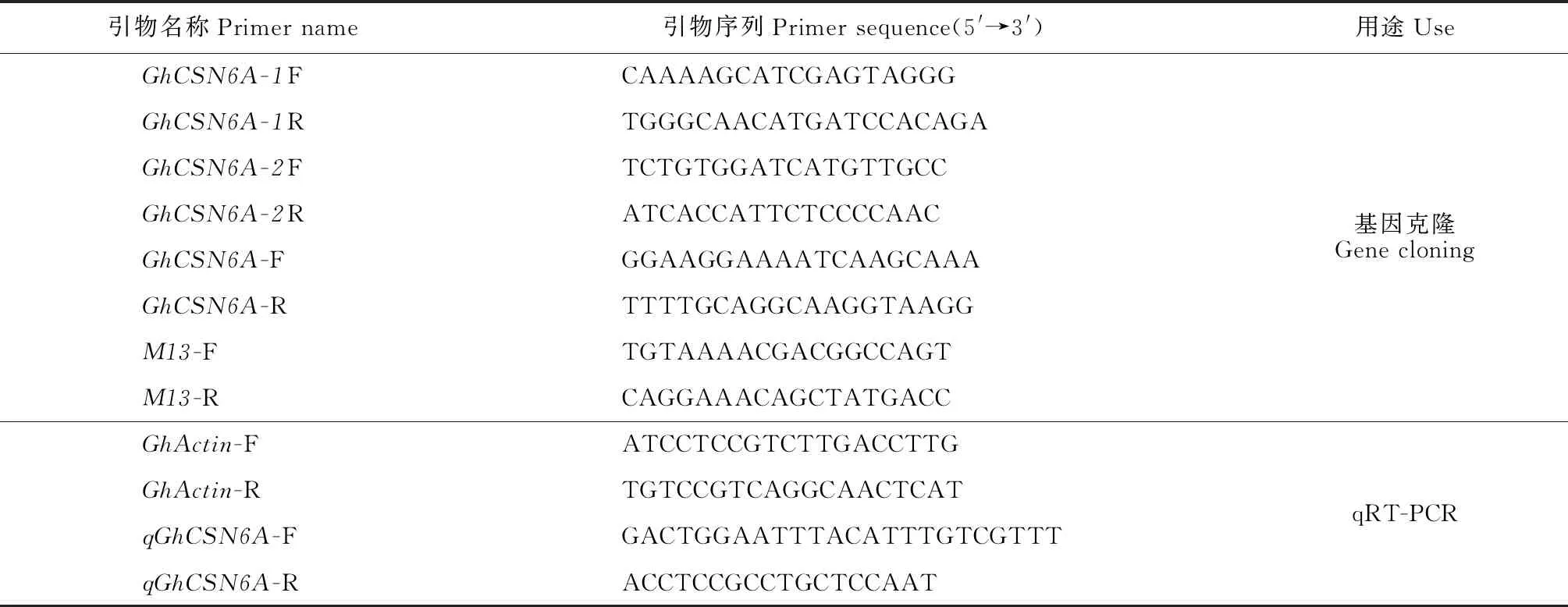
表1 实验所用引物Table 1 Application of primers in the experiment
2 结果与分析
2.1 低磷胁迫差异表达序列延伸
通过BLAST检索陆地棉EST数据库,获得ES816317相似的陆地棉ESTs,采用电子延伸的方法进行最大程度的序列延伸。然后利用NCBI上的ORF finder进行查找,发现拼接得到的序列全长为1 814 bp,开放阅读框为88~1 035共948 bp,编码315 个氨基酸。通过blastn及blastp检索全长序列,将该基因命名为GhCSN6A。
2.2 基因克隆与序列分析
以‘新陆早19’的基因组DNA为模板,GhCSN6A-1F和GhCSN6A-1R及GhCSN6A-2F和GhCSN6A-2R两对引物进行PCR扩增,得到的扩增产物如图1的第1、2条带,分别约为1 650 bp和1 780 bp,以‘新陆早19’总RNA反转得到的cDNA为模板,GhCSN6A-F和GhCSN6A-R为引物进行扩增,得到的产物如图1的第3条带,约1 190 bp。利用在线工具GSDS对分别以DNA和cDNA为模板扩增得到的已拼接的序列进行分析,可发现该基因含有9个外显子和8个内含子(图2)。

图2 GhCSN6A基因结构分析Fig.2 Analysis of GhCSN6A gene structure
2.3 GhCSN6A蛋白理化性质及结构分析
陆地棉GhCSN6A蛋白分子式为C1582H2491N435O471S11,相对分子量为35.49 kD,含32个酸性氨基酸(Asp+Glu)、30个碱性氨基酸(Arg+Lys),理论等电点为6.70,疏水性总平均值为-0.217,不稳定系数为46.49,属于亲水性不稳定蛋白。蛋白质二级结构预测表明,其二级结构由α-螺旋(alpha helix,44.13%)、无规则卷曲 (random coil,38.10%)、延伸链(extended strand,15.87%)和β-折叠(Beta turn,1.90%)组成。GhCSN6A蛋白三维结构同源建模显示,该蛋白包含COP9 信号体复合亚基 6,即COP9 信号体的晶体结构。利用CD-Search对GhCSN6A蛋白功能结构域进行分析,发现GhCSN6A蛋白属于MPN或MOV34超家族,具有MPN_CSN6结构域,是 COP9 信号体的 8 个亚基之一,与三级结构得到的结果一致。TMHMM 2.0预测结果显示,GhCSN6A蛋白无跨膜螺旋结构和信号肽。SignalP 3.0信号肽预测结果与TMHMM预测结果相同,即该蛋白无信号肽。NetPhos 3.1 Server预测结果显示,GhCSN6A蛋白含有36个能被磷酸化的位点(超过阈值线),其中包括Ser位点20个、Thr位点11个和Tyr位点5个,主要被unsp、PKC和cdc2等蛋白激酶磷酸化。GhCSN6A蛋白亚细胞定位预测,该蛋白定位于细胞核。

M.DL2000;1-2.DNA;3.cDNA图1 GhCSN6A的PCR扩增Fig.1 PCR amplification of GhCSN6A
2.4 GhCSN6A蛋白的同源比对及系统进化树分析
以GhCSN6A蛋白与拟南芥的10个AtCSN蛋白构建系统发育进化树,结果(图3)显示,GhCSN6A蛋白与拟南芥CSN6家族聚为一类,其中,与AtCSN6A蛋白相似性最高。从NCBI数据库下载CSN6A的同源蛋白氨基酸序列,并利用DNAMAN 对陆地棉与拟南芥(Arabidopsisthaliana)、烟草(Nicotianaattenuata)、木槿(Hibiscussyriacus)的CSN6A蛋白序列进行多重序列比对,结果(图4)显示,蛋白序列的一致性达到90.93%,由此可得MPN_CSN6结构域有一定的保守性。用NCBI数据库中BLASTp对GhCSN6A基因进行同源比对,结果显示该基因与木槿CSN6A基因的相似性最高,达95.87%,与哥伦比亚锦葵(Herraniaumbratica)CSN6A基因和榴莲(Duriozibethinus)CSN6A基因的相似性均达93%。利用MEGA5对GhCSN6A和其他植物的CSN6A蛋白进行构建系统发育进化树,结果(图5)表明,不同物种的GhCSN6A蛋白分为2大分支,其中陆地棉GhCSN6A与在同一分支上的澳洲棉CSN6A、木槿CSN6A、榴莲CSN6A和哥伦比亚锦葵CSN6A亲缘关系较近。

图3 基于GhCSN6A与拟南芥AtCSN蛋白氨基酸序列构建的系统发育进化树Fig.3 Phylogenetic tree constructed based on amino acid sequences of GhCSN6A and Arabidopsis thaliana AtCSN protein

AtCSN6A.拟南芥;NaCSN6A.烟草;HsCSN6A.木槿;GhCSN6A.陆地棉图4 GhCSN6A蛋白与同源蛋白氨基酸序列的多重比对结果AtCSN6A.Arabidopsis thaliana;NaCSN6A.Nicotiana attenuata;HsCSN6A.Hibiscus syriacus;GhCSN6A.Gossypium hirsutum L.Fig.4 Multiple alignment results of amino acid sequences between GhCSN6A protein and homologous proteins
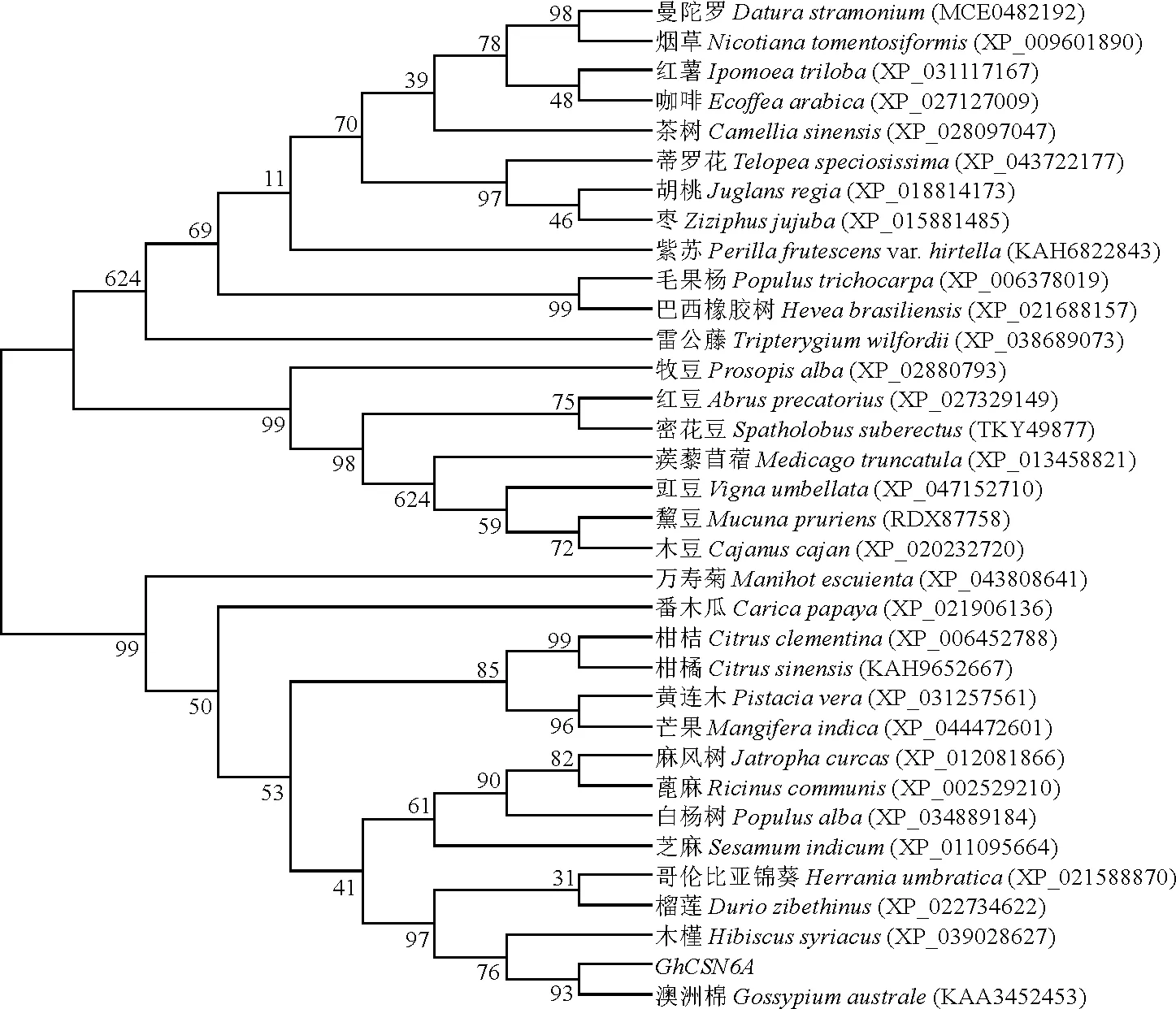
图5 基于GhCSN6A与其他植物的CSN6A蛋白氨基酸序列构建的系统发育进化树Fig.5 Phylogenetic tree constructed based on amino acid sequences of GhCSN6A and CSN6A proteins in other plants
2.5 GhCSN6A基因在不同组织及根部低磷胁迫处理的表达模式分析
以棉花的GhActin为内参基因,利用qRT-PCR技术检测GhCSN6A基因在根、茎、叶和花等4个组织中的表达情况,结果(图6)显示,GhCSN6A基因在根、茎、叶和花中均有表达,在叶中的表达量最高,显著高于花与茎中的表达量,其次是在根中的表达量较高,与茎中的表达量无显著差异,在花中的表达量最低(P<0.05)。对陆地棉整株植物进行低磷胁迫,以不同处理时间的陆地棉根部组织为材料进行qRT-PCR,分析GhCSN6A基因响应低磷胁迫的情况。结果(图7)表明,低磷处理0、4、12和24 h该基因的表达量比同期适磷处理的表达量低,低磷处理72 h时其表达量比适磷处理72 h的表达量高,是适磷处理的2倍。GhCSN6A基因在低磷胁迫0~72 h的总体变化趋势为:相对表达量在0~4 h内上调,在4~24 h从一个较高的水平(4 h)降落到一个最低水平(24 h),然后在72 h时又上升到最高水平。

不同字母表示在0.05水平差异显著(P<0.05)图6 GhCSN6A基因在不同组织的相对表达分析Different letters indicate significant differences at 0.05 level (P<0.05)Fig.6 The relative expression of GhCSN6A gene in different tissues
3 讨 论
COP9复合信号体在进化过程中是一种高度保守的蛋白复合物[19]。其中拟南芥COP9信号体的亚基6由一个家族的2个基因CSN6A和CSN6B编码,其蛋白具有87%的氨基酸相似,并在N端区域包含MPR1p和PAD1p N端(MPN)结构域。CSN6蛋白与CSN5具有相似性,均属于Mov34蛋白超家族[20-22]。本研究克隆的GhCSN6A基因编码的蛋白含有MPN结构域,属于Mov34蛋白超家族,与拟南芥、烟草、木槿的CSN6A蛋白序列相似性达90.93%,说明其在进化过程中具有一定的保守性。Gusmaroli等[23]发现,在拟南芥中,MPN结构域控制着CSN复台体的组装、复合体的稳定性及活性,并且还调控着一种泛素连接酶的稳定性。陆地棉GhCSN6A和拟南芥AtCSN6A蛋白具有相似的结构域,可能具有相似的功能。
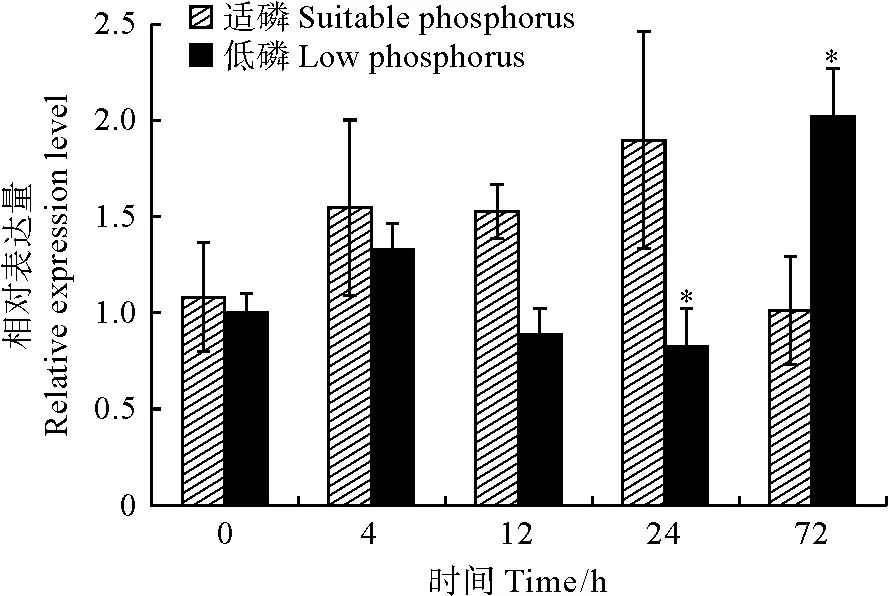
*表示同期适磷与低磷处理间存在显著差异(P<0.05)图7 GhCSN6A基因在根部低磷胁迫不同时间的表达模式* indicates significant difference between suitable and low phosphorus treatment at 0.05 level.Fig.7 The relative expression analysis of GhCSN6A gene with low phosphorus treatment at different time points in roots
COP9信号复合体亚基已从多种生物中被克隆分析,其功能也被陆续发现,主要参与植物激素信号传导、逆境胁迫应答、次生代谢活动等[24]。黄瓜绿斑驳花叶病毒CGMMV侵染黄瓜苗后,CsCSN5b基因表达量逐渐上升,说明该基因参与了病毒胁迫的应答机制响应CGMMV感染胁迫[25];CSN6参与水稻缺铁的早期反应,在缺铁的早期,由于CSN6和CSN活性的下调,IDEF1在水稻中积累。IDEF1蛋白的积累有助于通过增加水稻中铁吸收/利用相关基因的表达水平来克服缺铁胁迫[26]。本研究发现该基因在‘新陆早19’叶中的表达量最高,根、茎次之,在花中表达量最低。王彦平等[27]发现低磷胁迫下棉花的生长素在根、茎、叶的含量变化明显,尤其是叶中的含量变化最大。Stuttmann和Schwechheimer等[28-29]发现CSN与SCFTIRI互作参与植物生长素信号途径。本研究低磷胁迫处理下的表达模式结果表明,在4~24 h这个时间段里,该基因的表达量从一个较高的水平(4 h)到一个最低水平(24 h),然后又上升到最高水平(72 h),其趋势符合植物响应低磷胁迫反应的典型过程[9]。因为COP9复合体是作为一个整体的形式发挥作用的,通过与泛素系统相互作用来调控一系列的生物学功能。也就是说,生物体内的许多功能与泛素及COP9复合体有关。所以可以推测:在低磷胁迫的早期,生物体的功能会呈现出一种紊乱的状态,导致很多功能被抑制,所以与这些功能相关的分子,如泛素、COP9复合体,也会受到相应的影响,因此表达量会明显下降;但是当植物开始启动低磷胁迫响应时,相应的基因就会得到表达,从而要启动特殊的转录与调控机制,其中就包括COP9复合体在内的一些具有重要调控功能的基因就会加强表达,这个加强表达的时间就可能在72 h左右。因此,根据低磷胁迫表达模式的结果可推测:在72 h以后,COP9复合体的表达量明显大于4~24 h,说明植物在72 h左右的时候已经进入一种响应低磷胁迫且相对稳定的状态。在这个状态之中,有很多基因需要受到特异性的转录或表达调控,从而应答低磷胁迫。
依据该基因的组织表达分析及根部低磷处理不同时间的表达模式分析结果,推测陆地棉GhCSN6A基因在调控低磷胁迫方面具有重要作用。但是GhCSN6A基因作为COP9信号复合体亚基之一,其具体的生物学功能及在植物应答低磷胁迫的调控机制仍需深入研究。后续将通过酵母杂交、VIGS和转基因等功能验证手段分析GhCSN6A基因如何在低磷胁迫中发挥作用,并观察植株在低磷胁迫下的表现,从而揭示GhCSN6A基因在植物应答低磷胁迫的调控机制。

