基于iTRAQ蛋白组技术的沙门氏菌抗酸性机理的研究
张丽君,李琳琼,尹大成,高瑀珑
(南京财经大学食品科学与工程学院,江苏省现代粮食流通与安全协同创新中心,江苏南京 210023)
沙门氏菌是一种常见的的食源性致病菌,在自然界中分布较广,在适宜条件下可迅速生长繁殖,引起人类急性、慢性和隐性感染病,如食物中毒和肠胃炎等[1]。在食品的生产、储运和销售过程中,沙门氏菌极易反复受到极端温度、酸、碱、抗生素以及消毒剂等不适生长环境条件的胁迫作用,诱发其产生一系列的适应性变化,达到维持生存的目的[2]。沙门氏菌对食品加工中的消杀措施的适应性变化,潜在地增加了沙门氏菌感染的可能性[3]。已有相关研究表明,大多数微生物在不适生长的环境中会通过特定的抗性蛋白来保证其生存[4]。Chen等[5]研究发现,微生物处于高温、低温和高渗等一系列压力环境下其部分蛋白质的表达会发生变化,来稳定膜结构、维持细胞功能等。
近年来不断完善的蛋白组学技术已用于研究不同环境压力下对微生物产生的影响以及微生物在这些胁迫下的应激机制[6]。蛋白质组学是用于研究单个细胞或一种生物所表达全部蛋白质特征的技术,主要包括蛋白的表达水平、翻译的修饰、蛋白之间的相互作用等[7]。iTRAQ蛋白组技术具有通量高,灵敏度高,可重复性高,定量效果好,结果可靠,适用样品类别广泛等优点,是系统研究生物学规律和相关机制的有效工具[8]。Liu等[9]利用iTRAQ蛋白组技术,探讨沙雷氏菌在铬胁迫下的潜在机制,发现差异蛋白主要集中在碳水化合物代谢、应激反应、氨基酸代谢等方面。张佩佩[10]通过对甲型鼠伤寒沙门氏菌进行热处理,发现其产生抗热性,利用iTRAQ蛋白组技术分析其在热胁迫前后产生的差异蛋白,发现5%的差异蛋白参与热刺激适应,其中DNA连接酶(ligB)、逆境压力反应蛋白(dps)、分子伴侣(HdeB)以及未知蛋白ACN8920515和ynaF均下调,在热胁迫适应中发挥作用,使甲型鼠伤寒沙门氏菌在热胁迫下得以存活。但采用iTRAQ技术从蛋白质组学对鼠伤寒沙门氏菌的抗酸性机制的研究,国内外鲜见相关报道[11]。
本研究以食源致病菌鼠伤寒沙门氏菌为试材,采用iTRAQ技术对鼠伤寒沙门氏菌抗酸性菌株的蛋白组进行了分析,找出酸胁迫处理前后菌体之间的差异表达蛋白,通过GO功能注释和富集对差异蛋白进行生物过程、分子功能和细胞组分的聚类分析,通过KEGG注释和富集对差异蛋白参与的通路进行联合分析,筛选出与菌株抗酸性相关的差异蛋白,并揭示酸胁迫条件下鼠伤寒沙门氏菌在应激反应、双组分系统、ABC转运系统和能量代谢等过程中相关蛋白表达的差异,旨在揭示酸胁迫诱导鼠伤寒沙门氏菌产生抗酸性的机理,对优化食品加工条件和建立食品安全控制体系具有重要意义。
1 材料与方法
1.1 材料与仪器
鼠伤寒沙门氏菌CGMCC 1.1190 中国普通微生物菌种保藏管理中心;异丙醇、磷酸、柠檬酸、氯化钾、磷酸二氢钾、乙腈、甲酸、三氯乙酸、丙酮、溴化四乙铵、十二烷基硫酸钠、二硫苏糖醇、盐酸、三羟甲基氨基甲烷 南京慧杰诚生物科技有限公司;所有试剂 均为分析纯。
SAF-680T型酶标仪、SF-CF-2A型超净工作台上海三发科学仪器有限公司;AKTA Purifier 100型纯化仪、PHS-3G型pH计 上海仪电科学仪器股份有限公司;Easy nLC 1200型液相色谱仪、Q Exactive型质谱仪 赛默飞世尔科技有限公司;SM-650D型超声波破碎仪 南京舜码仪器设备有限公司;GL-21M型高速冷冻离心机 上海市离心机械研究所有限公司;LDZX-50FBS型立式压力蒸汽灭菌器 上海申安医疗器械厂;GNP-9160型隔水式恒温培养箱 上海三发科学仪器有限公司;ME55型塞多斯精密电子天平 北京塞多斯天平有限公司;SK-1型快速混匀器 金坛市杰瑞尔电器有限公司。
1.2 实验方法
1.2.1 菌种活化与预处理 实验菌株CGMCC 1.1190采用胰蛋白胨大豆肉汤(Trypticase Soy Broth,TSB)培养。供试菌经活化后,接入pH7.2的TSB培养基,于37 ℃、150 r/min摇床振荡培养18 h,为后续备用。
1.2.2 鼠伤寒沙门氏菌CGMCC 1.1190抗酸性菌株的获得 pH2.5柠檬酸胁迫处理:将1.2.1中活化的CGMCC 1.1190以体积分数1%转接至100 mL TSB培养基中,37 ℃振荡培养18 h,取10 mL培养液离心(5000 r/min,4 ℃,10 min),弃上清,重悬于柠檬酸调节pH2.5的TSB中进行酸胁迫处理,处理30 min,取体积分数为1%经pH2.5柠檬酸胁迫后的CGMCC 1.1190接种于100 mL TSB培养基中,37 ℃振荡培养18 h,再取10 mL培养液离心(5000 r/min,4 ℃,10 min),取沉淀,重悬于柠檬酸调节pH2.5的TSB中进行酸胁迫处理,处理30 min;以此类推,重复上述摇床振荡培养和酸胁迫处理12次,后续备用[12]。
1.2.3 蛋白组样品的制备 将1.2.1中活化的CGMCC 1.1190原始对照菌株及1.2.2节获得抗pH2.5柠檬酸的菌株分别接种于100 mL TSB中,摇床振荡培养(37 ℃,150 r/min,24 h),离心(5000 r/min,4 ℃,10 min),取沉淀,将菌体沉淀的离心管置于液氮中,速冻10 min,并标记样品号为对照组CK1、对照组CK2、处理组T1和处理组T2,置于-80 ℃冰箱保存,备用。
1.2.4 iTRAQ标记 采用SDT裂解法和TCA丙酮沉淀法提取CGMCC 1.1190菌体蛋白[13],用BCA蛋白浓度测定试剂盒测定蛋白样品浓度。取制备的蛋白样品各100 μg,用FASP法[14]进行酶解,酶解后采用C18 Cartridge对肽段进行脱盐,将上述样品真空冷冻干燥2 h,加入0.1%、40 μL的甲酸溶液复溶肽段。用iTRAQ试剂盒对肽段进行标记,室温静置2 h,标记信息分别为:CK1,113;CK2,114;T1,115;T2,116。等量混合经iTRAQ标记的4组肽段样品,根据强阳离子交换柱法对混合后的肽段进行预分离,之后进行液相分离和质谱分析。
1.2.5 实时荧光定量PCR验证 本研究选取CGMCC 1.1190菌株酸胁迫前后的8个显著差异蛋白的对应表达基因进行实时荧光定量PCR验证和分析。通过在线设计软件Primer 3.0来设计用于荧光定量PCR的引物,引物见表1。用HiScript® II 1st Strand cDNA Synthesis Kit试剂盒进行CGMCC 1.1190菌体第一链cDNA的合成。以cDNA链为模板,16S rRNA基因为内参基因,用ChamQTMSYBR® qPCR Master Mix试剂盒进行实时荧光定量PCR反应,采用ABI 7500 PCR分析荧光定量PCR反应中CGMCC 1.1190菌体对照菌株和抗酸菌株样品中8个待测基因的RNA相对表达含量,对照组为未经过酸处理的CGMCC 1.1190菌体RNA,设其表达水平为1,按照2-△△Ct法计算基因的相对表达量。

表1 RT-qPCR验证基因及引物设计Table 1 RT-qPCR verification gene and primer design
1.2.6 生物信息学分析 采用Mascot2.2和Proteome Discover1.4对质谱分析原始数据进行查库鉴定及质量分析[15]。通过对蛋白质相对分子质量、等电点、肽段序列长度、肽段序列覆盖度等方面来对蛋白质鉴定和定量的结果可信度进行评估。以P≤0.05对差异蛋白进行显著性筛选,倍数变化大于1.2或小于0.83的蛋白质被定义为差异蛋白,得到CGMCC 1.1190对照菌株和抗酸菌株之间的上调和下调蛋白的数目。
根据抗酸性CGMCC 1.1190菌株显著差异蛋白的表达特性,采用层次聚类算法对差异表达蛋白质进行分组归类,以热图的形式将表达趋势相同的基因进行聚类[16],同时与抗酸性CGMCC 1.1190菌株的生物学特性建立关联,筛选出符合抗酸性生物学特性的蛋白表进行重点研究。采用Blast2Go软件对CGMCC 1.1190酸胁迫前后筛选出的所有差异表达蛋白质进行GO功能注释[17],向GO数据库中对应功能的term映射,计算每个term中差异蛋白质的数量,然后应用Fisher精确检验CGMCC 1.1190抗酸性菌株与对照菌株差异蛋白的显著性[18],找出差异蛋白显著富集的GO条目,从而对CGMCC 1.1190菌株筛选出的差异蛋白行使的生物学功能进行分析。采用KAAS软件对CGMCC 1.1190菌株酸胁迫前后筛选出的差异表达蛋白质进行KEGG通路注释,通过Fisher精确检验CGMCC 1.1190抗酸性菌株与对照菌株差异蛋白的显著性,找出所有显著差异表达蛋白,并对抗酸性相关代谢的关键蛋白进行分析。
2 结果与分析
2.1 鼠伤寒沙门氏菌CGMCC 1.1190蛋白浓度的测定结果
利用BCA法测定所提取总蛋白的浓度,结果见表2。由表2可知,CGMCC 1.1190原始对照菌株(CK1,CK2)提取总蛋白浓度均为2.8 μg/μL,抗pH2.5菌株(T1,T2)提取总蛋白浓度分别为1.7和1.9 μg/μL,提取浓度能满足后续蛋白组测定相关的要求,样品的平行性和评价均较好。

表2 CGMCC 1.1190对照组和抗酸组菌株蛋白质样品浓度及评价结果Table 2 Concentration and valuation results of protein samples of CGMCC 1.1190 control strain and the acid-resistant strain
2.2 CGMCC 1.1190蛋白表达差异分析
2.2.1 CGMCC 1.1190蛋白表达差异统计 成功鉴定到的蛋白共2373个,已定量的蛋白占99.71%,CGMCC 1.1190对照组和抗酸组的差异蛋白总数为195个,其中包括95个显著下调蛋白和100个显著上调蛋白。蛋白质定量差异结果见图1。
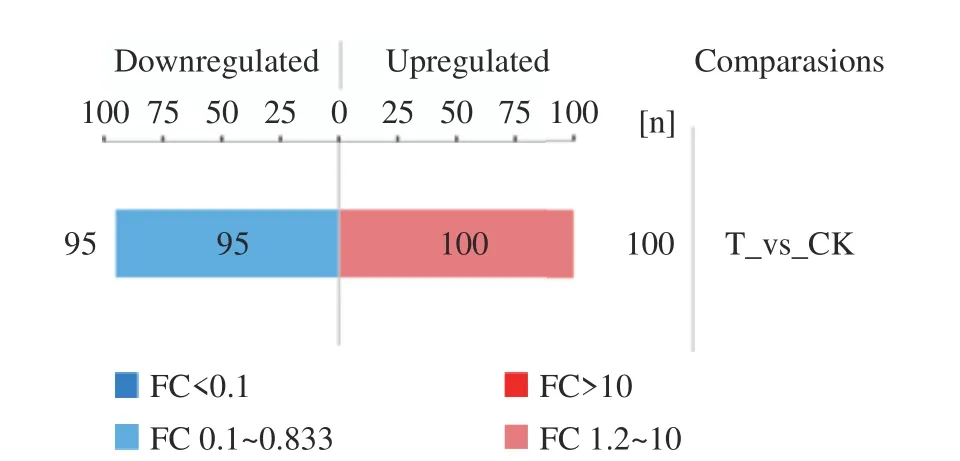
图1 CGMCC 1.1190对照组和抗酸组菌株的蛋白质定量差异结果柱状图Fig.1 Histogram of protein quantitative difference results of CGMCC 1.1190 control strain and the acid-resistant strain
2.2.2 CGMCC 1.1190蛋白表达模式聚类分析 本研究对CGMCC 1.1190对照菌株和抗酸性菌株的差异表达蛋白质进行分析,结果见图2,横坐标对样品进行分析,纵坐标对蛋白质进行分析。从图2中可以看出组内的数据相似性较高,而组间的数据相似性较低,说明对照组和抗酸组的差异蛋白质表达量变化说明酸胁迫处理对样本造成的影响显著,组内平行性较好。100个上调蛋白在CGMCC 1.1190抗酸性菌株中的表达量高于对照菌株,其中,RNA聚合酶σ因子(rpoS)、膜融合蛋白家族蛋白(STM4260)、外膜孔道蛋白(ompC)、鞭毛丝蛋白(fliC)的变化倍数分别为1.83、3.34、2.13、1.88;95个下调蛋白在CGMCC 1.1190抗酸性菌株中的表达量高于对照菌株,其中,LexA阻遏蛋白(lexA)、异柠檬酸脱氢酶(IcdA)、精氨酸脱亚胺酶(arcA)、鸟氨酸氨基甲酰转移酶(arcB)的变化倍数分别为0.75、0.70、0.55、0.57。
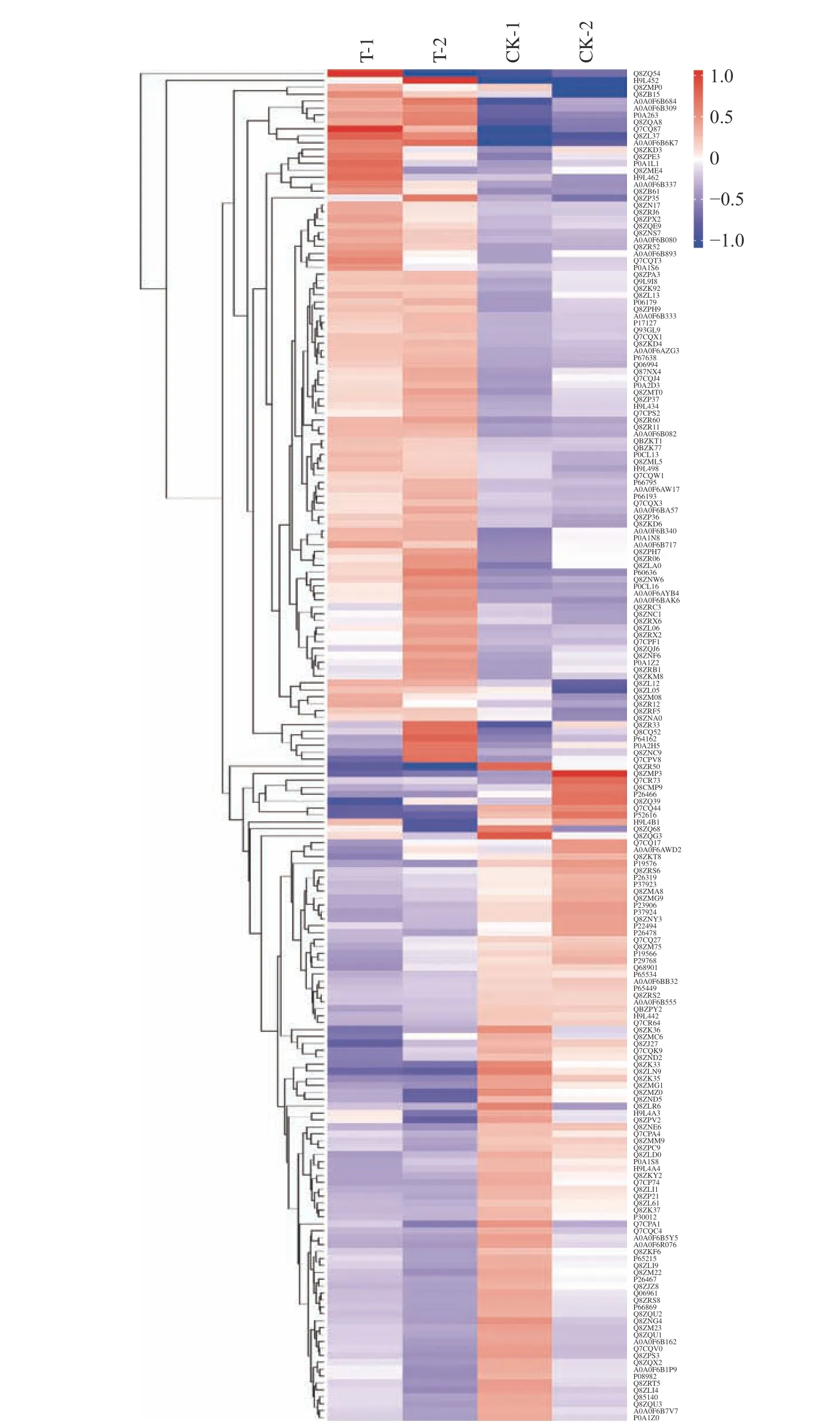
图2 CGMCC 1.1190对照组和抗酸组菌株的差异蛋白质聚类分析Fig.2 Cluster analysis of differential protein of CGMCC 1.1190 control strain and the acid-resistant strain
2.2.3 CGMCC 1.1190差异表达功能蛋白分类分析本研究对CGMCC 1.1190对照菌株和抗酸性菌株的蛋白质图谱进行对比分析,得到195个表达差异显著的相关蛋白。在这些差异蛋白中,大多数蛋白质主要参与CGMCC 1.1190内部调节代谢途径的生理功能,包括参与应激反应、细胞膜变化、鞭毛合成、转运系统、双组份系统相关的蛋白等,表明CGMCC 1.1190抗酸性菌株通过上述代谢途径相关的蛋白来调控适应酸胁迫环境。在195个显著差异表达的蛋白中,与应激反应、细胞膜变化、鞭毛合成、转运系统、双组份系统相关的蛋白的信息分别见表3~表8。
表3 应激相关蛋白信息Table 3 List of protein information related to stress

表8 与能量代谢相关蛋白信息Table 8 List of protein information related to energy metabolism
2.2.4 CGMCC 1.1190差异蛋白的GO富集分析GO分析结果如图3所示,根据生物过程注释,差异蛋白主要与菌体内代谢、应激调节反应有关;根据分子功能注释,差异蛋白主要是各种酶类、修复蛋白以及转运蛋白;根据细胞组分注释,差异蛋白主要涉及细胞膜组成蛋白,得到注释的蛋白中膜蛋白占32.7%。
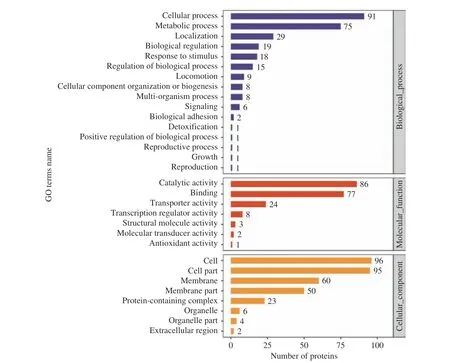
图3 CGMCC 1.1190对照组和抗酸组菌株差异表达蛋白质的GO功能注释及富集分析图Fig.3 GO function annotation and enrichment analysis map of differentially expressed proteins of Salmonella typhimurium CGMCC 1.1190 control strain and the acid-resistant strain

表4 细胞膜相关蛋白信息Table 4 List of protein information associated with membrane

表5 鞭毛相关蛋白信息Table 5 List of protein information related to flagella
2.2.5 CGMCC 1.1190差异蛋白的KEGG生物通路分析 KEGG分析发现CGMCC 1.1190对照菌株和抗酸性菌株的差异蛋白质富集于60条代谢通路上,显著性分析结果见图4~图5。由图4~图5可知,CGMCC 1.1190菌株的差异蛋白质主要影响了6个代谢通路,其中双组分调节系统途径参与的蛋白质有19个,三羧酸循环途径参与的蛋白质有10个,碳固定途径参与的蛋白质有10个,氧化还原途径参与的蛋白质有7个,丙酮酸代谢途径参与的蛋白质有7个,ABC转运系统参与的蛋白质有6个。与CGMCC 1.1190对照菌株相比,抗酸性菌株的蛋白质在双组分调节系统、三羧酸循环、碳固定等通路差异显著。
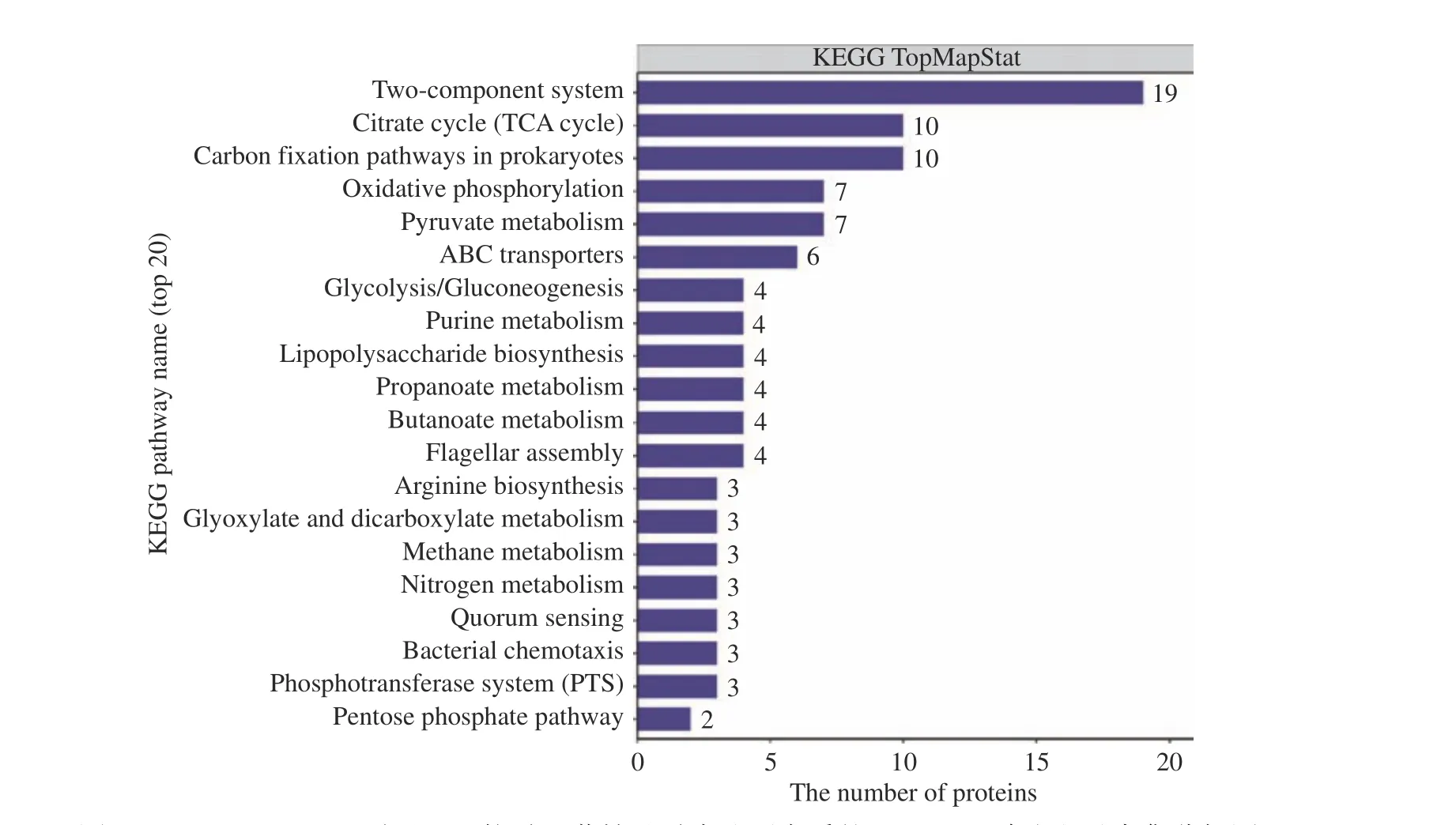
图4 CGMCC 1.1190对照组和抗酸组菌株差异表达蛋白质的KEGG通路注释及富集分析图Fig.4 KEGG pathway function annotation and enrichment analysis map of differentially expressed proteins of CGMCC 1.1190 control strain and the acid-resistant strain
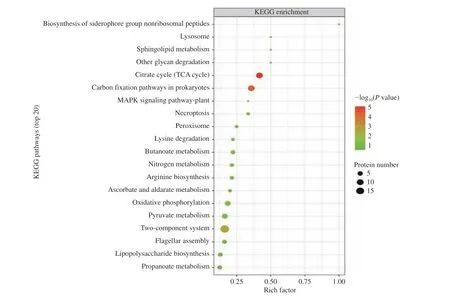
图5 CGMCC 1.1190对照组和抗酸组菌株差异表达蛋白质的KEGG显著性分析Fig.5 KEGG significance analysis of differentially expressed proteins between CGMCC 1.1190 control strain and the acid-resistant strain

表6 双组分系统相关蛋白信息Table 6 List of protein information related to two-component system

表7 ABC转运系统相关蛋白信息Table 7 List of protein information related to ABC transfer system
2.2.6 CGMCC 1.1190部分差异表达基因的RT-qPCR验证 为验证本研究中iTRAQ蛋白组数据的准确性,在经柠檬酸调节pH为2.5的TSB培养基反复胁迫后的CGMCC 1.1190中选取8个差异显著蛋白(5个上调蛋白和3个下调蛋白),进行RT-qPCR验证,结果见图6。8个目的基因的RT-qPCR结果与8个差异蛋白的iTRAQ蛋白组测定结果在表达幅度上有一定的差异,但其表达趋势基本一致。

图6 CGMCC 1.1190对照组和抗酸组菌株部分基因的RTqPCR验证Fig.6 RT-qPCR verification of some genes between CGMCC 1.1190 control strain and the acid-resistant strain
3 讨论
3.1 抗酸性鼠伤寒沙门氏菌CGMCC 1.1190与应激相关蛋白的分析
微生物常处于不适的环境中,应激蛋白指大多数菌体在遭受外界压力时合成的蛋白,在菌体迅速回应逆境等方面发挥重要作用[19]。对比分析CGMCC 1.1190对照菌株和抗酸性菌株的蛋白组,当菌体经过酸胁迫后,RNA聚合酶σ因子(rpoS)蛋白水平上调约1.8倍,其处于调控较上游的位置并控制休克蛋白表达,保护环境压力下的菌体,对其适应性有调控作用[20]。本研究发现,RNA聚合酶σ因子对CGMCC 1.1190菌体适应酸胁迫环境起到一定的调控作用。热休克蛋白是一种保护性蛋白,通过参与蛋白的正确折叠、维持细胞骨架等来提高细胞的耐热等应激能力,使菌体细胞内的生理过程保持稳定[21]。本研究发现,抗酸性CGMCC 1.1190的小热休克蛋白(ibpA)表达增加,表明其对菌株的耐酸能力也具有一定的调控作用,该调控作用可能与菌株产生耐热性的过程类似。RecA蛋白和LexA阻遏蛋白是与菌体遭受环境胁迫后与修复机制相关的蛋白,当SOS系统被激活,recA和单链DNA会结合生成recA-ssDNA,诱导LexA阻遏蛋白进行自我降解,增加重组酶和聚合酶的表达,修复损伤[22]。本研究发现,抗酸性CGMCC 1.1190的recA蛋白表达增加,lexA阻遏蛋白表达减少,表明在经过酸胁迫后,CGMCC 1.1190抗酸性与菌体内部SOS系统的激活有关,菌体通过相关蛋白的协同作用修复损伤来提高其在酸环境下的生存能力。
3.2 抗酸性CGMCC 1.1190与膜相关蛋白的分析
细菌膜蛋白在其生命活动中起着重要的作用,主要维持细胞膜的完整、流动性、参与菌体内营养物质的运输以及物质代谢[23]。本研究发现,抗酸性CGMCC 1.1190部分膜蛋白的表达量发生改变,其中ompC基因编码的外膜孔道蛋白C表达水平显著上调。戎建荣等[24]发现,在高渗环境中,大肠杆菌通过改变外膜孔道蛋白C的表达水平减少胆盐渗入菌体细胞;受到抗菌药物胁迫的细菌,其外膜孔道蛋白C蛋白表达有助于降低菌体内抗菌药物的浓度、增加耐受力。本研究结果表明外膜孔道蛋白C对CGMCC 1.1190的抗酸性起到一定的调控作用,通过改变细胞膜的通透性,如降低细胞膜对H+的通透性来帮助细菌适应酸胁迫环境。Tol-Pal系统是革兰氏阴性菌高度保守的膜蛋白系统之一,主要由Tol-Pal系统蛋白(tolB)、肽多糖相关蛋白(pal)、Tol-Pal系统蛋白(tolA)、Tol-Pal系统蛋白(tolR)和Tol-Pal系统蛋白(tolQ)组成,其中tolA、tolQ和tolR构成细菌的内膜蛋白,tolB和pal构成细菌外膜复合体,Tol-Pal系统维持革兰氏阴性菌细胞膜的完整性和稳定性。Prouty等[25]研究发现,鼠伤寒沙门氏菌耐受胆盐的能力与Tol-Pal系统的调控有关。本研究CGMCC 1.1190抗酸性菌株Tol-Pal系统蛋白均上调,表明酸胁迫后CGMCC 1.1190产生的抗酸性与其Tol-Pal膜蛋白系统的调控有关,可能通过增加细胞膜的完整性和稳定性来帮助其抵抗并适应极端酸胁迫环境。因此,膜蛋白表达量的变化可能是CGMCC 1.1190酸胁迫菌株比原始菌株更抗酸的原因之一。
3.3 抗酸性CGMCC 1.1190与鞭毛相关蛋白的分析
细菌的鞭毛是一种位于菌体表面的细丝状蛋白质附属物,主要与细菌的运动性和大分子蛋白质的转运有关[26]。Duan等[27]通过对比研究大肠杆菌fliC基因缺失株的运动能力和侵袭能力,发现大肠杆菌fliC缺失株的运动能力和侵袭能力显著低降低。本研究发现,抗酸性CGMCC 1.1190的鞭毛相关差异蛋白均上调,其中鞭毛丝蛋白(fliC)上调约1.9倍,表明抗酸性CGMCC 1.1190的运动能力对其抗酸性有重要影响。细菌鞭毛蛋白调控的运动性也是生物被膜形成初期和成熟期的决定性因素,大肠杆菌运动性的强弱与生物被膜形成能力呈正相关[28]。这与本研究结果一致,表明鞭毛蛋白调控的运动性增强可以促进菌体形成具有抵抗能力的生物被膜。本研究中鞭毛相关蛋白表达的变化表明抗酸性CGMCC 1.1190可能通过调控鞭毛蛋白的表达来增强运动能力,促进生物被膜的形成,使其抗酸性增强。
3.4 抗酸性CGMCC 1.1190与双组分系统相关蛋白的分析
双组分系统(Two-Component System,TCS)是存在于细菌内的一种信号传导系统,与细菌的群体感应、致病性、耐药性、毒力因子表达和生物被膜生成等生物学功能密切相关,是细菌适应环境变化并响应多种刺激的一种调节机制[29]。TCS与细菌的群体感应有关,群体感应是细菌之间信号传递的重要机制,当细胞密度增加达到一定阈值,细菌通过信号传递影响特定基因的表达,来调控微生物群体的生理特征[30]。何绿琴[31]通过构建转录调控蛋白(qseB)/感应蛋白(qseC)缺失株,发现副猪嗜血杆菌生物膜生成能力降低,抵御不良环境能力减弱,毒力下降。双组分系统组氨酸激酶传感蛋白(envZ)/DNA结合双转录调节因子(ompR)主要与细菌的渗透调节相关,通过对膜孔道蛋白进行正向或负向的调控来影响膜内外离子转运、平衡渗透压力[32]。双组分系统精氨酸脱亚胺酶(arcA)/鸟氨酸氨基甲酰转移酶(arcB)对厌氧和需氧生长下细菌的呼吸代谢、生物合成和运动能力有调控作用,主要影响菌体的能量代谢过程。武珊珊[33]研究发现沙门氏菌arcA/arcB缺失株对氨基糖苷类抗生素的抗性有所提高,但其引起的耐药性变化的机理尚未阐明。本研究发现,抗酸性 CGMCC 1.1190双组分系统病毒性传感器组氨酸激酶(phoP)/病毒性转录调节蛋白(phoQ)、qseB/qseC、envZ/ompR的蛋白水平均上调,双组分系统arcA/arcB蛋白水平下调,表明CGMCC 1.1190双组分系统与其抗酸性调控具有密切关系。
3.5 抗酸性CGMCC 1.1190与ABC转运系统相关蛋白的分析
ABC转运系统相关蛋白是指含有ATP结合区域的蛋白,主要功能包括营养物质的转运和排出有害物质等[34]。谢腾飞[35]研究发现,冷胁迫后的副溶血性弧菌通过抑制ABC转运蛋白的量来保护菌体免于低温损伤。MalEFGK是位于菌体内膜上的ABC转运蛋白复合体,麦芽糖/麦芽糊精导入ATP结合蛋白(malK)负责与运输系统的能量偶联;麦芽糖/麦芽糊精运输系统渗透酶蛋白(malF)负责跨膜转运;麦芽糖/麦芽糊精结合周质蛋白(malE)是麦芽糖摄取的关键,参与麦芽糖的ABC运输系统[36]。本研究发现,CGMCC 1.1190抗酸性菌株的菌体中构成MalEFGK复合物的3种蛋白(malK、malF、malE)表达水平均下调,表明CGMCC 1.1190在酸胁迫后可能通过改变细胞膜的通透性和控制转运物质来保护菌体、抵抗不良环境。
3.6 抗酸性CGMCC 1.1190与能量代谢相关蛋白的分析
微生物代谢需要的能量是菌体在酶的作用下通过其细胞内一系列氧化还原反应产生的,微生物提供能量的途径以三羧酸循环和糖酵解为主,能量代谢的正常进行是保证菌体正常生长的必要条件[37]。本研究探讨CGMCC 1.1190抗酸性菌株的蛋白组,发现参与三羧酸循环的蛋白表达多上调,包括异柠檬酸脱氢酶(IcdA)、异柠檬酸裂解酶(aceA)、柠檬酸裂合酶(citG)等,表明CGMCC 1.1190菌体在适应酸胁迫环境时消耗了大量能量来维持自身正常的生理代谢。本研究发现, 抗酸性CGMCC 1.1190丙酮酸分解代谢途径的磷酸烯醇式丙酮酸碳羧化酶(PckA)活跃,说明其分解产生了更多能量以维持酸胁迫下菌体细胞的稳态。此外,本研究还发现,CGMCC 1.1190菌体内氨基烷基膦酸酯N-乙酰转移酶(phnO)上调,该路径可以促进CoA生成。有研究报道,当生物体遭受外界胁迫作用,可以通过CoA激活体内的免疫应答来抵抗外部胁迫[38]。由此推测,酸胁迫下CGMCC 1.1190菌体内的CoA的表达变化激活了菌体内的应激反应,导致抗酸性增加。
4 结论
本实验通过ITRAQ蛋白组技术对鼠伤寒沙门氏菌CGMCC 1.1190在经柠檬酸调节pH为2.5的TSB基质中进行多次酸胁迫处理后的差异表达蛋白进行研究,共检测到2373个蛋白,其中195个差异蛋白,包括95个显著下调蛋白和100个显著上调蛋白。通过对CGMCC 1.1190菌株的差异蛋白进行GO分析和KEGG分析,发现抗酸性菌株高表达应激蛋白相关基因,可以通过合成一些应答外界刺激的蛋白质来帮助CGMCC 1.1190增强抗酸性;高表达细胞膜相关蛋白来增加CGMCC 1.1190菌体细胞膜的完整性;高表达鞭毛相关蛋白来增强CGMCC 1.1190菌体的运动能力和增加保护性生物被膜的形成;高表达双组分系统相关蛋白来响应酸信号,通过调控酸激蛋白的产生增强CGMCC 1.1190菌体的抗酸性;低表达ABC转运系统相关蛋白来降低细胞膜对H+的通透性,达到保护菌体的作用;高表达能量代谢途径相关蛋白来为CGMCC 1.1190菌体应对酸胁迫提供能量、维持稳态。综上可知,CGMCC 1.1190在经过pH2.5的柠檬酸胁迫,能通过蛋白水平的表达变化启动一系列应答机制并生存下来。

