利用CRISPR/Cas9技术突变BnaMPK6基因降低甘蓝型油菜的耐盐性
张文宣 梁晓梅 戴 成 文 静 易 斌 涂金星 沈金雄 傅廷栋 马朝芝
利用CRISPR/Cas9技术突变基因降低甘蓝型油菜的耐盐性
张文宣 梁晓梅 戴 成 文 静 易 斌 涂金星 沈金雄 傅廷栋 马朝芝*
华中农业大学作物遗传改良全国重点实验室 / 国家油菜工程技术研究中心 / 洪山实验室, 湖北武汉 430070
甘蓝型油菜是我国一种重要的油料作物。MPK6是一种丝裂原活化蛋白激酶, MAPK级联途径可以被各种胁迫激活, 进而调节植物对胁迫的响应, 在植物响应非生物胁迫过程中发挥着重要的作用, 但在甘蓝型油菜盐胁迫响应过程中的功能还尚不清楚。基因结构和蛋白序列分析表明, MAPK6在十字花科芸薹属植物种间分化相对保守, 基因结构相似, 蛋白之间均有相同的STKc_TEY_MAPK结构域, 与胁迫和生长发育相关。为探究其功能, 本研究利用CRISPR/Cas9技术对基因进行编辑, 通过农杆菌介导的方法将基因转入到甘蓝型油菜中, 获得了基因4个同源拷贝同时突变的材料:和。突变体表现出明显的盐敏感性: 在100 mmol L–1和150 mmol L–1浓度的NaCl溶液处理下, 功能缺失突变体长势明显缓慢, 并且株高和鲜重显著低于野生型植株, 但根长无明显差异。此外, 在盐胁迫下, 功能缺失突变体体内的活性氧和丙二醛含量的积累显著高于野生型对照, 脯氨酸含量也明显高于野生型植株。本研究结果表明,基因在盐水胁迫下正向调控植株的生长和发育, 影响甘蓝型油菜的耐盐性。本研究不仅为基因调控甘蓝型油菜盐胁迫的研究提供了理论基础, 而且为甘蓝型油菜耐盐性遗传改良提供了一定的技术支持。
甘蓝型油菜;; CRISPR/Cas9; 盐胁迫
油菜是中国重要的油料作物[1], 我国主要种植是甘蓝型油菜[2]。基于CRISPR/Cas9的基因编辑技术, 打破了传统育种依靠诱变获得突变体的限制, 可以定向改良作物的农艺性状, 已经成为最热门的生物技术之一[3]。CRISPR/Cas9技术通过设计特异的靶向性位点, 引导sgRNA与Cas9蛋白相结合, 进而对目标基因DNA序列进行剪切, 然后通过非同源末端连接的DNA修复方式, 造成序列的缺失、错配和插入, 使基因发生突变[4-5]。由于其载体构建简单、编辑效率高等优点, 已经广泛应用在水稻、拟南芥、玉米和油菜等作物中[6-9]。
植物在环境中不断受到外界刺激信号的影响, 包括对植物生长发育不利的胁迫信号。其中, 非生物胁迫是制约植物生长、造成农业生产下降、威胁粮食安全的主要原因之一。近年来, 由于极端天气的不断出现, 进一步加剧了非生物胁迫对农作物产量的影响[10]。土壤盐碱化不仅大面积的影响了耕地面积, 造成总产量的下降, 而且引起的盐离子毒害对作物的危害日益严重。盐胁迫会引起渗透胁迫、氧化胁迫和离子毒害, 影响植物细胞活性[11]。SOS途径是植物中的一种非生物胁迫信号转导途径[12], 与植物耐盐性密切相关, 其主要功能是负责胞内Na+外排[13]。在这个途径中, SOS3感知盐胁迫诱导的细胞内游离钙水平的增加, 并与SOS2相互作用形成SOS3-SOS2激酶复合体, 通过解除SOS1自抑制作用从而激活SOS1活性, 激活SOS1把Na+排出细胞外[14-15]。在SOS途径中, MAPK6 (丝裂原活化蛋白激酶6)也参与此信号传导途径, MAPK6与磷脂酸结合进而被激活, 活化的MAPK6结合并激活SOS1, 完成Na+排出细胞外这一活动[16]。植物的MAPK激酶家族成员很多, MAP3Ks被刺激的质膜受体激活, 激活MAP3Ks磷酸化并激活MAP2Ks, 进而磷酸化并激活MAPKs[17]。植物的MAPK级联途径在生物和非生物胁迫过程中发挥着重要的作用。近年来, 植物中有部分MAPK激酶信号级联途径已被阐明, 其中最具特征的是MAPK3、MAPK4和MAPK6, 它们参与逆境反应相关的各种信号级联[18], 例如非生物胁迫[19], 早期的研究结果表明, MAPK6在低温和盐胁迫处理下可以被上游的MKK2所激活, 并存在一条磷酸化信号转导途径[20]; 乙烯反应[21], 乙烯作为一种重要的植物激素, 也参与胁迫下的调控, 在植物生长发育阶段, 昆虫口器和病原体都会引起乙烯含量的增加, 2003年, Ouaked等[22]发现, MAPK6可以激活乙烯的表达, 2008年, Xu等[24]进一步确定了多种非生物胁迫和生物胁迫因素均可激活MAPK6的表达进而引起乙烯的产生[23]; 以及胚珠发育等, 表明植物信号转导网络中MAPK级联的复杂性。在拟南芥中的研究结果还表明MAPK6激酶可以被低温、触伤、渗透胁迫激活[25]。
虽然基因在植物应对各种胁迫过程中发挥着不同的调控功能, 但目前关于MAPK基因家族成员在甘蓝型油菜非生物逆境胁迫响应过程中的作用报道较少。本研究利用实时荧光定量PCR分析了在盐胁迫下的表达特征以及在各个组织中的表达模式, 同时采用CRISPR/Cas9技术对基因进行编辑, 通过农杆菌介导的方法将基因转入到甘蓝型油菜中研究其耐盐性, 为进一步研究基因在植物耐盐性中的作用奠定基础。
1 材料与方法
1.1 数据库及生物学软件
甘蓝型油菜基因组参考BnPIR (http://cbi.hzau. edu.cn/bnapus/index.php)泛基因组数据库; 白菜和甘蓝基因组参考BRAD (http://brassicadb.org/brad/)数据库; 拟南芥基因组参考TAIR (https://www.arabi-dopsis.org/); 基因序列重新预测其结构使用网站Fgenesh-M (http://linux1.softberry.com/berry.phtml); 在InterProScan (http://www.ebi.ac.uk/interpro/)网站对蛋白序列的保守结构域进行分析; 使用MEGA7.0进行序列比对和构建系统进化树; GeneDoc处理序列比对结果; 利用生物信息学软件TBtools完成部分结果可视化。
1.2 试验材料
以甘蓝型油菜(L.)品系‘Westar’为主要植物材料, 由华中农业大学油菜改良中心提供, 常规材料种植于华中农业大学试验田, 转基因材料种植于温室或转基因试验田, 所有材料按照田间管理办法种植。
所用的载体质粒骨架pKSE401由中国农业大学陈其军教授实验室提供, 用于甘蓝型油菜‘Westar’的遗传转化。本研究使用大肠杆菌宿主菌DH5α、农杆菌菌株GV3101购自北京全式金生物技术有限公司。由北京擎科生物科技有限公司合成引物, 由安诺优达基因科技有限公司完成测序。
1.3 植物叶片DNA提取
采用CTAB法[26]提取油菜材料的DNA, 所有材料均选取苗期鲜嫩叶片。用ddH2O调至50 ng μL–1储存在–20℃备用。
1.4 RNA提取和qRT-PCR分析
选取饱满的野生型种子进行萌发, 待种子萌发7 d后, 选取长势一致的幼苗, 将幼苗分为4组, 每组分成3个重复, 每个重复3株单株。将幼苗根部浸入100 mmol L–1NaCl的水溶液中进行盐水胁迫处理, 在0、1、3和6 h分别剪取叶片和根部进行RNA提取。利用SV Total RNA Isolation System (Z3100, Promega)试剂盒提取植物组织RNA, 利用Prime Script RT Reagent Kit with gDNA Eraser (RR047, TaKaKa)反转录获得cDNA, 反转录产物稀释50倍作为模板。使用Perl Primer设计引物,为内参基因。qRT-PCR反应体系为SYBR Realtime Mix 10 μL, 包含上下游引物各0.8 μL、cDNA模板8.4 μL。qRT-PCR反应程序为95℃预变性3 min; 95℃变性10 s, 60℃退火15 s, 72℃延伸30 s, 共45个循环。每个处理3个生物学重复, 并使用2–ΔΔCT方法计算基因的相对表达量。相关引物见表1。
1.5 CRISPR/Cas9载体构建
利用甘蓝型油菜基因组参考BnPIR (http://cbi. hzau.edu.cn/bnapus/index.php)泛基因组数据库进行blast比较分析, 鉴定出4个同源拷贝, 分别为()、()、()和()。采用靶点设计网站(http://crispr.hzau.edu.cn/CRISPR2/)分别在()和()的第4个外显子以及()和()的第5个外显子的保守序列位置设计2个sgRNA, 将2个靶点构建到载体中。
1.6 农杆菌介导的甘蓝型油菜的遗传转化
利用农杆菌介导的下胚轴暗光培养遗传转化方法, 将构建的CRISPR/Cas9载体导入到油菜的受体材料‘Westar’的下胚轴中, 经过组织培养的继代转化方式获得阳性苗[27]。
1.7 HI-TOM高通量测序确定突变单株的突变基因型
为进一步确定阳性苗的突变类型, 本研究利用HI-TOM (High-throughput Tracking of Mutations)测序。按照2轮PCR扩增的方式: 第1轮PCR使用靶点特异性引物扩增目标序列; 第2轮PCR以第1轮PCR产物为模版, 使用通用引物进行扩增(文章作者提供序列)[28], 最后将第2轮PCR产物混合并纯化回收, 送安诺优达公司进行二代高通量测序, 测序得到的数据通过在线网站Hi-Tom (http://www.hi-tom.net/hi- tom/)分析每个单株的具体突变类型[28]。相关引物序列见表2。
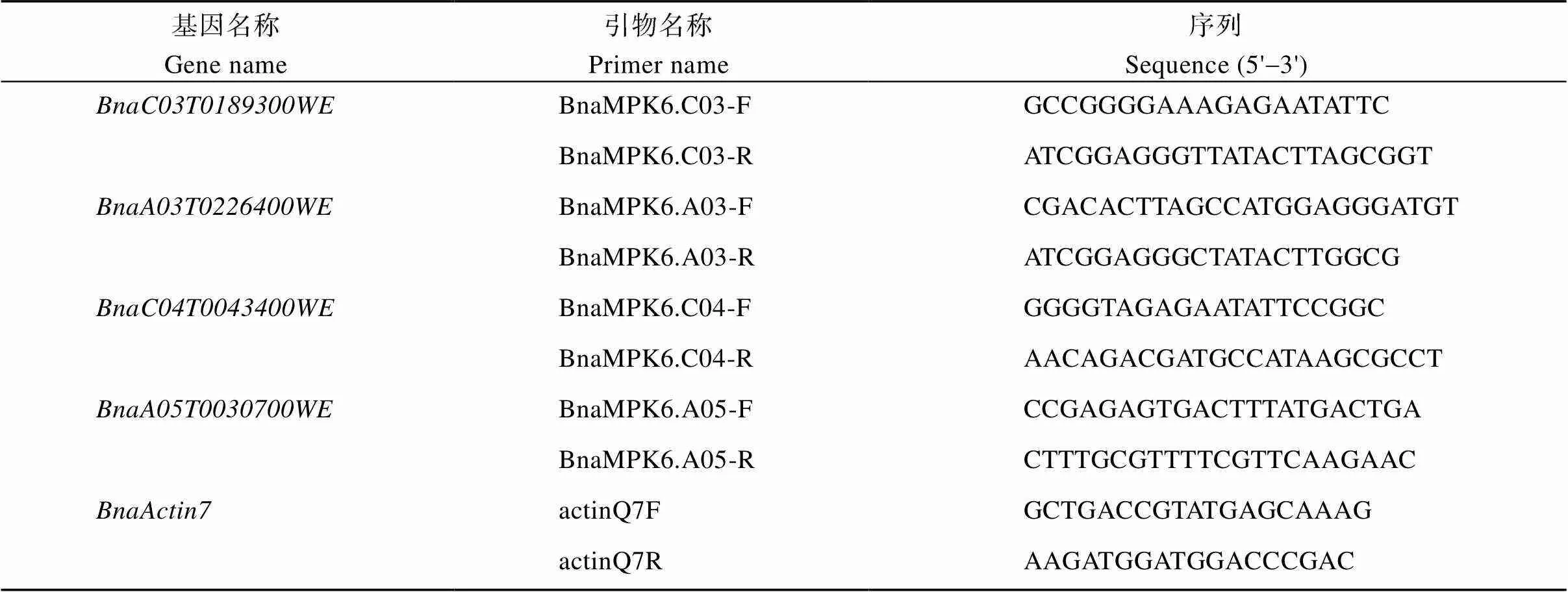
表1 qRT-PCR引物序列

表2 高通量测序引物序列
1.8 BnaMPK6功能缺失突变体在盐胁迫下生理指标的测定
1.8.1 丙二醛和脯氨酸含量测定 选取T1代功能缺失突变体和野生型对照种子同时萌发, 7 d后进行水培培养, 培养14 d后进行100 mmol L–1和150 mmol L–1浓度的NaCl溶液处理, 处理7 d后对其株高、根长和鲜重表型进行观察。之后, 取0.5 g植株叶片充分研磨后测定丙二醛和脯氨酸的含量[29]。
1.8.2 NBT染色步骤 称取0.05 g NBT粉末, 加入磷酸缓冲液(pH 7.5) 500 μL震荡溶解, 加水定容至50 mL。将叶片浸于NBT染液中并抽真空30 min, 室温下黑暗染色8 h, 在90%无水乙醇中80℃煮沸脱色。后置于光学显微镜下照相观察。
2 结果与分析
2.1 MAPK6基因结构分析和进化树构建
在拟南芥信息资源数据库TAIR查找到1个基因的核苷酸和蛋白质序列; 在油菜数据库BnPIR中获得Westar的全基因组数据(westar.all. v0); 在芸薹属数据库BRAD中下载白菜(Brapa_ genome_v3.0)和甘蓝(Brassica_oleracea_JZS_v2)全基因组数据。基于AtMPK6的蛋白序列, 使用TBtools软件的BLAST功能, 分析出油菜、白菜和甘蓝全基因组所有潜在的基因。用MEGA7.0使用最大似然值的分析方法构建系统进化树, 研究在拟南芥、甘蓝型油菜、白菜型油菜和甘蓝进化水平上的关系。从系统进化树可以看出, 甘蓝型油菜、白菜型油菜和甘蓝具有较高的同源性, 种间分化相对保守。基因结构可视化分析发现,基因在内含子和外显子数量上具有较大相似性, 其中、、、和有6个外显子,、和有5个外显子(图1)。

图1 拟南芥、甘蓝型油菜、白菜型油菜和甘蓝MPK6基因的系统进化树及基因结构
2.2 MAPK6蛋白质序列以及保守结构域分析
利用BnPIR数据库, AtMPK6蛋白序列在线BLAST冬性油菜Westar、白菜型油菜和甘蓝全基因组。将蛋白序列进行对比, 并定位保守结构域。对其保守结构域进行分析发现, MAPK6在甘蓝型油菜、拟南芥、白菜型油菜和甘蓝中具有相同的STKc_ TEY_MAPK结构域, 属于MAPK基因家族TEY亚型A亚家族, 是不稳定的脂溶性亲水酸性蛋白, 与逆境胁迫、生长发育和激素相关。统计结构域氨基酸残基差值发现, BnaC03.MPK6、BnaC04.MPK6、BnaA05.MPK6和BnaA03.MPK6与AtMPK6、BolMPK6、BraA03.MPK6和BraA05.MPK6的氨基酸残基差异仅在2%左右, 具有很微小的差异。对比蛋白全长序列, BnaC03.MPK6、BnaA03.MPK6、BolMPK6、BraA05.MPK6与AtMPK6相比仅有2%左右的差异, 缺失了3个氨基酸残基, BnaC04. MPK6、BnaA05.MPK6、BraA03.MPK6与AtMPK6相比仅有2%左右的差异, 缺失了5个氨基酸残基(图2)。
2.3 BnaMPK6基因表达模式分析
2.3.1基因在不同组织中表达模式分析 为研究不同组织中的表达情况, 以苗期‘Westar’多个组织的cDNA为模板, 通过qRT-PCR确定表达量。在各组织中均有表达。在各个营养组织中(根、茎、叶、芽),、以及均有较高的表达量, 其中在所有拷贝中表达量最高。在营养组织中表达量均比较低, 是4个拷贝中表达量最低的。总体来看,是一个泛表达的基因, 其中、和在各组织中均有高表达,在各组织中均维持较低表达水平(图3-A)。
2.3.2基因在不同的盐水处理时间下表达模式分析 为了解基因在盐水胁迫处理下表达量的差异, 以胁迫处理后幼苗cDNA为模版, 进行qRT-PCR试验分析其表达模式。结果发现,在根和叶中的表达量最高, 在根中不同处理时间下的表达量无显著差异, 在叶片处理3 h和6 h时, 表达量较未处理时有显著升高。在根和叶中的表达量均为最低, 在根和叶中不同处理时间下的表达量无显著差异。在根中处理1 h时, 表达量较未处理时有所降低, 但在3 h时表达量又升高到与未处理时相近的表达量, 在6 h时又降低到与3 h相近的表达量; 在叶片中处理1、3和6 h时, 表达量较未处理时有显著升高。在根和叶片中随着处理时间的变长, 表达量逐渐升高, 在根中处理6 h时显著升高, 在叶片中处理1、3和6 h时, 表达量较未处理时有显著升高。总体来看,基因在叶片中的表达量差异显著, 在根中差异不显著, 并且一直都有最高表达量。因此本研究推测,基因在盐水胁迫时主要影响植株的地上组织部分, 对盐水胁迫响应敏感(图3-B)。
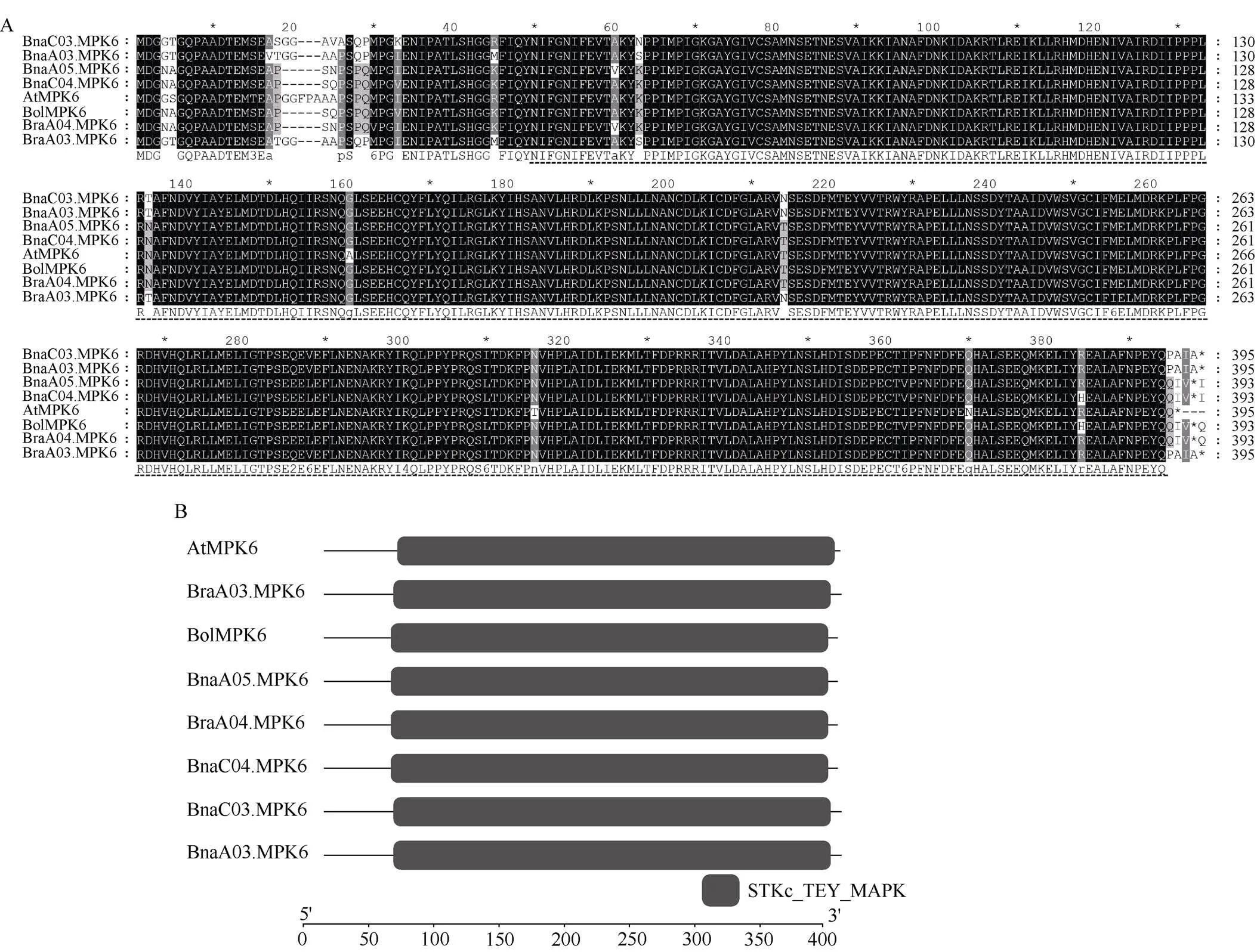
图2 拟南芥、甘蓝型油菜、白菜型油菜和甘蓝MPK6的蛋白序列和保守结构域
2.4 BnaMPK6功能缺失突变体的获得
为研究油菜的基因功能, 利用CRISPR/Cas9基因编辑技术对的4个拷贝进行敲除。基于Westar参考基因组, 将基因序列提交到CRISPR-P (http://crispr.hzau.edu.cn/ CRISPR2/), 筛选位置合适且得分高的sgRNA, 结果发现在a基因序列内有符合要求的靶点, 双靶点序列定位于和的第4个外显子以及和的第5个外显子上。高通量建库测序结果分析发现, T0代株系均为杂合突变, 其中和为杂合四拷贝突变体, 经过测序筛选出相对稳定遗传的T1代四突变体材料和, sgRNA处发生了碱基的缺失和插入, 编辑类型如图4-B。图4-C汇总了部分株系的的编辑情况。设计的靶点被成功编辑, 氨基酸序列翻译过程会提前终止(图4)。
2.5 BnaMPK6基因影响盐胁迫下植株长势
为探究基因正向调控盐水胁迫并影响植株发育, 进行了苗期不同浓度的盐水胁迫处理。苗期是植株发育的初期, 对盐水胁迫十分敏感, 研究盐水胁迫下苗期的植株发育对于植物的耐盐机制的研究具有重大意义。本试验选取饱满的突变体材料、和野生型种子进行萌发, 待种子萌发7 d后, 选取长势一致的幼苗, 在营养液中继续进行培养, 待长出4~5片真叶开始施加100 mmol L-1和150 mmol L-1浓度的NaCl溶液, 每组设置3个生物学重复, 处理7 d后, 拍照并对其长势进行观察, 统计株高、根长和鲜重。结果显示, 在100 mmol L-1和150 mmol L-1浓度的NaCl溶液处理下, 与野生型相比较,和植株受到胁迫, 植株矮小, 长势显著缓慢。尤其是地上部分, 随着盐浓度的升高, 株高逐渐受到抑制, 在150 mmol L-1NaCl溶液处理后,和株高比野生型降低约50%; 根长无显著变化, 长势基本一致; 鲜重明显降低, 仅是野生型植株的1/2 (表3)。由此推测对盐水响应敏感, 抑制其表达会影响盐胁迫下植株的生理发育(图5)。
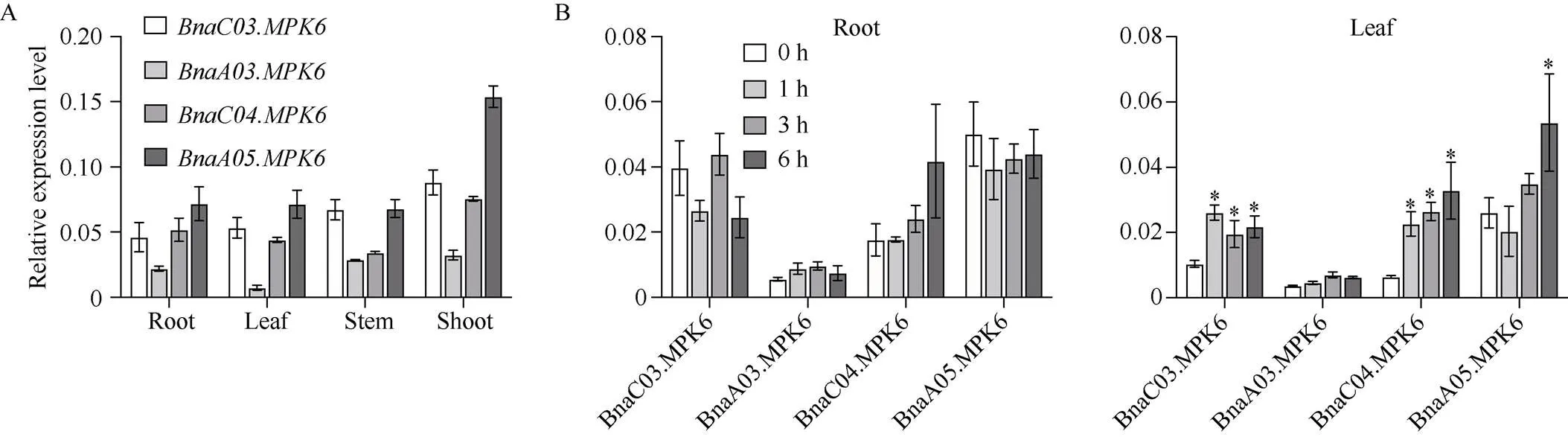
图3 BnaMPK6基因表达模式分析
A:在4个不同组织的表达模式; B、C:在不同的盐水处理时间下的表达模式。数据计算采取2-DDCt法, 每个数据3个重复,的相对表达量设定为200。以0 h处理为对照, 使用检验进行显著性分析, 星号(*)代表< 0.05时显著性情况。
A: the relative expression patterns of6 genes in four different tissues; B, C: the relative expression pattern ofgenes under different salt treatment time. The data was calculated in the light of the 2–DDCtmethod and the mRNA levels ofgenes set to 200. 0 hour is used as control, the asterisk (*) are significantly different (-test: *,< 0.05).
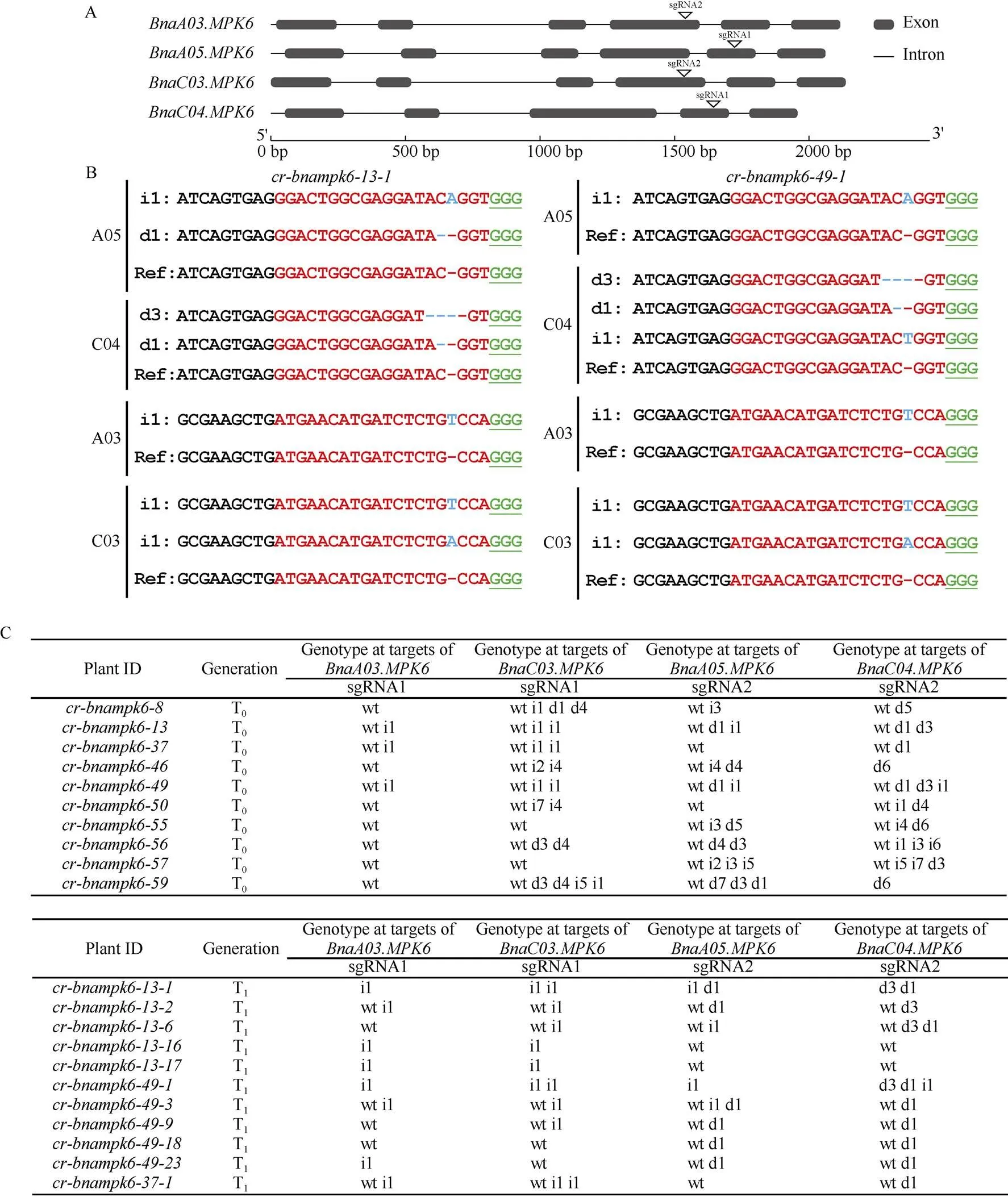
图4 cr-bnampk6突变体编辑情况
A:基因结构可视化和靶点位置; B:单株T1代基因靶点突变情况; C:株系T0代和T1代部分基因编辑类型统计。蓝色代表变异; 红色和绿色代表靶点序列。d#: 缺失#个碱基; i#: 插入#个碱基; wt: 野生型。
A: the visualization ofgene structure and the target sites; B: the mutation of targeted sites ofhomoulogous inT1plants; C: the partial editing results of T0and T1generation of. The blue parts indicate mutations; the red and green parts indicate target sites. d#: # of bp deleted from target site; i#: # of bp inserted from target site; wt: wild type.
2.6 BnaMPK6基因影响盐胁迫下植株体内活性氧和脯氨酸的含量
为探究盐水胁迫后抑制基因表达导致植物长势缓慢的可能因素, 测定了相关指标的变化。本试验对100 mmol L–1和150 mmol L–1浓度NaCl溶液处理后的植株进行活性氧、丙二醛和脯氨酸含量的测定表明, 在盐水胁迫处理后植株体内的活性氧大量积累, 相较于野生型,和叶片染色颜色加深, 表明功能缺失突变体植株体内有严重的氧化损伤发生。盐水胁迫后植株体内丙二醛(MDA)含量增, 说明膜质过氧化加剧, 与野生型做比较,和丙二醛(MDA)含量明显增加, 说明体内遭受了严重的盐胁迫并最终导致植株长势显著缓慢。与对照相比,和脯氨酸含量显著升高, 表明植物受到了盐水胁迫导致脯氨酸含量升高以响应渗透胁迫, 保护植物正常发育和生长(图6)。

表3 不同浓度NaCl溶液处理后cr-bnampk6表型分析
表中数据为平均值±标准差; 同一行的相同处理中不同小写字母表示在< 0.05水平存在显著差异(样本数= 20)。
Data indicate mean±SD; The different lowercase letters in the same treatment of the same line indicate the phenotypic difference is significant at the 0.05 probability level (=20). WT: wild type.
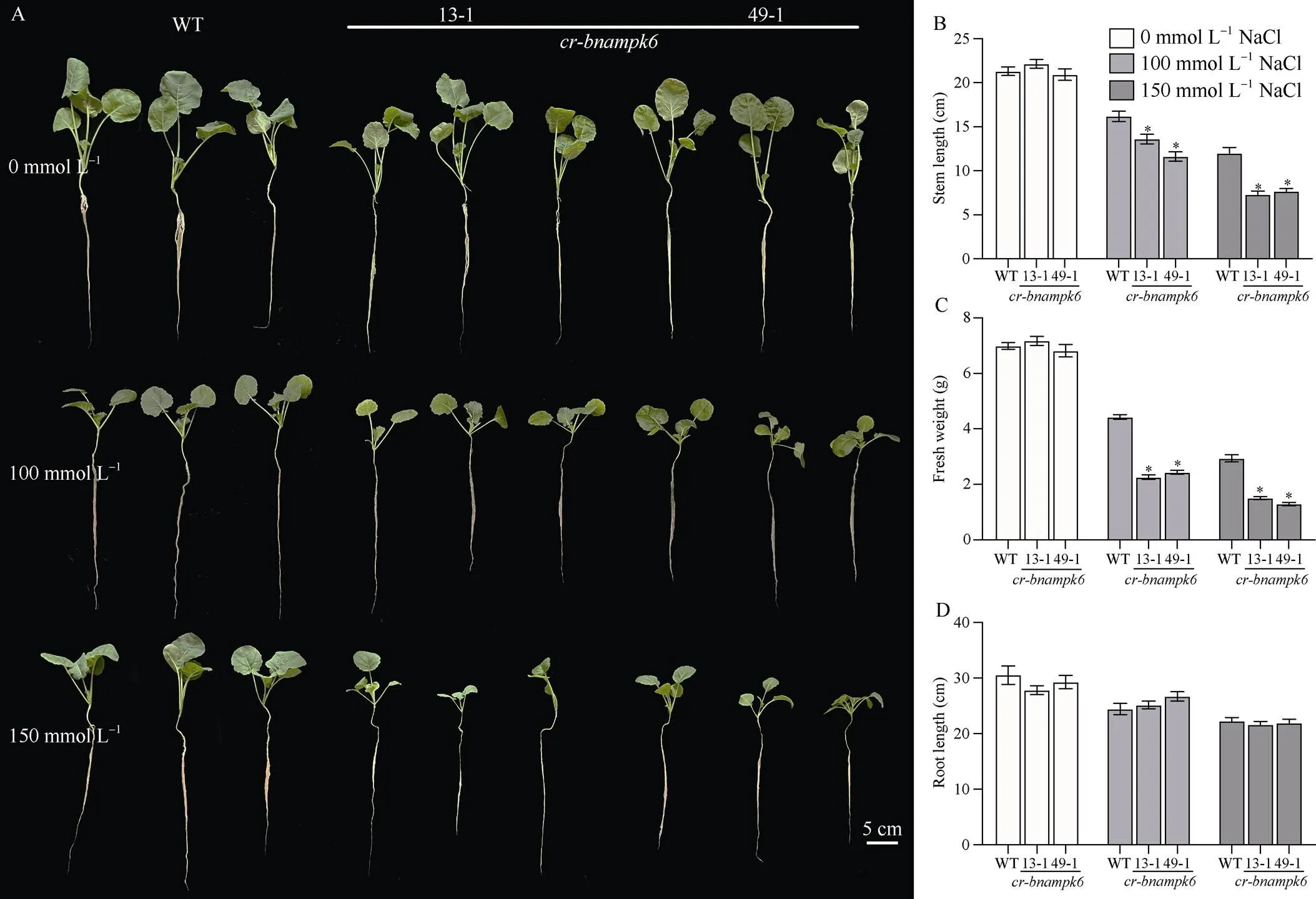
图5 不同浓度NaCl溶液处理后BnaMPK6基因耐盐性分析
A: WT和植株盐胁迫下的表型; B: WT和植株在盐胁迫下根长分析; C: WT和植株在盐胁迫下鲜重分析; D: WT和植株在盐胁迫下株高分析。以WT为对照, 使用检验进行显著性分析, 星号(*)代表< 0.05时显著性情况。
A:WT andplants phenotypes under salt stress; B:root length of WT andplants under salt stress; C:fresh weight of WT andplants under salt stress; D:plant height of WT andplants under salt stress. WT are used as control, the asterisk (*) are significantly different (-test: *,< 0.05).
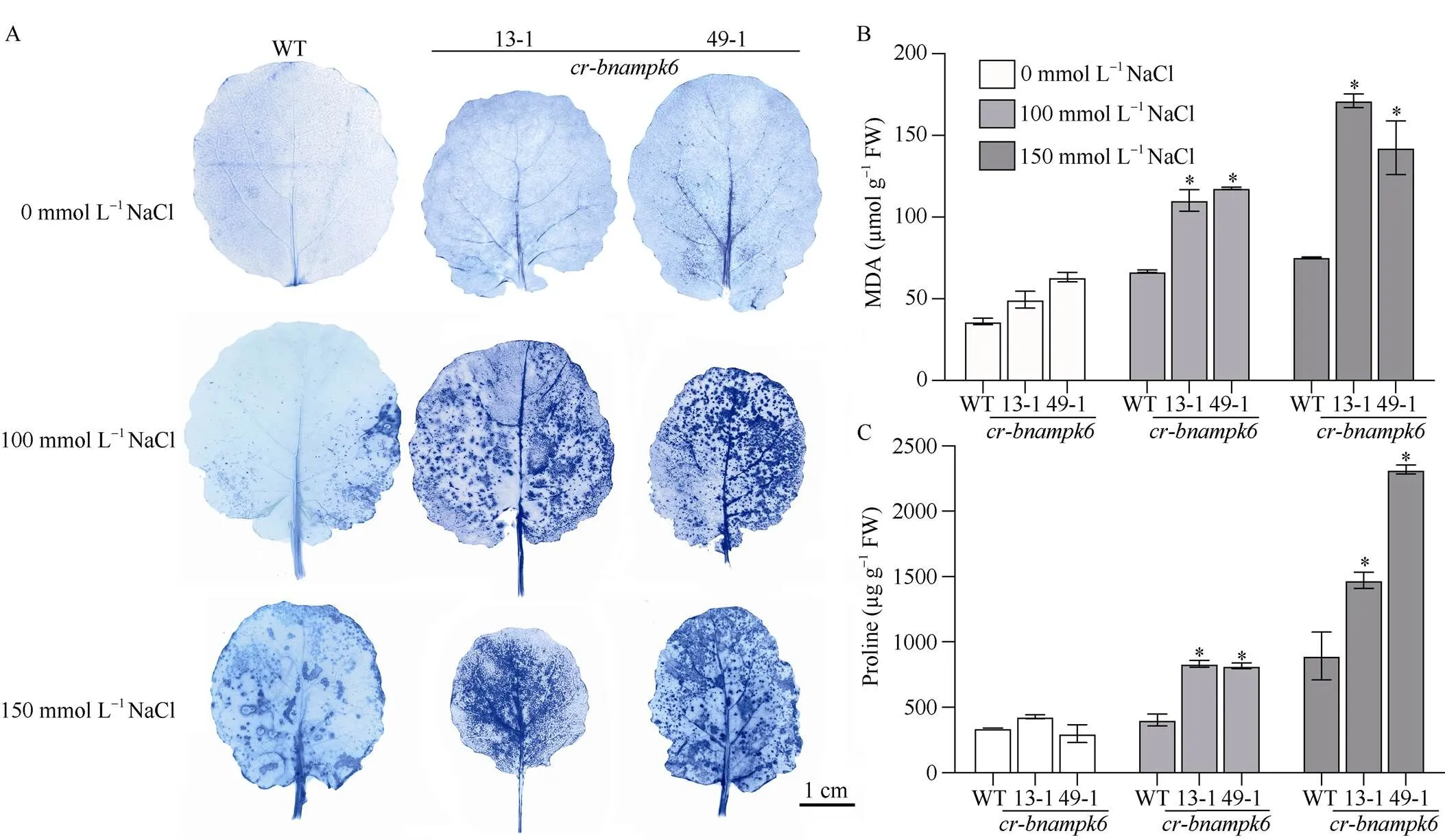
图6 不同浓度NaCl溶液处理后cr-bnampk6植株的活性氧和脯氨酸含量分析
A: NBT染色分析植株活性氧含量; B:植株丙二醛含量分析; C:植株脯氨酸含量分析。ROS: 活性氧; MDA: 丙二醛; Pro: 脯氨酸。以WT为对照, 使用检验进行显著性分析, 星号(*)代表< 0.05时显著性情况。
A:the accumulation of ROS ofplants by NBT; B:the accumulation of MDA ofplants; C:the accumulation of proline ofplants. ROS: reactive oxygen species; MDA: malondialdehyde; WT are used as control, the asterisk (*) are significantly different (-test: *,< 0.05).
3 讨论
近几年, CRISPR/Cas9基因编辑技术已经在各种农作物中得到广泛应用, 并且其创造的功能缺失突变体可以得到稳定遗传, 为基因功能研究提供了良好的技术支持。根据前人的研究, 发现利用基因编辑技术敲除基因可以创造出胁迫敏感突变体[30]。本研究利用Westar基因组中同源基因的保守序列选择了2个靶点, 分别靶向基因的4个同源拷贝的保守的外显子序列。高通量测序结果发现, 根据2个靶点设计的sgRNA实现了目标位点的编辑突变。本研究选择其中4个拷贝同时被编辑的单株和进行后续的试验, 并且在T1代获得了稳定遗传的纯合突变体材料。
土壤盐浓度过高对几乎所有重要的作物都会产生影响, 影响其生长发育[28], 因此植物进化了各种耐盐机制, 例如MAPK级联途径。丝裂原活化蛋白激酶(MAPK)在植物中广泛存在并表达, 在植物抵抗生物和非生物胁迫, 例如高盐、冷害、干旱和病害以及在植物胚胎发育等过程中发现了多个,其中对、和研究较多[19,30]。目前已经在多种作物中相继报道了敲除和过表达基因, 会影响植物应对不利环境[31-32], 例如在甘薯中过表达基因, 会提高植物对低温胁迫的耐受性[33]; 在拟南芥中敲除基因, 会降低植物对冷胁迫的耐受性[31]; 在UV-B处理下, 与野生型相比,敲除突变体表现出更强烈的耐受性[34]; 利用JA处理敲除突变体后, 植株根长长势缓慢, 表现出明显的不耐受性[35], 但是在甘蓝型油菜中的研究很少。因此, 研究油菜缺失基因的表达在盐水胁迫下相关的生理指标和生长情况的变化, 可以为研究植物的耐盐性提供新的遗传学基础。
本研究发现,基因在盐水处理后表达量上调。当盐水处理其功能缺失突变体后, 株系的长势以及鲜重等生理指标显著低于野生型。在和植株中还发现了氧化物质的积累和保护性物质的增加。在正常生长环境下, 植物会通过抗氧化机制和过氧化机制调节体内的ROS含量。但当植株处理逆境胁迫下时, 植物体内的抗氧化系统会被激活, 会导致活性氧(ROS)的积累, 进而导致氧化应激反应的发生和细胞损伤[36-37]。受到氧化胁迫后, 活性氧产生的损伤进而会导致植物体内的丙二醛(MDA)含量增加, 其含量的高低通常能代表植物细胞受到胁迫的水平[38]。但当植物处于胁迫环境中时, 植物为了维持自身生长发育的稳定性, 会积累大量的保护性物质来调节体内的渗透压, 脯氨酸可以作为渗透保护物质进行渗透调节, 维持植物在逆境下的稳定性, 因此脯氨酸的含量可以作为植物胁迫下的重要生理指标,可以在一定程度反映植物的抗逆性。可见,基因不仅会影响植物对盐胁迫的耐受性, 进而影响其生长发育, 还与植物体内抗氧化机制的活性有关。
4 结论
本研究利用CRISPR/Cas9技术有效编辑了基因的所有同源拷贝, 并验证了油菜缺失基因后, 会导致植物体内发生氧化损伤和保护性物质的增加, 进而抑制了植物苗期的生长发育。因此,基因在油菜中可能参与逆境胁迫过程, 正向调控植物的耐盐机制。本研究获得的盐敏感突变体材料, 对油菜抗逆相关的基础性研究和抗逆育种都具有重要意义。
[1] 李豪, 邹伟. 微生物在油菜秸秆资源化中的应用研究进展. 中国饲料, 2018, (23): 72–76.
Li H, Zou W. Progress on the applied research of microorganisms in the utilization of rape straw in China., 2018, (23): 72–76 (in Chinese with English abstract).
[2] 李利霞, 陈碧云, 闫贵欣, 高桂珍, 许鲲, 谢婷, 张付贵, 伍晓明. 中国油菜种质资源研究利用策略与进展. 植物遗传资源学报, 2020, 21: 1–19.
Li L X, Chen B Y, Yan G X, Gao G Z, Xu K, Xie T, Zhang F G, Wu X M. Proposed strategies and current progress of research and utilization of oilseed rape germplasm in China., 2020, 21: 1–19 (in Chinese with English abstract).
[3] Tyagi S, Kumar R, Kumar V, Won S Y, Shukla P. Engineering disease resistant plants through CRISPR-Cas9 technology., 2021, 12: 125–144.
[4] 单奇伟, 高彩霞. 植物基因组编辑及衍生技术最新研究进展. 遗传, 2015, 37: 953–973.
Shan Q W, Gao C X. Research progress of genome editing and derivative technologies in plants., 2015, 37: 953–973 (in Chinese with English abstract).
[5] 刘耀光, 李构思, 张雅玲, 陈乐天. CRISPR/Cas9植物基因组编辑技术研究进展. 华南农业大学学报, 2019, 40(5): 38–49.
Liu Y G, Li G S, Zhang Y L, Chen L T. Current advances on CRISPR/Cas9 genome editing technologies in plants., 2019, 40(5): 38–49 (in Chinese with English abstract).
[6] Mao Y, Zhang H, Xu N, Zhang B, Gou F, Zhu J K. Application of the CRISPR-Cas system for efficient genome engineering in plants., 2013, 6: 2008–2011.
[7] Xie K, Yang Y. RNA-guided genome editing in plants using a CRISPR-Cas system., 2013, 6: 1975–1983.
[8] Liang Z, Zhang K, Chen K, Gao C. Targeted mutagenesis inusing TALENs and the CRISPR/Cas system., 2014, 41: 63–68.
[9] Braatz J, Harloff H J, Mascher M, Steinb N, Himmelbach A, Jung C. CRISPR-Cas9 targeted mutagenesis leads to simultaneous modification of different homoeologous gene copies in polyploid oilseed rape ()., 2017, 174: 935–942.
[10] Fedoroff N V, Battisti D S, Beachy R N, Cooper P J, Fischhoff D A, Hodges C N, Knauf V C, Lobell D, Mazur B J, Molden D, Reynolds M P, Ronald P C, Rosegrant M W, Sanchez P A, Vonshak A, Zhu J K. Radically rethinking agriculture for the 21st century., 2010, 327: 833–834.
[11] Zhu J K. Salt and drought stress signal transduction in plants., 2002, 53: 247–273.
[12] Zhu J K. Genetic analysis of plant salt tolerance using., 2000, 124: 941–948.
[13] Qiu Q S, Guo Y, Dietrich M A, Schumaker K S, Zhu J K. Regulation of SOS1, a plasma membrane Na+/H+exchanger in,by SOS2 and SOS3., 2002, 99: 8436–8441.
[14] Zhu J K.Regulation of ion homeostasis under salt stress., 2003, 6: 441–445.
[15] Quan R, Lin H, Mendoza I, Zhang Y, Cao W, Yang Y, Shang M, Chen S, Pardo J M, Guo Y. SCABP8/CBL10, a putative calcium sensor, interacts with the protein kinase SOS2 to protectshoots from salt stress., 2007, 19: 1415–1431.
[16] Yu L, Nie J, Cao C, Jin Y, Yan M, Wang F, Liu J, Xiao Y, Liang Y, Zhang W. Phosphatidic acid mediates salt stress response by regulation of MPK6 in., 2010, 188: 762–773.
[17] Jiang C, Zhang X, Liu H, Xu J R. Mitogen-activated protein kinase signaling in plant pathogenic fungi., 2018, 14: e1006875.
[18] Asai T, Tena G, Plotnikova J, Willmann M R, Chiu W L, Gomez-Gomez L, Boller T, Ausubel F M, Sheen J. MAP kinase signalling cascade ininnate immunity., 2002, 415: 977–983.
[19] Kovtun Y, Chiu W L, Tena G, Sheen J. Functional analysis of oxidative stress-activated mitogen-activated protein kinase cascade in plants., 2000, 97: 2940–2945.
[20] Ichimura K, Casais C, Peck S C, Shinozaki K, Shirasu K. MEKK1 is required for MPK4 activation and regulates tissue-specific and temperature-dependent cell death in, 2006, 281: 36969–36976.
[21] Yoo S D, Cho Y H, Tena G, Xiong Y, Sheen J. Dual control of nuclear EIN3 by bifurcate MAPK cascades in C2H4signalling., 2008, 451: 789–795.
[22] Ouaked F, Rozhon W, Lecourieux D, Hirt H A. MAPK pathway mediates ET signaling in plants., 2003, 22: 1282–1288.
[23] Xu J, Li Y, Wang Y, Liu H, Lei L, Yang H, Liu G, Ren D. Activation of MAPK kinase 9 induces ethylene and camalexin biosynthesis and enhances sensitivity to salt stress in., 2008, 283: 26996–27006.
[24] Wang H, Ngwenyama N, Liu Y, Walker J C, Zhang S. Stomatal development and patterning are regulated by environmentally responsive mitogen-activated protein kinases in., 2007, 19: 63–73.
[25] Ichimura K, Mizoguchi T, Yoshida R, Yuasa T, Shinozaki K. Various abiotic stresses rapidly activateMAP kinases ATMPK4 and ATMPK6., 2000, 24: 655–665.
[26] Doyle J J. Isolation of plant DNA from fresh tissue., 1990, 12: 13–15.
[27] Cardoza V, Stewart C N. Increased Agrobacterium-mediated transformation and rooting efficiencies in canola (L.) from hypocotyl segment explants., 2003, 21: 599–604.
[28] Liu Q, Wang C, Jiao X, Zhang H, Song L, Li Y, Gao C, Wang K. Hi-TOM: a platform for high-throughput tracking of mutations induced by CRISPR/Cas systems.:, 2019, 62: 1–7.
[29] 王学奎, 黄见良. 植物生理生化实验原理和技术(第3版). 北京: 高等教育出版社, 2015. pp 1–324.
Wang X K, Huang J L. Principles and Techniques of Plant Physiological and Biochemical Experiments, 3rd edn. Beijing: Higher Education Press, 2015. pp 1–324 (in Chinese).
[30] Li C H, Wang G, Zhao J L, Zhang L Q, Ai L F, Han Y F, Sun D Y, Zhang S W, Sun Y. The receptor-like kinase SIT1 mediates salt sensitivity by activating MAPK3/6 and regulating ethylene homeostasis in rice., 2014, 26: 2538–2553.
[31] Ding Y, Li H, Zhang X, Xie Q, Gong Z, Yang S. OST1 kinase modulates freezing tolerance by enhancing ICE1 stability in., 2015, 32: 278–289.
[32] Liu C, Mao B, Yuan D, Chu C, Duan M. Salt tolerance in rice: physiological responses and molecular mechanisms., 2021,10: 13–25.
[33] 靳容, 刘明, 赵鹏, 张强强, 张爱君, 唐忠厚. 甘薯丝裂原活化蛋白激酶MPK6对低温胁迫的响应. 中国农业科学, 2021, 54: 4265–4273.
Jin R, Liu M, Zhao P, Zhang Q Q, Zhang A J, Tang Z H. IbMKP6, a mitogen-activated protein kinase, confers low temperature tolerance in sweetpotato., 2021, 54: 4265–4273 (in Chinese with English abstract).
[34] Gonzalez Besteiro M A, Bartels S, Albert A, Ulm R.MAP kinase phosphatase 1 and its target MAP kinases 3 and 6 antagonistically determine UV-B stress tolerance, in dependent of the UVR8 photoreceptor pathway., 2011, 68: 727–737.
[35] Takahashi F, Yoshida R, Ichimura K, Mizoguchi T, Seo S, Yonezawa M, Maruyama K, Yamaguchi-Shinozaki K, Shinozaki K. The mitogen-activated protein kinase cascade MKK3-MPK6 is an important part of the jasmonate signal transduction pathway in., 2007, 19: 805–818.
[36] Miller G, Suzuki N, Ciftci-Yilmaz S, Mittler R. Reactive oxygen species homeostasis and signaling during drought and salinity stresses., 2010, 33: 453–467.
[37] Kaye Y, Golani Y, Singer Y, Leshem Y, Cohen G, Ercetin M, Gillaspy G, Levine A. Inositol polyphosphate 5-phosphatase7 regulates the production of reactive oxygen species and salt tolerance in., 2011, 157: 229–241.
[38] Zhang Z, Liu H, Sun C, Ma Q, Bu H, Chong K, Xu Y. A C2H2 zinc-finger protein OsZFP213 interacts with OsMAPK3 to enhance salt tolerance in rice., 2018, 229: 100–110.
Genome editing ofgene by CRISPR/Cas9 for loss of salt tolerance inL
ZHANG Wen-Xuan, LIANG Xiao-Mei, DAI Cheng, WEN Jing, YI Bin, TU Jin-Xing, SHEN Jin-Xiong, FU Ting-Dong, and MA Chao-Zhi*
National Key Laboratory of Crop Genetic Improvement, Huazhong Agricultural University / National Engineering Research Center of Rapeseed / Hongshan Laboratory, Wuhan 430070, Hubei, China
L. is an important oil crop. MPK6 (Mitogen-Activated Protein Kinases 6) is activated by various stresses to control plant stress tolerance, which is the key gene in response to abiotic stresses. However, the function of MAPK6 instress tolerance is still unclear. Gene structure and protein sequence analysis showed thatgene structures were similar, and all proteins had the same STKc_TEY_MAPK domain in.To explore the potential role of, themutants were generated by CRISPR/Cas9 genome editing technology. Two quadruple mutants,and, were obtained.mutant lines were hypersensitive to salt treatment (100 mmol L–1and 150 mmol L–1), the growth of the mutant was strongly inhibited after salt treatment, and the plant height and fresh weight were significantly lower than those of wild-type plants, but there was no significant difference in root length. In addition, reactive oxygen species (ROS), malondialdehyde (MDA), and free proline were more accumulated inmutant lines. In conclusion, these results revealed thatgene positively regulated salt tolerance in, providing theoretical and technical supports for genetic improvement of salt tolerance in
L.;; CRISPR/Cas9; salt stress
10.3724/SP.J.1006.2023.24013
本研究由中央高校基本科研业务费专项基金(2662020ZKPY020)和国家重点研发计划项目(2020BBB061)资助。
This study was supported by the Fundamental Research Funds for the Central Universities (2662020ZKPY020) and the National Key Research and Development Program of China (2020BBB061).
马朝芝, E-mail: yuanbeauty@mail.hzau.edu.cn
E-mail: 18645272827@163.com
2022-01-10;
2022-06-07;
2022-07-08.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20220707.1125.003.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/)

