结合分子对接技术研究牦牛乳干酪苦味肽RK7和KQ7的α-淀粉酶抑制活性
李梦瑶,梁 琪,*,宋雪梅
(1.甘肃农业大学食品科学与工程学院,甘肃 兰州 730070;2.甘肃省功能乳品工程实验室,甘肃 兰州 730070)
α-淀粉酶是消化系统中的一种关键酶,抑制α-淀粉酶的活性可阻止碳水化合物水解为可吸收的单糖,降低餐后血糖水平,达到预防和治疗II型糖尿病(diabetes mellitus type 2,T2DM)的目的[1]。Ganesan[2]和Ummuhan[3]等发现阿卡波糖、米格列醇等药物可作为α-淀粉酶抑制剂用于治疗T2DM,但长期使用此类药物会导致腹痛腹泻等不良症状。近年来,国内外研究人员从天然食物蛋白水解物中提取的α-淀粉酶抑制肽成为治疗T2DM的研究热点,发现肽可通过占据α-淀粉酶的活性位点减少碳水化合物的消化[4-6]。
生物活性肽存在于蛋白质序列中,经酶解和微生物发酵等方式得以释放[7]。干酪成熟期间在发酵剂和凝乳酶的作用下,产生了具有多种功能的生物活性肽[8]。同时,干酪蛋白质的降解会产生大量苦味肽,研究表明肽的苦味源自Leu、Gln、Ile和Pro等疏水性氨基酸,这些疏水性氨基酸又与肽的生物活性密切相关[9-11]。牦牛乳干酪是高原地区居民传统食物,成熟时间可达6 个月至2 年,更易产生大量肽序列。课题组前期研究发现牦牛乳干酪苦味肽具有血管紧张素转化酶抑制活性、抗氧化活性及抑菌活性,然而关于牦牛乳干酪苦味肽的α-淀粉酶抑制活性及其作用机制的研究还处于空白阶段。
ExPASy-ProtParam、Innovagen和Biopep-UWM数据库等生物信息学工具和分子对接技术可以高效准确地预测肽的生物活性。根据肽的结构特征及基本理化性质研究其对生物活性的影响,并且能通过分子对接技术分析肽与受体蛋白之间的相互作用,阐明生物活性肽的作用机制[12-13]。杨保军等[14]利用生物信息学方法研究了牦牛乳源抑菌肽的分子机制。
本实验利用课题组前期鉴定的牦牛乳干酪苦味肽RK7和KQ7,通过生物信息学工具分析肽的理化性质及序列特征,结合分子对接阐明α-淀粉酶抑制肽的作用机制,并进行体外活性实验,为α-淀粉酶抑制肽的作用机制研究提供依据。
1 材料与方法
1.1 材料与试剂
RK7和KQ7由生工生物工程(上海)股份有限公司提供;α-淀粉酶、3,5-二硝基水杨酸 上海麦克林生化科技有限公司;阿卡波糖 石药集团欧意药业有限公司;可溶性淀粉、磷酸二氢钠、磷酸氢二钠、酒石酸钾钠、结晶苯酚、亚硫酸钠、氢氧化钠(均为分析纯) 天津市光复精细化工研究所。
1.2 仪器与设备
VersaMax酶标仪 美国Molecular Devices公司;VM-500S涡旋混合器 群安实验仪器有限公司;PHS-3C pH计 上海仪电科学仪器股份有限公司;FR224CN电子分析天平 奥豪斯仪器有限公司;HWS26恒温水浴锅上海一恒科技有限公司;Discovery Studio Client v16.1.0(DS) 美国Accelys公司;ChemBioDraw Ultra 11.0美国Cambridge Soft公司。
1.3 方法
1.3.1 生物活性及理化性质的预测
利用Innovagen、PepDraw和ExPASy ProtParam等生物信息学工具预测肽的分子质量、等电点、疏水性和疏水氨基酸比例等基本理化性质[15]。
1.3.2 配体和受体的准备
RK7与KQ7的结构式如图1所示。使用ChemBioDraw Ultra 11.0构建肽RK7和KQ7的二维结构,使用DS软件中CHARMm程序进行能量最小化处理,使用Prepare Ligands程序优化肽RK7和KQ7结构。

图1 RK7与KQ7的分子结构Fig.1 Molecular structures of RK7 and KQ7
α-淀粉酶三维结构图见图2。从RCSB Protein Date Bank(PDB,http://www.Pdb.org)中下载α-淀粉酶(PDB: 3BAJ)蛋白晶体的X射线三维结构[16]。使用Discovery Studio 2016软件打开PDB文件,删除其3D晶体结构的水分子和杂原子,通过Macromolecules模块中的“Clean Protein”优化蛋白质构象,去除蛋白多构象[17]。
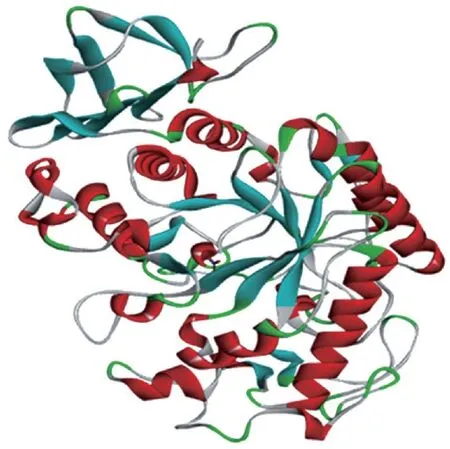
图2 α-淀粉酶三维结构Fig.2 Three-dimensional structure of α-amylase
1.3.3 分子对接
利用DS软件中的LibDock模块,选择阿卡波糖(阳性对照)的活性位点,坐标为(X:10.1522,Y:15.8388,Z:41.1172,R:10.2578 Å),将肽RK7和KQ7与α-淀粉酶(PDB:3BAJ)进行LibDock对接。
1.3.4 肽RK7和KQ7的合成及α-淀粉酶抑制活性的测定
采用固相合成法对肽RK7和KQ7进行合成,并通过液相色谱-质谱分析合成肽的纯度和分子质量。肽的合成委托生工生物工程(上海)股份有限公司完成。
α-淀粉酶抑制活性采用DNS比色法测定[18],并根据下式计算α-淀粉酶抑制率:
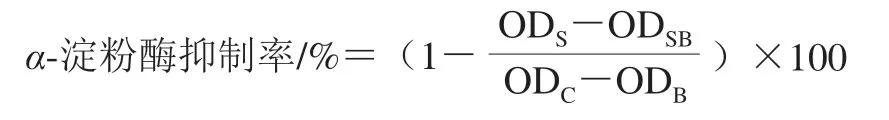
式中:ODS为样品组光密度值;ODSB为样品空白组光密度值;ODC为对照组光密度值;ODB为空白组光密度值。
1.4 数据处理
实验中每个样品重复3 次,使用SPSS进行数据统计,采用Origin 2018软件作图。
2 结果与分析
2.1 肽RK7与KQ7理化性质预测
如表1所示,RK7分子质量为875.07 Da,等电点为11.57,净电荷为3,RK7含有3 个疏水性氨基酸,分别为Pro(2)和Ile,疏水氨基酸比例为42.86%,疏水值为16.80 kcal/mol;KQ7分子质量为779.98 Da,等电点为9.84,净电荷为1,KQ7含有5 个疏水性氨基酸,分别为Val(2)、Pro(2)和Leu,疏水氨基酸比例为71.42%,疏水值为9.58 kcal/mol。肽的分子质量及氨基酸组成在α-淀粉酶抑制活性中起着至关重要的作用[19]。Miguel等[20]提出分子质量小于1 kDa的肽组分对α-淀粉酶的抑制作用更强。本实验的牦牛乳干酪苦味肽RK7和KQ7含有疏水性氨基酸Pro、Leu、Ile和Val。Ngoh等[19]研究斑豆蛋白水解产物发现疏水性氨基酸(Ala、Leu、Phe、Val、Pro和Gly)在抑制α-淀粉酶活性中占有重要地位。此外,具有芳香环的高分子质量氨基酸,如Arg、Phe、Trp和Tyr对α-淀粉酶活性位点的相互作用也至关重要[21]。

表1 肽的理化性质Table 1 Physicochemical properties of peptides
2.2 分子对接分析RK7、KQ7与α-淀粉酶相互作用
2.2.1 阿卡波糖与α-淀粉酶(3BAJ)
为了阐明肽和阿卡波糖与α-淀粉酶之间的相互作用,进行分子对接研究。由图3可知,阿卡波糖与α-淀粉酶(3BAJ)的氨基酸残基Glu233、Asp300、Thr163、Gln63和Lys200形成氢键作用力,其中氨基酸残基Asp300与阿卡波糖形成3 个氢键,键长分别为4.14、4.61 Å和5.04 Å;阿卡波糖与α-淀粉酶的氨基酸残基Ala198(5.15 Å)、His101(6.72 Å)和Leu162(4.84 Å)形成疏水相互作用,与Ala198(4.06 Å)、Asp197(4.26 Å/4.16 Å)和Gly104(3.04 Å)形成碳氢键,与His305、Trp58、Trp59、His201和Tyr62等形成范德华力。阿卡波糖通过与α-淀粉酶的相关氨基酸残基相互作用产生抑制效果。
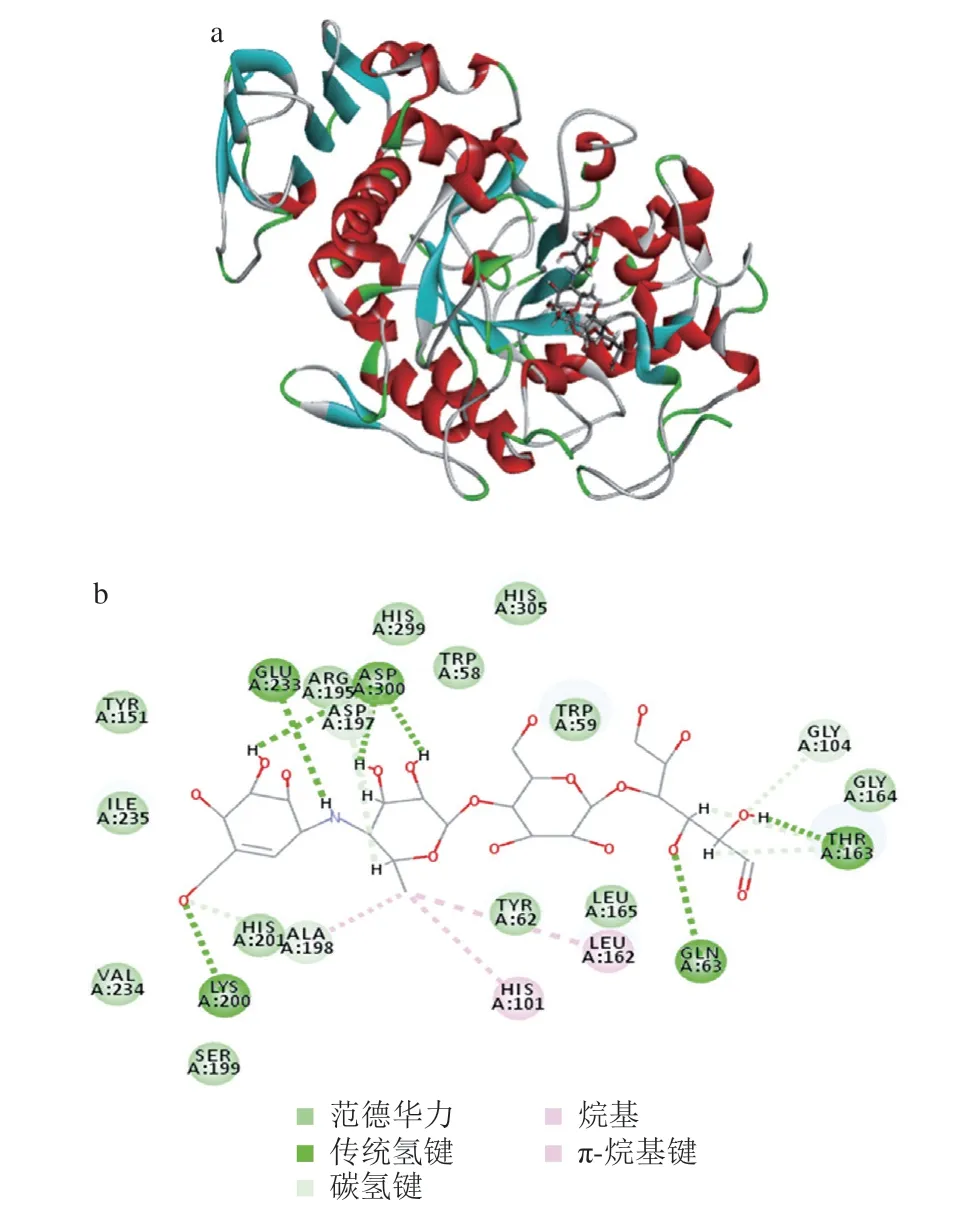
图3 阿卡波糖与α-淀粉酶的对接构象图Fig.3 Docking conformation of acarbose with α-amylase
2.2.2 RK7与α-淀粉酶(3BAJ)
将阿卡波糖作为阳性对照,分析RK7和KQ7在分子对接过程中与α-淀粉酶形成的结构,判断RK7和KQ7的α-淀粉酶抑制能力。如图4所示,RK7的Arg与3BAJ的Trp59形成氢键,键长为4.03 Å,RK7 N端第3位Lys与3BAJ的残基His305、Glu233和Ala307形成4 个氢键,其中与Glu233形成2 个较为紧密的氢键,键长分别为4.06 Å和3.11 Å,RK7的His和Ile分别与3BAJ的氨基酸残基Glu240和Gly304各形成1 个氢键,键长为5.43 Å和4.62 Å;与3BAJ氨基酸残基Tyr151(4.81 Å)、His305(3.77 Å)和Trp58(6.56 Å)形成属于疏水相互作用的π-π键和π-烷基键;与3BAJ的残基Glu240(8.18 Å)、Tyr151(4.36 Å)、His305(3.05 Å)、Asp197(4.98 Å/4.57 Å)、Asp300(7.87 Å)和Glu233(6.97 Å)形成静电相互作用。与3BAJ的氨基酸残基Gly306(4.04 Å)、His305(3.05 Å)、Aps300(4.54 Å)和Tyr62(3.79 Å)形成碳氢键,与Gly308、Gly309、Val234、Lys200、Ile235、Asp356、Thr163、Arg195、His101、Gln63、Leu165、Leu162、Glu60、Ala198、His201和Ser199之间形成范德华力。抑制剂通过与α-淀粉酶重要氨基酸的结合产生抑制效果,Ummuhan等[3]通过对含哌嗪和苯并咪唑结构的α-淀粉酶抑制剂的研究,发现抑制剂与残基Glu233、Asp197、Asp300、Trp58和Tyr62存在相互作用。分子对接显示出α-淀粉酶的残基在抑制机理中的重要地位。

图4 RK7与α-淀粉酶的对接构象图Fig.4 Docking conformation of RK7 with α-amylase
2.2.3 KQ7与α-淀粉酶(3BAJ)
如图5所示,KQ7中N末端Lys与3BAJ的氨基酸残基Thr163形成2 个氢键,键长分别为3.32 Å和5.12 Å,N端第2位Val分别于3BAJ残基Thr163和Gln63形成2 个氢键,键长为3.93 Å和5.34 Å,C端第2位氨基酸Pro与3BAJ残基Glu233形成氢键,键长为4.74 Å,C末端Gln与3BAJ氨基酸残基Lys200形成2 个氢键,键长为3.66 Å和5.13 Å,与Ile235形成1 个氢键,键长为3.78 Å;与3BAJ的氨基酸残基Ala198(4.71 Å)、Tyr62(3.78 Å)、Trp58(6.39 Å)、Trp59(4.70 Å)、Leu165(3.53 Å)、His305(5.22 Å)和Leu162(6.33 Å)形成疏水相互作用的烷基键和π-烷基键;与Asp147形成静电相互作用,键长为4.28 Å;与3BAJ残基Val234(4.04 Å)、His201(4.56、5.11 Å和4.52 Å)、Glu233(5.62 Å)、Ala198(3.47 Å)、Trp59(4.34 Å)、Thr163(3.03 Å)和Asp147(4.96 Å)之间形成碳氢键;与Ser199、Gly306、Ala307、Asp300、Arg195、Tyr151、Asp197、His101、Cys101、Asn105、Gly104、Ala106、Val107、Gly164和Ile148形成范德华力。本实验分子对接结果显示氢键、疏水相互作用、静电相互作用和范德华力等相互作用的存在,降低了α-淀粉酶的活性。Liu Fufeng等[22]发现氢键在配体与受体蛋白的相互作用中起重要作用,能形成更加稳定的结构,疏水相互作用、静电相互作用和范德华力均有效促进了多肽与受体蛋白的结合,达到降低酶活的目的。

图5 KQ7与α-淀粉酶的对接构象图Fig.5 Docking conformation of KQ7 with α-amylase
2.2.4 RK7、KQ7与阿卡波糖的对比分析
由于抑制剂与酶结合期间氢键稳定,因此氢键的数量和键长决定了肽的亲和力。由表2可以看出,RK7、KQ7和阿卡波糖分别与α-淀粉酶形成10、17 个和12 个氢键。其中RK7与Glu233形成2 个作用较强的氢键,距离为4.06 Å和3.11 Å,KQ7与Glu233形成1 个氢键和1 个碳氢键距离分别为4.74 Å和5.62 Å,阿卡波糖与Glu233形成1 个氢键距离为4.95 Å;RK7与3BAJ的残基Trp59形成的氢键键长为4.03 Å,KQ7与Trp59形成碳氢键,距离为4.34 Å,RK7形成的作用更强;KQ7和阿卡波糖与3BAJ的Lys200、Thr163和Gln63共同形成氢键相互作用,其中KQ7与Thr163形成4 个氢键,但阿卡波糖与之形成的氢键距离较短,作用更强。Subhiksha等[23]从鹰嘴豆蛋白水解物中得到的肽KMTAGSGVT(KT9)与3BAJ形成12 个氢键,对比发现RK7与KT9共同作用于3BAJ残基His305、Gly306和Gly304形成氢键,KQ7和KT9与残基His201和Lys200形成氢键。除拥有共同的氢键作用外,KQ7和阿卡波糖与残基Ala198和Leu162形成疏水相互作用,与Ser199、Arg195、Tyr151和Gly164形成范德华力,KQ7和阿卡波糖与3BAJ的结合有更多相同的相互作用,因此,KQ7与阿卡波糖具有一致的α-淀粉酶抑制机制。KQ7是三者中与3BAJ发生最多相互作用的抑制剂,但与3BAJ的重要氨基酸残基结合较少,抑制效果低于RK7和阿卡波糖。抑制剂与α-淀粉酶关键氨基酸Glu233、Asp300、Lys200、Gln63和Thr163形成氢键相互作用可以更加牢固的将抑制剂固定在活性部位[24]。Nurul等[25]从罗勒种子中得到的肽ACGNLPRMC可与α-淀粉酶的重要残基Glu233、Asp300、Lys200、Gln63发生相互作用产生抑制效果。RK7和KQ7与3BAJ的氨基酸结合情况可以看出,RK7和KQ7与3BAJ的重要残基形成了氢键、静电相互作用、疏水相互作用和范德华力等相互作用,使之与3BAJ形成稳定的复合物结构。KQ7与3BAJ的氨基酸残基形成的氢键数目更多,其与α-淀粉酶的结合更加稳定。研究发现Glu233、Asp300和Asp197是α-淀粉酶活性位点上的重要氨基酸[26]。RK7与这3 种氨基酸均产生了相互作用,而KQ7仅与Glu233相互作用,因此,RK7的活性强于KQ7。分子对接揭示了RK7、KQ7和阿卡波糖与3BAJ的作用机制,发现KQ7和阿卡波糖与3BAJ的结合有更多一致的相互作用,因而KQ7的作用机制与阿卡波糖一致。

表2 3BAJ氨基酸结合对照Table 2 Comparison of RK7,KQ7 and acarbose binding to α-amylase
肽抑制酶的作用机制之一是肽与底物竞争酶的催化位点,中断酶与底物之间的相互作用,从而达到抑制效果[27]。α-淀粉酶与底物相互作用的氨基酸残基包括Asp165、Asp197、Lys200、Glu233、Asp236、Asp300、Trp59、Tyr62、His101、Pro163、Ile235、Tyr58、His299、His305和Ala307[28]。RK7和KQ7通过与这些氨基酸残基的结合占据底物与3BAJ的结合位点抑制α-淀粉酶的活性。
2.3 肽RK7、KQ7的合成与α-淀粉酶活性验证
2.3.1 肽RK7、KQ7的合成
如图6所示,RK7理论分子质量为875.09 Da,实际分子质量为875.1 Da;KQ7理论分子质量为779.98 Da,实际分子质量为779.80 Da,分子质量均与理论值接近,RK7与KQ7纯度分别为98.18%和99.93%,合成结果达到预期标准。将经固相合成的牦牛乳干酪苦味肽RK7和KQ7用于α-淀粉酶活性的验证。

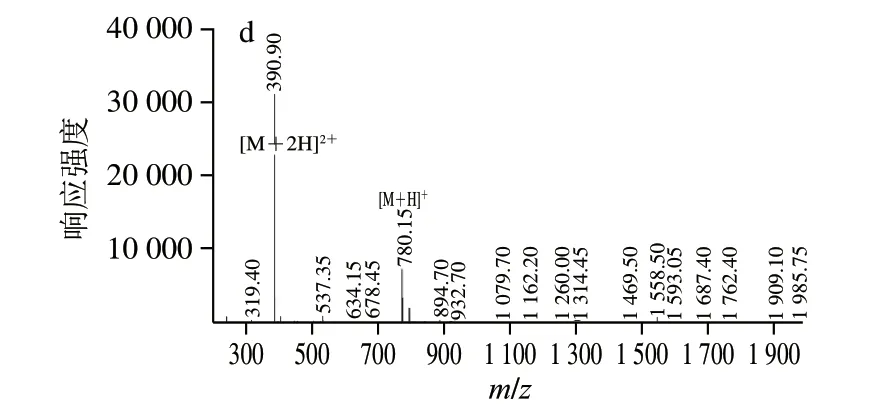
图6 合成肽RK7与KQ7的谱图Fig.6 HLPC chromatograms and mass spectra of synthetic peptides RK7 and KQ7
2.3.2 肽RK7与KQ7的体外活性验证
通过DNS比色法对苦味肽RK7和KQ7进行α-淀粉酶抑制率的测定,结果如图7所示。RK7、KQ7和阿卡波糖的α-淀粉酶抑制率随质量浓度的增加而增加,当质量浓度达到0.6 mg/mL时,抑制率的增速有所降低。阿卡波糖的α-淀粉酶抑制率曲线为y=-50.14x2+118.03x+14.39,R2=0.9763,质量浓度在0.2~1.0 mg/mL范围变化时,阿卡波糖的抑制率为37.08%~82.02%;肽RK7的α-淀粉酶抑制率曲线为y=-33.91x2+84.36x+18.74,R2=0.9926,肽质量浓度在0.2~1.0 mg/mL范围变化时,RK7的α-淀粉酶抑制率为34.18%~69.62%,KQ7的α-淀粉酶抑制率曲线为y=-17.30x2+57.87x+12.89,R2=0.9942,肽质量浓度在0.2~1.0 mg/mL范围变化时,KQ7的α-淀粉酶抑制率为24.19%~53.23%。根据抑制率曲线求得RK7与KQ7的IC50分别为0.45 mg/mL和0.86 mg/mL,阿卡波糖为0.36 mg/mL。RK7活性强于KQ7,且RK7与KQ7均强于Ramadhan等[29]发现的肽YSFR(10.82 mg/mL)和PGGP(4.23 mg/mL)具有良好的抑制活性。与分子对接结果一致,RK7与α-淀粉酶关键氨基酸结合数量更多,因此比KQ7具有更强α-淀粉酶抑制活性,但两者均低于阿卡波糖的α-淀粉酶抑制活性。体外活性验证实验进一步证明RK7和KQ7均对α-淀粉酶具有良好的抑制作用,且α-淀粉酶抑制效果随质量浓度的增加而增强。不同的氨基酸组成和肽序列影响肽的生物活性,疏水性氨基酸对α-淀粉酶的抑制活性有较大影响[30]。由于RK7与α-淀粉酶的重要氨基酸结合数相对较多,因此,RK7相较于KQ7具有更高的抑制活性。

图7 肽质量浓度对α-淀粉酶抑制率的影响Fig.7 Effect of peptide concentration on percentage of α-amylase inhibition
3 结论
通过生物信息学工具及体外活性实验探讨牦牛乳干酪苦味肽RK7和KQ7的α-淀粉酶抑制活性。与Biopep-UWM数据库对比发现肽RK7和KQ7是2 种新型的α-淀粉酶抑制肽,分子质量分别为875.07 Da和779.98 Da,均为低分子肽,肽序列中含有疏水性氨基酸Leu、Pro、Val和Ile有利于α-淀粉酶的抑制活性;分子对接揭示肽RK7和KQ7与α-淀粉酶形成氢键、范德华力、静电和疏水等相互作用,使之与α-淀粉酶稳定结合,达到抑制效果;体外活性实验证明RK7的IC50为0.45 mg/mL,KQ7的IC50值为0.86 mg/mL,α-淀粉酶抑制活性:RK7>KQ7。分子对接等生物信息学方法可高效快速预测肽的生物活性,并准确揭示牦牛乳干酪苦味肽RK7和KQ7抑制α-淀粉酶的作用机理,为牦牛乳源低分子肽生物活性的研究提供重要参考。

