理性设计和定点突变对提高海藻糖合酶TreSI热稳定性的影响
张丽霞,黄奕晴,吴嘉蕾,李元顺,王飞
(江西农业大学生物科学与工程学院/江西省农业微生物资源开发与利用工程实验室,江西 南昌 330045)
海藻糖(trehalose)是由2个吡喃环葡萄糖分子以α,α-1,1键连接而成的非还原性双糖[1]。海藻糖具有对生物大分子良好的保护作用,当细胞处于饥饿、干燥、高渗透压、脱水等胁迫环境时,海藻糖可以维持蛋白质的稳定,防止细胞膜损伤,从而提高生物的抗逆性[2]。海藻糖合酶(trehalose synthase,EC 5.4.99.16)可以麦芽糖为底物,通过转糖基作用催化生成海藻糖[3]。这一反应过程相对简单,转化率高,所用底物的成本比较低廉,因此在酶法生产海藻糖上受到广泛关注[4]。
1995年,Nishimoto等[5]首次从Pimelohactersp. R48中纯化得到海藻糖合酶,该酶最适反应pH值为7.5,最适反应温度为20 ℃,在较低的催化温度下,海藻糖得率为81.8%。来源于ThermusaquaticusATCC 33923的海藻糖合成酶活性为135 U·mg-1,最适反应pH值为6.5,最适反应温度为65 ℃,在30~40 ℃条件下,产率达80%。该酶在80 ℃保存60 min活性稳定,显示出较强的热稳定性[6],另一个由古细菌(Thermuscaldophilus)GK24产生的海藻糖合酶有着相似的比活性和温度稳定性[7]。来源于Pseudomonassp. F1的海藻糖合酶活性为41.2 U·mg-1,最适反应温度为45 ℃,在55 ℃条件下保存1 h依然热稳定[8]。其他人的研究结果显示:来源于G-细菌如Pseudomonasputida、Mycobacteriumsmegmatis、Arthrobacteraurescens、Enterobacterhormaechei、CorynebacteriumglutamicumATCC13032[9]等的海藻糖合酶大多对温度敏感,在60 ℃条件下保存1 h基本丧失酶活性;而来源于古细菌如Picrophilustorridus、Thermusthermophilus、Thermobifidafusca、Deinococcusradiodurans、Meiothermusruber等的海藻糖合酶则都具有较强的耐热性[10-11],Agarwal等[11]从温泉宏基因组中克隆得到的海藻糖合酶TreM也具有较高的热稳定性和催化能力。来源于G+菌如StreptomyceslavendulaeX33、Streptosporangiumroseum的海藻糖合酶在氨基酸序列上与嗜热古细菌的海藻糖合酶具有较高的一致性,但都对温度敏感[12]。
本实验室从黏细菌(Myxococcussp.)V11基因组中克隆到1个新的海藻糖合酶基因TreSI(GenBank:MW217472),其酶活性为172 U·mg-1,高于已报道的其他海藻糖合酶,并且不产生副产物葡萄糖,但该酶对热不稳定,在50 ℃条件下保存2 h后残留酶活性仅为20%[13]。蛋白质突变可导致结构变化,影响蛋白质功能,在蛋白质工程、药物设计或优化行业中,突变通常用于提高蛋白质的稳定性或在保持稳定性的同时改变蛋白质的性质。单一氨基酸突变可以改变蛋白的折叠自由能,从而改变蛋白质的结构稳定性[14]。许多研究已经使用机器学习来构建预测单点突变对蛋白质稳定性影响的模型。基于蛋白结构,利用三维结构信息提取二级结构、化学组成和原子间相互作用等特征,构建预测模型在提高蛋白的稳定性方面已取得不错的效果。如Liao等[15]利用PoPMuSiC对来源于Aspergillusniger的α-L-鼠李糖苷酶进行预测,并通过定点突变获得了热稳定性提高的突变体K573V-E631;Woo 等[16]利用SDM对来源于Pseudomonasputida的Baeyer-Villiger单加氧酶突变后的自由能进行预测,通过点突变获得了在氧化和热应激下变得更加稳定的突变株E6BVMO-C302L;Dotsenko等[17]利用mCSM工具预测Penicilliumcanescens的木聚糖酶E突变后的稳定性,并通过定点突变,使改良后的Xyl E变种的熔化温度(Tm)值增加了1.1~3.1 ℃,70 ℃半衰期增加了1.3~1.7倍。本文以几种不同的工具联合预测,并通过定点突变对TreSI相关的氨基酸残基进行改造,以期获得热稳定高的突变子,从而扩大其应用范围。
1 材料与方法
1.1 试验材料与仪器
克隆菌株EscherichiacoliDH5α、表达宿主菌株E.coliBL21(DE3)、表达菌株E.coliBL21(DE3)-pET29a-TreSI均保藏于江西省农业微生物资源开发与利用工程重点实验室。
LB培养基:酵母粉 5.0 g·L-1,胰蛋白胨 10.0 g·L-1,NaCl 10.0 g·L-1,pH值7.0~7.2。
质粒提取试剂盒、DNA凝胶回收试剂盒购自北京索莱宝科技有限公司;定点突变试剂盒Fast Mutagenesis System购自北京全式金生物科技有限公司。核酸测序及引物合成委托湖南擎科生物科技有限公司进行。仪器有PCR 扩增仪(Biometre T1)、Beckman台式高速冷冻离心机(Allegra X-22R)等。
1.2 氨基酸序列分析及突变位点的选择
将TreSI氨基酸序列在NCBI数据库中进行比对,查询并下载相似度高的参考蛋白PDB(protein data bank)文件。将参考蛋白以PoPMuSiC在线软件对每个氨基酸突变后的去折叠自由能变化(ΔΔG=ΔGWT-ΔGMT)进行预测[18],筛选对参考蛋白热稳定性有较大影响的氨基酸位点[19]。以BioEdit7.0软件分析TreSI的非保守区氨基酸位点。将选择的位点提交至DUET(http://biosig.unimelb.edu.au/duet/stability)进行在线预测[20],筛选对TreSI热稳定性影响最大的突变类型。以Alpha fold2进行蛋白三维结构模拟,以PyMoL软件进行可视化分析。
1.3 PCR介导的定点突变
PCR介导的定点突变按Fast Mutagenesis System试剂盒说明书进行,设计突变引物(表1),以pET-29a(+)-TreSI质粒为模板,进行重叠延伸PCR扩增,PCR体系:2×Trans Start Fast Pfu PCR Super Mix 12.5 μL,正向引物1.0 μL,反向引物1.0 μL,质粒DNA 1.0 μL,ddH2O 9.5 μL。PCR程序:95 ℃ 2 min,95 ℃ 20 s,55 ℃ 20 s,72 ℃ 1 min,30个循环。PCR产物以DMT酶(改良型的DpnⅠ限制性核酸内切酶)消化,去除非突变型质粒模板后,转化至E.coliDH5α感受态细胞[21]。提取突变克隆的质粒送湖南擎科生物科技有限公司测序,将突变位点正确的质粒热激转化至E.coliBL21(DE3)感受态细胞,构建突变表达菌株。

表1 TreSI定点突变PCR扩增所用引物Table 1 Primers used for TreSI mutations
1.4 重组酶的表达与纯化
将表达菌株接种至800 mL LB液体培养基(含有终浓度为50 mg·mL-1卡那霉素),37 ℃、180 r·min-1培养至D600为0.6,加入终浓度为0.2 mmol·L-1IPTG,16 ℃诱导培养24 h。6 000 r·min-1离心10 min,收集菌体,以PBS缓冲液(50 mmol·L-1,pH6.0)重悬洗涤菌体,超声波破碎,4 ℃、12 000 r·min-1离心20 min。上清液以Ni2+-NTA进行亲和层析,以含200 mmol·L-1咪唑的PBS 缓冲液(50 mmol·L-1,pH6.0)洗脱。SDS-PAGE电泳检测TreSI蛋白的表达和纯化情况,测定蛋白含量采用考马斯亮蓝染色法[22]。
1.5 酶活性测定
将重组酶加入至含0.03%(质量分数)麦芽糖的50 mmol·L-1PBS缓冲液(pH6.0)中,于40 ℃条件下反应20 min。加入1 mol·L-13,5-二硝基水杨酸(DNS)1 mL,沸水浴5 min,以蒸镏水稀释2倍后测定D540,计算酶活性。酶活性定义:40 ℃、pH 6.0反应条件下,以0.03%麦芽糖为底物,每分钟催化消耗 1 μmol 麦芽糖所需的蛋白量(mg)为一个活性单位(U)[21]。
1.6 重组酶的最适反应条件和热稳定性测定
最适温度测定:分别在30、35、40、45和50 ℃测定酶活性,把最适温度下的酶活性定为100%,计算其他温度下的相对酶活性。
最适pH测定:在PBS缓冲液(pH值5.0~7.5)中加入终浓度为0.03%麦芽糖底物,在最适温度条件下反应后测定酶活性,把最适pH值的酶活性定为100%,计算其他pH值的相对酶活性。
热稳定性测定:将稀释后的酶液在50和60 ℃条件下水浴保温,每隔一定时间取等量的样品,测定酶活性,将初始酶活性定义为100%,计算热处理不同时间的残余酶活性。
2 结果与分析
2.1 突变氨基酸位点的选择
将TreSI氨基酸序列提交至NCBI,选择protein data bank数据库进行比对,结果显示TreSI与来源于Thermobaculumterrenum的海藻糖合酶TtTs(PDB:5X7U)一致性为68%。将5X7U提交至PoPMuSiC,对其所有氨基酸位点突变后的折叠自由能变化预测,折叠自由能变化最高的突变位点见表2。

表2 参考蛋白TtTs自由能变化预测结果Table 2 Predicted stability change(ΔΔG)of TtTs
PoPMuSiC预测的结果中,当突变型蛋白的折叠自由能与野生型蛋白的差值ΔΔG(ΔGWT-ΔGMT)小于0时,表示该位点突变后稳定性将增加。溶剂可及性越低,则表示该位点位于蛋白分子内部。在表2所列出的氨基酸位点中,Glu244是活性中心残基之一,Glu15、Asp172、Gly30、Asp382都位于保守区(图1),这几个位点的突变可能会导致酶活性发生较大改变,因此不对它们进行突变。

图1 不同来源的海藻糖合酶的保守区Fig.1 Concerved regions of trehalose synthases from different sources DrTs:来源于Deinococcus radiodurans的海藻糖合酶;TcTs:来源于Thermomonospora curvata的海藻糖合酶;MtTs:来源于Mycobacterium tuberculosis的海藻糖合酶;MsTs:来源于Mycobacterium Smegmatis的海藻糖合酶;TtTs:来源于Thermobaculum terrenum的海藻糖合酶。DrTs:Trehalose synthase from Deinococcus radiodurans;TcTs:Trehalose synthase from Thermomonospora curvata; MtTs:Trehalose synthase from Mycobacterium tuberculosis;MsTs:Trehalose synthase from Mycobacterium Smegmatis;TtTs:Trehalose synthase from Thermobaculum terrenum.
蛋白质三维结构显示,TreSI的催化域由(α/β)8桶状结构组成,C端的结构域由6个反向平行的折叠片组成,N端由3个反向平行的折叠片和1个螺旋以及多个无规则卷曲形成的Loop环绕在活性中心周围。Loop区是蛋白中最为柔性的部分,其空间构象变化大,正好反映了其氨基酸的低保守性。因此选择这些柔性的氨基酸残基作为突变位点可以避免改变酶蛋白的保守结构,而且突变这些不稳定的氨基酸也可能会增加蛋白的稳定性[23]。在TreSI中,与TtTs上Gly515、Gln481、His549对应的3个氨基酸位点分别是Gly515、 Glu481、Ser549,这3个氨基酸残基都位于蛋白的C端结构域,远离活性中心(图2),这几个位点的突变对活性的影响可能较小,因此不作选择。与Asp193对应的氨基酸残基为Asn193,位于活性中心背面的2个螺旋相连的Loop附近,Glu100和Gln412都位于活性中心附近,Arg149位于底物入口处的Loop上,这4个位点的突变可能对稳定性有较大的影响,因此对这4个位点进行进一步分析。
将Alpha fold2模拟的TreSI结构pdb文件提交至http://biosig.unimelb.edu.au/duet/stability,以基于特定环境变量替换(site directed mutator,SDM)和突变终止扫描矩阵(mutation cutoff scanning matrix,mCSM),以及两者联合(Duet)进行预测,考察Asp193、Glu100、Ala412和Arg149几个位点饱和突变对热稳定性的影响,列出对热稳定性有促进作用的突变结果见表3。预测结果显示R149L、E100I和N193E的突变对热稳定性的提高有促进作用,而Gln412的所有突变均显示有减弱热稳定性的作用(未列出)。因此,本研究选取R149、E100和N193这3个位点进行突变。
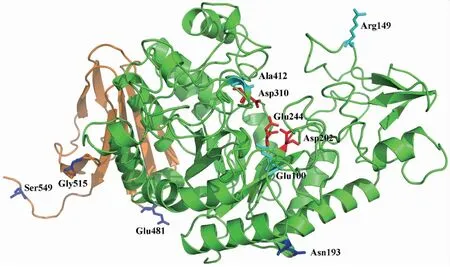
图2 几个氨基酸残基在蛋白三维结构中的位置Fig.2 The position of several amino acid residues in the protein’s three-dimensional structure 红色氨基酸残基为活性中心,绿色结构域为催化域,橙色结构域为Domain C。The red amino acid residues is the active center,the green Domain is the catalytic domain,and the orange domain is the Domain C.

表3 基于SDM、mCSM、Duet的突变体自由能变化预测结果Table 3 Predicted stability change(ΔΔG)based on SDM,mCSM and Duet
2.2 突变子的表达与纯化
以野生型pET-29a(+)-TreSI质粒为模板进行PCR扩增,参照定点突变试剂盒Fast Mutagenesis System说明书构建突变表达载体,转化至E.coliBL21(DE3)以构建表达菌株。将所设计的突变体全部同步进行诱导表达后,收集菌体,超声破碎后通过Ni-NTA进行纯化,SDS-PAGE电泳检测结果如图3所示。11个突变重组蛋白都可溶性表达并得到纯化,相对分子质量大小与野生型一致。
2.3 突变体的初步筛选
取纯化的突变体在40 ℃、pH6.5反应条件下进行酶活性测定,以野生型TreSI单位酶活性为100%,比较突变体与野生型TreSI的相对酶活性,结果如图4所示。TreSI的Glu100位点的3个突变体E100I、E100M和E100L的单位酶活性分别是野生型的66.49%、64.39%和61.20%,该位点邻近活性中心,可能参与了与底物结合作用,它的改变对活性的影响非常关键。R149L和N193E突变体的酶活性与野生型相比无显著差异,其余突变体的单位酶活性略有下降。
2.4 TreSI突变体和野生型的最适反应温度和最适反应pH值
如图5可知:突变体R149L和N193E的最适反应温度与野生型相比并没有发生变化,均为40 ℃,表明突变的氨基酸对TreSI的最适反应温度并没有影响。反应温度为35~45 ℃,TreSI野生型和2个突变体都保持了很高的酶活性(>70%)。TreSI突变体和野生型的最适pH值均为6.0,说明突变的氨基酸并没有影响TreSI的最适pH值,这可能是因为酶的pH值依赖性主要是由活性中心氨基酸残基的酸解离常数(pKa)值决定,而本研究所选择的突变氨基酸位点都在离TreSI的活性中心较远的位置。

图3 TreSI野生型与突变型SDS-PAGE电泳图Fig.3 SDS-PAGE electrophoresis of pure enzyme with site mutation M1、M2. 低分子量蛋白标准;Total. 上清液;WT. 野生型TreSI。下同。M1 and M2. Low molecular weight protein marker;Total. Total protein of E.coli BL21(DE3)harboring pET29a-tresI;WT. Wild type TreSI.The same as follows.
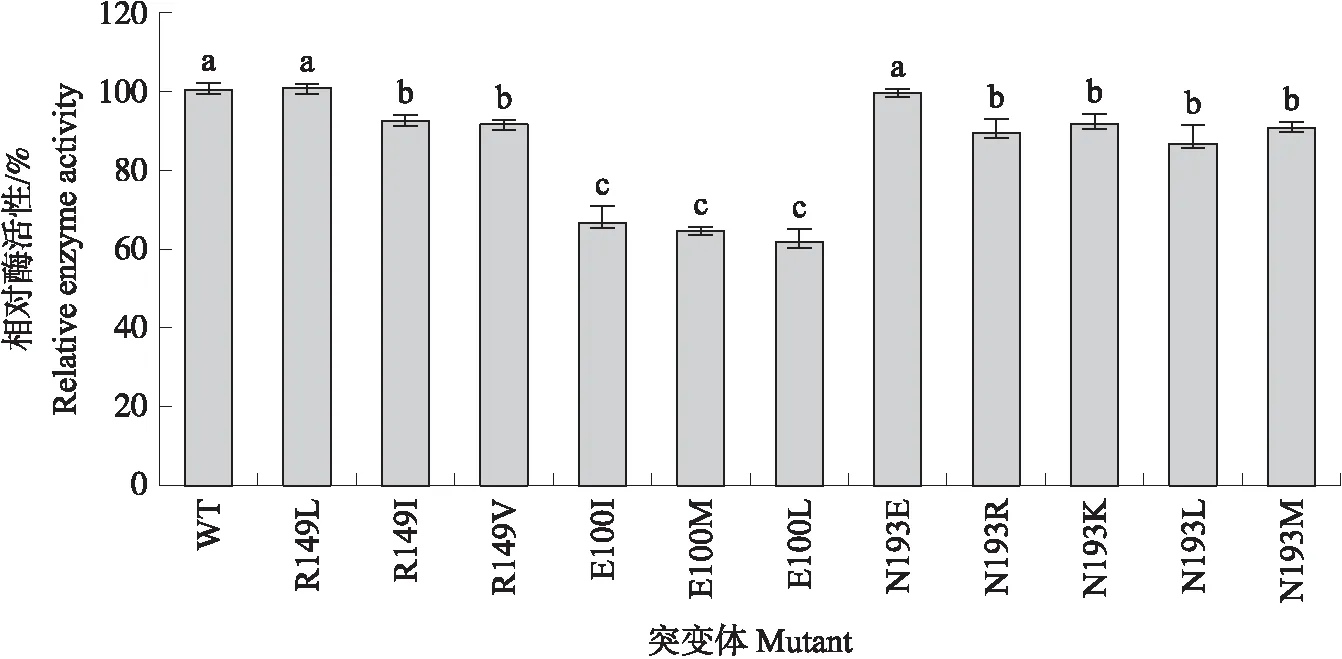
图4 TreSI突变型与野生型单位酶活性的比较Fig.4 Comparison of specific enzyme activity between mutant and wild type不同小写字母表示在0.05水平上差异显著。The different small letters mean significant differences at 0.05 level.
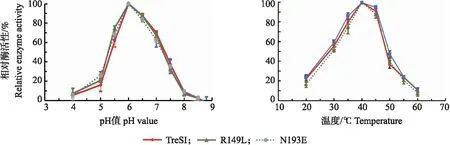
图5 TreSI和突变体(R149L和N193E)的最适反应条件Fig.5 Optimum reaction conditions of TreSI and mutants(R149L and N193E)
2.5 TreSI突变体和野生型的热稳定性比较
将TreSI和突变体R149L、N193E在50、60 ℃保温不同时间之后,测定它们的残余酶活性,结果如图6所示。在50 ℃处理1 h后,突变体R149L和N193E的相对酶活性分别为38.9%和70.9%,是野生型(28.3%)的1.37和2.5倍;在50 ℃处理2 h后,突变体R149L和N193E的相对酶活性分别为17.7%和50.2%,而野生型的酶活性则基本丧失;在60 ℃处理1 h后,突变体R149L和N193E的相对酶活性分别为24.4% 和43.7%,是野生型(10.1%)的2.41和4.32倍;在60 ℃处理2 h后,突变体N193E的相对酶活性为25.8%,野生型的酶活性彻底丧失,说明突变体R149L和N193E的热稳定性比TreSI野生型提高了。
突变体R149L和N193E热稳定性提高的原因可能是因为TreSI的149号精氨酸突变成亮氨酸,193号天门冬酰胺突变成谷氨酸,2个位点的氨基酸等电点分别由10.76下降到5.98(R149L),由5.41下降到3.22(N193E),从而导致蛋白的总体表面电势由-14下降到-17(图7)。2个位点的突变更有助于蛋白表面形成水化膜,保持胶体可溶性状态,从而提高其稳定性[24-25]。

图6 TreSI和突变体(R149L和N193E)的热稳定性Fig.6 Thermostability of TreSI and mutants(R149L and N193E)
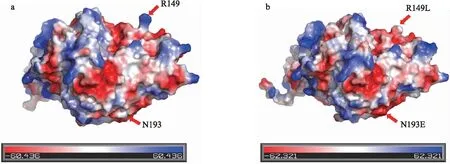
图7 TreSI和突变体(R149L和N193E)的表面静电势Fig.7 Electrostatic potential of TreSI and mutants(R149L and N193E)a. 野生型Wild type;b. 突变体Mutant(R149L and N193E)
3 讨论与结论
增强天然蛋白的热稳定性是蛋白质工程中的重要课题。目前国内外针对海藻糖合成酶进行理性设计的研究还不多,多数研究还集中于产海藻糖合成酶菌株的筛选,开发新酶以及菌株产酶发酵条件的优化等方面。Wang等[10]通过截短与融合方法对来源于Thermusthermoophilus的耐高温海藻糖合成酶(TtTS)的C-末端片段进行功能验证,发现TtTS特殊的C-末端对热稳定性具有重要的保护作用。Chou等[26]通过定点突变将Picrophilustorridus所产的海藻糖合酶(PtTS)的第503位天门冬酰胺替换成脯氨酸,突变体N503P-PtTS在65 ℃条件下保持2 h后,酶活性比野生型提高了39%。Chen等[27]以来源于Thermomonosporacurvata的海藻糖合酶(TcTS)为材料,以4种环化载体SpyTag-SpyCatcher、SpyTag-KTag、SnoopTag-SnoopCatcher、SnoopTagJR-DogTag构建N端与C端环化的重组蛋白,4种环化的TcTS变异体在55 ℃条件下的半衰期增加了2~3倍,且稳定性比G78D、V289L、G322A、I323L突变体提高更显著。本研究以来源于Myxococcussp. V11的海藻糖合酶TreSI为材料,率先采用PoPMuSiC,结合SDM、mCSM以及两者联合(Duet)预测的方法对TreSI进行理性设计,成功地提高了TreSI的热稳定性。后续工作中,将采用联合突变、融合古细菌来源的C端结构域等手段,进一步提高TreSI的热稳定性,使之符合工业化应用。本研究所获得的热稳定性更高的突变体R149L和N193E增加了TreSI的工业应用前景,为工业应用提供了新的酶资源,同时也为其他工业酶的理性设计提供了参考。

