肉中猪源性成分Real-time PCR定量检测技术
翟晓虎,李翎旭,陈小竹,蒋怀德,贺卫华,姚大伟
肉中猪源性成分Real-time PCR定量检测技术

1江苏农牧科技职业学院,江苏泰州 225300;2南京农业大学动物医学院,南京 210095
【】建立一种快速、准确的肉中猪源性成分定量检测方法。【】首先从GenBank数据库中筛选猪特异性的微卫星DNA,根据微卫星DNA核酸序列设计引物,对常见10种动物基因组DNA进行PCR扩增,通过有无扩增产物判断筛选的微卫星DNA对猪源性成分的特异性。然后根据微卫星DNA核酸序列,设计特异性引物和探针,建立猪源性成分Real-time PCR检测方法,采用双标准曲线分别对猪源性成分和总动物源性成分进行定量,计算猪源性成分的百分含量。【】筛选到猪特异性微卫星DNA(Accession EF172428),根据其序列设计的引物SEQ-sus2-F/R只能从猪基因组DNA中扩增出目的条带,其他动物的基因组均无目的条带扩增。建立的Real-time PCR检测方法灵敏度为0.02 ng/25 μL反应体系。该方法能够准确检测出混合DNA样品中猪源性成分和混合肉样品中猪源性成分,百分误差分别约为1.32%和1.06%—7.12%。【】本研究利用Real-time PCR技术建立的定量猪源性成分的检测方法可以用来检测猪源性成分在混合样品中的百分含量。
肉;猪;动物源性成分;Real-time PCR;定量
0 引言
【研究意义】动物源性商品广泛分布于食品、药品、保健品、化妆品、饲料和服饰等各种产品中。在动物源性商品的消费过程中经常会遇到不法商家为追逐经济利益在食品加工过程中对动物源性原料以次充好、掺杂使假等欺诈甚至违法行为。此外,动物源性商品还涉及一些民族的宗教信仰问题(如在清真食品中掺入猪肉)。因此,建立一种快速、准确的动物源性成分鉴别方法对于提高动物源性食品安全监管水平,确保食品质量安全具有积极的作用。【前人研究进展】目前,动物源性成分鉴别主要根据不同物种之间基因的差异,设计特异性引物,进行PCR检测。线粒体DNA具有较好的物种特异性、多拷贝性,在加工过程中不完全降解,成为动物源性成分鉴别的首选靶基因[1-3]。然而由于不同物种、不同组织中线粒体数量各有差异[4],在Real-time PCR定量检测时检测结果的Ct值与线粒体数量、动物组织的质量没有明确的对应关系,因此,以线粒体DNA作为靶基因进行定量检测会造成偏差,失去定量意义[5]。基因组DNA在每个细胞中的数量恒定,采用基因组DNA中特异的单拷贝基因建立Real-time PCR检测方法可以实现动物源性成分的定量检测[6]。然而,目前可利用的具有物种差异性的靶基因较少,常用的单拷贝基因有生长激素基因、复制蛋白A1基因、转化生长因子等[5,7-8],仍无法满足多种动物源性成分的检测需要。【本研究切入点】筛选高保守性、拷贝数恒定、低降解程度的目标基因来设计物种特异性引物和探针,将是动物源性成分Real-time PCR定量检测技术面临的首要问题。微卫星DNA大量存在于动物基因组中,其侧翼序列保守,在组织中的拷贝数恒定,片段较短,尽管样品降解仍能进行PCR扩增,这些特点非常适合作为Real-time PCR定量检测的靶基因[9-10]。【拟解决的关键问题】在筛选猪特异性微卫星DNA的基础上,设计引物和探针,绘制标准曲线,建立Real-time PCR定量方法,从而实现猪源性成分的定量检测。
1 材料与方法
试验于2021年在南京农业大学动物医学院临床应用分子生物学研究室进行。
1.1 材料与试剂
1.1.1 生鲜肉样 随机采购南京市各大超市、集贸市场生鲜猪肉样品(n=24),牛肉样品(n=24),羊肉样品(n=24),鸡肉样品(n=24),鸭肉样品(n=6),鹅肉(n=6),兔肉(n=6),驴肉(n=2),马肉(n=2),鹿肉(n=3),置于含有冰块的泡沫箱中保存,立即带回实验室-20℃保存,用于基因组DNA提取。
1.1.2 主要试剂 Taq DNA聚合酶、蛋白酶K、DL2000 DNA marker等购自天根生化科技(北京)有限公司;Rox Reference Dye Ⅱ、Premix Ex Taq购自宝生物工程(大连)有限公司;裂解液(10 mmol·L-1Tris-Cl,pH 8.0;20 mmol·L-1EDTA,pH 8.0;0.5% SDS);TE(10 mmol·L-1Tris-Cl,pH 8.0;1 mmol·L-1EDTA,pH 8.0)。
1.2 方法
1.2.1 基因组DNA提取 采用蛋白酶K消化和苯酚抽提的方法提取高分子质量DNA[11]。称取每种动物生鲜肉样0.3 g置于5 mL离心管中,用眼科剪剪碎成约1 mm大小,加入10倍体积(m/V)的裂解液,用IKA T 25数显型分散机均质。取400 μL均质液于新的离心管中,37℃温育1 h。加入2 μL 20 mg·mL-1的蛋白酶K,置于55℃水浴中消化3 h。加入等体积的Tris平衡酚,振荡混匀,12 000 r/min离心5 min。将上层水相转移至新的离心管中,加入0.2倍体积10 mol·L-1醋酸铵和2倍体积的无水乙醇,上下颠倒混匀,12 000 r/min离心5 min,收集沉淀,用70%的乙醇洗涤DNA沉淀1次,加入TE溶解DNA,用Nanodrop测定DNA浓度,分装后-80℃保存备用。
1.2.2 引物设计 在NCBI中查找猪的微卫星DNA,进行Blast比较分析,筛选种内通用、种间特异的微卫星DNA。采用Primer Premier 5.0软件根据选择的微卫星DNA序列设计猪特异性引物SEQ-sus1-F/R、SEQ-sus2-F/R(表1)。根据PCR特异性鉴定结果,采用Beacon designer 7软件重新设计引物和探针SEQ-sus2-probe-F/R、Sus-Taqman-probe(表1),另外根据脊椎动物保守序列设计动物通用的引物和探针SEQ-common296-F/R、Common-Taqman-probe(表1),引物和探针均由英潍捷基(上海)贸易有限公司合成。
1.2.3 PCR检测引物的特异性 引物的特异性采用PCR凝胶电泳进行验证,用设计好的引物以各种动物的基因组DNA为模板进行PCR检测,设置无模板阴性对照(NTC)。25 μL PCR反应体系:模板(200 ng·μL-1)1 μL,上、下游引物(2 μmol·L-1)各2.5 μL,dNTP(2.5 mmol·L-1)2 μL,MgCl2(25 mmol·L-1)1.5 μL,Taq(5 U·μL-1)0.3 μL,补水至25 μL。采用降落PCR反应程序:预变性94℃,3 min;变性94℃,15 s;退火65℃,15 s,每个循环降1℃;延伸72℃,30 s;10个循环。变性94℃,15 s;退火55℃,15 s;延伸72℃,30 s;25个循环。最后延伸72℃,5 min。PCR产物采用2%的琼脂糖凝胶电泳(内含Goldview 30 μL·L-1)进行检测,恒压120 V,电泳约30 min,置于Bio-Rad凝胶成像系统内拍照观察。

表1 引物、探针信息
1.2.4 引物和探针的特异性验证 用设计好的引物和探针以各种动物的基因组DNA为模板进行Real- time PCR检测,设置无模板阴性对照(NTC)。25 μL反应体系:模板(200 ng·μL-1)2.5 μL,上、下游引物(2 μmol·L-1)各2.5 μL,探针(2 μmol·L-1)2.5 μL,Rox Reference Dye Ⅱ 0.5 μL,Premix Ex Taq(2×)12.5 μL,补水至25 μL。反应程序:预变性95℃,30 s;变性95℃,5 s;退火60℃,34 s;重复40个循环。
1.2.5 Real-time PCR退火温度的优化 采用连续10倍稀释的猪基因组DNA作为模板(200、20、2和0.2 ng·μL-1)对退火温度(61.5、60、58.5、57和55℃)进行优化。比较各试验条件下的扩增效率、决定系数、最小Ct值(Ctmin)、非特异性扩增的Ct值、空白对照的Ct值等,筛选最佳的退火温度。
1.2.6 Real-time PCR引物和探针浓度的优化 采用连续10倍稀释的猪基因组DNA作为模板(200、20和2 ng·μL-1)对引物浓度(150、250和350 nmol·L-1)和探针浓度(150、200和250 nmol·L-1)进行优化。比较各试验条件下的扩增效率、决定系数、最小Ct值(Ctmin)、非特异性扩增的Ct值、空白对照的Ct值等,筛选最佳的引物和探针浓度。
1.2.7 标准曲线的绘制及混合样品中猪源性成分检测方法 基因组DNA提取方法同1.2.1,测定DNA的浓度,将DNA连续10倍稀释作为标准品,以每个反应中DNA质量的常用对数作为标准曲线的横坐标,以Ct值作为标准曲线的纵坐标,绘制标准曲线。猪源性成分检测的标准曲线回归方程s= ks·s+bs。s:以猪特异性引物进行Real-time PCR测定的Ct值;s:猪源性成分质量的常用对数;ks:以猪源性成分检测建立的回归方程的斜率;bs:以猪源性成分检测建立的回归方程的截距。样品总动物源性成分检测的标准曲线回归方程:T= kT·T+bT。T:以动物通用引物进行Real-time PCR测定的Ct值;T:总动物源性成分质量的常用对数;kT:以总动物源性成分检测建立的回归方程的斜率;bT:以总动物源性成分检测建立的回归方程的截距。混合样品中猪源性成分的含量(%)=10S/10T×100= 10(XS-XT)×100。
1.2.8 混合样品制备及猪源性成分含量检测 混合DNA样品制备,提取猪、牛、鸡基因组DNA,测定DNA浓度,然后按照质量1﹕1﹕1比例混合,按照上述方法,测定混合DNA样品中猪源性成分的含量。另外称取一定质量的猪肉和牛肉制备成混合肉样品,检测者在不知道猪肉比例的情况下,提取样品中的DNA,采用上述方法检测猪肉DNA在总DNA中的比例。
2 结果
2.1 猪特异性微卫星DNA筛选
猪的引物SEQ-sus1-F/R PCR特异性检测结果如图1所示,该引物对除了在猪的模板中扩增出目的条带外,在鸭、兔和驴的模板中也有扩增条带,说明该引物对猪的特异性不高。
猪的引物SEQ-sus2-F/R PCR特异性检测结果如图2所示,只有猪的模板中扩增出了约262 bp的目的片段,牛、羊、鸡、鸭、鹅、兔、驴、马和鹿的模板没有扩增出条带,说明该微卫星DNA只在猪基因组中存在,对猪具有特异性,根据该位点设计的引物SEQ-sus2-F/R可以特异地检测出猪源性成分。

1:DNA Marker;2:猪;3:牛;4:羊;5:鸡;6:鸭;7:鹅;8:兔;9:驴;10:马;11:鹿;12:空白对照。下同
2.2 猪源性成分Real-time PCR检测
2.2.1 引物和探针特异性检测 采用引物SEQ-sus2- probe-F/R和探针Sus-Taqman-probe对相同浓度不同动物的基因组DNA模板进行检测,结果如图3所示,猪基因组DNA模板扩增的Ct值为23(图3虚线箭头),其他物种的Ct值大于35(图3空心箭头)或未检测到扩增信号(图3实心箭头),NTC未检测到信号。

图2 猪引物SEQ-sus2-F和SEQ-sus2-R特异性检测结果

虚线箭头:猪DNA;空心箭头:其他动物DNA;实心箭头:NTC
2.2.2 退火温度优化 猪的引物SEQ-sus2-probe-F/ R Real-time PCR退火温度优化结果如表2所示,在扩增效率、空白对照扩增方面,各退火温度之间没有显著差异。综合分析比较,选择较高的决定系数、较小的Ctmin、非特异性扩增Ct值>35的退火温度,退火温度60℃同时满足上述条件,因此,后续试验中采用60℃进行Real-time PCR检测。
2.2.3 引物和探针浓度的优化 不同的引物和探针浓度优化结果如表3所示,综合分析,选择较高的扩增效率和决定系数,较小的最小Ct值,非特异性扩增Ct值>35,空白对照无扩增的引物和探针组合(表3),后续试验中采用探针浓度200 nmol·L-1,引物浓度250 nmol·L-1进行Real-time PCR检测。
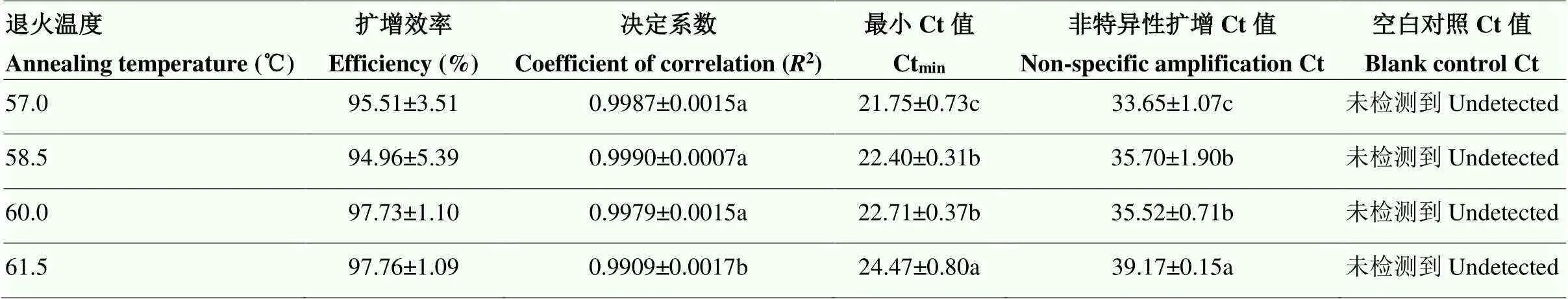
表2 猪引物退火温度的优化
不同字母表示差异显著(<0.05)。下同 Different lowercase letters indicate significant difference (<0.05). The same as below
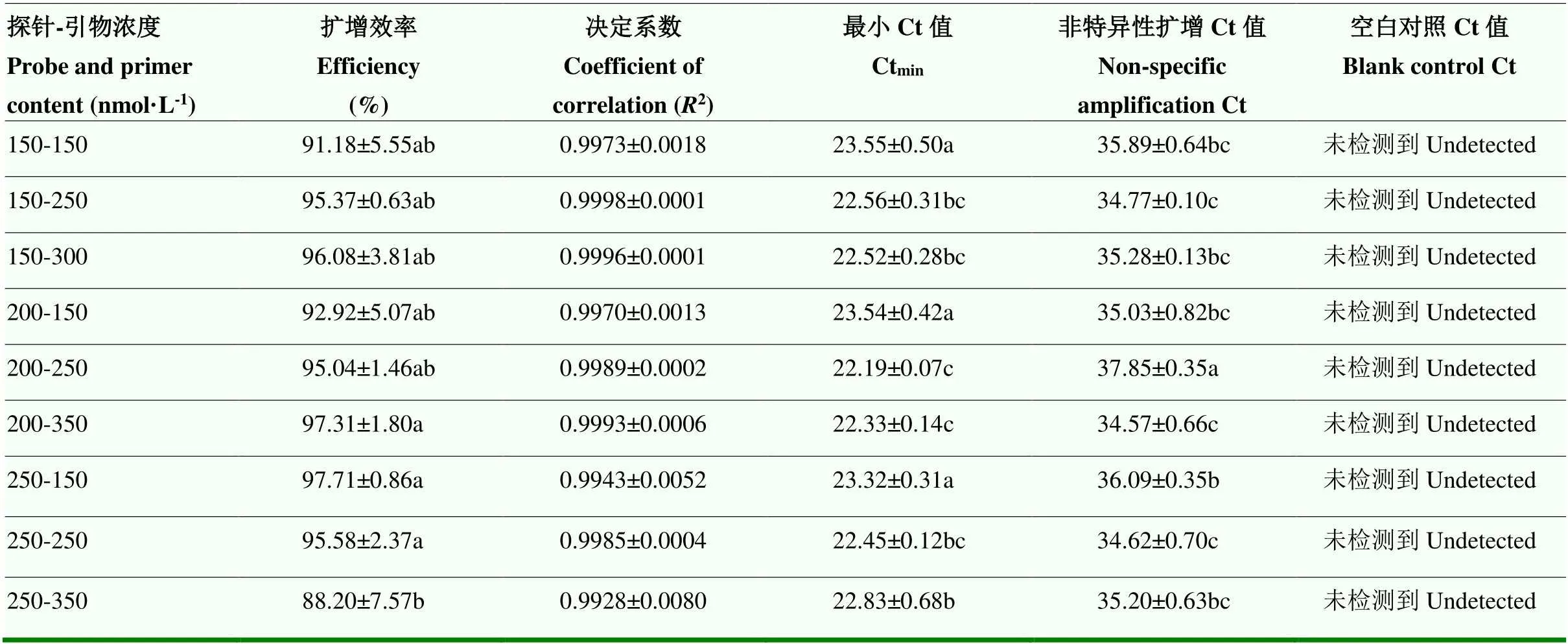
表3 猪引物和探针浓度优化
2.3 动物源性成分Real-time PCR检测
采用引物SEQ-common296-F/R对不同物种的基因组DNA模板进行检测,所有物种均有扩增,且Tm相似,NTC未检测到信号(图4)。综合分析,选择较高的扩增效率和决定系数,较小的Ctmin,非特异性扩增Ct值>35,空白对照无扩增的引物和探针组合,退火温度58.5℃具有较好的扩增效果,其次是60℃,考虑与猪源性成分扩增同时进行,因此选择与猪源性成分扩增相同的退火温度60℃(表4)。采用引物浓度350 nmol·L-1,探针浓度200 nmol·L-1(表5)进行Real-time PCR检测。
2.4 基因组DNA标准品的制备及标准曲线的绘制
采用连续稀释猪基因组DNA作为标准品,用猪的引物和探针进行Real-time PCR扩增,绘制标准曲线如图5所示,标准曲线为=-3.29+29.04,2=0.9985。DNA含量在0.02—200 ng/25 μL反应体系范围内标准曲线呈良好的线性关系,检测的灵敏度达0.02 ng/25 μL反应体系。
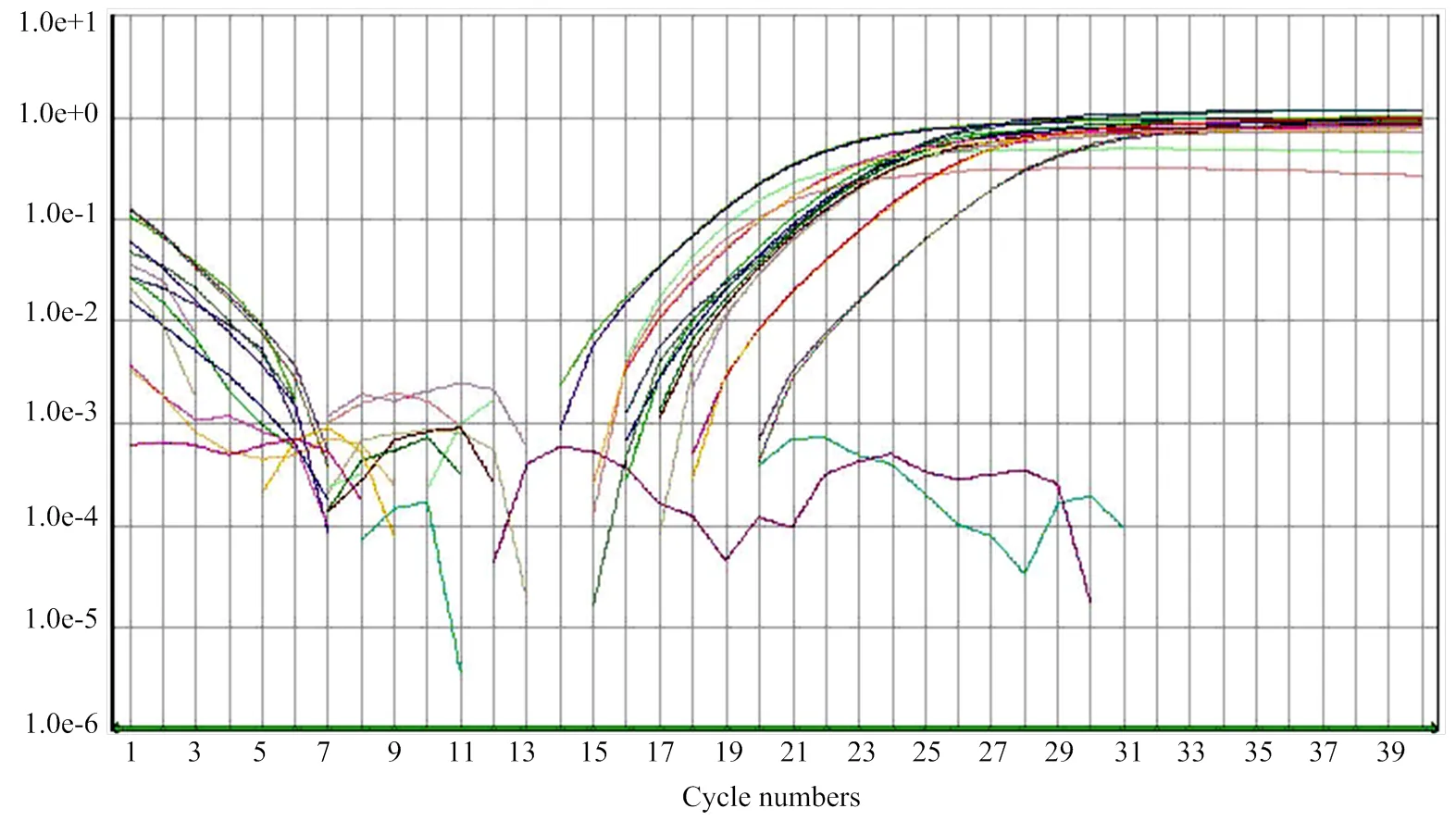
图4 动物源性成分real-time PCR扩增

表4 通用引物退火温度的优化

表5 通用引物和探针浓度优化

图5 猪源性成分基因组DNA定量标准曲线
采用连续稀释猪基因组DNA作为标准品,用动物通用的引物和探针进行Real-time PCR扩增,绘制标准曲线如图6所示,标准曲线为=-3.32+29.93,2=0.9968。DNA含量在0.02—200 ng/25 μL反应体系范围内标准曲线呈良好的线性关系,检测的灵敏度达0.02 ng/25 μL反应体系。

图6 动物源性成分基因组DNA定量标准曲线
2.5 混合DNA样品中猪源性成分Real-time PCR检测
当混合3种相同浓度的基因组DNA样品(猪、牛、鸡)时(每种成分质量百分含量33.33%),以猪引物和探针进行Real-time PCR测得的Ct值为24.16,以通用引物和探针进行Real-time PCR测得Ct值为23.44,根据公式(3)计算的猪源性成分百分含量为33.77%,百分误差为1.32%。
当混合不同质量的牛肉和猪肉,检测样品中的猪源性成分,实际值和检测值如表6所示,检测的最小百分误差为1.06%,最大百分误差为7.12%,百分误差大于单纯DNA混合样品的检测。

表6 混合肉样中猪源性成分检测结果
3 讨论
3.1 特异性基因筛选
动物源性成分PCR/Real-time PCR鉴别技术的关键在于特异性基因的筛选,基于线粒体基因建立的物种特异性PCR/Real-time PCR检测方法已广泛应用于动物源性成分的定性检测。但是对于定量检测,需要筛选基因组上拷贝数恒定的特异性基因进行Real- time PCR检测[5-6]。微卫星DNA在基因组中数量庞大,在猪的基因组中,目前已研究鉴定了878 967个多态性的微卫星DNA[12]。而且相关文献中已经报道了微卫星DNA扩增所需要的引物序列,无需自行设计引物,可以直接进行PCR验证,大大缩短了筛选和验证的时间。本研究筛选的猪特异性微卫星DNA(Accession EF172428)通过Blast比较分析,在GeneBank数据库中只有一条猪的核酸序列数据,未在其他动物核酸数据库中发现相似的序列。根据其基因序列设计的特异性引物只能从猪的基因组DNA模板中扩增出目的条带,其他9种动物中没有扩增条带,因此该基因在猪的基因组DNA中具有特异性。虽然本试验挑选的另一个微卫星DNA(Accession AF375760)不具有特异性,但是从两个微卫星DNA中就可以筛选出一个特异性的微卫星DNA,也说明本试验筛选特异性微卫星DNA的方法具有较高的成功概率,可以采用此方法开发更多的物种特异性的微卫星DNA用于动物源性成分的定性和定量检测。
3.2 Real-time PCR反应优化
影响Real-time PCR检测方法的因素主要有退火温度、引物浓度和探针浓度。针对上述因素,本试验绘制标准曲线,从扩增效率、回归方程的相关系数、最小Ct值、非特异性扩增的Ct值以及空白对照的Ct值等方面考察上述因素对Real-time PCR扩增效果的影响。最终优化的试验条件下扩增效率均高于96%,相关系数大于0.99,均符合Real-time PCR检测的相关要求(扩增效率90%—105%,相关系数大于0.98)[13]。虽然对其他9种动物源性成分检测时也出现了非特异性扩增,但是Ct值均大于35,一般在动物源性成分检测中,判断结果时定义临界点Ct值为35,大于35视为未检测出动物源性成分[14-15]。通过优化退火温度、引物和探针浓度,本研究建立的Real-time PCR检测方法的检测限为0.02 ng/25 μL反应体系,结果与巫坚等[16]和Chen等[17]报道的检测灵敏度(0.03 ng/25 μL、0.025 ng/25 μL)相似。
3.3 定量检测分析
Real-time PCR技术最大的优势是可以通过标准曲线对未知样品中的DNA含量进行定量检测,因此,其在食品检测领域广泛应用于转基因成分检测、食源性致病菌的检测、食品真伪检测[18-20]等方面。在转基因检测中,最多的是双标准曲线相对定量法,即将标准物质的DNA溶液梯度稀释,对内、外源基因分别生成标准曲线,依据标准曲线分别定量待测样品内、外源基因的拷贝数,以待测样品的内、外源基因的拷贝数之比来近似代表样品中的转基因质量分数[21]。Ren等[22]采用鸡-干扰素基因、猪-actin基因、牛生长激素基因作为靶基因,肌肉生长抑制素基因作为内参基因进行定量,检测混合样品中鸡、猪和牛肉的成分,检测误差在0.69%—22.66%,低于25%的检测误差水平,认为可以接受。本研究采用双标准曲线相对定量法,分别对猪源性成分和总动物源性成分进行定量。采用筛选出的特异性基因检测猪源性成分,采用脊椎动物基因组中超级保守序列[23]检测总动物源性成分,分别定量二者DNA含量,通过计算猪源性成分和总动物源性成分的比值,获得猪源性成分的百分含量。通过测试,检测混合DNA样品中猪源性成分的百分误差较小(1.32%)。而实际混合肉样的检测误差高于DNA混合样品,百分误差在1.34%—7.12%,低于25%的检测误差水平,检测的准确性与Wang等[24]报道的相对误差相似(1.64%—18.43%)。产生误差的主要原因在于不同的肉样中,单位质量肉中的细胞数目存在差异,相应的造成单位质量肉中的DNA质量存在差别[8],所以Real-time PCR最终检测出的是猪源性成分DNA含量的百分比,而不是猪肉质量的百分比。另外,不同动物肉的DNA提取效率可能存在差别,也可能对最终结果造成影响。
4 结论
本研究筛选猪特异性微卫星DNA,设计引物和探针建立了猪源性成分Real-time PCR检测方法,检测灵敏度达0.02 ng/25 μL反应体系,具有较好的特异性和敏感性。采用双标准曲线法分别定量肉中的猪源性成分和总动物源性成分,计算样品中猪源性成分的百分比,误差在1.06%—7.12%,可初步实现猪源性成分的定量检测。
[1] 史艳宇, 王莹, 石虹, 李天雨, 华蕾. 微滴数字PCR方法检测畜肉食品中鸭源性成分. 食品安全质量检测学报, 2018, 9(3): 583-588.
SHI Y Y, WANG Y, SHI H, LI T Y, HUA L. Detection of duck-derived materials in meat products by droplet digital PCR. Journal of Food Safety and Quality, 2018, 9(3): 583-588. (in Chinese)
[2] SUL S, KIM M J, LEE J M, KIM S Y, KIM H Y. Development of a rapid on-site method for the detection of chicken meat in processed ground meat products by using a direct ultrafast PCR system. Journal of Food Protection, 2020, 83(6): 984-990. doi: 10.4315/jfp-19-583.
[3] KIM M J, KIM H Y. A fast multiplex Real-time PCR assay for simultaneous detection of pork, chicken, and beef in commercial processed meat products. LWT-Food Science and Technology, 2019, 114: 108390. doi: 10.1016/j.lwt.2019.108390.
[4] 陈念, 赖小平. 线粒体基因组: 结构特点和基因含量进化. 生物学杂志, 2011, 28(1): 70-73, 17. doi: 10.3969/j.issn.1008-9632.2011.01. 070.
CHEN N, LAI X P. Mt-genome revolution: Structure and gene content. Journal of Microbiology, 2011, 28(1): 70-73, 17. doi:10.3969/j.issn. 1008-9632.2011.01.070. (in Chinese)
[5] 陈传君, 金鹭, 林华, 胡滨, 韩国全, 陈世界, 张婧, 安微, 杨苗. 食品中羊肉源性成分微滴数字PCR定量方法的建立. 食品与发酵工业, 2020, 46(6): 229-237.
CHEN C J, JIN L, LIN H, HU B, HAN G Q, CHEN S J, ZHANG J, AN W, YANG M. Quantification of mutton-derived ingredients in food by droplet digital PCR. Food and Fermentation Industries, 2020, 46(6): 229-237. (in Chinese)
[6] BALLIN N Z, VOGENSEN F K, KARLSSON A H. Species determination-Can we detect and quantify meat adulteration? Meat Science, 2009, 83(2): 165-174. doi: 10.1016/j.meatsci.2009.06.003.
[7] WANG Z C, WANG Z Y, LI T T, QIAO L, LIU R, ZHAO Y, XU Z Z, CHEN G, YANG S M, CHEN A L. Real-time PCR based on single- copy housekeeping genes for quantitative detection of goat meat adulteration with pork. International Food Science and Technology, 2020, 55(2): 553-558. doi: 10.1111/ijfs.14350.
[8] 刘立兵, 陈敏娜, 孙晓霞, 张亦琴, 付琦, 钱云开, 周巍, 郭春海, 王建昌. 微滴式数字聚合酶链式反应对香肠制品中鸡、猪、牛源性成分的定量分析. 肉类研究, 2020, 34(8): 51-56.
LIU L B, CHEN M N, SUN X X, ZHANG Y Q, FU Q, QIAN Y K, ZHOU W, GUO C H, WANG J C. Quantitative analysis of chicken-, porcine- and bovine-derived ingredients in sausage products by droplet digital polymerase chain reaction. Meat Research, 2020, 34(8): 51-56. (in Chinese)
[9] TABERLET P, WAITS L P, LUIKART G. Noninvasive genetic sampling: Look before you leap. Trends in Ecology and Evolution, 1999, 14(8): 323-327. doi: 10.1016/S0169-5347 (99)01637-7.
[10] SELKOE K A, TOONEN R J. Microsatellites for ecologists: a practical guide to using and evaluating microsatellite markers. Ecology Letters, 2006, 9(5): 615-629. doi: 10.1111/j.1461-0248.2006. 00889.x.
[11] 萨姆布鲁克 J, 拉塞尔 D W, 黄培堂. 分子克隆实验指南. 三版. 北京: 科学出版社, 2002.
SAMBROOK J, RUSSELL D W, HUANG P T. Molecular Cloning: A Laboratory Manual. 3rd ed. Beijing: Science Press, 2002. (in Chinese)
[12] WU Z Z, GONG H F, ZHANG M P, TONG X K, AI H S, XIAO S J, PEREZ-ENCISO M, YANG B, HUANG L S. A worldwide map of swine short tandem repeats and their associations with evolutionary and environmental adaptations. Genetics Selection Evolution, 2021, 53(1): 39. doi: 10.1186/s12711-021-00631-4.
[13] KRALIK P, RICCHI M. A basic guide to real time PCR in microbial diagnostics: Definitions, parameters, and everything. Frontiers in Microbiology, 2017, 8: 108. doi: 10.3389/fmicb.2017.00108.
[14] DOOLEY J J, PAINE K E, GARRETT S D, BROWN H M. Detection of meat species using TaqMan real-time PCR assays. Meat Science, 2004, 68(3): 431-438. doi: 10.1016/j.meatsci.2004.04.010.
[15] MAYER W, HOCHEGGER R. Discrimination of two alleles of the melanocortin receptor 1 gene to discern European wild boar () and domestic pig () in meat products by real-time PCR. European Food Research and Technology, 2011, 232(4): 687-692. doi: 10.1007/s00217-010-1402-8.
[16] 巫坚, 黄晓韵, 王海华, 陈小聪. 实时荧光PCR法检测肉制品中猪源性成分. 食品安全质量检测学报, 2021, 12(9): 3715-3720.
WU J, HUANG X Y, WANG H H. CHEN X C. Detection of porcine- derived components in meat products by real-time fluorescence PCR. Journal of Food Safety and Quality, 2021, 12(9): 3715-3720. (in Chinese)
[17] CHEN X Y, LU L X, XIONG X H, XIONG X, LIU Y J. Development of a real-time PCR assay for the identification and quantification of bovine ingredient in processed meat products. Scientific Reports, 2020, 10(1): 2052. doi: 10.1038/s41598-020-59010-6.
[18] LIU G Q, LUO J X, XU W L, LI C D, GUO Y S, GUO L. Improved triplex real-time PCR with endogenous control for synchronous identification of DNA from chicken, duck, and goose meat. Food Science and Nutrition, 2021, 9(6): 3130-3141. doi: 10.1002/fsn3.2272.
[19] LI J, GAO H F, LI Y J, XIAO F, ZHAI S S, WU G, WU Y H. Event-specific PCR methods to quantify the genetically modified DBN9936 maize. Journal of Food Composition and Analysis, 2022, 105: 104236. doi: 10.1016/j.jfca.2021.104236.
[20] LABRADOR M, GIMÉNEZ-ROTA C, ROTA C. Real-time PCR method combined with a matrixprocedure for the quantification of listeria monocytogenes in meat products. Foods, 2021, 10(4): 735. doi: 10.3390/foods10040735.
[21] 黄文胜, 邓婷婷, 韩建勋, 吴亚君, 陈颖. 转基因定量检测的不确定度研究. 中国生物工程杂志, 2012, 32(1): 49-55.
HUANG W S, DENG T T, HAN J X, WU Y J, CHEN Y. Estimate the uncertainty on quantification of GMO by the fluorescence real-time PCR method, China Biotechnology, 2012, 32(1): 49-55. (in Chinese)
[22] REN Y F, LI X, LIU, Y M, YANG L T, CAI Y C, QUAN S, PAN L W, CHEN S S. A novel quantitative real-time PCR method for identification and quantification of mammalian and poultry species in foods. Food Control, 2017, 76: 42-51. doi: 10.1016/j.foodcont.2017. 01.003.
[23] BEJERANO G, PHEASANT M, MAKUNIN I, STEPHEN S, KENT W J, MATTICK J S, HAUSSLER D. Ultraconserved elements in the human genome. Science, 2004, 304(5675): 1321-1325. doi: 10.1126/ science.1098119.
[24] WANG W J, WANG X Y, WEI T, ZHANG Q D, ZHOU X, LIU B. A multiplex Real-time PCR approach for identification and quantification of sheep/goat, fox and murine fractions in meats using nuclear DNA sequences. Food Control, 2021, 126: 108035. doi: 10.1016/j.foodcont. 2021.108035.
Quantitative Detection Technology of Porcine-Derived Materials in Meat by Real-time PCR

1Jiangsu Agri-Animal Husbandry Vocational College, Taizhou 225300, Jiangsu;2College of Veterinary Medicine, Nanjing Agricultural University, Nanjing 210095
【】The aim of this study was to develop a rapid and accurate quantitative method for identifying porcine-derived materials.【】Porcine microsatellites DNA were selected from GenBank nucleotide database. Primers specific for porcine were designed based on the sequences of microsatellite DNA. Genomic DNA from 10 kinds of common animals was amplified by PCR method. The specificity of selected microsatellite DNA to porcine-derived materials was judged by the amplification products. According to the microsatellite sequence, the specific primers and probes were designed to establish a Real-time PCR method for identifying porcine-derived materials. The double standard curve was used to quantify the porcine-derived materials and total animal-derived materials, respectively, and the percentage content of porcine-derived materials was calculated. 【】Porcine specific microsatellite DNA with the accession number EF172428 was selected. Only porcine DNA gave a fragment through PCR assay, while there was no amplification for other non-target animal species DNA. The limit of detection was 0.02 ng in a 25 μL reactive system using the Real-time PCR method. This method could accurately detect porcine-derived components in mixed DNA samples and mixed meat samples with 1.32% percent error and 1.06%-7.12% percent error, respectively. 【】The quantitative detection method of porcine-derived materials by Real-time PCR in this research could be used to detect the percentage content of porcine-derived materials in mixed samples.
meat; swine; animal-derived materials; Real-time PCR; quantitation

2022-03-26;
2022-11-01
国家自然科学基金青年基金(30471225)、泰州市科技支撑计划社会发展(指导性)项目(TS202019)
翟晓虎,E-mail:zhaixiaohu010@163.com。李翎旭,E-mail:2020107106@njau.edu.cn。翟晓虎和李翎旭为同等贡献作者。通信作者姚大伟,E-mail:yaodawei@njau.edu.cn
(责任编辑 赵伶俐)

