奶牛产奶量性状相关基因知识图谱的研究与构建
胡 浩,高 静,刘振羽
1.内蒙古农业大学 计算机与信息工程学院,呼和浩特010018
2.内蒙古自治区农牧业大数据研究与应用重点实验室,呼和浩特010018
随着领域数据的爆发式增长,目前科研人员在寻找与特定表型相关的基因时,需要在不同的基因或表型数据库中人工整理基因及表型间的关系,以此确定某一基因型对表型的不同影响,这就需要构建大量的特定物种的特定表型与基因型的关系库。目前主流的构建数据库的方式有两种,一种是基于测序数据[1],另外一种是基于文献挖掘。
对于传统的基因变异数据库而言,数据主要来源于通过高通量测序等一系列生物分子技术实验得到的原始数据和大型数据库已有数据[2]。例如:Kamiński等人[3]为了处理和编目所有关于直接、间接或潜在与牛奶蛋白生物合成相关的单核苷酸多态性(SNPS)的可用信息。建立了一个包含39个基因中的339个SNPs的数据库。在339个SNPs中,发现了316个单核苷酸取代、8个缺失、5个重复、7个插入和3个插入。该研究中SNPs的收集可作为研究牛奶蛋白质生物合成变异的遗传测定和经过广泛的种群筛选后的可靠资源,也可用于奶牛的亲子鉴定和进化研究。Ogorevc等人[4]构建开发了一个关于牛奶生产和乳腺炎的候选基因和遗传标记的牛数据库来作为一个综合的研究工具,该数据库包含943个参与乳腺发育和功能的基因和基因标记,作为进一步功能研究的候选对象,候选基因点被绘制在基因图上,之后利用七种不同研究方法通过多个独立分析发现的4个基因被认为是最有前途的候选基因,并进一步分析出硅泌乳乳腺的表达水平、基因变异和功能网络的最优生物学功能。
与此同时,还有一大部分科研成果以文献或报告的形式存在于不同的文献数据库中,要在大量文献中检索出针对特定表型被哪些基因型及变异影响,就需要进行繁琐复杂的人力工作,将会耗时较长,严重影响研究进展[5]。而如果能够对文献中的基因表型关联知识进行收集和整理,将隐含在文献中的信息有逻辑地建立关系,就能极大地减少科研人员收集和检索的时间,为知识的有效利用奠定相应的基础。
因此,为满足领域相关科研人员快速了解当前研究领域现状,基于文献挖掘的数据库成为了研究的热点。例如:佟凡[6]针对当前自然语言处理技术精准面对医学知识库的构建时,仍因模型、算法存在诸多局限和不足而导致难以满足实际需求的情况,基于生物医学文献库及现有知识库资源,结合自然语言处理技术,优化了基因、变异和疾病命名实体识别算法,构建了满足“基因-变异-疾病”文献挖掘任务需求的关系类型和关系抽取语料库,设计了适用于“基因-变异-疾病”关系抽取的新算法,开发出可集成不同来源知识的“基因-变异-疾病”知识图谱构建、编审、可视化检索利用和展示的平台。2020年付义报[7]基于共现的方法,从PubMed历年文献中筛选出生缺陷与表型/症状、基因、致畸物/药物等共同出现的句子。首先,对这些句子进行假阳性处理,根据远程监督的思想,利用UMLS定义的关系,人工给句子中两个实体标注关系,从而建立起出生缺陷相关的语料库。然后,利用人工标注好的语料库训练了三种不同的深度学习关系抽取模型并依据这三种模型的预测结果,采用多数表决和高置信度的方法,生成最终的关系抽取预测模型以得到<实体,关系,实体>三元组,之后将三元组相关信息存储在图数据库Neo4j中,构建了出生缺陷领域的知识图谱。最后,利用训练好的预测模型对新句子进行预测从而推断出句子中实体之间的关系,并将新得到的三元组添加到知识图谱中。
随着高通量测序技术的不断发展,基于大规模的生物信息学数据积累,筛选出与提高牛奶产量和质量的相关基因或者物理环境的影响因素成为生物学家与相关畜牧业专家研究奶产量领域的重中之重。伴随领域数据的爆发式增长,单纯依赖人工采集、编审的传统知识库构建策略,由于其耗时长、无法自动更新等原因已不再适合,从文献中自动抽取信息和挖掘潜在知识成为近年的研究重点与应用热点[8]。
针对现有生物领域数据库的研究中还没有基于奶牛产奶量-基因而构建的专题知识库,且现有生物数据库大多使用关系型数据库存储数据,在对数据进行多对多关系建模时并不合适。本文在文献挖掘的基础上,基于知识图谱构建各类奶牛产奶量-基因知识库,通过多实体识别得到基因和物理影响因素的信息,再进行产奶量-基因的关系抽取,经过人工审核后,采用Neo4j图数据库以图形化方式更加直观地描述奶牛产奶量-基因的关系[9],形成基因-表型知识库。为科研人员进一步筛选与表型相关的主效基因提供基础知识库的支撑。
1 知识图谱构建方法
知识图谱(knowledge graph)于2012年由谷歌公司首次提出,与传统存储结构不同,知识图谱本质上是一种语义网络,是以图的形式来描绘客观事物。这里的图即指数据结构中的图,是由节点和边构成,其中的节点表示实体或者概念,即现实或者抽象出来的事物,而边表示事物的关系和属性,事物的内部特征用属性来表示,外部联系用关系来表示[10-11]。
在以往的搜索引擎核心中,都是将要搜索的内容看作字符串,将其与数据库中数据进行比对,按照匹配度从高到低的形式展现给用户。将知识图谱的技术加入后,可以将搜索内容看作客观世界的事物,也就是单独的个体后进行语义搜索,去查找所需的本体或者本体关联的信息。因此知识图谱在智能问答、语义搜索、推荐系统等方面具有广泛的应用前景。
基于知识图谱构建奶牛产奶量-基因知识库的构建方法主要分为四个阶段如图1所示。
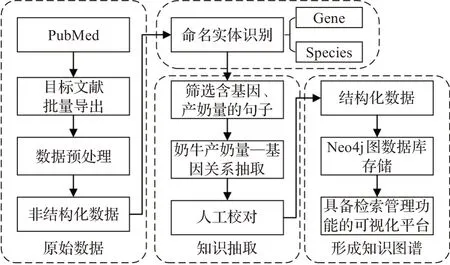
图1 奶牛产奶量-基因知识图谱的构建方法Fig.1 Construction method of knowledge graph of gene-production traits in dairy cows
(1)数据的采集和预处理:为确保数据具有权威性,数据采集阶段以目前国际上公认的生物医学文献数据库PubMed为来源,PubMed是一个免费的文献搜索引擎,其中相关文献的摘要部分一般包括研究目的、方法、结果和结论,能够基本涵盖论文主旨。本实验以“产奶量”“基因”为关键字,对在PubMed中检索出的700多篇文献进行筛选整理后,将符合条件的170余篇文献作为实验数据的来源。
通过引擎自动相关文献摘要的功能获取原始数据,采用自然语言处理技术对文本进行预处理,删除作者详细信息及出版商信息等数据,保留文献URL、标题、摘要以及实验样本等关键信息,为后续知识抽取工作做准备。
(2)基因/物种命名实体识别:随着生物医学的快速发展,相关文献数量也以爆发式的速度在增长,而从生物医学文献到结构化的数据是非常耗时且繁琐的,这就使得生物医学研究很难及时顺利地开展[12]。因此,通过自动文本挖掘工具帮助定位相关物种、基因、性状等成为了帮助相关人员研究工作的利器。
PubTator是一个基于Web的系统,区别于现有的注释和文献挖掘工具[13],PubTator具有几个优秀的特性。首先,PubTator是一个集成式工具,可以为相关研究者从搜索和检索相关文献到注释选定文章提供一站式服务。第二,用户在PubTator中的输入既可以是搜索查询,也可以是PubMed文章的列表。在完成手动管理后,用户就可以轻松地下载和导出其注释文件。第三,PubTator的界面设计与PubMed类似,使得相关生物研究人员可以轻松地使用操作。第四,PubTator中集成了目前多种已经在生物医学领域具有较高精确度的文本挖掘工具[14],可以自动地识别关键的基因、物种、疾病等相关实体,如表1所示。
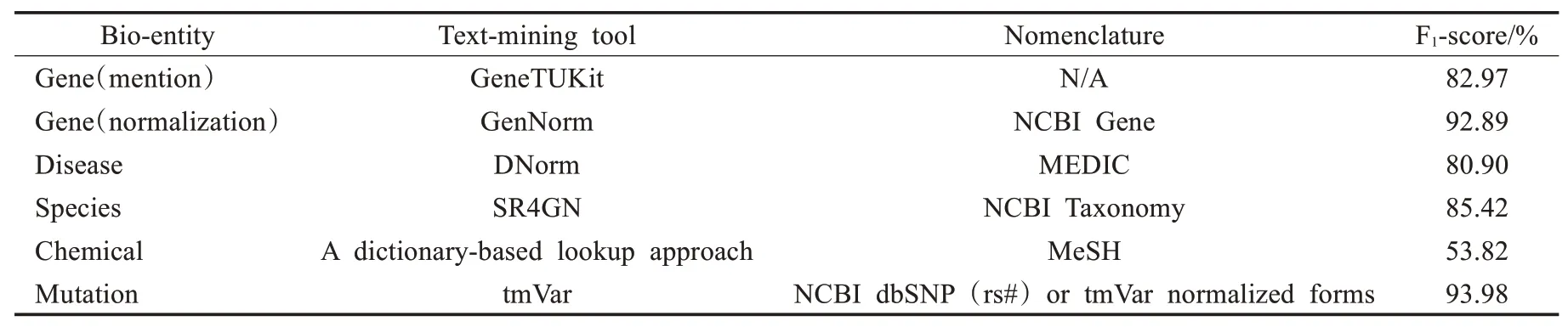
表1 Pubtator集成工具展示Table 1 Pubtator integration tool presentation
(3)生产性状-基因关系抽取:在构建知识图谱时,关系的抽取直接影响着之后生物研究人员对于检索信息的筛选和判断。所需的信息即为文献中直接提到的基因型与相关生产性状之间的表达关系。由于目前没有大规模人工注释的表型-基因型的语料库。为此本文采用Open IE关系抽取工具进行半自动筛选。Open information extraction(Open IE)能够从纯文本中提取开放域关系三元组,用以表示关系的主体、关系和客体[15]。
(4)关系表示、存储和可视化:本文的目的是通过三元组来构建实体及实体间的关系,因此采用Neo4j图数据库表示和存储数据。Neo4j是一种高性能NOSQL图形数据库,它将结构化数据存储在网络上而不是表中,是一个嵌入式的,基于磁盘存储的,具备完全的事务特性的Java持久化引擎[16]。在Neo4j中,一般通过两种三元组关系建立联系,一种是实体-关系-实体,用以表达基因和生产性状之间的关系[17]。另一种是实体-属性-值,用以表达目标基因所涉及文献的相关属性,以便于研究人员快速找到对应文献或关键信息,进行更深入的研究。
2 产奶量相关表型-基因知识图谱的构建
2.1 数据来源
以PubMed文献库为数据来源,按照关键词“Milk Yield”“gene”进行检索,共得到700余篇相关文献,之后根据文章摘要部分对文献进行筛选,得到170余篇在文章摘要中包含奶牛基因及产奶量相关关系的文献。利用PubTator对目标文献进行导出,其中涵盖了文章的PMID、文章出版时间、标题、作者信息、摘要等相关信息。利用脚本对文章进行预处理,删除其中的作者信息、出版时间等,方便之后对于文章摘要部分进行命名实体识别以及关系抽取等关键步骤。
2.2 基因/物种命名实体识别
利用Pubtator命名实体识别工具对170余篇文献进行处理,剔除掉疾病、化学因素等与本文没有联系的实体,发现Pubtator对于基因实体的识别度相对较高,能有效地挖掘文献文本中存在的基因种类,在此基础上,再通过阅读文献对上述基因进行进一步的筛选,选出其中对产奶量能够产生影响的基因实体,并进行统计整理后,得到结构化数据,以便构建知识图谱。相对于基因实体,Pubtator对于物种的识别度较低,大多只能识别出cattle、Bovine等科级门类,而对下一等级的属、种难以识别,如表2所示。例如,本文将文献涉及样本精确到了牛的具体种类或状态如:China Holstein(中国荷斯坦奶牛)、Italian Holstein(意大利荷斯坦奶牛)、Jersey cattle(泽西牛)、wild yak(牦牛)、Egyptian buffaloes(埃及水牛),calving patterns(产犊状态)等。对于无法识别的具体种类,在经过数据清洗并通过人工审核筛选后最终得到139个基因实体,6个物理环境影响因素(heat stress(HT)and photoperiod(热应激和光周期)、milking frequency(挤奶频率)等)。基于以上实验数据,Pubtator对于基因的识别准确率约为82.35%,能够满足前期对于初始数据的较为高效的筛选,极大减少了人力工作,提高了研究进展。值得一提的是,Pubtator是一个基于网络的辅助生物医学文本挖掘工具,对于不同需求种类的文本有着不一样的准确率,且差异化明显。因此要想使Pubtator能够较好地适用于自身需求,需要不断地完善和丰富自身语料库,这也是后期需要不断完善和改进的地方。经过校对和整理后,文献库中得到了140个对应文献的实验样本。
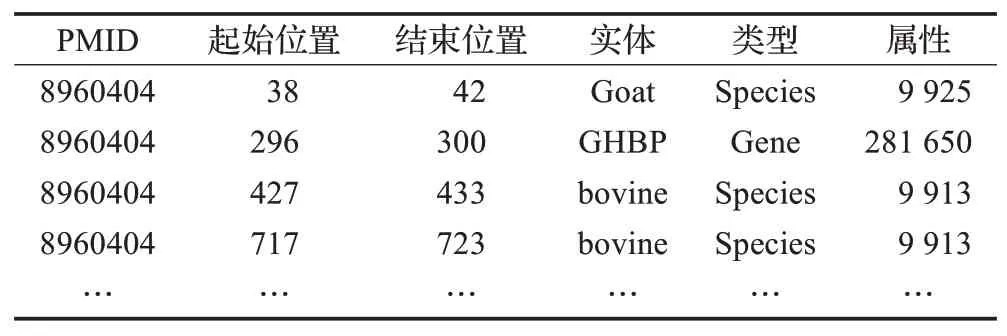
表2 命名实体识别结果数据Table 2 Named entity recognition
2.3 奶牛产奶量-基因关系抽取
利用Stanford Open IE对已经经过命名实体识别后的文献文本进行实体关系的抽取,经过Open IE抽取后以{'subject:','relation:','object:'}的三元组形式输出,由于Open IE是通用领域关系抽取工具,抽取结果会以多可能性的方式展现,因此在抽取后还需进一步经过人工筛选校对,抽取及校对后得到的关系示例如下:
(1)奶牛产奶量-基因关系:对于包含关系的语句“These results would suggest that LPIN1 is having an effect on yak milk fat synthesis.”在经过抽取后得到若干条可能存在的关系,如表3所示。经过筛选后保留三元组{'subject':'LPIN1','relation':'having an effect on','object':'yak milk fat synthesis'}。

表3 基因-生产性状关系抽取结果数据Table 3 Result data extracted from gene-production trait relationship
(2)奶牛产奶量-物理环境因素关系:对于包含语句“Heat stress(HT)and photoperiod affect milk production and immune status of dairy cows.”在经过抽取后得到若干条可能存在的关系,如表4所示。经过筛选后保留三元组{'subject':'Heat stress(HT)and photoperiod',
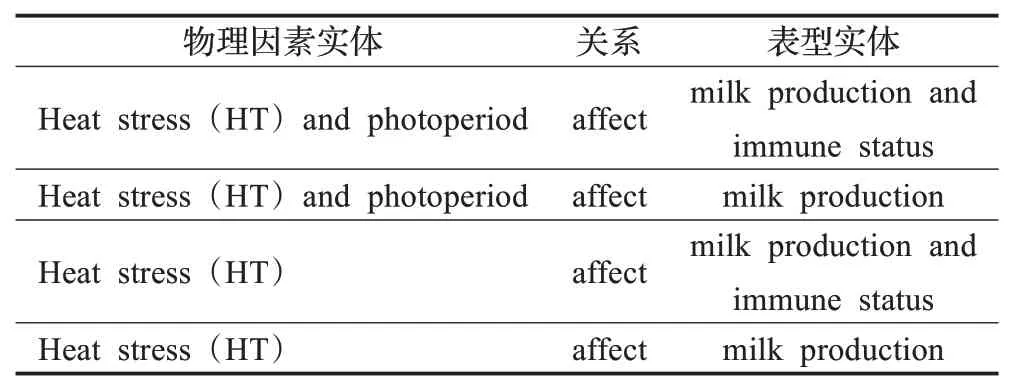
表4 奶牛产奶量-物理环境因素关系抽取结果数据Table 4 Extracting result data from relationship between physical environmental factors and production traits
'relation':'affects','object':'milk production and immune status'}。经整理校对后,共得到生产性状-基因关系139条,生产性状-物理环境因素6条,如表5所示,整理为结构化数据后,方便下一步知识图谱的构建。

表5 奶牛产奶量-基因/物理环境因素实体关系展示Table 5 Partial gene/physical environment factor-production trait entity relationship display
OpenIE实现了对海量信息中可能存在的关系的抽取,该种方法不需要前期手工标注训练集,也不局限于特定的领域,而是通过自动的学习和统计来实现关系的抽取。在上述实验的过程中,OpenIE能够较好地完成对含有基因表型实体的特定语句的关系抽取,且准确率随着实验数据的积累不断提高。但同时也存在着部分关系抽取不明确,可能存在关系较多需要后期人工筛选等的情况出现。并且,作为自然语言处理领域较为常见的指代消减等问题也同样存在,需要在自动抽取的基础上进一步筛查校对,保证关系的准确性和对生物医学文献中所存在关系的最精确的保留和引用。后期,随着文献库中数据量的不断扩大和新的表型性状的不断加入,构建一个关于奶牛的基因表型特定语料库对于关系抽取的准确性和精确性会有进一步的提高。
2.4 利用Neo4j图数据库存储数据
对经过结构化处理后的数据进行整体分析,创建了7种实体类型,构建出如图2的关系结构图。
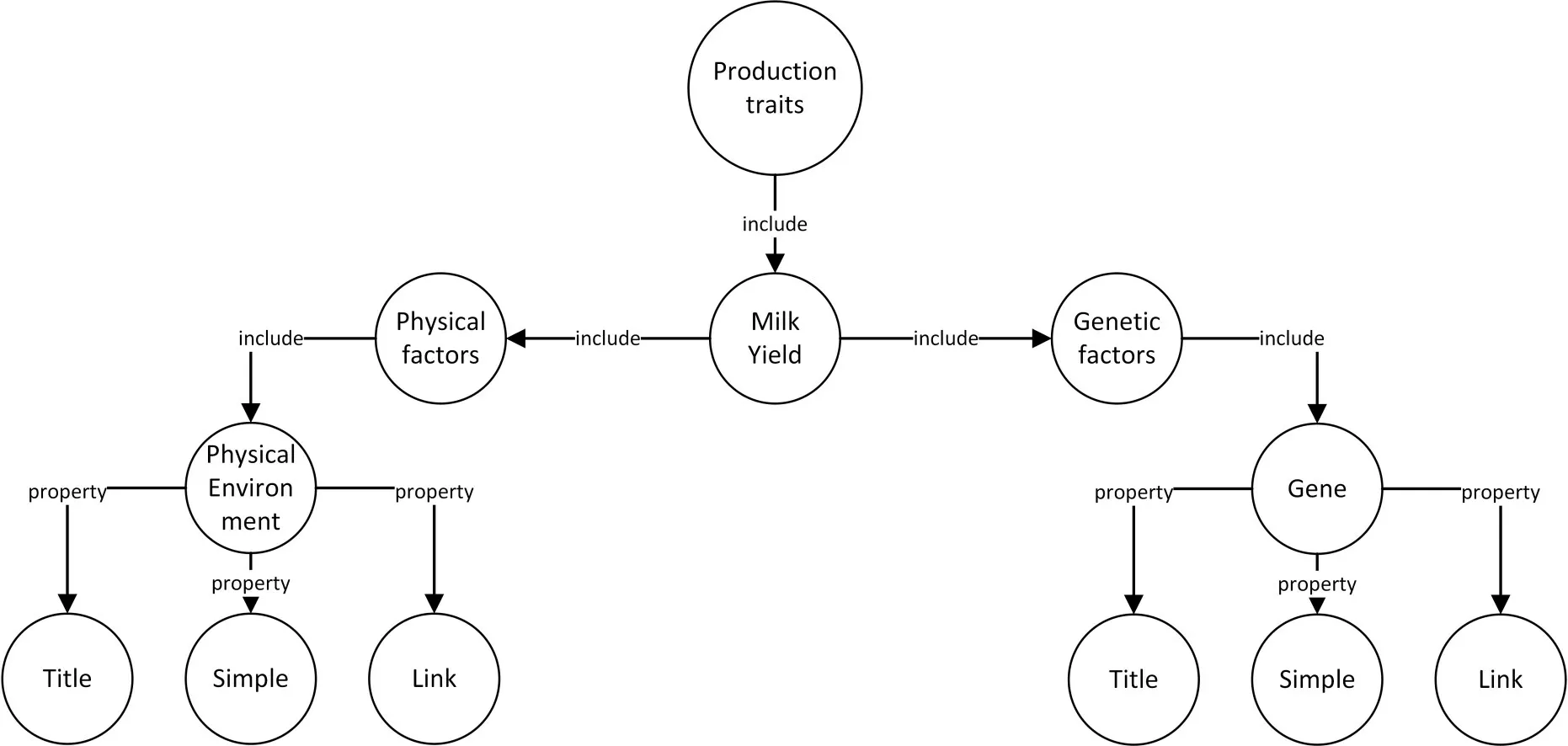
图2 奶牛产奶量-基因知识库实体关系结构图Fig.2 Structure diagram of entity relationship between milk yield of dairy cows and gene knowledge base
结合奶牛产奶量的实体关系结构,确定实体与实体以及实体与属性间的关系类型。Production traits实体与Milk Yield实体间,Milk Yield实体与Physical factors实体及Genetic factors实体间,Physical factors实体与Physical Environment实体,Genetic factors实体与Gene实体间的关系均为include,Physical Environment实体及Gene实体与Title、Simple和Link实体间的关系均为property,如表6所示。

表6 实体关系类别Table 6 Entity relationship category
为了使基因与表达性状间的关系表达更为准确,本文直接采用文献中对于关系的描述,没有做人为的更改。例如:medium effect on,was associated with decreased,had no significant effect,shows a statistically significant relationship(P=0.05),have potential effects on,had significant(P<0.05)effect on等140余种不同的在原文献中对于关系的表达。按照实体与实体或者实体与属性的关系将奶牛产奶量与基因、物理环境因素以及其所在文献的标题、链接、实验样本等相关信息一次建立关系,方便后续的检索和审查。在建立关系时,首先将前期整理好的结构化数据以CSV格式储存,再通过Python读取,按照前期设计的关系结构,以父子节点的方式存储,存储时首先检查库中是否存在该种实体节点,如存在则合并,如不存在则创建新的节点,建立联系。将定义好的实体节点和关系存储到Neo4j图数据库中用到了Python语言提供的py2neo接口实现。最终构建的知识图谱部分数据展示如图3。
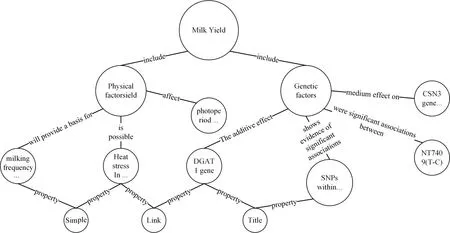
图3 奶牛产奶量-基因知识图谱Fig.3 Milk yield of dairy cows-genetic knowledge graph
3 知识图谱的可视化平台
奶牛产奶量-基因的可视化平台搭建使用了前后端分离的技术,后端搭建以目前较为流行的Python Web框架Flask为基础,通过导入py2neo包连接Neo4j数据库,将用户输入的字符串利用json模块转化为字典,结合提前写好的Cypher查询语句框架,对数据库中已有内容进行查询检索。检索平台前端通过同样流行的开源框架Vue实现,方便后期功能的完善和管理的同时具备了较好的兼容性。检索系统目前主要包含了以下三类功能:
(1)查询奶牛产奶量-基因相关文献信息功能。分别支持按照基因Genetic factors或生产性状Phenotype两种关键词的搜索方式进行检索,检索关键词不区分大小写,检索结果以生产性状类型、相关联基因、具体基因型,以及文献的标题、实验所涉及样本和文章在PubMed中的具体链接的形式返回给用户,方便研究人员直接找到对应文献进行更为深入的查阅和研究。如图4所示,在类型选择下拉列表中选择Phenotype,在对应的名称输入框中输入“Milk Yield”关键字,点击“搜索”按钮,即可以表格的形式获得与产奶量有关的所涉及的全部基因以及其文献资料的相关详细信息。
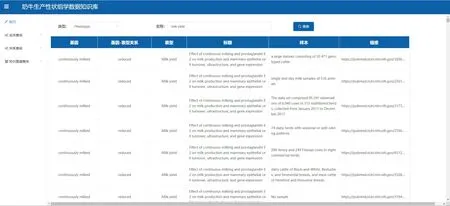
图4 奶牛基因-生产性状知识库界面Fig.4 Dairy cow gene-production traits knowledge base interface
(2)查询奶牛产奶量-基因关系图查询功能。该功能以单个目标基因为主体,方便在上一个功能的基础上查询特定的基因,在输入特定基因后检索系统可以将特定基因与生产性状的关系以力向导图的形式展现给用户。用户还可以根据自身需求,对关系图进行缩小、放大、旋转和拖动,当用户将光标移动到某一节点上时,该节点以及对应的关系和关系节点都将高亮显示,同时,其他节点变为灰度显示。同时,关系图还提供了下载保存到本地的功能。
(3)奶牛产奶量-基因知识图谱的可视化功能。如图5所示,该功能可向用户以力向导图的形式展示现有数据库中所有存在的约600个实体节点和500余条存在的关系,同样支持放大、缩小、拖动和旋转功能方便用户查看。
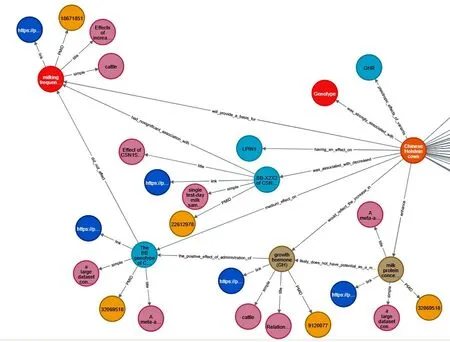
图5 奶牛产奶量-基因知识图谱可视化Fig.5 Visualization of dairy cow gene-production trait knowledge graph
奶牛产奶量性状相关基因知识图谱及平台支持研究人员在海量文献库中快速检索挑选影响奶产量的重要文献,锁定目标基因与相关表型的关系,能够在确定相关表型的基础上,快速对该种表型的其他影响基因进行深入了解,或在已知特定基因的基础上,查询与之相关联的基因,确定多种基因之间是否存在共同影响作用,并且对于将基因与表型、基因与基因间关系进行非结构化的呈现等多种实际用途。具有直观、清晰、易于操作的特点,为研究人员进一步筛选与奶牛奶产量表型相关的主效基因提供知识库支撑。
4 结语
本文详细阐述了奶牛产奶量-基因知识图谱的构建,前期共涉及文献700余篇,经整理筛选后得到与生产性状的相关文献170余篇,共筛选出约140个关联基因和130余条关联关系,这些数据为相关研究人员提供了方便,对奶牛生产性状,尤其是产奶量这一表型的深入研究具有重要意义。同时,也存在着前期人工筛选耗时,命名实体识别与关系抽取准确率有待提高等问题,针对这些问题,后续将继续深入完善优化,力争实现垂直领域的特定语料库,将在大大提高识别准确率的同时进一步解放劳动力,而着力于数据量的扩展和数据库字段类型的丰富。此外,后续还将继续对检索系统做进一步的完善优化,增加用户意见以及词条修正等功能,使广大专业领域研究人员能够积极参与到提高基因-表型关系精确度的工作中来。

