腺嘌呤碱基编辑器ABE8e多聚体的形成机制研究及抑制策略
周 项, 朱海霞, 黄 强
复旦大学 生命科学学院, 上海 200438
单碱基编辑是一种由Cas蛋白主导的基因编辑技术[1],具有不依赖于DNA链的DSB(Double strand break)的特点,可以对靶位点上的单碱基进行替换[2-3]。近年来,该技术已广泛应用于动植物模型的建立、疾病发病机制以及微生物的研究等领域[4-7]。在应用过程中,单碱基编辑技术展现出了高效、高靶向性等特点,因此该技术备受研究人员的关注,有望在基因治疗中大放异彩。根据碱基的转换类型,单碱基编辑技术可以分为两大类,分别是胞嘧啶碱基编辑器(Cytosine base editors, CBE)和腺嘌呤碱基编辑器(Adenine base editors, ABE)。在一些动植物的基因编辑实验中,ABE比CBE表现出了更好的特异性、有效性和安全性[5, 8],因此,ABE具有广阔的应用前景。通过多代定向进化优化,人们获得了迄今最为优秀的一个腺嘌呤碱基编辑器:ABE8e[2]。
目前,ABE8e的结构已被解析,结构显示其天然形态为二聚体结构[9]。但是,我们在ABE8e的体外纯化制备过程中却发现其多聚体比例较高。在其它相关文献中,ABE8e的制备缓冲体系中含有一定量的还原剂[10]。当蛋白质混合液中含有大量多聚体时,会导致蛋白质性质不稳定,进而使结构发生改变[11]。因此,在蛋白质的体外制备过程中需要尽可能地控制多聚体的比例,以保持样品的稳定性[12]。
在本文中,为了降低ABE8e多聚体的比例,我们首先分析了ABE8e的三维结构,了解其多聚体形成的分子根源。然后,在结构信息的指导下推测分子间的聚集源于ABE8e二聚化界面中疏水区域的曝露。为验证理论推测的正确性,我们构建了相应的ABE8e突变体(ABE8e-T),对其进行表达和纯化。ABE8e-T的纯化结果证实了多聚体成因的理论推测。在此基础上,我们有针对性地优化了缓冲体系,从而降低了ABE8e多聚体的比例,有效地提高了纯化制备的效率。
1 材料与方法
1.1 材料
1.1.1质粒与菌株
本研究使用的ABE8e及ABE8e-T质粒由课题组保存。DH5α、Rosetta (DE3)大肠杆菌感受态细胞购自天根生化科技(北京)有限公司。
1.1.2主要试剂
表达:氨苄青霉素、IPTG、氯霉素及LB Broth购自生工生物工程(上海)股份有限公司。纯化:咪唑、Tris、KCl、精氨酸及盐酸购自生工生物工程(上海)股份有限公司。醋酸、SDS、考马斯亮蓝、甲醇购自沪试实验室器材(上海)股份有限公司。电泳胶配制缓冲液购自天根生化科技(北京)有限公司。突变体构建:质粒小提试剂盒、点突变试剂盒及感受态细胞购自天根生化科技(北京)有限公司。
1.1.3主要设备
表达:恒温摇床购自上海天呈实验仪器制造有限公司。纯化:AKTA Avant 25购自格来赛生命科技(上海)有限公司。电泳仪购自北京六一生物科技有限公司。凝胶成像系统购自上海勤翔科学仪器有限公司。
1.1.4主要耗材
纯化:Ni-NTA Agarose购自凯杰生物工程(深圳)有限公司。凝胶层析柱为Superdex 200 Increase 10/300及HiLoad Superdex 200 16/600,均购自格来赛生命科技(上海)有限公司。
1.2 方法
1.2.1表达步骤
将复苏好的菌液添加至含有氨苄及氯霉素的LB培养基中,进行扩大培养。扩大培养的温度设为37 ℃,转速设为200 r/min,培养4 h~5 h后检测OD值。当OD值为0.8~1.2 时,进行表达培养。表达培养的温度设置为20 ℃,转速设置为200 r/min,培养12 h~14 h后停止培养。将菌液进行离心,收集菌体,保存至-80 ℃,以备后用。
1.2.2纯化步骤
Ni柱亲和层析的实验步骤依次为:平衡1、平衡2、上样、淋洗及洗脱。其中,平衡1及洗脱步骤的缓冲体系为:20 mmol/L Tris,500 mmol/L KCl,300 mmol/L咪唑pH 8.0;平衡2与淋洗步骤的缓冲体系为:20 mmol/L Tris,500 mmol/L KCl,10~20 mmol/L咪唑pH 8.0。平衡1冲洗体积大于5个柱体积,平衡2的冲洗体积大于5个柱体积;淋洗体积大于5个柱体积;洗脱体积大于3个柱体积,在洗脱的过程中可以分管收集洗脱液,根据SDS-PAGE电泳结果分析各泳道蛋白的分子量、浓度以及纯度,并进行下一步骤。
凝胶层析的实验步骤依次为:平衡、上样、再平衡。其中,优化前的缓冲体系为:20 mmol/L Tris,500 mm/L KCl pH 8.0,优化后的缓冲体系为:20 mmol/L Tris,0.5 mol/L精氨酸盐酸,0.5 mol/L KCl pH 8.0。此外,还设置了对照缓冲体系:20 mmol/L Tris,0.5 mol/L 精氨酸盐酸 pH 8.0。流速设置为0.5 mL/min~1.0 mL/min,平衡与再平衡的体积为1个柱体积。
1.2.3突变体突变流程
采用点突变的方式进行质粒构建。实验流程为首先提取ABE8e的质粒,其次对进行点突变。突变完成后,转化入细胞内并挑选单克隆,最后对其进行测序确认序列的正确性。
1.2.4PyMOL及Rosetta软件的应用
运用PyMOL软件观察ABE8e的三维结构,观察的主要内容是:疏水性氨基酸在三维结构中所处的位置,分子之间的静电势等信息。从观察结果中更深入地分析分子之间的相互作用,从而了解多聚化的原因及确认可能的氨基酸突变位点。
用Rosetta软件中的fixbb模块对需要突变的氨基酸位点进行随机氨基酸突变,经排列组合后,得到多种突变体。根据fixbb输出的5 000个突变体结果,通过Python编程剔除序列完全相同的重复突变体,最后根据Rosetta能量由低到高排序,从而选出能量最低的突变设计结构。
2 结果与讨论
2.1 多聚体成因的理论分析
核酸酶大多为碱性蛋白质,在中性pH条件下,表面氨基酸带有大量正电荷,与带有负电荷的靶标DNA相互吸引并结合,捕获靶标DNA。但是,在体外制备核酸酶的过程中,一般不直接添加靶标DNA。因此,在缓冲体系中缺乏负电荷的情况下,核酸酶的结构有可能会不稳定。所以,在核酸酶制备缓冲体系中,通常使用高盐缓冲体系提供一定的负离子环境。以SpCas9为例,在高盐缓冲体系中,该蛋白质以单体为主;而在相同的缓冲体系中,ABE8e则是含有34.2%的多聚体。
为了抑制多聚体的形成并降低其比例,首先分析了ABE8e的结构,了解多聚体的成因。蛋白质分子结构的形成与分子间的相互作用取决于静电作用力、疏水作用力、氢键与范德华力的共同影响,其中静电作用力和疏水作用力起到了非常关键性的作用。多聚体的产生往往因为蛋白质自身之间发生了非正常的结合,通常与静电作用力和疏水作用力相关[13-15]。因此,我们详细分析了ABE8e的三维结构、TadA-8e的氨基酸序列、疏水性氨基酸分布、电荷分布等性质,以了解多聚体的成因。具体如下:
(1) TadA-8e氨基酸序列分析。ABE8e主要由两部分组成,分别是nCas9和TadA-8e。其中nCas9仅在野生型SpCas9上突变了一个氨基酸(D10A)。我们和其他人的研究表明,野生型SpCas9的纯化结果显示其仅含有少量的多聚体(结果未显示)。因此,首先考虑分析TadA-8e的氨基酸序列。
TadA-8e是从TadA野生型(wtTadA)定向进化而来。从图1可知,进化后所得的TadA-8e一共突变了19个氨基酸,其中三个疏水性氨基酸突变为亲水性氨基酸(W23R、A109S、F149Y),5个为亲水性氨基酸突变为疏水性氨基酸(H36L、P48A、R51L、E155 V、T166I)。相较于野生型TadA,TadA-8e新增了两个疏水性氨基酸。所以,由表1可知,TadA-8e共有166个氨基酸,其中70个氨基酸为疏水性氨基酸,疏水性氨基酸占比42%。

表1 TadA-8e疏水性氨基酸分布概况表

(a) wtTadA 和 TadA-8e 序列对比图
当疏水性氨基酸含量较高时,分子形态的改变可能会造成更多的疏水区域曝露,从而造成分子间聚集,产生大量的多聚体。考虑到ABE8e疏水性氨基酸占比较高,推断其存在易于聚集的风险。但是,当疏水性氨基酸位于分子内部时,将有助于分子结构的稳固,并不会引起分子间聚集。因此,需要进一步观察TadA-8e的疏水性氨基酸在结构中的分布。
(2) TadA-8e疏水性氨基酸分布分析。TadA-8e序列如下:
MSEVEFSHEYWMRHALTLAKRARDEREVPVGAVLVLNNRVIGEGWNRAIGLHDPTAHA
EIMALRQGGLVMQNYRLIDATLYVTFEPCVMCAGAMIHSRIGRVVFGVRNSKRGAAGS
LMNVLNYPGMNHRVEITEGILADECAALLCDFYRMPRQVFNAQKKAQSSI
图2(a)展示了TadA-8e的疏水性氨基酸分布,从图中可以观察到,其疏水性氨基酸主要分布于分子内部、柔性部位及分子表面,其中柔性部位也大多位于分子表面。疏水性氨基酸密集地位于蛋白质分子表面时,易造成分子间的聚集。图2(c)展示了TadA-8e疏水性氨基酸分布较密集的区域。由图可知,柔性部位的疏水性氨基酸虽分布在分子表面,但分布较松散。分子表面的疏水性氨基酸主要密集地分布在两个α螺旋的外表面,占比约17.1%(表2)。然而,这部分的区域为TadA-8e的二聚化界面,当TadA-8e呈二聚体形态时,二聚化界面内的疏水性氨基酸聚拢在分子间,有助于稳固二聚体的构象。图2(b)展示了TadA8e的电势分布,可以看到:在二聚化界面中,除了疏水作用力,界面中的静电势相互吸引,界面三维结构呈互补形态,二聚化界面结合的非常紧密。综上,我们推断:当带有TadA-8e的ABE8e分子以二聚体形态存在时,分子表面密集的疏水性区域较少,不易造成聚集。

表2 TadA-8e二聚化界面疏水性氨基酸概况表
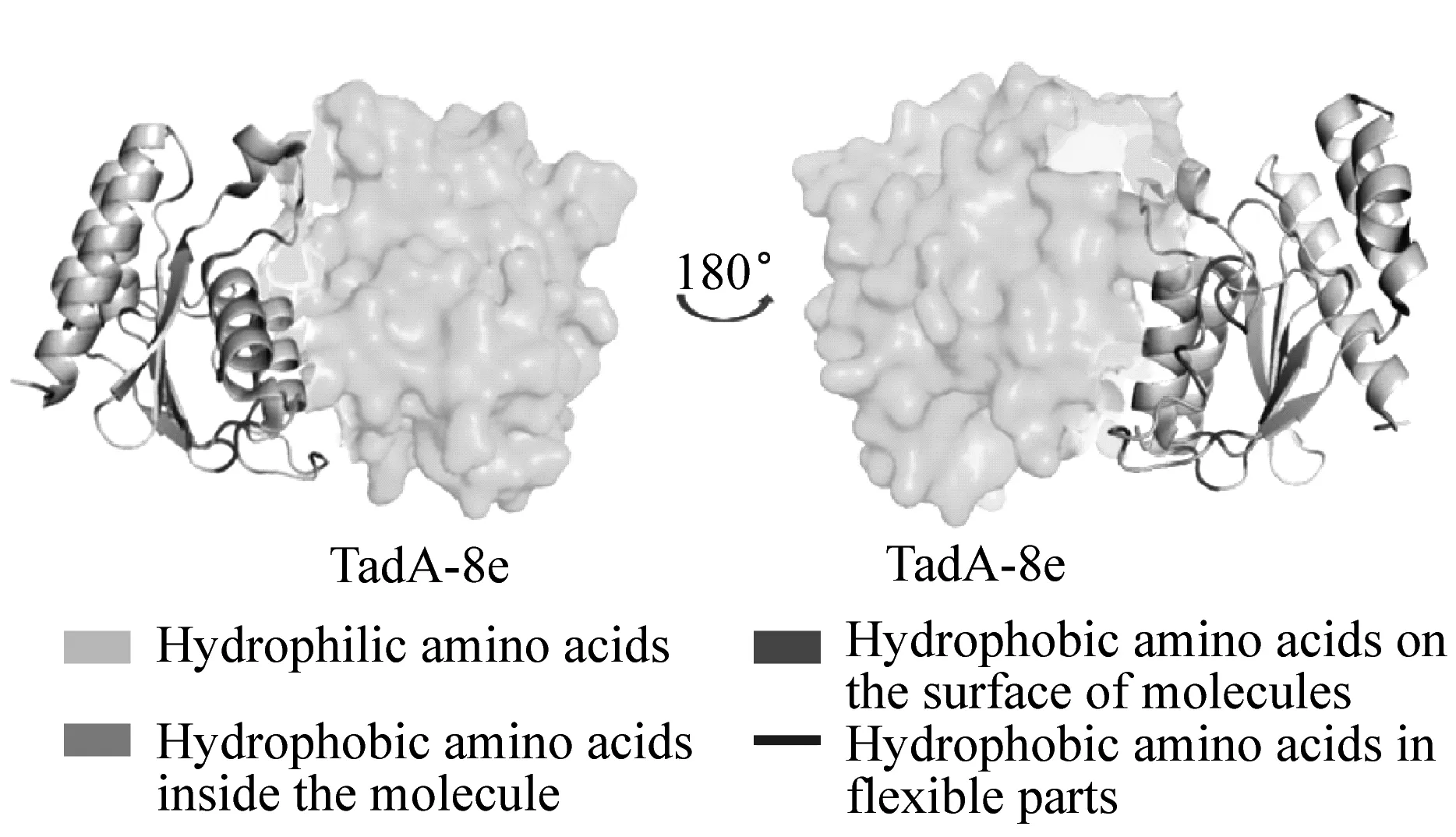
(a) TadA-8e 疏水性氨基酸分布图
(3) ABE8e三维结构分析。根据上述理论分析结果可知,ABE8e以二聚体为主要结构形态时,多聚体较少。但是,ABE8e的体外纯化结果显示:蛋白混合液中约含有34.2%的多聚体。因此,我们进一步考察ABE8e的三维结构。
由图2(d) ABE8e的三维结构可以发现:nCas9的分子较大,当TadA-8e通过linker连接上nCas9后,增加了蛋白质的刚性。分子刚性的增加限制了TadA-8e的移动,减少了TadA-8e二聚化界面碰撞结合的机会。当二聚化界面没有相结合时,相关界面中的疏水性区域可能会和蛋白质其他区域的疏水性氨基酸相互结合,特别是分子表面及柔性部位。这种结合将会造成多聚体的形成。
综上,本研究提出关于多聚体形成的假说:由于TadA-8e二聚化界面中疏水性区域的曝露,造成了分子间的聚集,从而产生大量的多聚体。当ABE8e无法形成二聚体结构时,其更倾向形成多聚体,而非单体。
2.2 多聚体成因验证实验
为了验证多聚体的形成是否与二聚化界面的打开有关,我们突变了TadA-8e二聚化界面的关键氨基酸,以阻碍其二聚化。观察二聚化界面进一步打开后,多聚体的比例是否会发生改变。若多聚体的比例增加,则可证明以上假说的正确性。
(1) 突变体的构建。为了寻找二聚化界面的关键氨基酸,运用PyMOL软件分析了该界面中特异性较高的部分。从图3(a) ABE8e二聚化界面的三维结构细节图可以发现:64位和74位的精氨酸位于此部位,且精氨酸带有正电荷,与对面的负电荷相互吸引。可以推断:这两个位点的氨基酸是二聚化界面中影响二聚化的核心氨基酸。
在确定需要突变的氨基酸后,使用Rosetta软件中的Fixbb模块对ABE8e的64及74位氨基酸进行突变优化。Fixbb模块共输出5000个结果,先通过Python程序剔除重复结果,再以蛋白质的能量值由低到高进行排序,然后选出能量最低的突变体。这样,最终确定的突变体中R64突变为L;R74突变为E。我们将此突变体命名为ABE8e-T。从图3(b)突变体的测序图中可以看出,64位的核酸序列为CTG;74位的核酸序列为GAA,说明突变体ABE8e-T的表达质粒构建成功。
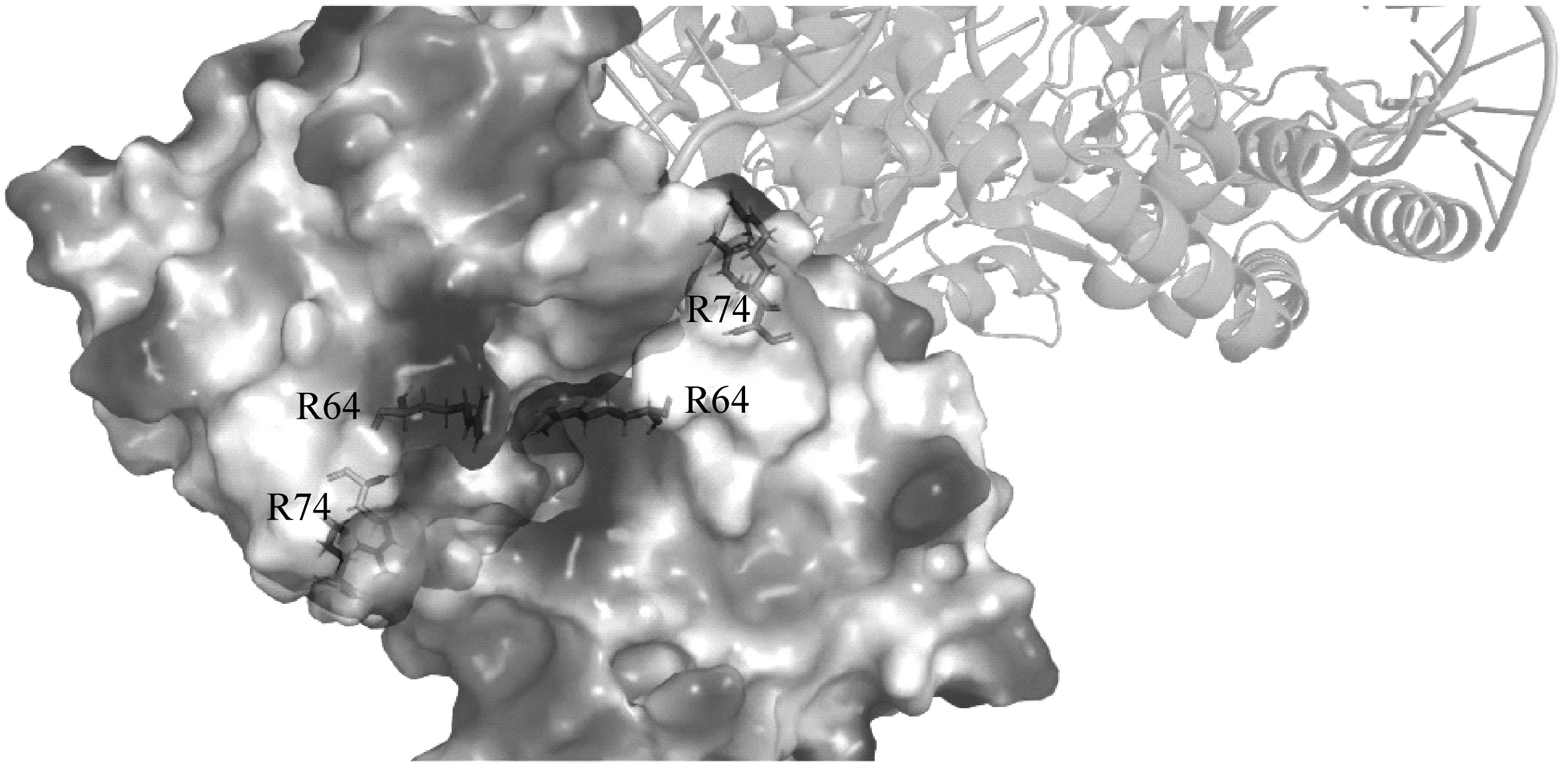
(a) ABE8e突变位点图
(2) 突变体的纯化。 为了观察ABE8e-T的结构形态,采用平台工艺条件对ABE8e-T进行表达纯化。先进行Ni柱纯化,再取其Ni柱层析收获的洗脱液进行凝胶层析纯化,分离多聚体和二聚体。ABE8e-T的Ni柱亲和层析纯化结果见图4(b)。由图可知,ABE8e-T经Ni柱纯化后纯度较高。ABE8e-T的凝胶层析纯化结果如图4(c)所示。从分子筛的实验结果见图4(c)中可知,在20 mmol/L Tris 0.5 mol/L KCL 缓冲体系中,F1的出峰位置约在7.2 mL,F2的出峰位置约为9.3 mL。根据Superdex的说明书可知,660 kDa的标准蛋白出峰位置约在8.0 mL,440 kDa的标准蛋白出峰位置约在8.8mL,66 kDa的标准蛋白的出峰位置约在12.5 mL。ABE8e的单体理论分子量为182 kDa,二聚体理论分子量为364 kDa。因此,我们判断,F1为多聚体,占比为34.2%,F2为二聚体,占比为43.8%。凝胶层析实验结果显示ABE8e-T的多聚体占比为71.6%,比例远高于二聚体的占比27.1%。对比ABE8e-T与ABE8e的凝胶层析图谱见图4(d)后发现:ABE8e-T的多聚体比例大幅度上升,二聚体比例大幅度下降。由此说明,在二聚化界面打开后,ABE8e-T没有倾向变成单体,而是倾向于多聚体。
综上,ABE8e-T的体外纯化实验结果验证了2.1中对ABE8e多聚体形成的理论分析假说。
(3) 缓冲液优化。在蛋白质制备的过程中,通过优化缓冲溶液的组分,可以有效地防止及抑制蛋白质聚集倾向[12]。在确认了ABE8e聚集体的成因后,本研究拟通过此方法来抑制多聚体的比例,从而帮助ABE8e恢复其二聚体结构。精氨酸因其对蛋白质分子有良好的保护作用,已经广泛地应用于制剂及下游纯化工艺中[16-18],其中精氨酸盐酸体系抑制分子间聚集的效果最佳[19, 20]。探究其机理,有两方面可能的影响因素:其一,精氨酸侧链能够与某些氨基酸侧链产生有利的相互作用,特别是对疏水性氨基酸上的芳香烃类的侧链[17]。其二,精氨酸侧链中的胍基能以游离基团的形式存在于缓冲体系中,并与蛋白质分子表面产生相互作用[21],减弱分子间的作用力。因此,精氨酸可以在不影响蛋白质二级及三级结构折叠的情况下,减少蛋白质与蛋白质之间的疏水作用力,从而有效地抑制蛋白质之间的聚集[19, 21]。所以,本研究在原缓冲体系中添加了0.5 mol/L 精氨酸,将缓冲体系优化为:20 mmol/L Tris,0.5 mol/L KCl,0.5 mol/L 精氨酸盐酸 pH 8.0,以抑制ABE8e多聚体的形成。
从图4(c)和图4(d)的凝胶层析实验结果可知,优化了缓冲体系后,ABE8e的二聚体比例上升到52.8%,多聚体比例降至18.2%。对比两种缓冲体系下凝胶层析谱发现:在保持其它条件不变的情况下,仅在原缓冲体系中添加0.5 mol/L 精氨酸盐酸后,ABE8e的二聚体比例上升了8.8%,多聚体比例下降了16.0%。可见,精氨酸能有效地抑制多聚体的形成。

图4 ABE8e-T及ABE8e缓冲体系优化纯化实验结果
此外,我们还尝试了仅含有20 mmol/L Tris 0.5 mol/L精氨酸pH 8.0的缓冲体系。在这个缓冲体系下,负电荷相对减少了一定数目,因而ABE8e的多聚体比例也提升到40.6%,二聚体占比为35.4%。这再次表明,在缺少负电荷的环境下,ABE8e不稳定,容易形成多聚体。
3 结论与讨论
前期研究表明,ABE8e在提纯的过程中发现了出现多聚体比例偏高的现象。为降低多聚体的比例,提高二聚体的含量,本研究首先探究了多聚体产生的原因。通过研究与分析TadA-8e疏水性氨基酸分布、电荷分布及ABE8e的三维结构,推测ABE8e分子在未形成二聚体的情况下,曝露了二聚化界面中疏水性区域,这些区域倾向与分子表面的其它疏水性氨基酸结合。这种倾向加剧了分子之间的结合力,从而导致了多聚体的形成。
为了验证理论推测的正确性,本研究构建了可进一步阻碍二聚体形成的相应突变体ABE8e-T。ABE8e-T的纯化结果证实了ABE8e多聚体形成的理论推测。在找到多聚体的成因后,本研究通过优化缓冲体系,在缓冲液中添加0.5 mol/L 精氨酸盐酸后,有效地降低了多聚体的比例。优化后的缓冲体系可以为其它碱基编辑器的制备提供了一种新的技术方案。
在常规缓冲体系优化的过程中,一般通过试错的方式来摸索工艺条件。这样的方式费时费力,效率低下。在本研究中,使用PyMOL建模软件分析了蛋白质的结构信息,可以快速精准地寻找引起聚集的关键氨基酸。这样,在确认具体原因后再寻找解决方案可以有效地提高了缓冲体系的优化效率。不难预见,当其它碱基编辑器遇到类似聚集问题时,也可考虑应用相同的方式解决,这为纯化制备工艺的开发提供了有用的思路。
还有,现有的ABE定向进化优化主要方向是通过增强其与底物DNA 的结合力来实现提高编辑活性及降低脱靶效应。本文的结果提示,当蛋白质不易发生聚集时,将有助于提高蛋白质的稳定性,增加其编辑活性。所以,本研究的结果也可以为ABE的定向进化优化提供了新的方向。

