LEF1基因在巴什拜羊不同毛色皮肤组织中的DNA甲基化及mRNA表达水平分析
侯晨曦,洪文娟,何宗龙,决肯·阿尼瓦什
(新疆农业大学动物科学学院,乌鲁木齐 830000)
0 引 言
【研究意义】羊毛是重要的纺织原材料[1]。毛色是绵羊重要的外观表型特性,在品种鉴定、特征描述等起着重要作用[2]。新疆绵羊品种,巴什拜羊是具有代表性的优质品种之一,其毛色主要为棕红色,少量为青灰色、白色等颜色,虽为粗毛羊,但具有产毛量大,绒毛含量高,被毛光泽好等优点,具有较高的毛用价值[2]。研究巴什拜羊毛色的形成,对更好利用天然毛色提高其经济价值具有重要意义。【前人研究进展】绵羊毛色形成过程比较复杂,但其主要受遗传因素影响。目前已经发现了300多个基因座和150多个已识别的毛色相关基因[4]。这些毛色相关基因通过调控黑色素细胞产生的真黑素和褐黑素的分布和数量来影响绵羊的被毛颜色[5]。Wnt/β-catenin信号通路是毛色形成的重要通路,能够抑制黑色素的合成以及运输[6]。淋巴增强因子-1(Lymphoid enhancer factor 1,LEF1)是这条通路中重要的转录因子,Van Genderen C等[7]研究发现,敲除小鼠皮肤组织LEF1后,小鼠由于毛囊中黑色素生成失败而呈现浅色。LEF1基因在毛囊发育与再生过程中均有表达,对毛色的形成有重要影响。在正常细胞中,DNA确保和调节基因的适当表达以及稳定基因的沉默,调节基因组的功能,改变遗传表现,对毛色的形成过程也有重要的调控作用[8]。【本研究切入点】目前对于在毛色基因的研究比较深入,但对LEF1基因的研究,特别对棕红色与青灰色间LEF1基因的表达研究均较少。需分析LEF1基因在红棕色与青灰色巴什拜羊皮肤组织中DNA甲基化与mRNA的表达水平。【拟解决的关键问题】采用亚硫酸氢盐修饰分析巴什拜羊皮肤组织中LEF1基因的甲基化水平,分别从表观遗传和分子遗传两个方面来分析皮肤组织中LEF1基因的DNA甲基化水平与mRNA表达量之间的关系,探讨LEF1基因的DNA甲基化水平对不同毛色的影响,了解DNA甲基化对LEF1基因转录的调控作用,筛选与毛色相关的表观遗传标记。
1 材料与方法
1.1 材 料
1.1.1 试验羊
试验动物选取新疆塔城地区裕民县红棕色与青灰色周岁巴什拜羊各12只,个体间体重相近,健康无疾病,取其左肩胛部的皮肤组织,大小约为0.6 cm2。将皮肤组织样本置于液氮中运输保存,用于RNA和DNA的提取。
1.1.2 主要试剂
Trizol总RNA抽提试剂、Trizol总RNA抽提试剂、PrimeScriptTMRT reagent Kit with gDNA Eraser(Perfect Real Time)试剂盒、TB Green Premix Ex TaqTM(Tli RNaseH Plus)试剂盒、琼脂糖、EZ DNA Methylation-GoIdTMKit、胶纯化试剂盒、pMD19-T Vector Cloning Kit、DH5α感受态细胞等。
1.2 方 法
1.2.1 总DNA的提取与亚硫酸氢盐修饰基因组DNA
取巴什拜羊皮肤组织10~30 mg于研钵中研磨至粉末状,采用酚-氯仿抽提法对不同毛色巴什拜羊的皮肤样组织提取总DNA。将纯度与浓度检测合格后的DNA按照EZ DNA Methylation-GoIdTMKit试剂盒说明书对基因组DNA进行处理。
1.2.2 CpG岛预测及引物设计
使用Chormas软件打开巴什拜羊LEF1基因亚硫酸氢盐处理后的测序峰图,对测序结果与LEF1基因原始序列进行比对,统计LEF1基因甲基化位点中的胞嘧啶(C)转换为胸腺嘧啶(T)的位点。通过在线软件MethPrimer(http://www.urogene.org/methprimer/)对LEF1基因甲基化位点的预测分析。根据NCBI上的GenBank(https://www.ncbi.nlm.nih.gov/gene/)数据库中查询的绵羊β-actin、LEF1的基因序列,使用Primer Premier 5.0软件、Methprimer在线软件与BLAST在线网页进行荧光定量引物、BSP的设计,确保以保证引物的特异性后,最终由生工生物工程(上海)股份有限公司合成。表1

表1 荧光定量与BSP扩增引物
1.2.3 PCR扩增及产物纯化
将EZ DNA Methylation-GoldTMKit试剂盒修饰后的DNA进行PCR扩增反应。对PCR扩增产物进行凝胶电泳检测,确定片段大小后,使用PCR产物纯化试剂盒(天根)对产物进行纯化。纯化后的产物与pMD19-T Vector连接,转化DH5α感受态细胞,37℃培养12~16 h,挑取培养板上的白色单菌落,在1.5 mL的含Amp的LB液体培养基中200 r/min过夜培养至培养基浑浊;对菌液进行PCR检测,鉴定其产物片段与目的片段一致后,将样本送至公司测序。表2,表3
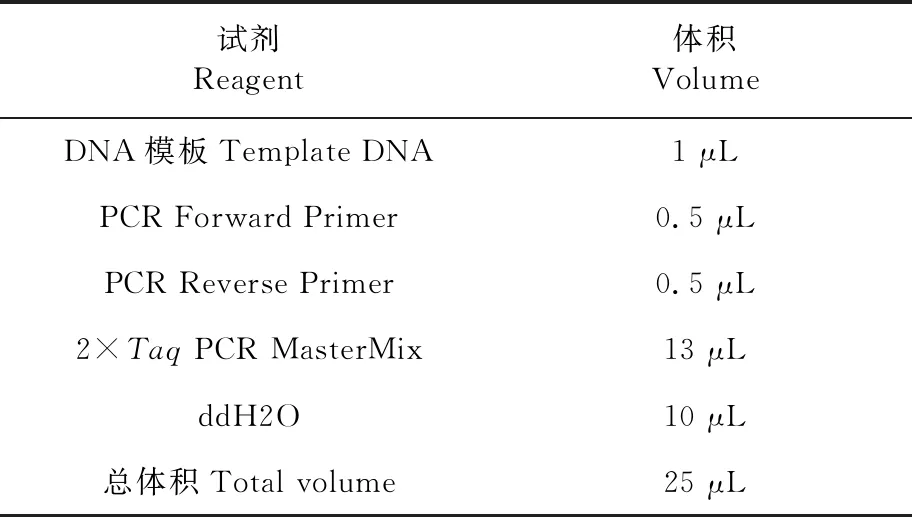
表2 PCR扩增反应体系
1.2.4 LEF1基因在不同毛色巴什拜羊皮肤组织中的mRNA表达
试剂与用量为cDNA 2 μL,PCR Forward/Reverse Primer(10 μmol/L)各1 μL,TB Green Premix ExTaqII 12.5 μL,Rnase-Free ddH2O 8.5 μL。扩增程序为95℃ 3 min,95℃ 5s,最适温度58℃ 30s,40个循环,95℃ 10s,65℃ 5s,95℃ 5s。扩增结束后,用琼脂糖凝胶检测产物片段大小是否与预期的基因片段大小一致。
1.3 数据处理
用Excel 2019对数据进行整理,将每个样品的3次重复中差异较大的数值去除,运用相对表达量计算公式F=2-△△CT计算相对表达量,并运用SPSS统计分析软件单因素方差分析目的基因在红棕色与青灰色巴什拜羊皮肤组织中的表达差异,使用Graph Prime 8软件绘制柱形图。

表3 PCR扩增反应程序
2 结果与分析
2.1 总DNA与RNA质量检测
研究表明,用酶标仪对提取出红棕色与青灰色巴什拜羊皮肤组织总RNA进行浓度与纯度的检测,检测结果显示,总RNA的浓度在100 ng/μL以上,纯度均在1.8~2.0,通过结果可知总RNA的浓度与纯度均较好;用琼脂糖凝胶电泳对总RNA进行完整性检测,总RNA完整性较好,可以进行反转录试验。图1
2.2 PCR扩增结果
研究表明,利用LEF1基因的甲基化引物以甲基化修饰后的DNA为模板进行PCR扩增,通过2%琼脂糖凝胶电泳对PCR产物检测,产物的片段大小为160 bp,与目的片段大小一致。图2
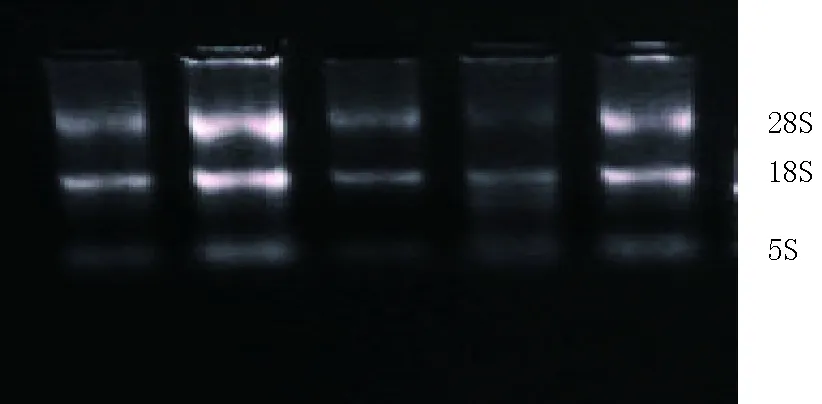
图1 总RNA检测结果
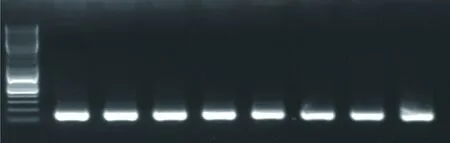
注:M:1 kb Plus DNA Marker;1~8:LEF1基因
2.3 测序结果统计与序列比对
研究表明,巴什拜羊LEF1基因启动子区的DNA序列存在1个从1 642 bp~1 801 bp的甲基化密集区域,长度为160 bp,CPG位点为14个,分别在目的片段的第31、39、44、57、74、91、94、101、103、105、114、123、129和136 bp处。图3
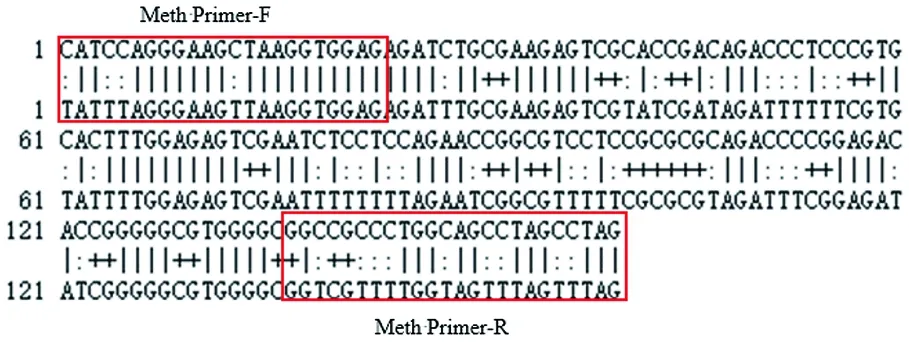
图3 LEF1基因的甲基化区域预测序列
2.4 LEF1基因的DNA甲基化模式
研究表明,共有7个CpG位点。LEF1基因在两种毛色巴什拜羊中的甲基化概率均较低,红棕色巴什拜羊皮肤组织中的甲基化率为4.29%,在青灰色巴什拜羊皮肤组织中的甲基化率为2.86%。不同毛色的启动子区甲基化CpG位点不同,红棕色巴什拜羊的启动子区CpG位点为CpG4、CpG6、CpG14,青灰色为CpG1、CpG3、CpG7、CpG14。图4,图5
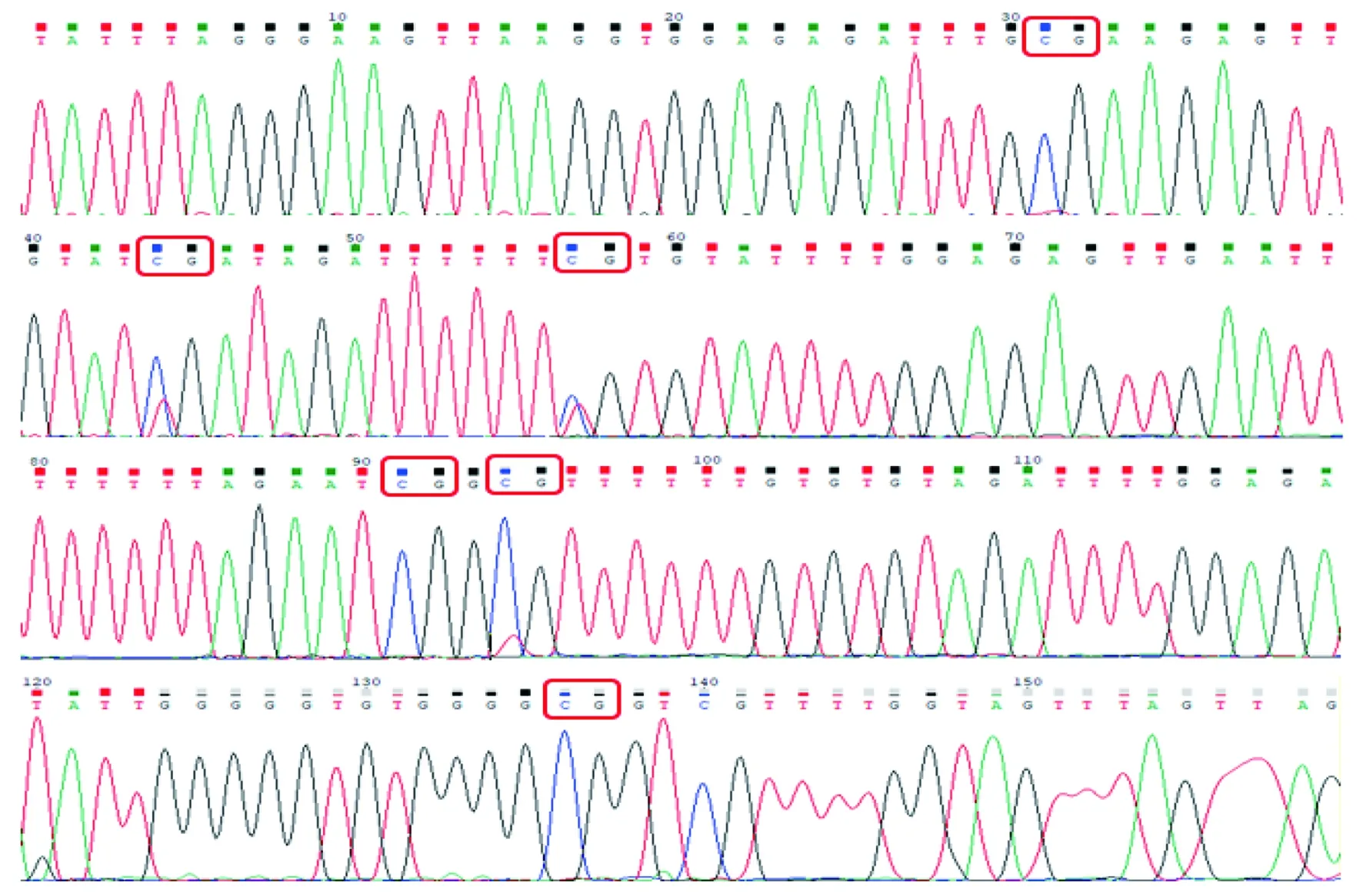
图4 LEF1基因甲基化区域测序峰
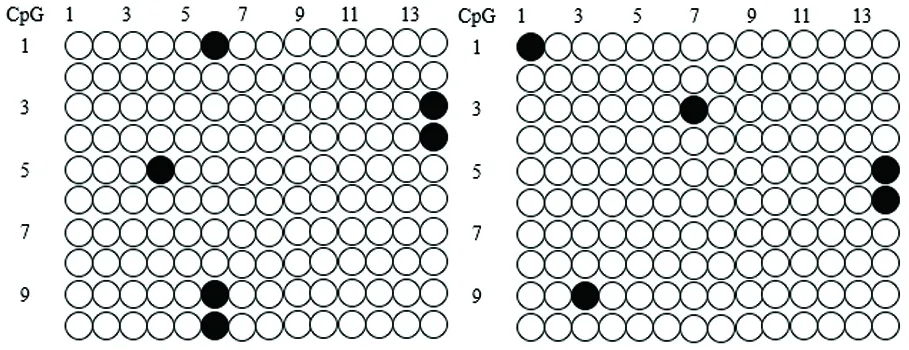
注:●:表示发生甲基化;○:表示未发生甲基化
2.5 LEF1基因在红棕色与青灰色巴什拜羊皮肤组织中表达量
研究表明,LEF1基因在青灰色巴什拜羊皮肤组织中的表达量极显著高于红棕色(P< 0.01),并且LEF1基因mRNA表达趋势与甲基化水平呈显著负相关(P< 0.05),表明LEF1基因的DNA甲基化对其表达量具有一定的负调节作用。图6
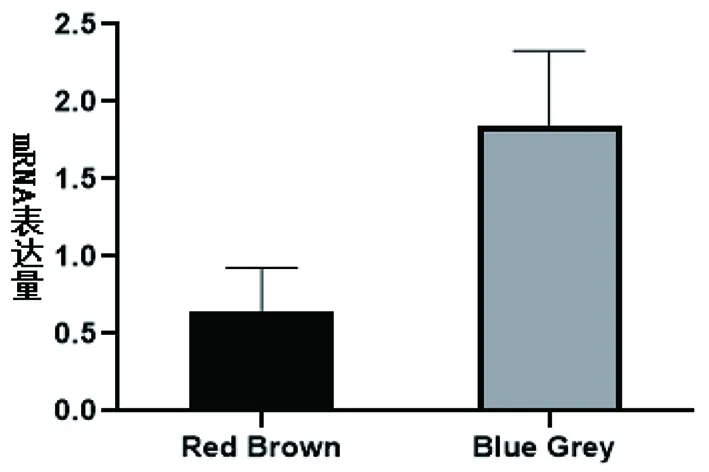
图6 红棕色与青灰色巴什拜羊羊皮肤组织中LEF1基因的表达量
3 讨 论
哺乳动物毛色的变化是由褐黑素和真黑素的生化功能、数量、分布和可用性决定的,而黑色素细胞的数量通常是不变的[9]。褐黑素使哺乳动物产生红棕色和黄色的毛色或肤色表型,而真黑素使哺乳动物产生黑色和棕色的毛色或肤色表型[10]。LEF1基因参与两种黑色素的生成过程,在毛囊发育与再生过程中均有表达,当敲除LEF1时,小鼠在毛发和胡须的形成方面会有缺陷[11]。Catherine A等[12]研究发现,当淋巴增强子结合因子(LEF1)的结合位点改变时,会降低培养LEF1反应性和增强子活性,从而使小鼠在毛发色素沉着方面表现出显著差异。Adam M.M等[13]通过转录组学对高度多型毒蛙颜色和模式的研究发现,LEF1基因参与黑素细胞分化和发育以及黑色素的生成。Song等[14]研究发现,LEF1基因黑色水貂中的表达量低于白色水貂,表达水平经qRT-PCR验证,结果与RNA-seq分析一致,说明LEF1基因与水貂两种毛色的形成有关。研究中LEF1基因在青灰色巴什拜羊皮肤组织中的表达量高于红棕色巴什拜羊皮肤组织中的表达量且高于红棕色表达量的3倍,这个结果与杨磊等[15]对不同毛色羊驼皮肤组织中LEF1的表达量结果基本一致。
DNA甲基化是表观遗传修饰中最为常见的修饰方式,普遍发生于动植物等真核生物体内以及细菌等原核生物体内[16]。随着DNA甲基化研究的不断深入,DNA甲基化的应用领域也不断增多,如基础研究中的癌症、发育生物学与干细胞分化等方面的研究,对改良畜禽新品种有重要意义[17]。基因启动子区的甲基化水平对基因表达量具有调节作用[18]。曼则热·朱尔丁等[19]研究发现哈萨克羊脂肪组织中的MSTN基因甲基化概率高于肌肉组织。刘亮等[20]对胶质瘤组织与正常脑组织中miR-133a的表达与甲基化水平的研究,发现DNA甲基化对miR-133a的表达具有负调节作用。CHEN等[21]通过对非肿瘤组织与肾细胞癌组织中miR-766-3p的比较,发现启动子区高度甲基化的肾细胞癌组织中,miR-766-3p的表达量低于非肿瘤组织,表明DNA的甲基化抑制其表达。黄永震[22]对中国地方黄牛IGF2和ZBED6基因的研究发现,在2个发育时期6中组织中mRNA表达量和甲基化水平呈现负相关。研究中LEF1基因在红棕色与青灰色皮肤组织中甲基化水平均较低,但红棕色甲基化水平高于青灰色,而红棕色皮肤组织中LEF1基因的表达量低于青灰色巴什拜羊,这与DNA启动子区甲基化水平与基因表达呈负相关结果一致,推测巴什拜羊皮肤组织中LEF1基因通过DNA甲基化修饰调节基因的表达,调控巴什拜羊的毛色。
4 结 论
共有14个CPG位点,两种毛色的甲基化概率分别为4.29%与2.86%,同时LEF1基因在青灰色的表达量极显著高于红棕色(P< 0.01),LEF1基因启动子区的DNA甲基化水平与mRNA表达量呈显著的负相关(P< 0.05),DNA甲基化水平对红棕色与青灰色巴什拜羊的毛色形成具有调节作用,可作为一个候选的毛色遗传标记。
参考文献(References)
[1] 杨广礼. 动物毛色形成的调控机制研究[J]. 黑龙江畜牧兽医, 2014,(5): 45-48.
YANG Guangli. Study on the regulation mechanism of animal hair color formation [J].HeilongjiangAnimalHusbandryandVeterinary, 2014,(5): 45-48.
[2]Gebreselassie G, Liang B, H Berihulay, et al. Genomic mapping identifies two genetic variants in the MC1R gene for coat colour variation in Chinese Tan sheep [J].PLoSONE, 2020, 15(8): e0235426.
[3]新疆家畜家禽品种志编写委员会. 新疆家畜家禽品种志[M]. 乌鲁木齐: 新疆人民出版社, 1985.
Compilation Committee of Xinjiang Livestock and Poultry Breeds.SpeciesoflivestockandpoultryinXinjiang[M]. Urumqi: Xinjiang People's Publishing House, 1985.
[4]Cieslak M, Reissmann M, Hofreiter M, et al. Colours of domestication [J].BiologicalReviews, 2011, 86(4): 885-899.
[5]徐伟, 封竣淇, 黄兰, 等. TYR基因研究进展[J]. 中国畜牧杂志, 2017, 53(4): 23-27.
XU Wei, FENG Junqi, HUANG Lan, et al. Research progress of TYR gene [J].ChineseJournalofAnimalHusbandry, 2017, 53(4): 23-27.
[6]Sethi J, Vidalpuig A. Wnt signalling and the control of cellular metabolism [J].BiochemicalJournal, 2010, 427(1): 1-17.
[7]Van Genderen C, Okamura R M, Farias I, Quo R G, et al. Development of several organs that require inductive epithelial-mesenchymal interactions is impaired in LEF-1-deficient mice[J].GenesDev. 1994, 8(22): 2691-703.
[8]Ozren, Bogdanovi, Ryan, et al. DNA methylation and the preservation of cell identity-Science Direct [J].CurrentOpinioninGenetics&Development, 2017, 46: 9-14.
[9]Ito S, Wakamatsu K. Chemistry of mixed melanogenesis: pivotal roles of dopaquinone [J].PhotochemistryandPhotobiology, 2008, 84(3): 582-592.
[10]Bonaventure J, Domingues M J, Larue L. Cellular and molecular mechanisms controlling the migration of melanocytes and melanoma cells [J].PigmentCell&MelanomaResearch, 2013, 26(3):316-325.
[11]Dasgupta R, Fuchs E. Multiple roles for activated LEF/TCF transcription complexes during hair follicle development and differentiation [J].Development, 1999, 126(20): 4557-4568.
[12]Guenther,C.A, Tasic,B, Luo,L., et al. A molecular basis for classic blond hair color in Europeans [J].NatureGenetics, 2014, 46(7): 748-752.
[13]Stuckert A, Moore E, Coyle K P, et al. Variation in pigmentation gene expression is associated with distinct aposematic color morphs in the poison frog Dendrobates auratus [J].BMCEvolutionaryBiology, 2019, 19(1): 85.
[14]Song X, Xu C, Liu Z, et al. Comparative Transcriptome Analysis of Mink (Neovison vison) Skin Reveals the Key Genes Involved in the Melanogenesis of Black and White Coat Color [J].ScientificReports, 2017, 7(1): 12461.
[15]杨磊, 王海东, 王祎, 等. Lef-1基因在棕色和白色羊驼皮肤差异表达[J]. 中国生物化学与分子生物学报, 2011, 27(11): 1061-1066.
YANG Lei, WANG Haidong, WANG Yi, et al. Differential expression of LEF-1 gene in brown and white Alpaca Skin [J].ChineseJournalofBiochemistryandMolecularBiology, 2011, 27(11): 1061-1066.
[16]蒋雪, 臧艺玫. 表观遗传概述[J]. 生物学教学, 2015, 40(4): 2-4.
JIANG Xue, ZANG Yimei. Overview of Epigenetics [J].BiologyTeaching, 2015, 40(4): 2-4.
[17]钟焱, 徐慧, 彭凤兰. DNA甲基化在基因表达调控中的意义及研究进展[J]. 中国医药导报,2019, 16(14): 33-36.
ZHONG Yan, XU Hui, PENG Fenglan. Significance and research progress of DNA methylation in gene expression regulation [J].ChinaMedicalHerald, 2019, 16(14): 33-36.
[18]龙鑫. 乌珠穆沁羊与呼伦贝尔短尾羊尾椎形态、Brachyury(T-box)基因型特征及甲基化研究[D]. 呼和浩特: 内蒙古农业大学, 2019.
LONG Xin.Studyonmorphology,Brachyury(T-box)genotypeandmethylationofWuzhumuqinsheepandHulunbeiersheep[D]. Hohhot: Inner Mongolia Agricultural University, 2019.
[19]曼则热·朱尔丁, 姚力丹, 包敖敦格日乐,等. 哈萨克羊MSTN基因的DNA甲基化分析[J]. 新疆农业科学, 2018, 55(7): 1326-1332.
Manzere Zhuerding, YAO Lidan, Baoaodun, Gerile, et al. DNA methylation analysis of MSTN gene in Kazakh sheep [J].XinjiangAgriculturalSciences, 2018, 55(7): 1326-1332.
[20]刘亮, 朱正权, 阿满·哈迪尔别克, 等. DNA甲基化对miR-133a表达及胶质瘤细胞增殖及凋亡的影响[J]. 现代肿瘤医学, 2021, 29(8): 1296-1300.
LIU Liang, ZHU Zhengquan, Aman Hadierbuick, et al. Effect of DNA methylation on mir-133a expression and proliferation and apoptosis of glioma cells [J].ModernOncology, 2021, 29(8): 1296-1300.
[21]Chen C, Xue S, Zhang J, et al. DNA‐methylation‐mediated repression of miR‐766‐3p promotes cell proliferation via targeting SF2 expression in renal cell carcinoma [J].InternationalJournalofCancer, 2017, 141(9): 1867-1878.
[22]黄永震. 黄牛肌肉生长发育相关基因甲基化鉴定及IGF2和ZBED6基因的转录调控研究[D]. 杨凌:西北农林科技大学, 2014.
HUANG Yongzhen.IdentificationofmethylationandtranscriptionalregulationofIGF2andzbed6genesincattlemusclegrowthanddevelopment[D]. Yangling: Northwest University of agriculture and forestry science and technology, 2014.

