基于PMV-LSTM的中文医学命名实体识别
陈雪松,朱鑫海,王浩畅
(1.东北石油大学 电气信息工程学院,黑龙江 大庆 163318;2.东北石油大学 计算机与信息技术学院,黑龙江 大庆 163318)
0 引 言
命名实体识别(named entity recognition,NER)作为信息抽取[1]的关键性任务,在关系抽取[2]、文本摘要[3]、机器翻译[4]等任务中发挥着重要作用。医学领域命名实体识别是指从海量的医学数据中识别出有价值的信息的过程,包括症状信息、药物信息、身体部位信息、疾病诊断信息等。有效的命名实体识别过程可以挖掘出医学文本关键信息,对于电子病历等文本的规范化和标准化有着重要的作用。
近年来有学者使用深度学习的方法[5-7]处理医学领域命名实体识别任务,然而目前大多数医学领域命名实体识别模型单独使用字符级别向量或者词汇级别向量,而忽略了将字符级别和词汇级别特征相结合作为句子特征表示的问题。Yue Zhang等[8]创造性地提出一种Lattice LSTM模型,以字符级别的中文命名实体识别模型为基础,融入大规模词表匹配信息,在4个通用数据集上验证引入词汇信息之后效果可以有比较好的提升。为解决中文医学文本结构复杂,且单纯使用字符级别特征或者词汇级别特征所导致的语义信息缺失问题,本文提出PMV-LSTM(pre-trained medical vectors long short term memory)方法,通过对大规模医学领域文本进行词向量和“字符分割”形式的字向量训练,然后将字向量送入Lattice LSTM中,通过Lattice结构巧妙地将词向量特征和字向量特征融合到一起,得到句子的特征表示,最后对特征表示经过CRF层进行解码,得到最终的实体标注结果。经过实验验证,PMV-LSTM模型与同类型模型相比,在不同医学领域文本命名实体识别任务中取得了优异效果。
1 算法模型
1.1 基本架构
基于深度学习的命名实体识别一般被看作是序列标注问题。通过特征抽取器对句子特征进行编码,然后使用解码器进行解码来进行端到端的处理,得到标注结果。虽然深度学习的技术一定程度上解决了NER任务对词典等外部信息的依赖,但只使用基于字符或者基于词汇的NER方法也会影响到命名实体识别的效果。为此,根据中文命名实体识别的特点,设计出PMV-LSTM方法来对中文医学文本进行命名实体识别。
本文所提出的PMV-LSTM模型结构分为3部分,嵌入层、Lattice LSTM层和CRF层。模型结构如图1所示。首先通过对输入的医学文本进行词级别和字符级别的Word2Vec训练,利用“字符分割”的方式增加字符级别向量的数量,所谓“字符分割”指的是将医学文本按字符进行切分,字符与字符之间以空格隔开,来达到字符分词的效果。然后将得到的字符级别特征输入到Lattice LSTM中进行编码,在Lattice LSTM中巧妙地利用Lattice结构将词级别特征结合到一起,最后输入到CRF层中进行解码,从而得到医学文本命名实体识别的标注结果,整个模型处理过程属于端到端的过程,不需要进行特征工程处理。

图1 PMV-LSTM模型结构
1.2 嵌入层
Word2Vec是一种新的词向量表示方法[9],通过Skip-gram和CBOW两种可选的训练方式将词汇之间的关系转化成空间中向量的距离问题,距离越近的向量所代表的词汇越相似。例如,医学文本中,“关节炎”和“风湿病”的相似度和相关度明显比和“新冠肺炎”要高。
本文采用Word2Vec中的Skip-gram模型学习得到词向量,PMV-LSTM模型需要将字符级别特征和词汇级别特征都学习到。另外,为了保证字符级别特征的质量,所以将词向量和字向量分开训练。首先从网络中爬取医学文本语料,对其进行去停用词处理,利用pkuseg分词工具[10]进行医学领域文本分词,同时为了保证分词质量,加入自定义的医学领域词典,然后利用gensim工具包[11]进行词向量训练,维度为300维。将得到的词向量中字符级别向量单独取出,余下的向量作为词汇级别向量使用。字符级别向量训练过程类似,不同的是医学文本不需要进行分词处理,而是对每个字符以空格隔开,得到类似于字符级分词的效果,然后再进行Word2Vec训练,得到的向量全部都是模型中可以使用的字符级向量。此时得到医学领域向量有542 554条,包括词汇级别向量534 427条,字符级别向量8127条。最后将字向量和词向量输入到Lattice LSTM中融合,以提高模型的性能。
1.3 Lattice LSTM
不同于英文文本的命名实体识别,中文文本的命名实体识别通常需要以字符级别为单位进行序列标注。这主要是因为中文文本的特点,并没有像英文那样以空格作为天然的分隔符,需要进行分词处理。然而中文分词由于文本的领域不同和模型准确率的原因会存在误差,导致基于字符级别的命名实体识别模型效果要优于基于词汇级别的命名实体识别模型。但并不是NER任务不需要词汇信息作为辅助,在NER模型中加入词汇信息不仅可以强化命名实体的实体边界,也可以看作是一种数据增强方式,以词汇信息作为辅助信息来使模型学习到更多的句子深层语义特征。
本文提出的PMV-LSTM模型,引入Lattice LSTM,重新训练适用于医学领域的词向量,以词汇作为外部资源信息,Lattice结构能够动态引入词汇信息,用特殊的门控机制来控制词信息流,对句子中所有可能存在的词和词典进行匹配来编码。

(1)
(2)

首先Lattice LSTM以字符级别的LSTM模型为基础,如果句子s中字符cj在词典中没有相匹配的词,也就是说当前位置j没有对应的词序列,则在此位置Lattice LSTM就是基于字符级别的LSTM模型。

(3)
(4)
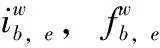

(5)
其中,WlT和bl为模型训练参数。
(6)
(7)
(8)
(9)
在NER模型训练过程中,反向传播参数Wc,bc,Ww,bw,Wl和bl,使模型动态的关注标注过程中更相关的词,选出最适合当前的字词表征。
最后加入CRF层,通过计算CRF中的发射分数和转移分数来对标注结果进行约束,学习到相邻标签的结果,保证最终预测结果的准确性。
2 实验设计
2.1 语料获取
训练出合适的词向量是PMV-LSTM模型的基础,本文用来训练词向量的医学文本语料有两部分,第一部分是采用爬虫技术从39健康网、99健康网、A+医学百科爬取的身体部位、疾病症状、诊断治疗等信息。第二部分是cMedQA中文社区医疗问答数据和互联网中搜集到的中文医疗对话数据集,涉及到疾病诊断、身体部位、治疗药物等信息,共约100万个问答对。最后经过数据清洗得到的有效医学文本语料约144万行,语料规模为650 MB大小。
2.2 分 词
分词是利用Word2Vec进行词向量训练的关键,在NLP领域中,一般使用jieba分词工具[12]进行分词操作,在通用领域取得了较好的效果。但是在中文医学领域,通用的分词工具在医学文本上并不一定有较好的效果。所以,本文使用北京大学开发的具有领域分词功能的pkuseg分词工具,该分词工具可以根据待分词文本的特点选择对应领域的分词模型。
本文在使用了医学领域的分词模型之后,为了确保分词的准确性,另外添加了一个自定义的医学词典。所构造的词典是从互联网中搜集到的符合ICD10标准[13]的疾病名称、药名、症状词典和医院用电子病历词库[14,15],共77 487条医学词汇。
2.3 Word2Vec训练
经过上述的分词处理之后,本文使用Python中的gensim工具包进行训练词向量。由于Lattice LSTM的特点,需要使用句子的词向量信息和字向量信息,首先对上述分词之后的医学文本进行普通的词向量训练,得到大规模的医学词向量539 809条,其中词汇级别向量534 427条,字符级别向量5382条。这里只选择词汇级别的向量作为所需要的词向量。然后对字符级别向量进行训练,所使用的方法和之前类似,将获取的医学文本以字符为单位用空格分隔,相当于分词操作,然后对此时的文本进行Word2Vec训练,得到字符级别的向量,共8127条。此时得到最终的医学领域词向量有542 554条,包括词汇级别向量534 427条,字符级别向量8127条。
在训练过程中,Word2Vec模型选择Skip-gram算法,窗口大小为5,最小词频选择1,采用层次softmax技巧进行训练,字词向量的维度选择300维。为了更直观体现出字嵌入和词嵌入的效果,分别选取“痛”和“心脏病”来对其字嵌入相关性和词嵌入相关性进行分析。如图2所示,可以看出通过Word2Vec训练得到的两个词,在向量空间上距离相近时,实际词义也相关。
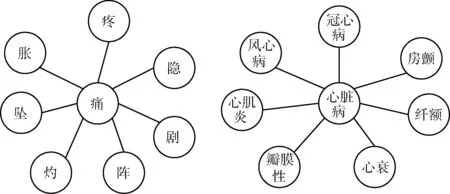
图2 字词嵌入相关性
2.4 实验环境及评价指标
本文实验使用Python语言进行编写,深度学习框架选择Pytorch,在Ubuntu 5.4.0系统,Intel(R) Core(TM) i7-9700K处理器,Nvidia RTX2080 Super显卡环境下进行本实验。医学领域命名实体识别的性能评估采用精确率(precision,P)、召回率(recall,R)和F1值3个指标。
2.5 实验数据集
为了验证本文模型对医学领域命名实体识别任务的普适性,使用了3个医学领域的数据集来进行实验,涉及到医学电子病历领域和医学社区问答领域,分别是CCKS2017命名实体识别数据集[16]、cEHRNER命名实体识别数据集和cMedQANER命名实体识别数据集[17]。
CCKS2017(China conference on knowledge graph and semantic computing 2017)为2017年全国知识图谱与语义大会的评测任务数据,有4种类型的电子病历,一般项目、病史特点、诊疗经过和出院情况,其中有身体部位、症状和体征、疾病和诊断、检查和检验、治疗5种类型的医学领域实体需要识别出来,实体数量分别为20 077个、14 583个、5205个、19 959个和5456个,标注模式为BIO模式。
cEHRNER为从中国电子健康记录(Chinese electronic health records)标记的数据集。有症状、手术、疾病和诊断、解剖部位、药物、实验室检验、影像检查7种医学领域实体,标注模式为BIO模式。其中训练集914条电子病历数据,验证集44条电子病历数据,测试集41条电子病历数据。
cMedQANER为从中文医学社区问答中标记的数据集。有症状、患者类型、疾病、药物、治疗、身体部位、特征、测试、时间、科室、生理机能11种医学领域实体,标注模式为BIO模式。其中训练集1673条医学问答数据,验证集175条医学问答数据,测试集215条医学问答数据。
3 实验结果分析
3.1 实验1 分词工具和词向量维度对医学实体识别的影响
词向量的优劣对于本文模型的效果有着重要的影响。为了得到合适的医学领域词向量,首先需要对医学语料进行文本分词,然后再进行词向量的训练。本文从所使用的分词工具、词向量训练的维度两个角度进行实验,充分验证不同的方式对于医学命名实体识别效果的影响,寻找到使得模型效果最好的参数。
本文选取目前自然语言处理中常用的jieba分词和具有领域分词功能的pkuseg分词两种工具,并且从50维、100维、200维、300维4个维度进行对比,在CCKS2017数据集上进行实验验证。此时所使用的词汇级别向量和字符级别向量为Word2Vec直接训练得出,从中提取出字符级别的向量为字符向量,共5382条,其余部分为词汇级向量,共534 427条。不同分词工具和词向量维度的医学实体识别结果如图3所示。

图3 不同分词和词向量维度实体识别结果对比
从图3中可以看出,词向量维度为50维、100维、200维和300维情况下,pkuseg和jieba均对医学实体识别的效果有所影响。当选择迭代次数为50次时,随着迭代次数的增加,医学实体识别的F1值在各个维度均处于先上升然后趋于平缓的状态。词向量维度在50维、100维、200维及300维时,使用pkuseg分词工具进行分词,然后进行实体识别的效果要好于使用jieba分词的结果,最好的结果处于使用pkuseg分词工具,并且词向量维度为300维时。因此,本文选取pkuseg分词工具进行分词,以确保分词效果,词向量维度选择300维,可以更大程度上体现出语义信息。
3.2 实验2 模型对比
为了进一步探讨PMV-LSTM模型的有效性,本文在多个医学领域数据集上进行验证,并且与几组基线模型进行对比。
表1中列出了几种对比模型的具体信息,其中字符向量训练方式中分开训练指Lattice LSTM字符向量是利用Giga-Word经过大规模标准分词后的语料[18]训练得到,词汇向量是使用CTB6.0(Chinese treebank 6.0)语料库[19]训练得到;联合训练是指利用Word2Vec在医学文本语料上将字符和词汇向量一起训练得到;字符分割训练指字符级别向量是在医学文本语料上利用字符分割的方式训练得到,词汇级别向量取联合训练中的词汇级向量。
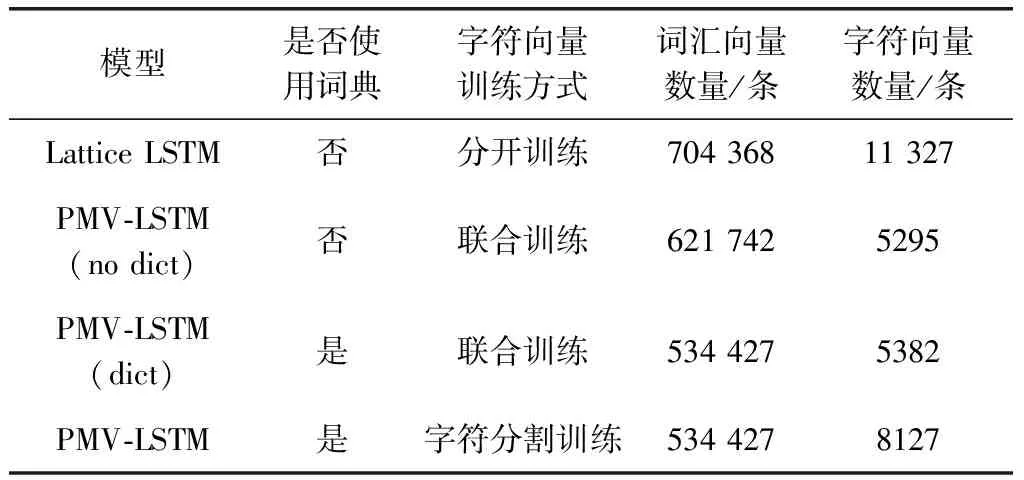
表1 对比模型信息
另外,BiLSTM、LSTM-CRF和BiLSTM-CRF是经典的命名实体识别模型,BiLSTM模型通过利用词向量信息,然后将其输入到BiLSTM中进行中文医学命名实体识别。LSTM-CRF模型使用单向LSTM进行编码,然后使用CRF进行解码,BiLSTM-CRF在LSTM-CRF的基础上使用了双向LSTM,能更好捕获双向的语义依赖信息。以上3种命名实体识别模型均采用随机初始化词序列特征表示,利用NCRFpp框架[20]进行复现。
PMV-LSTM模型在CCKS2017、cEHRNER和cMedQANER数据集的测试集结果见表2~表4,与其它经典模型比较的结果见表5。

表2 CCKS2017上识别效果对比

表3 cEHRNER上识别效果对比

表4 cMedQANER上识别效果对比
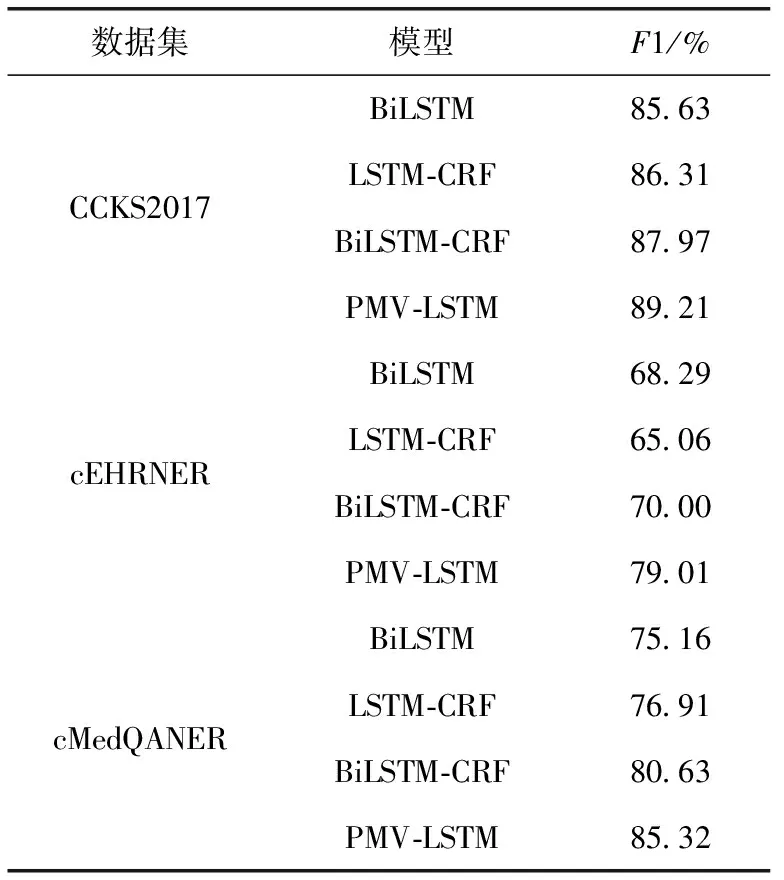
表5 与其它经典研究比较结果
由表2可知,在CCKS2017数据集上PMV-LSTM(dict)模型比PMV-LSTM(no dict)模型的F1值高了0.3%,表明在分词过程中加入自定义的医学领域词典对词向量的训练有着积极的作用。与PMV-LSTM(dict)模型相比,PMV-LSTM模型F1值也有着0.16%的效果提升,表明使用“字符分割”来得到字符级向量的方式,能够增加医学词汇的匹配度。同时PMV-LSTM模型比Lattice LSTM基线模型效果提升0.92%,说明PMV-LSTM模型在医学领域专门训练词向量,使用“字符分割”来得到字符级向量的方式,能够提升医学领域命名实体识别的效果。
进一步对比表3和表4,在cEHRNER和cMedQANER数据集中PMV-LSTM(dict)模型比PMV-LSTM(no dict)模型同样都有一定的提升,F1值提升幅度分别为0.93%和0.1%,这也说明词典作为额外的资源对医学领域复杂的专业名词分词有着帮助作用。PMV-LSTM模型比PMV-LSTM(dict)模型在两个数据集中F1值分别提升了2.47%和0.39%,进一步说明字符级向量的数量会影响模型的结果。通过对医学文本语料进行空格分隔操作,可以得到更多的字符级向量,增加数据集中字符的匹配度,减少遇到词库外字符的情况,提升模型的识别效果。总体来看cEHRNER上PMV-LSTM模型比Lattice LSTM基线模型提升幅度巨大,精确率、召回率、F1值分别提升了32.49%、39.54%、37.34%。主要有两个原因,首先在此数据集中,“手术”、“疾病和诊断”两类实体相对于其它实体而言长度较长,平均长度分别为13个字符和7个字符,而在用Word2Vec训练词向量时会有窗口大小限制,没有学习到窗口外的上下文信息,所以会影响“手术”、“疾病和诊断”这两类实体的识别准确性,进而影响整体识别效果;其次Lattice LSTM基线模型中的词向量在此数据集上没有得到很好的匹配,导致出现了较多“未登录词”的情况,所以识别效果较差,而用医学文本训练出来的词向量可以达到更高的匹配度,缓解OOV(out of vocabulary)的问题。cMedQANER上PMV-LSTM模型相较于Lattice LSTM基线模型识别效果也有所提升,精确率、召回率、F1值分别提升了2.33%、3.36%、2.87%。在此数据集上达到的F1值为85.32%,相较于CCKS2017数据集的F1值低,这表明实体类别的增加对实体识别也会有所影响,但PMV-LSTM模型效果能够在Lattice LSTM基线模型基础上有所提升,说明特定领域的词向量和大量的字向量的引入能够有效解决实体类别多的问题。由表5可知,与其它经典研究相比,在CCKS2017上BiLSTM-CRF模型取得了87.97%的F1值,与BiLSTM模型相比提升2.34%,说明CRF可以在BiLSTM的基础上进一步学习到相邻实体的信息,而LSTM-CRF模型取得了86.31%的F1值,均低于PMV-LSTM模型识别效果。在cEHRNER和cMedQANER上BiLSTM-CRF模型分别取得了70.00%和80.63%的F1值,均高于BiLSTM模型和LSTM模型效果,而PMV-LSTM模型在两个数据集上的F1值相较于BiLSTM-CRF模型分别提升了9.01%和4.69%,表明PMV-LSTM模型在使用了词汇级特征和字符级特征之后,比单纯使用词级特征效果要好。
综上所述,本文提出的PMV-LSTM方法将字符级命名实体识别方法进行改进,通过Lattice结构巧妙地将词汇信息动态引入字符级命名实体识别方法中,在文本表示时既考虑字符信息,也考虑到词汇信息,打破了纯字符级命名实体识别和纯词汇级命名实体识别方法不能很好地编码医学文本语义信息的缺陷。另外,通过对医学语料进行专门训练,得到面向医学领域的词向量,并且使用了“字符分割”方法来增加字符级别向量的数量,使得训练出来的向量能够更好地匹配医学领域文本,较好地适用医学领域命名实体识别任务,通过实验验证了本文模型的有效性。
4 结束语
本文针对目前中文医学领域命名实体识别大多只基于字符级别或者只基于词汇级别进行实体识别的缺陷,提出PMV-LSTM模型,重新训练医学领域词向量,将医学文本中的词汇信息通过Lattice结构引入基于字符级别的模型中,自动选择适合当前上下文语义的词汇信息,并且利用“字符分割”的方法得到更多的字符级别向量,提升医学文本的匹配度。通过实验验证,本文提出的方法在3个医学领域数据集上均取得了不错的效果,并且相较于Lattice LSTM模型效果有所提升,能够有效解决字符级别模型和词汇级别模型的语义信息缺失问题,更好提取上下文信息。由于医学文本命名实体识别非常依赖上下文的信息,本文下一步的研究将会探索使用更先进的预训练语言模型进行命名实体识别任务。

