结直肠癌的关键癌症驱动基因筛选及其生物学功能分析
王锦淼,王颖,周博昊,王雷,穆伟斌
1 齐齐哈尔医学院医学技术学院,黑龙江 齐齐哈尔 161003;2 常熟理工学院计算机科学与工程学院
癌症已经成为全球主要死亡原因之一[1-2]。癌症是一种由体细胞突变和克隆选择导致的细胞恶性增殖的基因疾病,可直接导致癌症发生的基突变即为“驱动突变”[3],而不会造成癌细胞增殖的无直接影响基突变则为“乘客突变”,“乘客突变”对癌症的驱动作用很小[4],因此,识别出对癌症具有重要影响的“驱动基因”是目前癌症发生机制的研究重点[5-6],包含驱动突变的基因则为“癌症驱动基因(Cancer Driver Genes,CDGs)”。CDGs可为癌症预防、诊断和精准治疗提供关键信息[7]。结直肠癌(Colorectal carcinoma,CRC)是全球最常见的恶性肿瘤之一,发病率逐年上升[8]。CRC 的发病与多种因素相关,随着高通量测序技术的进展,多组学基因检测技术得到快速发展,但检测常产生成千上万CRC 相关基因,无法识别CRC的CDGs[9-11]。2021年6月起,我们采用多组学数据的组合优化方法,筛选CRC 的关键癌症驱动基因(Cancer Driver Genes,CDGs),并分析其生物学功能。现将结果报告如下。
1 材料与方法
1.1 CRC 基因数据和基因表达数据的下载及预处理 从癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库(https://cancergenome. nih. gov/)中搜索并下载CRC 基因[13],一共获得612 份样本转录组分析数据,其中CRC 患者568 例、正常人44 例,癌基因56 753 个。从国际肿瘤基因组协作组数据库
(the International Cancer Genome Consortium,ICGC)
(https://dcc. icgc. org/)中搜索并下载CRC 基因表达数据[14],一共获得548 份数据,癌基因20 888 个。对下载的数据库数据进行质量控制,对实验数据进行预处理,剔除异常值数据,去除信息不全、重复和可能存在错误的样本和突变频率过高或过低基因[15],提高数据可靠性、准确性。
1.2 CRC的关键癌症驱动基因筛选
1.2.1 构建CRC 基因突变矩阵与CRC 突变基因表达矩阵 从基因芯片中提取出样本原始突变基因和表达基因,运用“python”软件将数据整理为基因-样本形式的矩阵,将其分为癌症组和对照组,将CRC突变基因数据整理成突变矩阵(CRC 基因突变矩阵),将突变基因表达数据构建成基因表达矩阵(CRC突变基因表达)。
1.2.2 构建CRC 高维突变基因加权网络模型 从STRING 数据库(Search Tool for the Retrieval of Interacting Genes)[17]中获取CRC 基因的蛋白质相互作用(PPI)网络,运用python 软件将CRC 基因突变矩阵、CRC突变基因表达矩阵和PPI数据整合交集,以突变基因和PPI 网络的score 值分别作为节点和边,score值代表了基因之间这种相互作用(既包括蛋白质之间直接的物理的相互作用,也包括蛋白质间连接功能的相关性),节点属性为突变因子分数,两者结合建立高维突变基因加权网络,共包含14 388个基因。
1.2.3 CRC 癌症驱动基因筛选 根据高维突变基因加权网络模型的结构特征,通过每个基因在网络中相邻基因的突变影响,计算最大基因影响分数,基因节点影响分数最大值即基因最大突变影响分数得分,根据基因最大突变影响分数得分最终得到CRC癌症驱动基因。
1.2.4 CRC 关键癌症驱动基因筛选 CGC(The Cancer Gene Census)[19]数据库的网址为:https://cancer. sanger. ac. uk/census,其收录基因是已被医学界和生物界所认定与癌症相关的驱动基因。从CGC 数据中提取已经证实的结直肠癌症基因[20-21]与“1.2.3”得到CRC 癌症驱动基因比较,得到CRC 关键癌症驱动基因。
1.3 CRC 关键癌症驱动基因的生物学功能分析 采用STRING(https://string-db. org)数据库构建CRC 关键癌症驱动基因的蛋白质相互作用网络(PPI),互动分数设置为中等置信度0.4 分,该图由节点和边组成,节点代表蛋白,边代表关系,不同的颜色代表不同的数据来源。利用STRING在线分析工具生物注释将排名前100的显著差异基因进行基因本体(gene ontology,GO)分析和京都基因和基因组数据库(Kyoto Encyclopedia of Genes and Genomes,KEGG)信号通路富集分析。GO 功能富集分析是对基因进行注释和生物学功能分析的重要工具,Go 功能主要分成三大类:生物学过程(BP)、分子功能(MF)和CC细胞组成(CC),KEGG 信号通路富集分析可从大规模分子数据集中了解基因的富集通路。
2 结果
2.1 CRC 关键癌症驱动基因 最终筛选出22 个CRC关键癌症驱动基因,其中排名前20分别为ATM、TTN、PCDHGB3、LRP1B、PCDHA6、PIK3CA、SYNE1、PCDHGB2、KMT2C、BRAF、BMPR1A、PCDHGA8、PCDHGA5、FAT4、PCDHA8、APC、PCDHGA7、PCDHA10、PCDHA9 及FBXW7。基因最大突变影响分数得分分别为37 146.55、37 146.55、34 319.47、33 546.18、33 546.18、32 235.49、32 008.97、31 207.03、30 492.44、30 362.73、30 340.14、29 289.54、29 289.54、28 121.02、26 733.33、23 042.20、21 811.26、20 764.04、20 764.04、20 394.35。CRC 关键癌症驱动基因的相互作用图见图1。
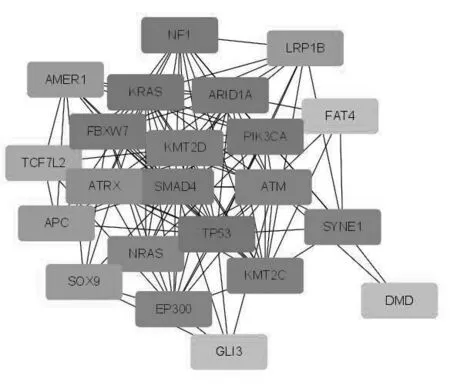
图1 CRC关键癌症驱动基因的相互作用图
2.2 CRC 关键癌症驱动基因的生物学功能 CRC关键癌症驱动基因的PPI 网络包含100 个节点,241条边,平均节点度为4.82,局部聚类系数为0.474,PPI 富集P值<1.0e-16。GO 功能富集结果显示,CRC 关键癌症驱动基因的分子功能主要集中在钙离子结合、阳离子结合、金属离子结合、离子结合、捆绑、β-连环蛋白结合、组蛋白甲基转移酶活性(h3-k4特异性)等;生物过程主要集中在通过质膜黏附分子的同源性细胞黏附、通过质膜黏附分子的细胞-细胞黏附、细胞黏附、细胞间黏附、神经系统发育、解剖结构发展、心脏发育、系统开发、多细胞生物发育、解剖结构形态发生等;细胞组成主要集中在质膜、质膜的组成部分、膜、肌原纤维、肌膜、肌节、膜的组成部分、质膜有界细胞投射、肌原纤维附着点、超分子纤维等。
KEEG 信号通路富集分析结果发现,CRC 关键癌症驱动基因的信号通路主要集中在大肠癌、子宫内膜癌、肝细胞癌、慢性粒细胞白血病、FoxO 信号通路、调节干细胞多能性的信号通路、胃癌、前列腺癌、乙型肝炎、癌症中的微小RNA。
3 讨论
癌症的发生和发展与基因组学、转录组学、蛋白质组学、表观组学及代谢组学等多组学数据息息相关[12]。以往通常是在单个的大样本数据中找到一些突变率显著很高的基因作为候选基因,这样的筛选造成癌症通路中存在基因之间强异质性的问题,所以如果单纯的对其中一种组学数据来进行数据挖掘和生物研究会存在明显的不足与缺陷,那么通过整合多组学数据并进行综合分析对癌症得到了更深层次和更全面的探索,利用生物信息学技术鉴定出关键基因及其相关通路,从病理发生的分子机制角度对CRC 进行理解,找到潜在可深入研究的用于诊断生物标志物以及治疗CRC的分子八项标志物。
本研究采用多组学数据的组合优化方法,整合体细胞突变数据、基因表达数据以及蛋白质相互作用网络三种组学数据。首先基于多统计学方法的CDGs生物特征提取,计算基因突变因子和皮尔逊相关系数,分别构建出突变矩阵和基因表达矩阵。然后以基因的突变频率和基因表达水平相关性为节点和边,建立高维突变基因加权网络模型,基于该网络模型利用重力学模型计算基因与邻居节点的突变影响分数,根据节点的影响分数最大值进而得出基因的突变影响因子大小,并在兼顾基因网络结构的条件下,以突变影响因子大小为根据运用一种综合的基因打分方法,最终得出驱动基因的预测集。
本研究中构建的基因相互作用网络信息更加全面,对于以往单一组学数据研究的缺陷进行了弥补,在多个体细胞突变数据集上进行评估,优先选择潜在的癌症驱动基因,对识别出的驱动基因进行CGC富集对比分析并能很好富集到CGC 列表中,利用本方法识别出的CRC排名靠前出现在CGC基因列表中的 前10 个 包 括:ATM、TTN、PCDHGB3、LRP1B、PCDHA6、PIK3CA、SYNE1、PCDHGB2、KMT2C 和BRAF,在CRC 组织中均明显表达增高,说明了这10个基因均与癌细胞的发生发展密切相关,这些关键基因可以作为CRC诊断和治疗的潜在靶标,也使我们更加全面的了解CRC的发病机制和发展机理,对于CRC的预防和早期诊断、治疗具有重要的临床意义。
对CRC 的关键癌症驱动基因进行GO 本体论分析和KEGG 通路生物功能分析,结果发现,CRC关键癌症驱动基因的分子功能主要集中在钙离子结合、阳离子结合、金属离子结合、离子结合、捆绑、β-连环蛋白结合、组蛋白甲基转移酶活性(h3-k4 特异性)等;生物过程主要集中在通过质膜黏附分子的同源性细胞黏附、通过质膜黏附分子的细胞-细胞黏附、细胞黏附、细胞间黏附、神经系统发育、解剖结构发展、心脏发育、系统开发、多细胞生物发育、解剖结构形态发生等;细胞组成主要集中在质膜、质膜的组成部分、膜、肌原纤维、肌膜、肌节、膜的组成部分、质膜有界细胞投射、肌原纤维附着点、超分子纤维等。驱动基因都是与质膜、肌膜和离子结合炎症等生命正常运行时有密切关联的,说明这些驱动基因具有重要的生物学功能。对KEGG 信号通路与关键基因关联的分析,本研究中CRC 关键癌症驱动基因的信号通路主要集中在大肠癌、子宫内膜癌、肝细胞癌、慢性粒细胞白血病、FoxO信号通路、调节干细胞多能性的信号通路、胃癌、前列腺癌、乙型肝炎、癌症中的微小RNA,说明CRC 关键基因的基因调控通路可能导致癌症的发生发展,同时同一驱动基因在不同肿瘤之间产生致癌作用,说明癌症的发病存在共通之处。
综上所述,成功筛选出22个CRC关键癌症驱动基因,如ATM、TTN、PCDHGB3等。CRC 关键癌症驱动基因的分子功能主要集中在钙离子结合、阳离子结合、金属离子结合等;生物过程主要集中在通过质膜黏附分子的同源性细胞黏附、通过质膜黏附分子的细胞-细胞黏附、细胞黏附等;细胞组成主要集中在质膜、质膜的组成部分、膜等;信号通路主要集中在大肠癌、FoxO 信号通路、调节干细胞多能性的信号通路等。深入了解CRC 关键癌症驱动基因有助于研究CRC 发生发展机制,同时可为CRC 提供新的治疗靶点。同时,多组学数据分析方法能够高精度预测疾病的驱动基因,为其他癌症的诊疗研究提供了一种新方法。

