番茄DIR基因家族鉴定及其对非生物胁迫响应的分析
陈凤琼,陈秋森,林佳昕,王雅亭,刘汉林,梁冰若诗,邓艺茹,任春元,张玉先,杨凤军,于高波,魏金鹏,王孟雪
番茄DIR基因家族鉴定及其对非生物胁迫响应的分析
陈凤琼,陈秋森,林佳昕,王雅亭,刘汉林,梁冰若诗,邓艺茹,任春元,张玉先,杨凤军,于高波,魏金鹏,王孟雪
黑龙江八一农垦大学园艺园林学院,黑龙江大庆 163319
【目的】鉴定番茄DIR基因家族所有成员,并对其基因结构、编码蛋白的理化性质、系统进化、染色体定位、共线性、启动子元件、表达模式、互作转录因子及内源竞争RNA预测等进行了解析,为探究DIR在番茄生长发育和逆境胁迫中的作用提供参考。【方法】采用生物信息学方法,基于全基因组数据对番茄DIR基因家族成员进行鉴定,运用Phytozome、MEME、PlantCARE、opsRNATarget和plantcircnet等在线网站获取染色体位置、保守基序、顺式作用元件、存在互作的转录因子、miRNA和circRNA等信息。利用Mapteace、TBtools、Cytoscape、OmicShare等作图软件及工具绘制染色体定位图、进化树、DIR与转录因子关系图、ceRNA网络等。采用NCBI的基因数据库结合转录组测序及qRT-PCR试验研究DIR基因家族在逆境下的表达情况。【结果】共鉴定到番茄中27个DIR基因,将其命名为—,分别位于12条染色体上,且大部分基因位于染色体末端,其基因结构、基序和结构域相对保守,其中22个具有一个外显子的经典结构。番茄DIR基因家族同拟南芥的共线性关系远高于水稻和大豆。基于系统进化关系将27个番茄DIR成员分为3个不同的亚家族。转录组数据表明大部分DIR基因在番茄根部具有较高的表达量。此外,DIR基因的启动子区域含有多个与干旱、低温等非生物胁迫,以及MeJA、ABA、SA等激素诱导相关的顺式作用元件,且预测到与激素、生长发育、非生物胁迫相关的ERF、E2F/DP、MYB等互作转录因子。结合转录组数据分析,分别有5、10、10和13个在农药、干旱、盐和冷胁迫后显著上调表达,其中,受到以上4种胁迫的诱导表达,而和特异性响应冷胁迫的诱导,特异性响应盐胁迫。番茄DIR的ceRNA调控表明,miR-156与靶基因可能共同作用调控番茄的逆境胁迫。【结论】共鉴定出番茄DIR家族基因27个,不均匀地分布在12条染色体上,在根部有较高的表达量。、和等具有MeJA、ABA、SA等激素响应元件,其中,仅具有MeJA元件,仅具有SA响应元件。另外,、等参与干旱、盐、低温等多种逆境胁迫,其中在不同胁迫处理下均可被激活。此外,DIR基因和转录因子、非编码RNA相互作用,共同参与调控番茄植株的逆境胁迫。
DIR蛋白;番茄;基因家族;生物信息学
0 引言
【研究意义】Dirigent(DIR)基因家族几乎存在于所有维管植物中,主要参与细胞壁木质素和木脂素的生物合成,对植物抗逆防御体系的形成起着重要作用。然而,迄今为止,关于番茄DIR基因家族的相关研究鲜有报道。本研究通过生物信息学方法鉴定番茄的DIR(SlDIR)基因家族,并进一步针对其在番茄应对逆境胁迫的响应与生长发育中的作用开展研究,为揭示番茄DIR的功能研究奠定基础。【前人研究进展】DIR基因家族首次于1997年在美国金钟连翘中发现[1]。经典的DIR基因结构没有内含子,含有一个DIR保守结构域,占据了大部分蛋白质序列[2]。近年来,在多种植物中也发现了数量不等的DIR基因家族。目前已分别在拟南芥[3]、云杉[4]、水稻[5]、五味子[6]和毛竹[7]等作物中发现了26、31、54、34和43个DIR成员。PANIAGUA等[3]将DIR蛋白家族成员分成5个亚家族,分别是DIR-a、DIR-b、DIR-c、DIR-d和DIR-e亚家族。随着DIR蛋白家族成员的增多,DIR-b和DIR-d亚家族聚合在了一起,并衍生出DIR-f和DIR-g亚家族。已有研究表明,DIR对植物响应生物胁迫及非生物胁迫起着重要作用,如棉花通过提高棉花的木质化程度来抵抗黄萎病菌的侵染[8],三七能够响应外源MeJA的诱导,并在茄腐镰刀菌侵染后表达量升高[9]。而辣椒的沉默会导致植株对病原体和盐胁迫的耐受性降低[10]。【本研究切入点】目前关于番茄的DIR(SlDIR)基因家族的系统鉴定与分析尚未见报道。【拟解决的关键问题】本研究拟通过生物信息学方法鉴定出番茄DIR基因家族所有成员,并对其基因结构、编码蛋白理化性质、系统进化关系、染色体定位、共线性分析、启动子元件、表达模式、转录因子及内源竞争RNA预测进行解析,结合已有逆境胁迫转录组数据与其在农药等逆境胁迫中的响应进行测定分析。
1 材料与方法
1.1 番茄DIR 基因家族鉴定
基于Pfam数据库的隐马尔可夫模型,搜索全长转录组中包含Dirigent结构域(Pfam 03018)的基因,并按照阈值条件为E-value<1×10-20将可信度较高的候选基因筛选出来。利用这些基因进行番茄DIR基因家族的隐马尔可夫模型的构建。使用新构建的模型重新对全长转录组进行搜索,并按照阈值条件为E-value<1×10-3对结果进行过滤。同时,搜集NCBI中植物DIR蛋白Ref-seq模块内的序列,通过blast将搜集到的蛋白序列同全长转录组序列进行比对,并按照阈值条件为E-value<1×10-10将可信度较高的基因筛选出来[11]。最后,取两种方法鉴定结果的交集,作为最终的番茄DIR候选基因,并通过NCBI的Batch Web CD-Search Tool(https://www.ncbi.nlm. nih.gov/Structure/bwrpsb/bwrpsb.cgi)验证候选基因的保守结构域。
1.2 番茄染色体定位和共线性分析
从Phytozome数据库中得到番茄DIR基因家族成员在染色体上的位置利用Mapteace软件绘制基因染色体定位图。从前人报道中得到拟南芥[3]、大豆[12]、水稻[5]的DIR基因登录号,从Gramene(http://www. gramene.org/)获取基因组长度信息、从phytozome得到DIR蛋白的位置信息,从PGDD(http://chibba.pgml. uga.edu/duplication/)中检索番茄、拟南芥、水稻和大豆之间的关系对,最后利用Tbtools进行DIR共线性分析。
1.3 番茄DIR基因家族蛋白理化性质分析及亚细胞定位
利用Phytozome查找番茄DIR基因的蛋白序列,结合利用ExPASy在线网站的ProtParam工具(https:// web.expasy.org/protparam/)对番茄DIR蛋白序列进行分析,获取氨基酸长度、相对分子质量、理论等电点、分子式、亲水性、脂肪系数、不稳定系数。利用在线网站Wolf PSORT(https://wolfpsort.hgc.jp/)对番茄DIR蛋白进行亚细胞定位预测[13]。
1.4 番茄DIR 基因家族蛋白保守基序分析
利用MEME在线网站(https://meme-suite.org/ meme/)鉴定番茄DIR基因家族蛋白序列的保守基序,motif数量选择13;motif长度范围选择6—50个氨基酸[14]。利用TBtools对保守基序进行可视化[15]。
1.5 番茄DIR基因家族系统进化树构建
拟南芥、番茄、棉花和辣椒的DIR蛋白序列通过MAFFT v7.475进行比对[16],并利用MEGAX 软件对比对后的序列构建邻接(Neighbor Joining)树[17],利用iTOL(https://itol.embl.de/)对进化树进行绘制[18]。
1.6 番茄DIR基因家族在不同组织中的表达分析
从番茄信息资源数据库(https://solgenomics.net/)得到栽培番茄(cv. Heinz)根、叶、花芽、完全开放的花和1 cm果实的转录组数据,使用TBtools绘制番茄不同组织部位DIR基因表达的热图。
1.7 番茄DIR基因家族的启动子顺式元件和胁迫表达模式分析
从Phytozome下载番茄DIR基因上游1 500 bp序列,利用在线网站PlantCARE(http://bioinformatics. psb.ugent.be/webtools/plantcare/html/search_CARE.html)[19]进行顺式作用元件分析。从NCBI的GEO DataSets下载冷、盐和干旱胁迫下的番茄转录组数据,并利用TBtools绘制热图。
1.8 番茄DIR相关转录因子预测
使用PlantRegMap(http://plantregmap.gao-lab.org/ index-chinese.php)在线网站预测与番茄DIR基因相关的转录因子,并利用Cytoscape软件构建番茄DIR基因与相关转录因子的关系图。
1.9 番茄DIR基因竞争性内源性RNA(ceRNA)调控网络分析
使用在线网站http://plantgrn.noble.org/psRNATarget/和http://bis.zju.edu.cn/plantcircnet/ index.php预测和识别与番茄DIR相关的miRNA和circRNA,通过OmicShare工具可视化ceRNA网络(http://www.omicshare. com/tools)。
1.10 转录组测序
本试验在黑龙江八一农垦大学生物技术中心人工智能气候室中进行,番茄品种为‘浙杂205’,由浙江省农科院提供。番茄常规育苗,待番茄幼苗长至6—7片叶时进行农药处理。试验以清水为对照(CK),以11.2 mmol∙L-1百菌清为农药处理,每个处理 3 次重复,将番茄叶片正反面均匀喷施相应的处理试剂,并于处理24 h后采集植株形态学上端第2片完全展开的功能叶,用于RNA提取,液氮速冻保存。样品送至百迈客生物科技有限公司进行测序。样品总RNA提取参考Axygen RNA 提取试剂盒说明书。构建文库后进行检测,库检合格后,用HiSeq X-ten 进行高通量测序。
1.11 番茄DIR基因的qRT-PCR表达分析
百菌清处理同1.10以300 mmol∙L-1NaCl溶液浇灌土壤为盐胁迫处理,以20%的PEG溶液浇灌土壤为干旱胁迫处理,以清水浇灌为对照(CK)。以4℃为低温处理,以常温25℃为对照(CK),每个处理3次重复,并于处理24 h后采集植株形态学上端第2片完全展开的功能叶,用于RNA提取。样品总 RNA 提取参考Axygen RNA 提取试剂盒说明书,并使用Prime ScriptRT Reagent(TaKaRa Japan)反转录试剂盒进行cDNA合成。使用SYBR(Roche.Switerland)进行qRT-PCR反应,使用软件Primer 5.0设计引物(附表1)。
1.12 数据处理
基因的相对表达量采用2-Δ∆Ct法[20]计算;数据处理使用Excel 2019软件;柱状图的绘制使用Origin 2017软件。
2 结果
2.1 番茄DIR家族成员的鉴定、染色体定位和理化性质分析
利用拟南芥的26个DIR蛋白的氨基酸序列为查询序列,在Phytozome进行BLAST同源比对,并对得到的DIR蛋白进行保守结构域鉴定,得到27个DIR蛋白。根据基因登录号将其命名为—(附表2),番茄DIR基因不均匀地分布在12条染色体上,且大部分位于染色体末端,其中第10条染色体上分布最多,含有7个DIR基因(图1)。对番茄DIR蛋白的理化性质进行预测(附表2),分析发现SlDIR蛋白质的大小介于60()—499()个aa之间,平均长度约为200 aa。SlDIR蛋白的相对分子质量介于6 433.74 kD()—41 413.18 kD(),等电点介于4.47()—9.85(),其中有12个酸性蛋白和15个碱性蛋白,这突出了它们结构的多样性,9个定位于细胞外区域(附表2)。

图1 番茄DIR基因染色体定位
2.2 番茄DIR 基因家族系统进化树分析
利用拟南芥、番茄、CM334品系辣椒和陆地棉的DIR蛋白序列构建NJ树,参考拟南芥DIR的分类,根据同源性分析,可将番茄DIR家族成员分成3个亚家族,分别为DIR-a、DIR-c和DIR-e,其中DIR-c亚家族的数量最多,为15条,DIR-e亚家族数量次之,有8个基因,DIR-a亚家族成员最少,仅包含和。另外,茄科作物辣椒的亚家族与番茄类似,且DIR-c、DIR-e和DIR-a亚家族基因数目分别为8、6和6条。拟南芥的DIR成员分为5个亚家族,分别为DIR-a、DIR-b、DIR-c、DIR-d和DIR-e,分别包含6、2、7、5和6条DIR基因。陆地棉的DIR亚家族分类与拟南芥类似,均有5个亚家族,但不同亚家族成员数量明显多于番茄、辣椒和拟南芥。综上,茄科作物番茄与辣椒中DIR-a和DIR-e亚家族中DIR数目与拟南芥中相似,说明这两个亚家族的DIR基因并没有在基因组中大范围变化。然而,番茄DIR-c亚家族数目约为拟南芥中的2倍,且与拟南芥相比,番茄缺失DIR-b和DIR-d亚家族成员(图2)。
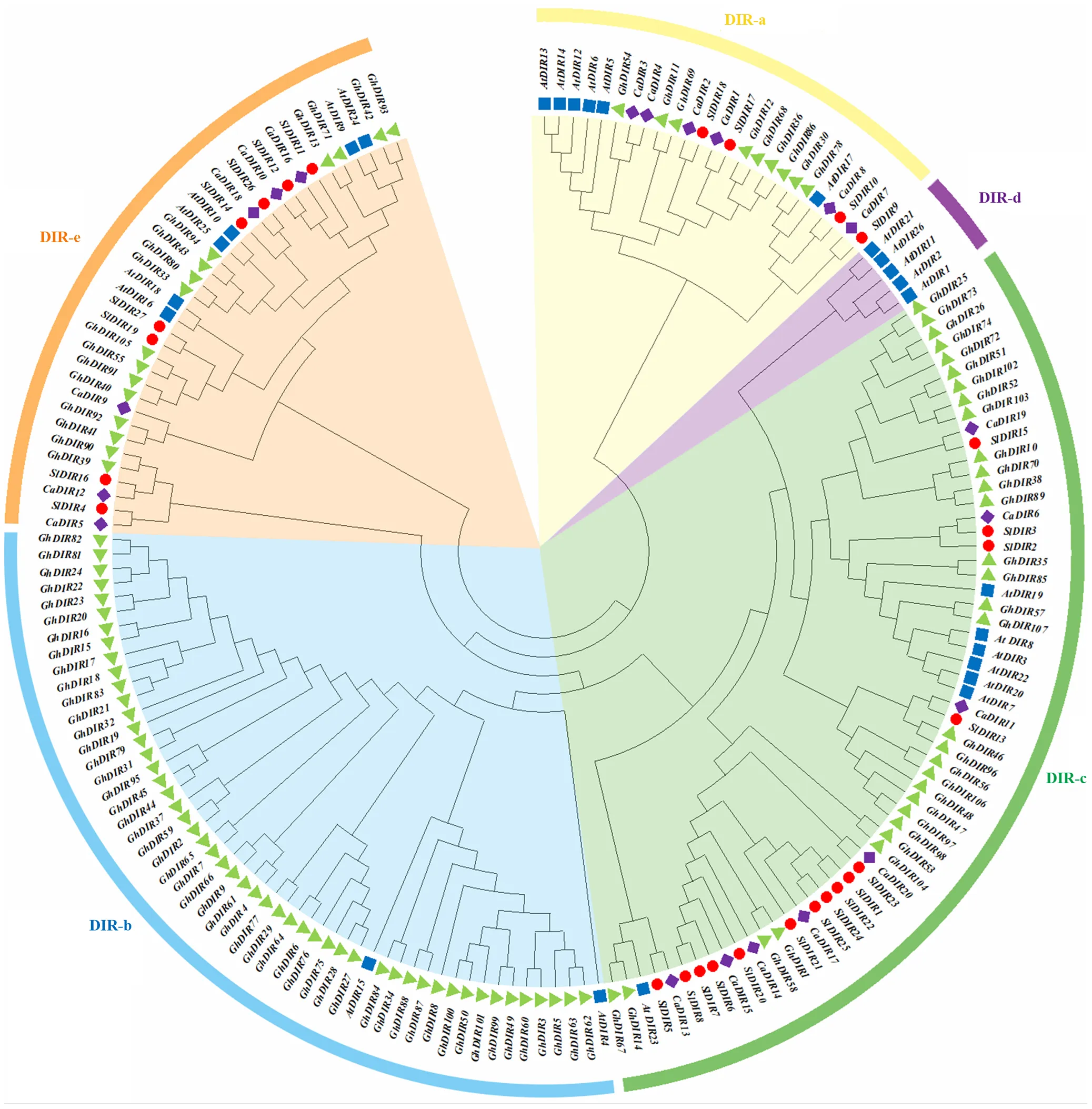
图2 拟南芥、番茄、辣椒和棉花DIR家族的系统发育树
2.3 番茄DIR基因家族蛋白保守基序分析
大多数包含一个外显子的经典DIR基因结构,但是、和包含两个外显子和一个内含子(图3)。此外,27个番茄DIR蛋白搜索到13个motif,其中3个亚家族共享motif 1、motif 2和motif 3,说明motif 1、motif 2和motif 3在番茄DIR蛋白质序列中的分布较为广泛,并具有较强的保守性。同一亚家族中的大多数SlDIR成员也共享一些保守的基序,说明这些蛋白质的功能保守。但也存在部分基序特异性地存在于某些亚家族中:motif 5、motif 6、motif 8和motif 11特异性存在于DIR-c亚家族中,而motif 9、motif 12和motif 13仅出现在DIR-e亚家族,motif 10仅在DIR-a亚家族中被发现,这表明在不同的亚家族中存在功能多样性(图3)。
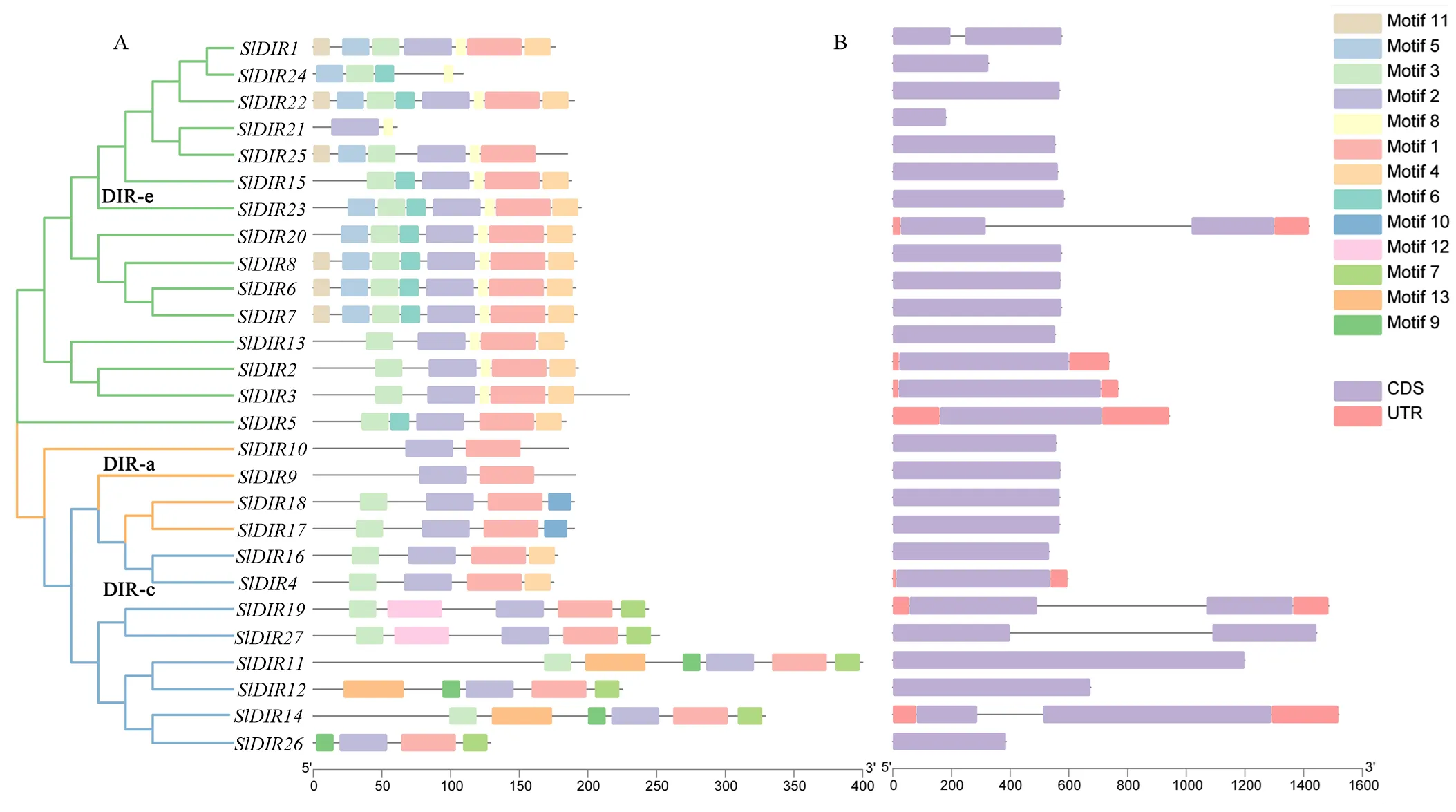
图3 番茄DIR基因家族系统进化树、蛋白基序和基因结构
2.4 番茄与不同作物的共线性分析
根据番茄与大豆、水稻、拟南芥DIR基因的共线性分析结果,在番茄的种内共线关系中发现和均发生片段复制,且片段复制使基因数目增多,尤其是DIR-e和DIR-c亚家族,而DIR-a亚家族成员未发生片段复制,基因数目最少。在对番茄、大豆、水稻和拟南芥4个物种的直系同源基因进行比对分析中发现,和在拟南芥染色体1、2、3和4上具有同源基因,同源基因对数量为12对,多于番茄与水稻(3对)、大豆与拟南芥(2对)、拟南芥与水稻(1对)和大豆与水稻(1对)的同源基因对。说明番茄和拟南芥的DIR基因家族具有更近的同源进化关系,且这些基因可能具有相似的功能,然而DIR基因在番茄和大豆中不存在同源基因对(图4)。
2.5 番茄DIR基因家族在不同组织部位的表达模式分析
为了进一步阐明在番茄生长发育中的潜在作用,在番茄信息资源数据库(https://solgenomics.net/)得到栽培番茄(cvHeinz)5个组织部位(根、叶、花芽、完全开放的花和1 cm的果实)的转录组数据,得到27条番茄DIR基因的表达情况。如图5所示,DIR-c亚家族在5个组织部位均有表达,其中在根及花芽中表达较为显著;DIR-a亚家族在组织部位中的表达与DIR-c亚家族类似;然而DIR-b亚家族仅在根及花芽有表达,其中在根部的表达最为显著。总体而言,番茄中共有17个在根中显著表达,且高于其他组织部位,而在叶和果实中的表达量相对很低,表明该基因的表达可能对番茄根部生长及逆境响应较为重要。
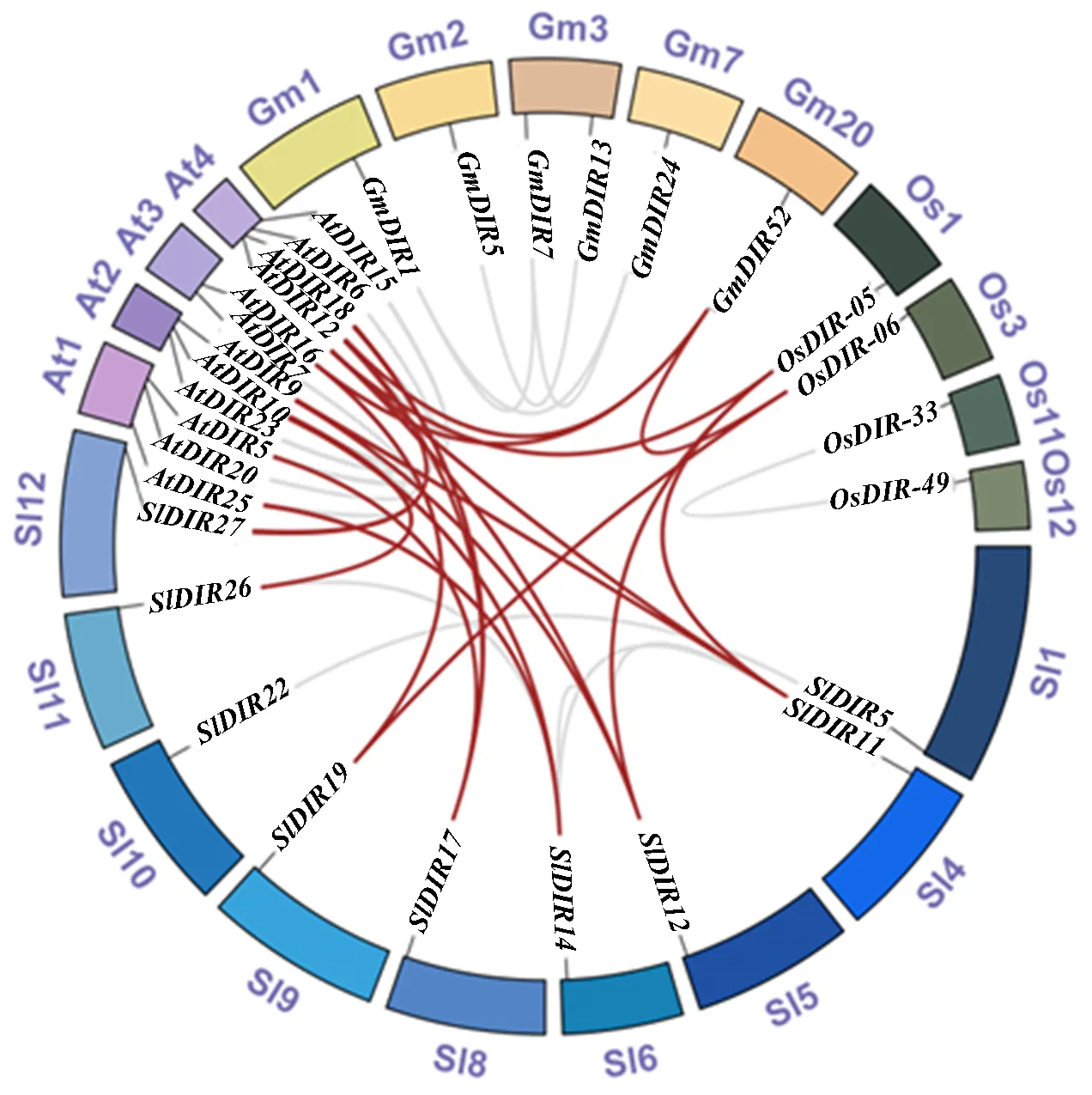
红色弧线表示物种间存在同源性,灰色弧线表示物种内存在同源性

图5 番茄DIR基因家族在不同组织部位中的表达分析
2.6 番茄DIR基因的启动子顺式元件分析和不同胁迫条件下的表达模式分析
启动子中含有大量激素应答元件、胁迫响应元件和光反应元件等(图6)。对所有顺式元件及其位点数进行分类和统计分析发现,是含有启动子顺式元件最多的成员,其次是,而仅含有2个顺式作用元件,说明不同的对激素处理或逆境胁迫等的应答存在较大的区别。在启动子序列预测到了多个激素响应元件,包括生长素、赤霉素、ABA、MeJA和SA。其中15个启动子序列含有MeJA作用元件,13个启动子序列含有ABA作用元件,10个启动子序列含有SA作用元件,含有生长素和赤霉素响应元件的均有5个。因此,多种激素可参与调控DIR的表达,不同成员之间存在着一定的协同或拮抗作用。与此同时,在启动子序列中还预测到了与厌氧诱导相关的响应元件ARE、与低温胁迫相关的响应元件LTR、与干旱相关的应答元件MBS等响应逆境胁迫的元件。此外,还发现与光反应相关的应答元件MRE和Circadian;与类黄酮合成相关的应答元件MBSI,与叶片衰老有关的应答元件CCAAT-box等。因此,预测表达在番茄上述调控过程中起到一定的作用。

图6 番茄DIR基因上游1500 bp启动子顺式元件分析
为了进一步探究对逆境胁迫的响应,通过转录组测序试验及数据库中的胁迫相关转录组数据分析,发现了27个SlDIR家族成员在4种非生物胁迫条件(农药百菌清、盐、干旱和冷)下的相对转录丰度(图7)。结果发现在百菌清、盐、干旱和冷胁迫后,在DIR-e亚家族中,分别有4、4、4和8个相对表达量显著上调;DIR-c亚家族中分别有0、4、6和4个被诱导表达;DIR-a亚家族分别有1、2、1和1个受到胁迫响应。其中,受到这4种胁迫的诱导表达,和在盐、干旱和冷胁迫后显著上调,和受到干旱和冷胁迫的诱导表达,和显著响应盐和冷胁迫。另外,也在百菌清和冷胁迫后显著上调,而、和特异性响应冷胁迫的诱导。此外,特异性响应盐胁迫。这些结果表明在番茄抵御各种非生物胁迫的过程中可能具有重要作用,且不同基因对胁迫存在着特异性的响应。

图7 番茄DIR基因在百菌清、盐、干旱和冷胁迫下的转录丰度(A)及番茄DIR基因在干旱胁迫(B)、盐胁迫(C)、冷胁迫(D)和百菌清胁迫(E)下的qRT-PCR验证
为了证实以上结果,从番茄转录组数据中选择基因表达量差异较为显著的8个DIR基因(和),通过荧光定量qRT-PCR检测其对干旱胁迫、盐胁迫、冷胁迫和百菌清胁迫24 h后的表达响应,并对干旱胁迫6 h、盐胁迫8 h、冷胁迫12 h和百菌清胁迫24 h的转录组数据和qRT-PCR数据进行log2处理分析。结果发现在百菌清(CHT)、盐、干旱和低温胁迫处理后,分别有5、4、3和6个表达量上调,分别有3、4、5和2个表达量下调。这与转录组测序结果基本一致,进一步验证了参与了番茄抵御逆境胁迫的过程。
2.7 番茄DIR相关转录因子预测
预测到共有30个转录因子(TFs)家族与存在互作关系。在所有中,预测到与存在互作的转录因子最多,有11个,分别为E2F/DP(E2 factor)、AP2、生长素响应因子(ARF)、BBR-BPC、Dof(DNAbinding with one finger)、ERF、GATA、GRAS、MYB、NAC)和三胺酸环延伸(TALE)(图8)。另外,有11个预测到5个及以上的互作转录因子,主要与植物的生长发育和响应逆境胁迫有关,这说明DIR基因在番茄的生长发育和逆境胁迫中扮演着重要的角色。在所有TF中,乙烯响应元件结合因子(ERF)和MYB(v-myb avian myeloblastosis viral oncogene homolog)均与14个存在互作关系,其中,与ERF有关的包括和;与MYB有关的是和,其次是GATA与8个存在互作关系。以上结果说明可能受这些转录因子的调控,与其互作参与植株的生长发育调控与逆境响应。

图中红色表示基因,绿色表示转录因子 Red indicates gene and green indicates transcription factor
2.8 番茄DIR基因的ceRNA网络分析
miRNA可与非编码RNA相互作用形成ceRNA网络,参与植物生理活动的调控。因此,预测了与有靶向关系的miRNA,以及与miRNA相关的circRNA。27个中有11个预测到了有靶向关系的12个miRNA和20个circRNA,并构建了50条ceRNA调控途径(图9)。大多数可作为1—2个miRNA的靶点,而可作为11个miRNA的靶点。此外,slsly-miR1917和y-miR5302b-3p预测的14个circRNA可以与靶基因(和)形成至少15个ceRNA网络。综上表明,可能受多个miRNA的调控,形成具有circRNA-miRNA模式的ceRNA网络,参与多种生理功能。

图9 番茄DIR基因的ceRNA网络分析
3 讨论
3.1 番茄DIR基因家族的鉴定
DIR基因在基因组进化过程中发生了广泛的变化,在不同作物中DIR数量不同,拟南芥、绿豆[21]、苜蓿[22]、丹参[23]和油菜[24]中分别含有26、45、34、33和29个成员,本研究在番茄中鉴定出27个DIR成员。其大多数亚族同样从其他作物中被鉴定出来[25],但一些亚族在进化过程中发生丢失,番茄仅包含6个DIR亚家族中的3个,包括DIR-a、DIR-c和DIR-e,而DIRb/d、DIR-g和DIR-f在番茄进化过程中丢失。生化试验表明,参与木质素合成的DIR蛋白都属于DIR-a亚家族[1],本研究中番茄、、和属于DIR-a亚家族,因此,推测这4个基因可能参与木质素合成。DIR基因的典型结构包含一个外显子[2],所有的DIR-a亚家族成员都含有典型的基因结构,表明DIR-a亚家族在3个亚家族中较为保守,但是DIR-e亚家族的、和DIR-c亚家族的、、包含两个外显子和一个内含子。另外,在所有的中都存在一些共有的基序,例如motif1、motif 2、motif 3和motif 4,表明具有较高的保守性。然而,一些基序仅存在于特定的亚家族中,其中motif 10仅在DIR-a亚家族中被发现,表明在不同的亚家族中表现出高度的功能多样性。另外,在番茄与大豆、水稻、拟南芥的同源关系比对中发现,除番茄与大豆外,与其他物种间均存在一定数量的同源关系对。拟南芥、番茄同属于双子叶植物,两者间DIR家族成员存在大量的共线性关系,而与单子叶植物水稻仅1个成员有共线性关系,这一结果与双子叶和单子叶植物间的进化关系一致。但同为双子叶植物的番茄和大豆DIR家族成员间不存在共线性,这表明DIR在不同植物中存在多样性。此外,本研究发现17个在根中显著表达,这与GUO等[26]和PANIAGUA等[3]在甘蔗和拟南芥的研究结果一致。许多DIR成员的功能主要涉及木脂素和木质素的形成,以及植物对非生物和生物胁迫的抗性。因此,推测番茄在抵御病原菌等生物及非生物逆境胁迫时,可能通过根部DIR基因合成更多的木脂素和木质素以增强其抵抗能力。
3.2 番茄DIR基因家族响应不同的逆境胁迫
本研究针对上游1 500 bp序列中的顺式作用元件分析发现,该家族中的大部分成员响应多种激素调控,包括MeJA、ABA、JA等参与植物胁迫的信号转导的激素,说明SlDIR家族在番茄生长发育和胁迫响应中发挥着重要作用。其中,有55.6%和48.1%的分别响应MeJA和ABA的调控。已有研究表明MeJA[27-28]和ABA[29-30]等激素还可以通过提高植物的抗氧化酶活性,清除自由基,增加渗透调节物质含量来减轻植物受到的胁迫损伤。因此,推测可能通过响应MeJA、ABA等激素应答,参与非生物胁迫的调控作用,从而增强植物对逆境胁迫的耐受性,但其具体机制有待深入研究。另有研究证实黄瓜[31]能够通过增加农药霜霉威胁迫下黄瓜的可溶性糖含量、提高POD酶活性,减缓霜霉威引起的活性氧对质膜的伤害。本研究的转录组测序结果与qRT-PCR数据分析也表明部分番茄受到农药百菌清胁迫的显著诱导。百菌清(CHT)作为一种广谱性、非系统性的杀菌剂,可用于保护蔬菜和作物免受真菌病原体的侵害,但其消散的速度极其缓慢,即使是低剂量的有毒成分也会对人体健康造成直接危害[32]。本研究还发现,启动子中存在与低温、干旱和热胁迫等非生物胁迫响应相关的顺式元件,转录组数据分析也表明分别有13、10、10和5个显著响应了冷、干旱、盐和百菌清胁迫,qRT-PCR的试验验证了这一结果。此外,GUO等[26]在甘蔗中已验证了参与H2O2、PEG和NaCl胁迫,THAMIL等[24]在油菜中也发现,受到低温胁迫后,表达量显著上调,提高酸溶性木质素含量,从而抵抗逆境胁迫。YADAV等[33]研究葫芦科作物甜瓜、西瓜和黄瓜的DIR基因对白粉病的抗性,发现DIR基因在抗病品种和非抗病品种中均有表达,其中随着接种时间的延长,表达量逐渐增加。这表明DIR基因家族在植物抵抗非生物逆境胁迫的过程中起着重要的作用。
由于植物基因的表达受到转录因子及非编码RNA等多种途径的复杂调控[34],其对植物生长发育及逆境胁迫的应答也可能是通过与转录因子互作、ceRNA网络调控等多种方式实现的。转录因子作为基因表达的驱动因素,在所有生物的基因表达调控中都发挥着核心作用。本研究发现与30个转录因子家族存在互作关系,其中,有14个DIR基因与ERF转录因子存在着复杂的关系网络。有研究发现菘蓝[35]中的可通过结合启动子区域的GCC-box,从而使表达水平显著上调,激活木脂素代谢,增强菘蓝对NaCl和H2O2的抵抗能力。另外,MARIAM等[36]研究表明,马铃薯感染茄花镰刀菌时,的过表达会激活葡聚糖酶(PR2)、几丁质酶(PR3)等编码基因的表达,从而使马铃薯对茄花镰刀菌产生抗性。因此,可能通过与这些转录因子的互作参与到逆境胁迫的调控过程。另外,本研究还构建了一个包括24个miRNA、21个circRNA和11个mRNA的ceRNA网络,并探索了一些与应激相关的非编码RNA,如sly-miR156a、sly-R156b和sly-miR156c。有研究表明,逆境条件下,miR156表达量上调,引起靶基因表达下调,负调控与花青素合成相关的作用因子DFR,使其表达增加,从而提高花青素的合成,抵抗逆境胁迫[37-38]。此外,在拟南芥中过表达miR156,转基因植株表现出较强的抗盐胁迫能力[39]。本研究发现作为sly-miR156a、sly-R156b和sly-miR156c的靶基因,可能与miRNA156存在互作关系,在逆境胁迫中扮演着重要的作用。
4 结论
从番茄中鉴定到27个,分为DIR-a、DIR-c和DIR-e三个亚家族,不均匀地分布在12条染色体上,并多数在根部有较高的表达量。22个具有1个外显子的经典结构,且3个亚家族共享motif 1、motif 2和motif 3。番茄DIR基因家族与拟南芥的共线性关系远高于水稻和大豆。、和等基因具有MeJA、ABA、SA等激素响应元件,仅具有MeJA响应元件,仅具有SA响应元件。另外,、参与干旱、盐、低温等多种逆境胁迫响应,尤其是在不同胁迫处理下均可被激活。此外,DIR基因和转录因子(MYB、ERF等)、非编码RNA(sly-miR156a、sly-miR156b等)相互作用,共同参与调控番茄植株的逆境胁迫。
[1] DAVIN L B, WANG H B, CROWELL A L, BEDGAR D L, MARTIN D M, SARKANEN S, LEWIS N G. Stereoselective bimolecular phenoxy radical coupling by an auxiliary (dirigent) protein without an active center. The Journal of Supercomputing, 1997, 275(5298): 362-366. doi: 10.1126/science.275.5298.362.
[2] CORBIN C, DROUET S, MARKULIN L, AUGUIN D, LAINÉ É, DAVIN L B, CORT J R, LEWIS N G, HANO C. A genome-wide analysis of the flax (L.) dirigent protein family: From gene identification and evolution to differential regulation. Plant Molecular Biology, 2018, 97(1/2): 73-101. doi: 10.1007/s11103-018- 0725-x.
[3] PANIAGUA C, BILKOVA A, JACKSON P, DABRAVOLSKI S, RIBER W, DIDI V, HOUSER J, GIGLI-BISCEGLIA N, WIMMEROVA M, BUDÍNSKÁ E, HAMANN T, HEJATKO J. Dirigent proteins in plants: Modulating cell wall metabolism during abiotic and biotic stress exposure. Journal of Experimental Botany, 2017, 68(13): 3287-3301. doi: 10.1093/jxb/erx141.
[4] RALPH S G, JANCSIK S, BOHLMANN J. Dirigent proteins in conifer defense II: Extended gene discovery, phylogeny, and constitutive and stress-induced gene expression in spruce (spp.). Phytochemistry, 2007, 68(14): 1975-1991. doi: 10.1016/j.phytochem. 2007.04.042.
[5] 穰中文, 周清明. 水稻dirigent基因家族生物信息学分析. 湖南农业大学学报(自然科学版), 2013(2): 111-120.
RANG Z W, ZHOU Q M. Bioinformatic analysis of the dirigent gene family in rice. Journal of Hunan Agricultural University (Natural Sciences Edition), 2013(2): 111-120. (in Chinese)
[6] 董雨青, 强亭燕, 刘久石, 李斌, 魏雪苹, 齐耀东, 刘海涛, 张本刚. 五味子DIR基因家族鉴定及特征分析. 中国中药杂志, 2021, 46(20): 5270-5277. doi: 10.19540/j.cnki.cjcmm.20210723.101.
DONG Y Q, QIANG T Y, LIU J S, LI B, WEI X P, QI Y D, LIU H T, ZHANG B G. Identification and characterization of DIR gene family in. China Journal of Chinese Materia Medica, 2021, 46(20): 5270-5277. doi: 10.19540/j.cnki.cjcmm.20210723.101. (in Chinese)
[7] 陈家璐, 张智俊, 刘笑雨, 朱丰晓. 毛竹Dirigent基因家族的全基因组鉴定与分析. 植物生理学报, 2019(9): 1406-1417.
CHEN J L, ZHANG Z J, LIU X Y, ZHU F X. Genome-wide identification and analysis of Dirigent gene family in moso bamboo (). Plant Physiology Journal, 2019(9): 1406-1417. (in Chinese)
[8] 郭宝生, 师恭曜, 王凯辉, 刘素恩, 赵存鹏, 王兆晓, 耿军义, 华金平. 黄萎病菌侵染下陆地棉Dirigent-like蛋白基因表达差异分析. 中国农业科学, 2014, 47(22): 4349-4359. doi: 10.3864/j.issn.0578- 1752.2014.22.001.
GUO B S, SHI G Y, WANG K H, LIU S E, ZHAO C P, WANG Z X, GENG J Y, HUA J P. Expression differences of dirigent-like protein genes in upland cotton responsed to infection by. Scientia Agricultura Sinica, 2014, 47(22): 4349-4359. doi: 10.3864/j.issn.0578-1752.2014.22.001. (in Chinese)
[9] 关瑞攀. Dirigent基因参与三七—茄腐镰刀菌互作的分子机理研究[D]. 昆明: 昆明理工大学, 2018.
GUAN R P. Molecular mechanism of Dirigent genes involved in the interaction of Panax notoginseng -[D]. Kunming: Kunming University of Technology, 2018. (in Chinese)
[10] KHAN A, LI R J, SUN J T, Ma F, ZHANG H X, JIN J H, ALI M, HAQ S U, WANG J E, GONG Z H. Genome-wide analysis of dirigent gene family in pepper (L.) and characterization of CaDIR7 in biotic and abiotic stresses. Scientific Reports, 2018, 8(1): 5500-5521.
[11] MCGINNIS S, MADDEN T L. BLAST: at the core of a powerful and diverse set of sequence analysis tools. Nucleic Acids Research, 2004, 32: W20-W25. doi: 10.1093/nar/gkh435.
[12] LI N H, ZHAO M, LIU T F, DONG L D, CHENG Q, WU J J, WANG L, CHEN X, ZHANG C Z, LU W C, XU P F, ZHANG S Z. A novel soybean dirigent genecontributes to promotion of lignan biosynthesis and enhances resistance to. Frontiers in Plant Science, 2017, 8: 1185.
[13] Horton P, PARK K J, OBAYASHI T, FUJITA N, HARADA H, ADAMS-COLLIER C J, NAKAI K. WoLF PSORT: Protein localization predictor. Nucleic Acids Research, 2007, 35: W585- W587.
[14] BAILEY T L, ELKAN C. Fitting a mixture model by expectation maximization to discover motifs in biopolymers. Proceedings. International Conference on Intelligent Systems for Molecular Biology, 1994, 2: 28-36.
[15] CHEN C, CHEN H, ZHANG Y, THOMAS H R, FRANK M H, HE Y, XIA R. TBtools: An integrative toolkit developed for interactive analyses of big biological data. Molecular Plant, 2020, 13(8): 1194-1202. doi: 10.1016/j.molp.2020.06.009.
[16] KATOH K, STANDLEY D M. MAFFT multiple sequence alignment software version 7: Improvements in performance and usability. Molecular Biology and Evolution, 2013, 30(4): 772-780. doi: 10.1093/molbev/mst010.
[17] KUMAR S, STECHER G, LI M, KNYAZ C, TAMURA K. MEGA X: Molecular evolutionary genetics analysis across computing platforms. Molecular Biology and Evolution, 2018, 35(6): 1547-1549. doi: 10.1093/molbev/msy096.
[18] LETUNIC I, BORK P. Interactive Tree Of Life (iTOL) v4: Recent updates and new developments. Nucleic Acids Research, 2019, 47(W1): W256-W259. doi: 10.1093/nar/gkz239.
[19] LESCOT M, DÉHAIS P, THIJS G, MARCHAL K, MOREAU Y, VAN DE PEER Y, ROUZÉ P, ROMBAUTS S. PlantCARE, a database of plant cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences. Nucleic Acids Research, 2002, 30(1): 325-327. doi: 10.1093/nar/30.1.325.
[20] LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2 (-Delta Delta C(T)) method. Methods, 2001, 25(4): 402-408.
[21] XU W Y, LIU T, ZHANG H Y, ZHU H. Mungbeangene subfamilies and their expression profiles under salt and drought stresses. Frontiers in Genetics, 2021, 12: 658148. doi: 10.3389/fgene. 2021.658148.
[22] SONG M, PENG X Y. Genome-wide identification and characterization of DIR genes in. Biochemical Genetics, 2019, 57(4): 487-506. doi: 10.1007/s10528-019-09903-7.
[23] 马金洋, 杨瑾冬, 李卿, 张磊. 丹参Dirigent基因家族的发现与生物信息学分析. 基因组学与应用生物学, 2017, 36(4): 1594-1610.
MA J Y, YANG J D, LI Q, ZHANG L. Discovery and bioinformatics analysis of dirigent multigene family in. Genomics and Applied Biology, 2017, 36(4): 1594-1610. (in Chinese)
[24] THAMIL ARASAN S K, PARK J I, AHMED N U, JUNG H J, HUR Y, KANG K K, LIM Y P, NOU I S. Characterization and expression analysis of dirigent family genes related to stresses in. Plant Physiology Biochemistry, 2013, 67: 144-153. doi: 10.1016/j.plaphy. 2013.02.030.
[25] KIM K W, MOINUDDIN S G A, ATWEL K M, COSTA M A, DAVIN L B, LEWIS N G. Opposite stereoselectivities of dirigent proteins inandspecies. The Journal of Biological Chemistry, 2012,287(41): 33957-33972.
[26] GUO J L, XU L P, FANG J P, SU Y C, FU H Y, QUE Y X, XU J S. A novel dirigent protein gene with highly stem-specific expression from sugarcane, response to drought, salt and oxidative stresses. Plant Cell Reports, 2012, 31(10): 1801-1812. doi: 10.1007/s00299-012-1293-1.
[27] 卫昭君, 牛冰洁, 王永新, 赵祥, 朱慧森, 郭秀萍, 乔栋. 茉莉酸甲酯对盐胁迫下偏关苜蓿种子萌发和幼苗生长的影响. 草地学报, 2020, 28(4): 998-1005. doi: 10.11733/j.issn.1007-0435.2020.04. 017.
WEI Z J, NIU B J, WANG Y X, ZHAO X, ZHU H S, GUO X P, QIAO D. Effect of methyl jasmonate on seed germination and seedling growth of'pianguan'under salt stress. Acta Agrestia Sinica, 2020, 28(4): 998-1005. doi: 10.11733/j.issn.1007- 0435.2020.04.017. (in Chinese)
[28] 樊晓培, 邢津溥, 魏铁锁, 李欣洋, 苍晶, 徐庆华, 张达. 外源MeJA对低温胁迫下冬小麦冷响应基因表达的影响. 麦类作物学报, 2020, 40(3): 292-299.
FAN X P, XING J P, WEI T S, LI X Y, CANG J, XU Q H, ZHANG D. Effect of exogenous MeJA on the expression of cold response genes in winter wheat under low temperature stress. Journal of Triticeae Crops, 2020,40(3): 292-299. (in Chinese)
[29] 李红利, 孙振元, 赵梁军, 韩蕾, 巨关升. 茉莉酸类物质对植物生长发育及抗性的影响. 中国农学通报, 2009, 25(16): 167-172.
LI H L, SUN Z Y, ZHAO L J, HAN L, JU G S. Effect of jasmonic acid and methyl jasmonate on the plant development and resistance. Chinese Agricultural Science Bulletin, 2009, 25(16): 167-172. (in Chinese)
[30] 朱菲菲, 刘奕清, 陈泽雄, 兰建彬. 外源脱落酸对盐胁迫下灰毡毛忍冬幼苗生理特性的影响. 中药材, 2013, 36(7): 1043-1046.
ZHU F F, LIU Y Q, CHEN Z X, LAN J B. Effect of abscisic acid on physiological characteristics inseedlings under salt stress. Journal of Chinese Medicinal Materials, 2013, 36(7): 1043-1046. (in Chinese)
[31] WANG Y L, MA F W, LI M J, LIANG D, ZOU J. Physiological responses of kiwifruit plants to exogenous ABA under drought conditions. Plant Growth Regulation, 2011, 64(1): 63-74.
[32] 刘春红. 黄瓜低霜霉威残留性相关基因CsDIR16的鉴定及功能分析[D]. 哈尔滨: 东北农业大学, 2017.
LIU C H. Identification and functional analysis of the propamcarb- related gene CsDIR16 in cucumber [D]. Harbin: Northeast Agricultural University, 2017. (in Chinese)
[32] YU G B, CHEN R N, CHEN Q S, CHEN F Q, LIU H L, REN C Y, ZHANG Y X, YANG F J, WEI J P. Jasmonic acid promotes glutathione assisted degradation of chlorothalonil during tomato growth. Ecotoxicology and Environmental Safety, 2022, 233: 113296.
[33] YADAV V, WANG Z y, YANG X z, WEI C h, CHANGQing X, ZHANG X. Comparative analysis, characterization and evolutionary study of dirigent gene family in Cucurbitaceae and expression of novel dirigent peptide against powdery mildew stress. Genes, 2021, 12(3): 326.
[34] GUTTMAN M, DONAGHEY J, CAREY B W, GARBER M, GRENIER J K, MUNSON G, YOUNG G, LUCAS A B, ACH R, BRUHN L, YANG X, AMIT I, MEISSNER A, REGEV A, RINN J L, ROOT D E, LANDER E S. LincRNAs act in the circuitry controlling pluripotency and differentiation. Nature, 2011, 477(7364): 295-300. doi: 10.1038/nature10398.
[35] 陈瑞兵. Dirigent蛋白催化菘蓝有效成分木脂素生物合成的机制研究[D]. 上海: 中国人民解放军海军军医大学, 2018.
CHEN R B. Study on the mechanism of the Dirigent proteins tocatalyze lignans in Isatisindigotica [D]. Shanghai: Naval Military Medical University of the Chinese people’s Liberation Army, 2018. (in Chinese)
[36] MARIAM C, MARIAM S, SAFA C, DONIA B, RADHIA G B. Ectopic expression of StERF94 transcription factor in potato plants improved resistance toinfection. Plant Molecular Biology Reporter, 2019, 37(5/6): 450-463. doi: 10.1007/s11105-019- 01171-4.
[37] GOU J Y, FELIPPES F F, LIU C J, WEIGEL D, WANG J W. Negative regulation of anthocyanin biosynthesis inby a miR156- targeted SPL transcription factor. The Plant Cell, 2011, 23(4): 1512-1522. doi: 10.1105/tpc.111.084525.
[38] HSIEH L C, LIN S I, SHIH A C, CHEN J W, LIN W Y, TSENG C Y, LI W H, CHIOU T J. Uncovering small RNA-mediated responses to phosphate deficiency inby deep sequencing. Plant Physiology, 2009, 151(4): 2120-2132.
[39] CUI L G, SHAN J X, SHI M, GAO J P, LIN H X. The miR156-SPL9-DFR pathway coordinates the relationship between development and abiotic stress tolerance in plants. The Plant Journal, 2014, 80(6): 1108-1117.
Genome-Wide Identification of DIR Family Genes in Tomato and Response to Abiotic Stress
CHEN FengQiong, CHEN QiuSen, Lin JiaXin, WANG YaTing, LIU HanLin, LIANG BingRuoShi, DENG YiRu, REN ChunYuan, ZHANG YuXian, YANG FengJun, YU GaoBo, WEI JinPeng, WANG MengXue
College of Horticulture and Landscape Architecture, Heilongjiang Bayi Agricultural University, Daqing 163319, Heilongjiang
【Objective】The aim of this study was to identify all members of tomato DIR gene family, and to predict and analyze their gene structures, physicochemical properties of proteins, phylogenetic relationship, chromosome location, collinearity analysis, promoter elements, expression pattern, prediction of interacting transcription factors and endogenous competitive RNA, so as to provide a reference for exploring the role of DIR in tomato growth and development and response to environmental stresses. 【Method】The family members of DIR were identified in the whole genome level by bioinformatics methods. The chromosome location, conserved motif, cis-acting element, transcription factors, miRNA and circRNA were analyzed with Phytozome, MEME, PlantCARE, opsRNATarget and plantcircnet, respectively. Chromosome location map, evolutionary tree, relationship map between DIR and transcription factors, ceRNA network, and etc., were drown using Maptea, TBtools, Cytoscape and Omicshare. The expression level of DIR under environmental stresses was studied by NCBI gene database, transcriptome sequencing and qRT-PCR. 【Result】27 members of DIR family were identified from the whole genome of the tomato, which were named-, and DIR genes of the tomato were located on 12 chromosomes, most of which were located at the end of chromosomes. The gene structures, motifs and domains were relatively conservative, and the classical structure of one exon existed in 22genes. The collinear relationship of DIR genes between tomato andwas much higher than that of rice and soybean. Based on the phylogenetic relationship, 27 DIR members of tomato were divided into three different subfamilies. The tissue specific expression analysis revealed that transcription levels of DIR members of tomato were higher in root. In addition, the promoter region of these genes contained multiple cis-acting elements related to abiotic stress, including drought, low temperature, and hormone induction, such as MeJA, ABA and SA. Hormone, growth and abiotic stress related to transcription factors (ERF, E2F / DP and MYB) were also predicted. Combined with our transcriptome data of pesticide stress and published transcriptome data analysis, the expression level of 5, 10, 10 and 13genes was significantly up-regulated after pesticide, drought, salt and cold stress. Andwas induced by all the four stresses, while,andwere specifically responded to the induction of cold stress, andspecifically responded to salt stress. Finally, the ceRNA regulation of tomato DIR showed that miR-156 might interacted with the target geneto regulate tomato against stresses.【Conclusion】A total of 27 DIR gene family members were identified from the tomato genome and unevenly distributed on 12 chromosomes, with high expression in roots. The expression of,,and othergenes was included by MeJA, ABA and SA and other hormone response elements, among which6 only contained MeJA element, and27 only contained SA response element. In addition,2,14,23 and other genes were participated in drought, salt, low temperature and other stresses. Especially for, it could be activated under different stress treatments. In addition, DIR genes interacted with transcription factors and noncoding RNA to regulate the responses of tomato plants under stresses.
DIR protein;; gene family; bioinformatics

10.3864/j.issn.0578-1752.2022.19.010
2021-12-19;
2022-03-21
黑龙江省博士后科研启动项目(LBH-Q20052)、黑龙江八一农垦大学博士科研启动项目、黑龙江省“揭榜挂帅”科技攻关项目(2021ZXJ05B02)、黑龙江省留学归国项目、国家自然科学基金(31301769)、黑龙江省自然科学基金(QC2018023)
陈凤琼,E-mail:1922925586@qq.com。通信作者魏金鹏,E-mail:weijp81@163.com。通信作者王孟雪,E-mail:wangmengxue@163.com
(责任编辑 赵伶俐)

