基因数据的交互依赖特征选择算法
张 俐
(江苏理工学院计算机工程学院 江苏 常州 213001)
过去几十年,在生物信息领域产出大量基因数据[1-2]。这些基因数据普遍具有样本小、维度高和高噪声等特点[3]。如何处理这些不相关和冗余特征给数据降维带来重大挑战。常见的数据降维包括特征提取[4]和特征选择[5]两类。特征选择由于可以删除无关和冗余特征,同时保留相关原始特征,因此引起许多关注。
在特征选择中主要有数据层面(过滤式方法)和算法层面(包装器方法和嵌入式方法)[6-8]两方面的研究。过滤式特征选择算法凭借其计算成本低、与具体分类器分离及应用领域广等优点,逐渐成为特征选择技术中的研究热点。常见的基于信息论的过滤式特征选择算法包括采用平均冗余策略的特征选择算法(MID[9]、MIQ[9]、JMI[10]和CFR[11]等)和采用“最大最小”极端标准的特征选择算法(CMIM[12]、JMIM[13]和DWUR[14]等)。然而这些算法存在忽视对交互依赖特征相关性和冗余性判断的问题。
因此,本文提出一种利用联合互信息和互信息判断特征与类标签之间相关性和冗余性的特征选择算法(joint feature relevance and redundancy, JFRR)。该算法利用联合互信息计算在已选特征下候选特征与类标签之间的相关性;通过互信息计算已选特征和候选特征的冗余性;通过在9 个基准基因数据集的实验对比,该算法(JFRR)优于其他特征选择算法(MID、MIQ、CMIM、JMIM、CFR 和CMIMRMR[15])。
1 联合互信息的一些概念
设X、Y和Z是3 个 离 散 型 变 量[16],其 中,X=

2 JFRR 算法的提出
通过以上描述可知,传统的特征选择算法通常使用最小化冗余项和最大化相关项选择特征子集S。但是由此产生如下问题:1) 当已选特征量增加时,冗余项的大小也会随着相关项的增加而增加。这就存在一些冗余特征可能被选中;2) 在冗余项中,只考虑已选特征和候选特征之间互信息的计算,而忽视类标签,可能会造成已选特征和候选特征共享信息,意味着它们之间存在冗余信息。事实上,它们可能与类标签集合C之间共享不同信息。
以上问题可能会高估某些候选特征的重要性[17-19]。因此需要考虑,如何在已选特征集合S规模不断增加的情况下,解决S与类标签集合C的相关性,同时解决候选特征fk与S的冗余性,以及解决在S条件下,候选特征fk与 类标签C的相关性的问题。
为此,本文提出一种基于信息论的特征选择算法(JFRR)。该算法充分利用了线性累计加和的方式,具体如下:

式中,设F是原始特征集合,S⊂F;J(·)代表评估标准;fi∈S,fk∈F−S。
通过式(4)可知,JFRR 算法利用联合互信息和互信息原理充分考虑S与C之 间的相关性,fk与S的冗余性以及在S条件下,fk与C之间的相关性。JFRR 算法的具体描述如下。
输入:原始特征集合F={f1,f2,···fn},类标签集合C,已选特征子集S,阈值K

从式(4)可知,JFRR 算法采用前向顺序搜索特征子集。JFRR 算法主要分为3 部分。第1 部分为1)~7),主要是初始化S集合和计数器k;将选择出最大的特征fk加 入S集合,同时fk变成已选特征fi。第2 部分为8)~13),分别计算I(fi;C)、I(fk;fi)和I(fk,fi;C)的值。第3 部分为14)~19),根据式(4)的选择标准选择fk,一直循环到用户指定的阈值K就停止循环。
3 实验验证与分析
3.1 实验环境设置
本节将JFRR 与MID、MIQ、CMIM、JMIM、CFR 和CMI-MRMR 算法进行对比。具体分类器为:决策树(C4.5)和支持向量机(support vector machine, SVM)。本 文 的 实 验 环 境 是Intel-i7 处 理器,16 GB 内存,仿真软件是Python2.7。实验数据集选择ASU 和UCI 基因数据集[9,20],详细描述见表1。其中,这9 个数据集包含不同的样本数、特征数和类数。样本范围为50~569,特征范围为31~9 712,类的范围为2~12,数据类型涉及连续型和离散型。采用6 折交叉验证方法进行实验验证。为保证实验公平,分别通过分类评价指标fmc(F1_micro)和pcm(Precision_micro)来评价预测性能。

表1 数据集描述
3.2 特征选择算法性能比较与讨论
为了比较JFRR 与MID、MIQ、CMIM、JMIM、CFR 和CMI-MRMR 算法之间的优劣性,将它们所选的特征子集放到同一个分类器(C4.5 和SVM)进行比较,特征子集的规模设置为30。表2 选择C4.5 分类器。表3 选择SVM 分类器。在表2~表3 中,粗体代表该数据集下特征选择算法中最高平均分类预测值。“Wins/Ties/Losses”描述JFRR算法分别与MID、MIQ、CMIM、JMIM、CFR 和CMI-MRMR 算法之间的优/平/输个数。

表3 SVM 分类器的平均fmc 性能比较 %
3.2.1 特征选择算法的fmc 性能比较
在表2 中,7 个特征选择算法的平均fmc 精度值分别为82.459%、80.24%、68.122%、75.356%、68.695%、73.047%和77.296%。JFRR 算法获得最高fmc 值。同时,从WINS/TIES/LOSSES 行的统计结果得出JFRR 分别优于MID、MIQ、CMIM、JMIM、CFR 和CMI-MRMR 算 法9、9、9、9、8 和6 次。
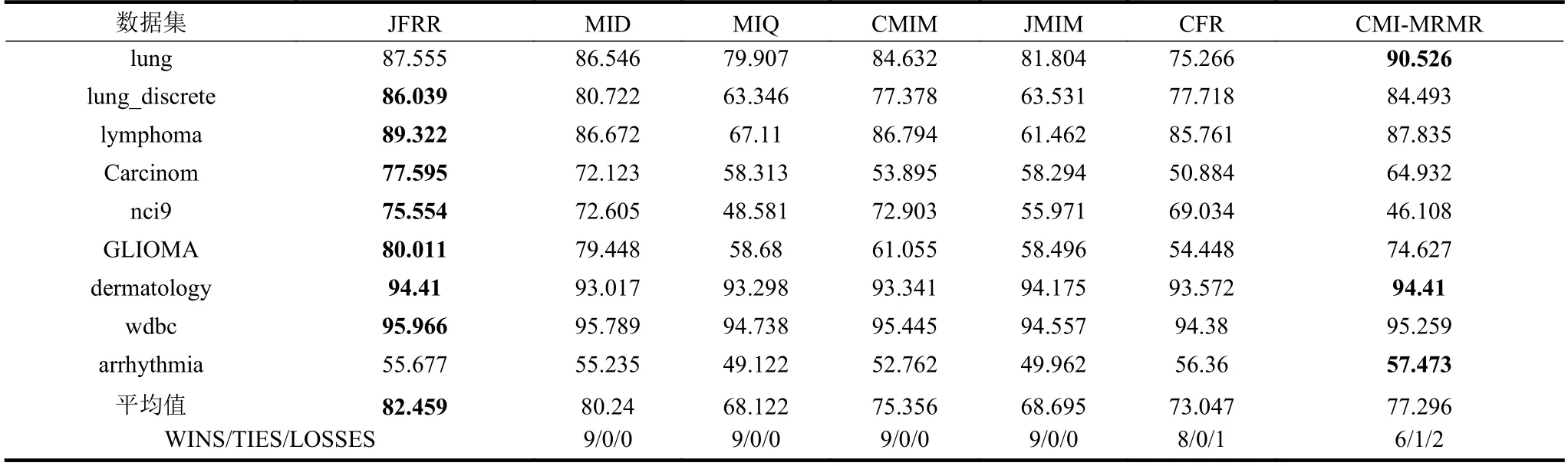
表2 C4.5 分类器的平均fmc 性能比较 %
在表3 中,7 个特征选择算法的平均fmc 精度值分别为80.985%、79.925%、67.712%、77.631 7%、68.329%、75.302%和76.461%。JFRR 算法获得最高fmc 值。同时,从WINS/TIES/LOSSES 行的统计结果得出JFRR 分别优于MID、MIQ、CMIM、JMIM、CFR 和CMI-MRMR 算 法6、8、8、8、7 和6 次。
为了进一步比较特征子集对fmc 值的影响,图1 和图2 分别给出部分数据集的fmc 性能差异。当数据的维数不断增加时,JFRR 算法通过动态调整特征间的相关性和冗余性提升了特征子集的数据质量。图1 和图2 的实验结果显示,JFRR 算法对分类提升的效果明显。并且,JFRR 明显优于MID、 CMIM、 MIQ、 JMIM、 CFR 和CMIMRMR。
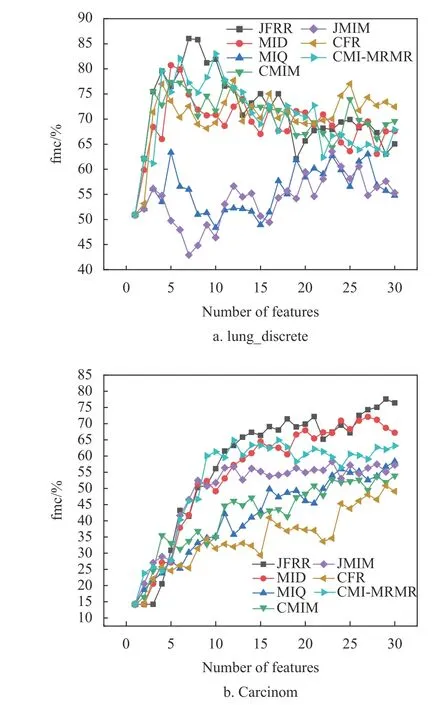
图1 C4.5 在高维数据集上的性能比较
图1 是C4.5 在高维数据集上的性能比较。在图1a 中,JFRR 算法的分类fmc 值为86.039%,是7 种分类算法中最高的,分别比MID、MIQ、CMIM、JMIM、CFR 和CMI-MRMR 高出5.317%、22.693%、8.661%、22.508%、8.321%和1.546%。在图1b 中,JFRR 算法的分类fmc 值为77.595%,也是7 种分类算法中最高的,分别比MID、MIQ、CMIM、JMIM、CFR 和CMI-MRMR 高出5.472%、19.282%、23.7%、19.301%、26.711%和12.663%。图2 是SVM 在高维数据集上的性能比较。在图2a中,JFRR 算法的分类fmc 值为95.102%,是7 种分类算法中最高的,分别比MID、MIQ、CMIM、JMIM、 CFR 和CMI-MRMR 高出0.0%、24.361%、1.389%、29.931%和3.143%和0.0%。在图2b 中,JFRR 算法的分类fmc 值为94.91%,是7 种分类算法中最高的,分别比MID、MIQ、CMIM、JMIM、CFR 和CMI-MRMR 高出0.347%、4.233%、0.53%、4.058%、0.351%和4.577%。
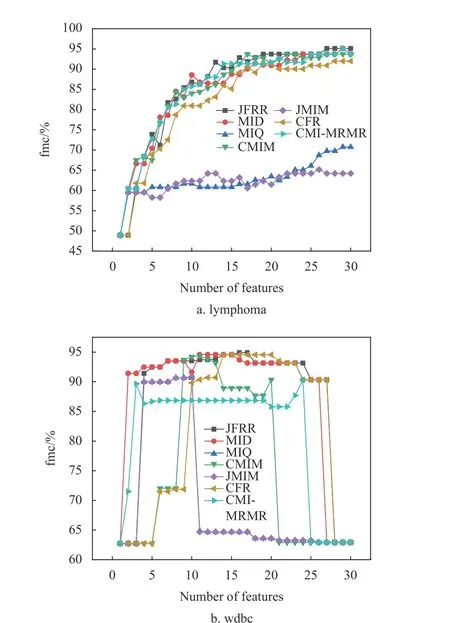
图2 SVM 在高维数据集上的性能比较
3.2.2 特征选择算法的pcm 性能比较
图3 为pcm 盒图。从图3a 中可以得出,在C4.5 分类器的pcm 盒图中,使用JFRR 算法选择出的特征集合在五位数(最小值、四分位数(第25 个百分位数)、中位数、四分位数(第75 个百分位数)和最大值)中体现出的分类效果都是最优。同时,从图3b 中也可以得出,在SVM 分类器的pcm 盒图中,使用JFRR 算法选择出的特征集合在五位数(最小值、四分位数(第25 个百分位数)、中位数和四分位数(第75 个百分位数))中体现出的分类效果都是最优的效果。

图3 C4.5 分类器和SVM 分类器的pcm 盒图
综上,不同分类器表现出的分类结果不尽相同。但是,JFRR 算法在fmc 和pcm 的评价指标值在大多数数据集上都是最好。从C4.5 和SVM 分类器表现结果中可知,C4.5 分类性能明显优于SVM分类性能。
3.3 算法的运行时间分析
计算特征选择算法的运行时间也是衡量特征选择算法重要性的标准之一。JFRR、MID、MIQ、CMIM、JMIM、CFR 和CMI-MRMR 算法在9 个数据集上进行特征排序后得出的运行时间如表4 所示。可以看出,JFRR 算法的运行时间在可接受的范围之内。

表4 不同特征选择算法运行时间比较 s
3.4 实验算法比较
本节分析JFRR 与MID、CMIM、MIQ、JMIM、CFR 和CMI-MRMR 之间在交互特征依赖相关性和冗余性的差异。从表5 可以得出,与JFRR 相比,MID、MIQ、CMIM 和CFR 将I(fk;C)定义为衡量特征相关性的标准。CMI-MRMR 将I(fi,C|fk)定义为衡量特征相关性的标准。只有JFRR 和JMIM 将I(fk,fi;C)定义为衡量交互特征依赖性动态变化标准。但是,JMIM 算法却忽视特征冗余性变化。因此,得出JFRR 与其他特征选择算法差异明显。

表5 算法比较
4 结 束 语
随着基因数据中高维特征数据的不断增多,特征间的关系变得越来越复杂(包含大量无关特征和冗余特征)。而传统的特征选择算法往往忽视特征间的相关性和冗余性之间的联系。本文提出一种基于联合互信息的JFRR 算法。该算法利用互信息和联合互信息间的关系动态分析和调整特征间以及特征与类标签间的相关信息和冗余信息,从而达到删除无关特征和冗余特征的目的,以此提高特征子集的数据质量。为了全面验证JFRR 算法的有效性,实验在9 个基因数据集上进行。分别通过使用分类器(C4.5 和SVM)和分类准确率指标(fmc 和pcm)全面评估所选特征子集的质量。实验结果证明JFRR 明显优于MID、MIQ、CMIM、JMIM、CFR和CMI-MRMR 等6 种特征选择算法。
但在一些基因数据中,JFRR 算法仍旧存在选择出的特征子集不理想的情况。未来的工作将进一步研究和改进互信息和联合互信息的关系,并以此优化JFRR 算法,同时在更广泛的基因数据集中对算法进行验证,以此提高分类预测精度。

