高、低剩余采食量滩羊瘤胃微生物的差异
和东迁,陶金忠
(宁夏大学农学院, 宁夏 银川 750021)
饲料成本占养殖总成本的65%~70%,增加养殖经济效益,提高饲料效率是关键。剩余采食量(residual feed intake, RFI)为基于动物体况和生产性能的实际饲料摄入量和其预期饲料摄入量之间的差异[1],被广泛应用于评估各种家畜动物饲料效率,RFI 较低的个体饲料效率更高。然而RFI 表型受许多因素影响,如日粮、喂养方式(如喂养的持续时间和次数)、瘤胃发酵、体况状况(脂肪和蛋白质)、代谢过程、运动和饲喂时所产生的热增耗[2]。目前鲜有研究报道性别与饲料效率间的差异关系,仅Ahmed 等[3]发现性别影响不同RFI 肉牛瘤胃上皮基因差异表达,对生长性能和瘤胃细菌没有显著影响。
瘤胃是瘤胃微生物的主要发酵场所,短链脂肪酸是瘤胃发酵的最终产物,是反刍动物能量的主要供应来源。因此,瘤胃功能与饲料效率密切相关。瘤胃发酵中会产生一些不必要的能量损失。甲烷是造成温室效应的主要气体之一,反刍动物瘤胃产生的CH4约7.7 × 107t,绝大部分CH4气体来源于反刍动物。据报道,反刍动物胃肠道CH4消耗占总能量摄入的5%~10%[4]。肉牛甲烷的日产率与RFI 正相关,低RFI 个体甲烷排放量少。因此,低RFI 动物个体的选育不仅可以降低动物的养殖成本,还可以减小养殖对环境的污染。Guan 等[5]报道了瘤胃微生物群及其发酵参数与牛的饲料效率之间的潜在联系,低RFI 较高RFI 个体拥有特异性菌群。Deborah 等[6]通过16S rRNA 测序技术评价内洛尔肉牛瘤胃微生物群与饲料效率的关系发现,低RFI 和高RFI 牛的细菌组成差异主要与Lachnospiraceae、Ruminococcaceae、Christensenellaceae 以及Prevotella的成员有关。对不同RFI 绵羊的16s RNA 测序发现,低RFI 绵羊表现出更丰富和更多样化的微生物菌群[7]。瘤胃微生物代谢网络异常也与饲料效率密切相关[8]。对不同RFI 肉牛瘤胃微生物18s rRNA 测序发现,原虫群落的未分配类群和系统发育多样性的差异可能是造成肉牛饲料效率表型差异的原因[9]。Liang 等[10]发现,低RFI 动物拥有更低的pH,并且低RFI 瘤胃环境比高RFI 更稳定,有利于饲料发酵。目前研究表明瘤胃菌群与饲料效率密切相关。因此,本研究选择大群体公母滩羊,对其进行不限定运动饲养,探究不同RFI 公母滩羊瘤胃微生物差异,确定影响饲料效率的关键微生物,为滩羊选育实践提供理论支持。
1 材料与方法
1.1 试验羊饲喂与管理
试验在宁夏盐池滩羊育种选育场进行。分别选择6 月龄健康和具有良好系谱的滩羊公羊163 只,母羊182 只。试验采用单栏颈夹饲喂,在采食期间采用颈夹固定羔羊,采食完成后松开颈夹,让其自由运动[11]。试验饲粮配方和加工饲粮营养水平参考肉羊饲养标准[12],试验饲粮按配方加工为颗粒饲料。日粮组成及营养水平与陈丽尧等[13]所报道一致(表1)。试验过渡期15 d,正试期50 d,试验期间饲喂同一种饲料。在试验开始前记录绵羊初始体重,并且每10 d 称重一次至试验结束。
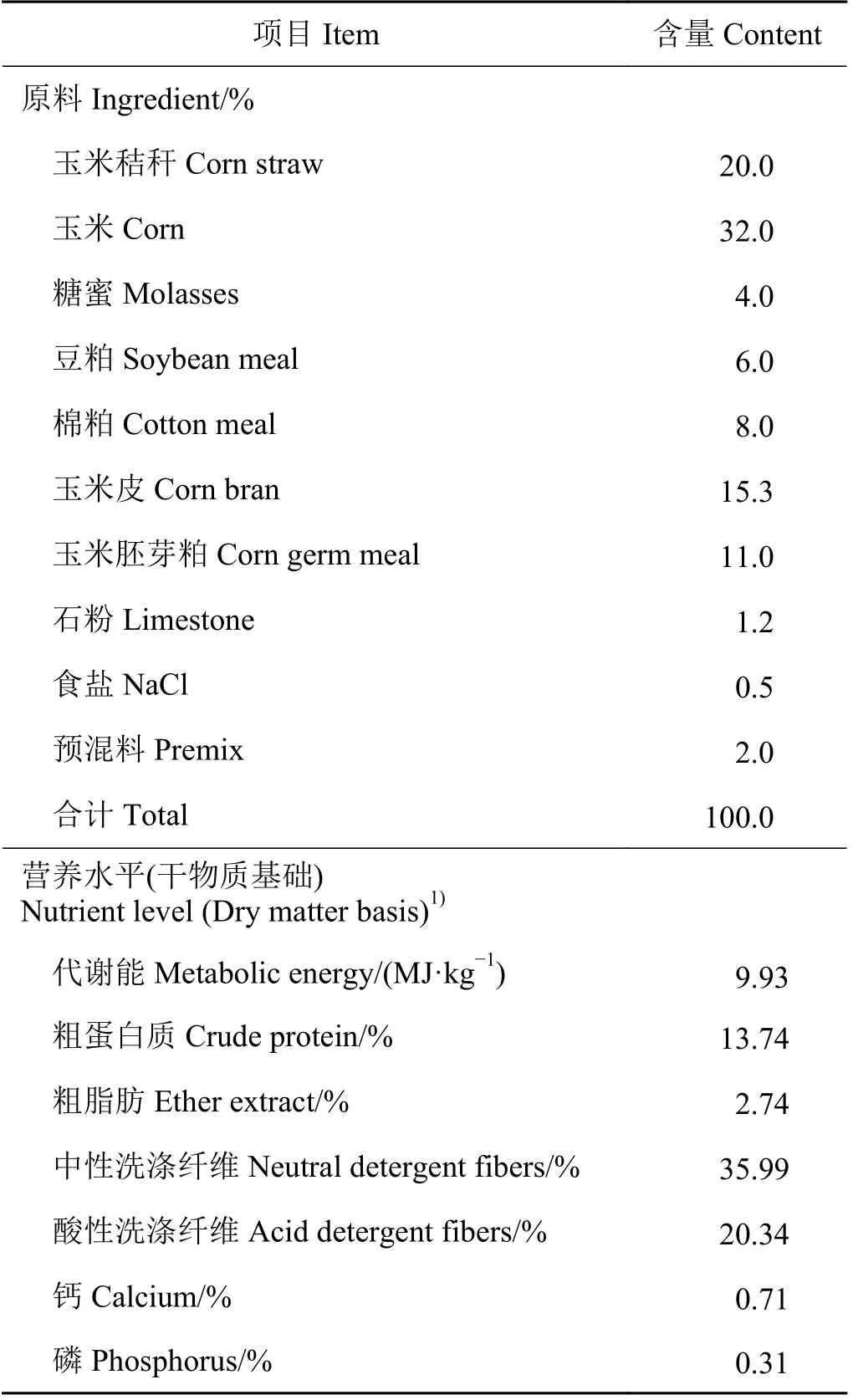
表1 基础日粮组成及营养水平Table 1 Composition and nutrient levels of basal diets
1.2 RFI 表型计算
RFI 通过回归模型计算,所用模型[13]如下:
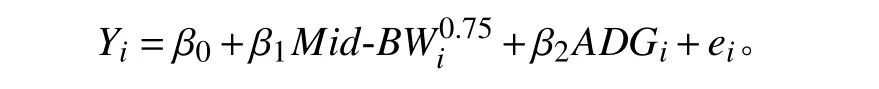
式中:Yi为第i只动物的DMI(干物质采食量);β0为回归截距;β1为Mid-BW0.75对应的回归系数;β2为ADG(日增重)对应的回归系数;ei为第i只动物的随机误差,Mid-BWi0.75为第i只动物的中期代谢体重,ADGi为第i只动物的平均日增重。
公、母滩羊的回归方程依次为:
DMI预测= - 0.244 + 1.669Mid-BW0.75+ 0.079ADG。
DMI预测= 0.103 + 2.126 ×Mid-BW0.75+ 0.051ADG。
RFI=RFI实际-RFI预测。
当个体RFI≥RFI平均值+0.5 时,划分为高RFI组;当个体RFI≤RFI平均值-0.5 时,划分为低RFI 组。
1.3 瘤胃液采集
饲养试验结束后选取极高RFI 母羊和公羊各7 只,极低RFI 母羊和公羊各8 只。在第66 天晨饲前采用绵羊瘤胃导管体外抽取瘤胃液,并将瘤胃液分管装入50 mL 冻存管,置于-20 ℃条件下保存,用于瘤胃微生物DNA 的提取。
1.4 瘤胃液DNA 提取
采用CTAB 法[9]提取样本基因组DNA,然后利用琼脂糖凝胶电泳和NanoDrop 2000 分光光度计检测DNA 纯度和浓度,取适量样品于离心管中,使用无菌水稀释样品至1 ng·μL-1。
1.5 16S rDNA V4 区PCR 扩增子测序
根据测序区域选择,使用带Barcode 的特异引物。16S V4 区域引物为515F-806R,上游引物F:CCTAYGGGRBGCASCAG/下游引物R:GGACTACN NGGGTATCTAAT。使用New England Biolabs 公司的Phusion® High-Fidelity PCR Master Mix with GC Buffer。对高效和高保真的酶进行PCR,确保扩增效率和准确性。PCR 产物使用2%浓度的琼脂糖凝胶进行电泳检测。根据PCR 产物浓度进行等浓度混样,充分混匀后使用1 × TAE 浓度2%的琼脂糖胶电泳纯化PCR 产物,选择主带400~450 bp 的序列,割胶回收目标条带。使用Illumina 公司TruSeq DNA PCR-Free Library Preparation Kit 建库试剂盒进行文库的构建,构建好的文库经过Qubit 定量和文库检测,合格后,使用NovaSeq 6000 进行上机测序。
1.6 信息分析与数据统计
剔除DNA 降解或不合格样本后,获得极高组RFI 母羊6 只和公羊5 只、极低组RFI 母羊6 只和公羊7 只的原始数据。测序得到的原始数据,存在一定比例的干扰数据,为了使信息分析的结果更加准确可靠,对原始数据进行拼接过滤,得到有效数据。基于有效数据进行OTUs (operational taxonomic units)聚类和物种分类分析,根据OTUs 聚类结果,对每个OTUs 的代表序列做物种注释,得到对应的物种信息和基于物种的丰度分布情况。同时,对OTUs 进行丰度、Alpha 多样性计算、Venn 图,以得到样品内物种丰富度和均匀度信息、不同样品或分组间的共有和特有OTUs 信息。对OTUs 进行多序列比对并构建系统发生树,进一步得到不同样品和分组的群落结构差异,通过主坐标分析(PCoA)和样品聚类树进行展示。为进一步挖掘分组样品间的落结构差异,选用T-test 和LEfSe 统计分析方法对分组样品的物种组成和群落结果进行差异显著性检。
利用Uparse 软件(Uparse v7.0.1001,http://www.drive5.com/uparse/)对所有样本的全部 Effective Tags进行聚类,默认以97%的一致性将序列聚类成为OTUs。对OTUs 序列进行物种注释,用Mothur 方法与SILVA138 (http://www.arb-silva.de/)的SSUrRNA数据库进行物种注释分析(设定阈值为0.8~1)。使用MUSCLE (Version 3.8.31, http://www.drive5.com/muscle/)软件进行快速多序列比对,得到所有OTUs代表序列的系统发生关系。使用Qiime 软件(Version 1.9.1)计算Observed-OTUs,Chao1,Shannon,Simpson,ace,Goods-coverage,PD_whole_tree 指数,使用R 软件(Version 2.15.3)绘制稀释曲线、rank abundance 曲线、物种累积曲线,并使用R 软件进行α 多样性指数组间差异分析;α 多样性指数组间差异分析进行非参数检验,选用agricolae包的wilcox 检验。试验数据使用 Excel 2010 进行整理,应用SPSS 17.0 进行独立样本 t 检验,P< 0.05 时认为差异显著,结果均以平均值 ± 标准误(means ± SEM)形式列出。PCoA基于unweighted unifrac 距离来分析。
2 试验结果
2.1 菌群测序量及序列长度分布
对瘤胃液样本的肠道菌群DNA 片段进行双端测序,筛选过滤后共产生2 063 628 条原始读段,平均每个样本85 970 条原始读段。经质控筛选过滤后共产生2 028 835 条有效读段,平均每个样本84 535 条有效读段。其中Q20 和Q30 (碱基质量值≥20 或30所占的比例)平均值分别为98%和94% (附表1)。
2.2 OUT 聚类分析
OTUs 是人为设置的微生物分类单元,按照序列相似度≥97%归为同一个OTU,即同一微生物物种的标准进行聚类,经测序后,基于97%物种相似性,将24 组瘤胃液样品中获得的序列进行OTU 聚类,对样品进行OTU 的统计,剔除稀有OTU 后,Venn 图显示(图1),极低组公羊发现1 812 个OTUs,极高组公羊发现1 610 个OTUs,极低组母羊发现1 677 个OTUs,极高组母羊发现1 698 个OTUs。极高组公羊和极低组公羊两组共有1 274 个OTUs。极高组母羊和极低组母羊共有1 327 个OTUs,极高组公羊和母羊共有1 303 个OTUs,极低组公羊和母羊共有1 377 个OTUs。
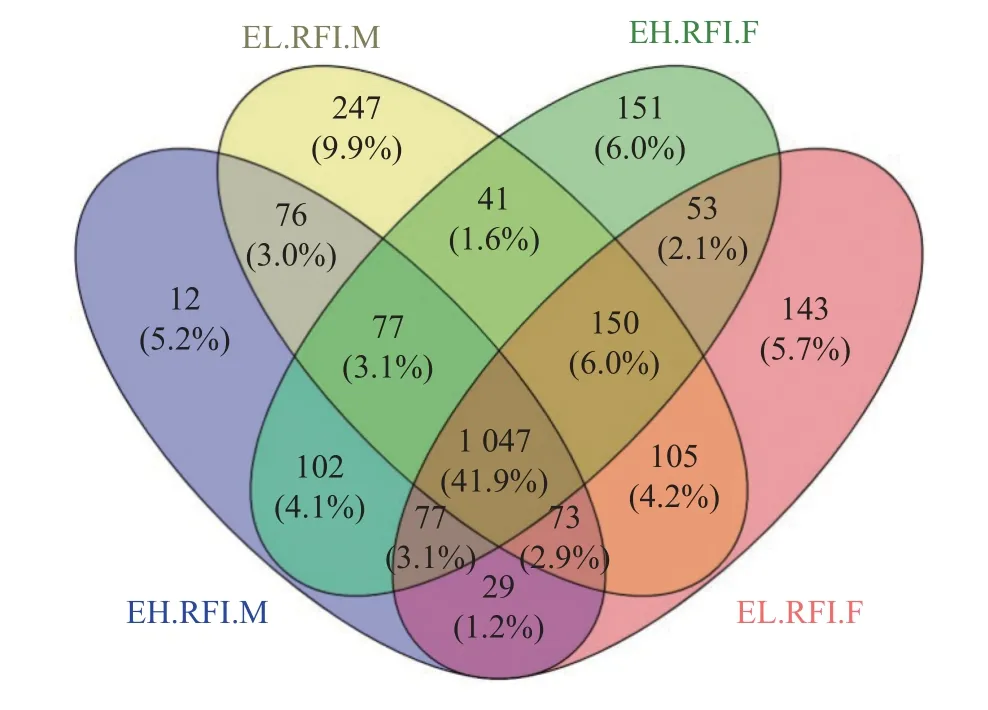
图1 不同RFI 组OTUsVenn 图Figure 1 OTUs Venn diagram of different RFI groups
2.3 微生物群落Alpha 多样性分析
所有组观测到的物种数、Shannon-Wiener 指数、Simpson 指数、Chao1 指数、ACE 指数和基因文库覆盖率均无显著差异(P> 0.1),EHM 与ELM 的PD_whole_tree 差异显著(P< 0.1) (表2)。但是极高组公羊的PD_whole_tree 明显高于其他组别。表明极高低组RFI 公羊和极高低组母羊之间菌群多样性差异较小,菌群趋于一致性。
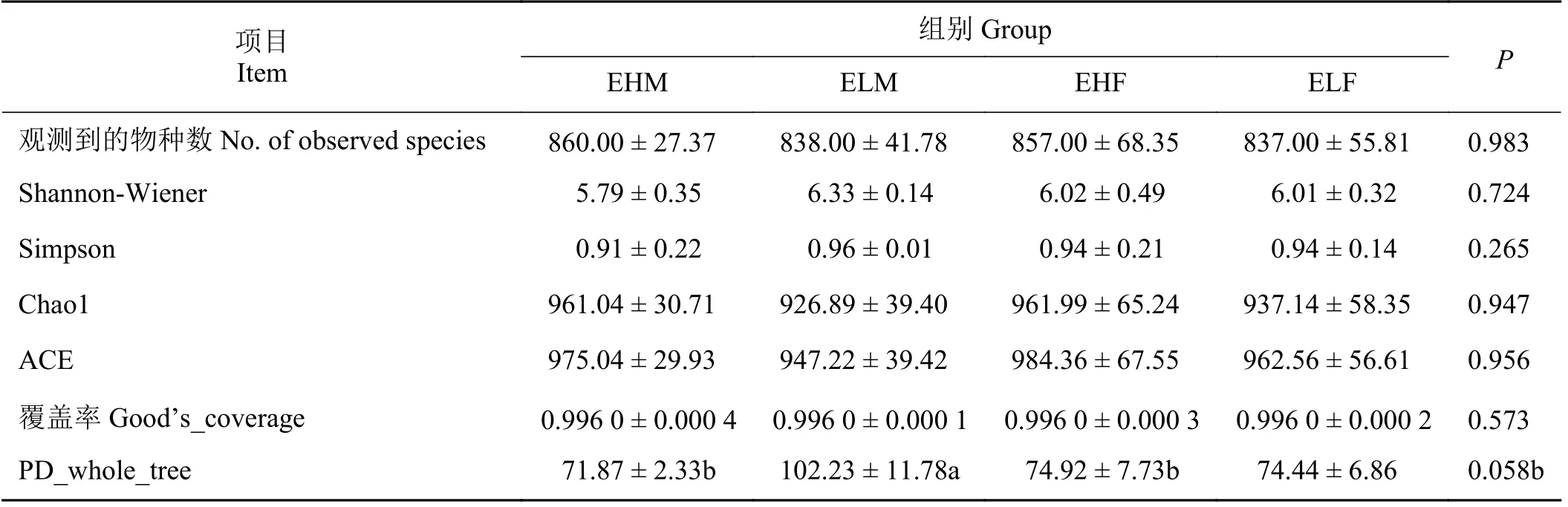
表2 不同剩余采食量对滩羊瘤胃微生物多样性影响Table 2 Different residual feed intake on rumen microbial diversity of Tan sheep
2.4 微生物群落多样性曲线
当测序量达到3 500 时,不同RFI 组物种曲线趋向平坦,说明测序数据量渐合理。等级聚类曲线可直观地反映样本中物种的丰富度和均匀度。在水平方向上,物种的丰富度逐渐变宽,物种的丰富度升高,曲线在横轴上的跨度越大;在垂直方向上,曲线逐渐变平缓,物种分布均匀性良好(图2)。

图2 不同RFI 瘤胃微生物群落多样性曲线Figure 2 Rumen microbial community diversity curve of Tan sheep with different residual feed intake
2.5 微生物群落β 多样性分析
β 多样性组间差异分析的箱型图(图3)表明,公羊极高组与极低组之间存在极显著差异(P< 0.01),母羊极高组与极低组之间差异不显著(P> 0.05)。极低组公羊和母羊之间差异极显著(P< 0.01),极高组公羊和母羊之间差异不显著(P> 0.05)。

图3 不同 RFI 公母羊组间微生物群落β 多样性指数Figure 3 Box diagram of microbial community β diversity index between male and female sheep groups with different RFI
2.6 主坐标分析
滩羊不同RFI 组间样本具有部分重叠,但没有明显的分离,表明样本组间和组内微生物群落差异性较小(图4)。
2.7 UPGMA 聚类
极高RFI 组公羊4 个样本聚集在一起,聚类效果较好,其他RFI 组样本聚类效果不明显(图4)。从整体结构来看,不同RFI 组微生物结构相似。

图4 不同RFI 组微生物群落PCoA 得分图Figure 4 PCoA score of microbial communities in different RFI Tan sheep groups
2.8 滩羊瘤胃微生物组成
滩羊瘤胃微生物在门和属水平上公母羊优势菌属不同,但是整体结构相似。在门水平上,公羊优势菌门是拟杆菌门和厚壁菌门,母羊优势菌门是厚壁菌门和拟杆菌门 (表3)。在属水平上,公羊优势菌属为普雷沃氏菌属(Prevotella)、韦荣球菌UCG-002(Erysipelotrichaceae_UCG_002), 母 羊 优 势 菌 属 是Olsenella和普雷沃氏菌属(表4)。
在门水平上(表3),拟杆菌门公羊极高组与极低组之间差异显著(P< 0.05),极高组公羊显著高于母羊。放线菌门公羊极低组明显高于极高组,极高组母羊明显高于公羊。广古菌门公羊极高组显著低于极低组(P< 0.05)。变形菌门极高组公羊显著高于其他组(P< 0.05)。在属水平上(表4),Olsenella菌属极高组母羊明显高于公羊,极低组公羊明显高于母羊。韦荣球菌科UCG-002 极高组公羊显著高于其他组别(P< 0.05)。甲烷短杆菌极高组公羊与极低组公羊明显高于其他组别。
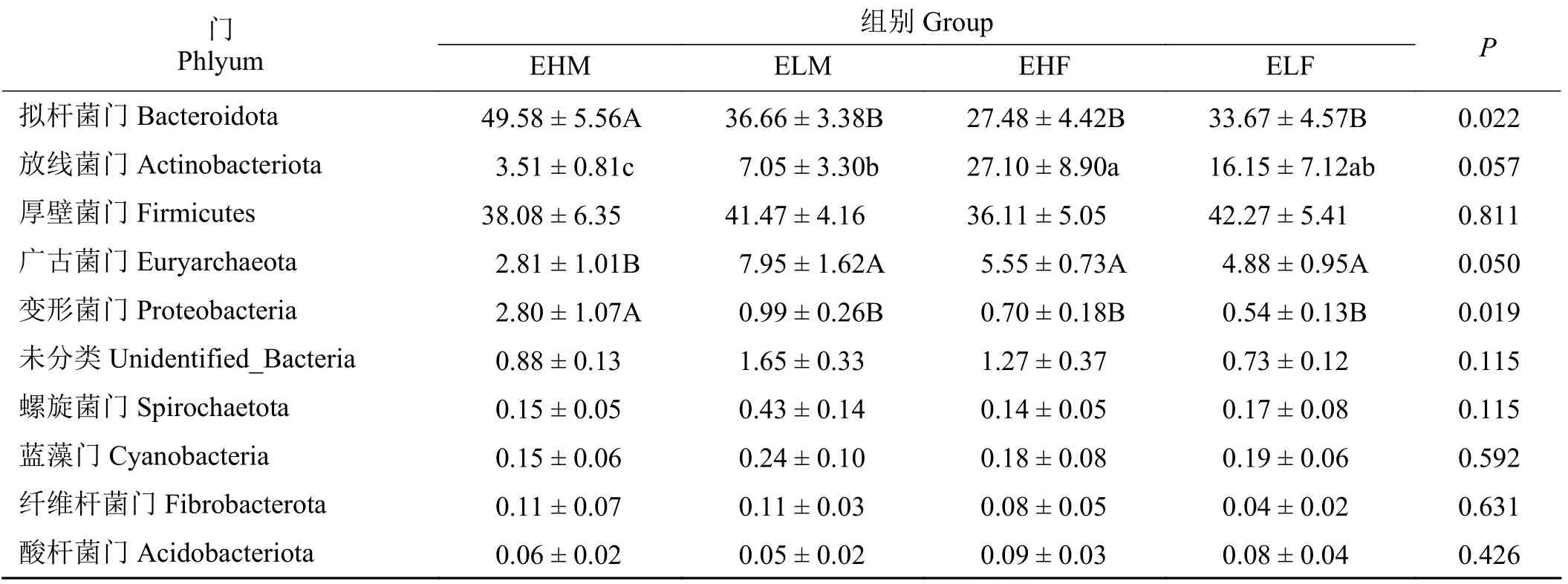
表3 微生物相对丰度在门分类水平上排名前十的物种Table 3 Top 10 species at phylum taxonomic level in relative abundance of microorganisms

表4 微生物相对丰度在属分类水平上排名前十的物种Table 4 Top ten species at genus taxonomic level in relative abundance of microorganisms
2.9 差异微生物的LEfSe 及功能富集分析
LEfSe 可以在各个水平上比较微生物丰度差异,以线性判别分析(LDA) 得分 > 4 为符合生物标记物的菌落。极高组公羊与极低组公羊比较发现梭菌纲、古菌域、广古菌门、甲烷杆菌科、甲烷杆菌纲、甲烷杆菌目和甲烷短杆菌属(Methanobrevibacter)在极低组公羊中高表达,Prevotella_DJF_cp65、Bacteria、普雷沃氏菌科、韦荣球菌科UCG-002 和韦荣球菌科SG0102 在极高组公羊中高表达(图5)。极高组母羊和极高组公羊比较发现, Bacteroidia、Bacteroidales、B a c t e r o i d o t a、 P r e v o t e l l a c e a e、 P r e v o t e l l a、Succinivibrionaceae_UGG_001、 Aeromonadales、Succinivibrionaceae 在极高组公羊中高表达,双歧杆菌属(Bifidobacterium)、双歧杆菌目、双歧杆菌科、放线菌纲、Olsenella.umbonata、Olsenella、Atopobiaceae、Coriobacteriales、Coriobacteriia和Actinobacteriota在极高组母羊中高表达(图5)。比较发现,月形单胞菌属 (Selenomonas)在极低组公羊中高表达,梭状芽胞 杆 菌UGG.014 (Clostridia_UGG_014)、Olsenella_umbonata在极低组母羊中高表达(图6)。
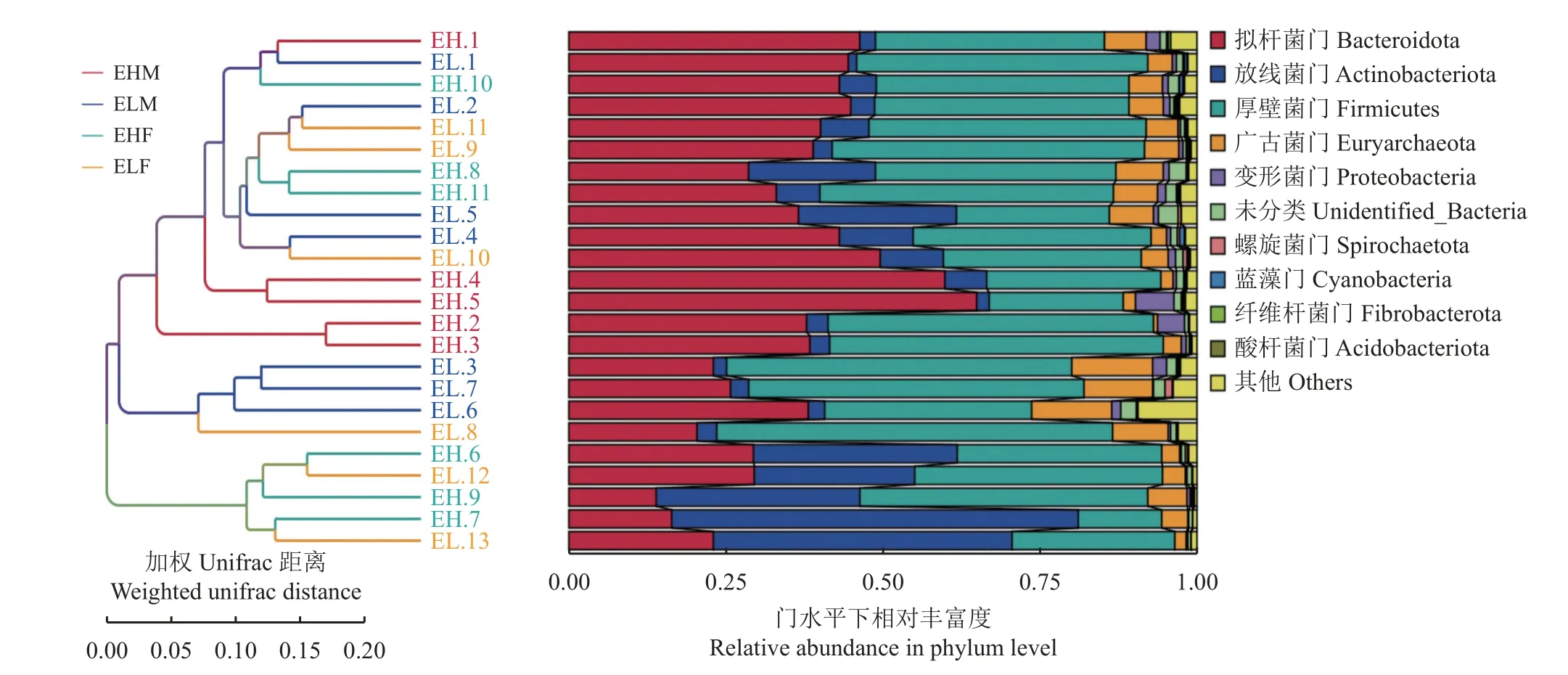
图5 不同RFI 组微生物群落UPGMA 图Figure 5 UPGMA of microbial communities in different RFI Tan sheep groups
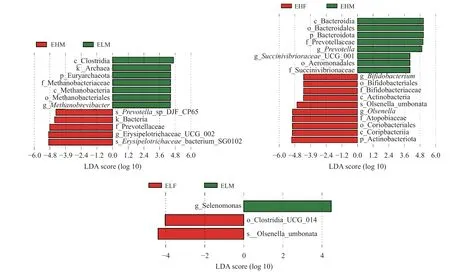
图6 不同RFI 组滩羊LEfSe 功能富集分析Figure 6 LEfSe function enrichment analysis of Tidal sheep in different RFI groups
3 讨论
本研究选择较大的滩羊群体,具有较高的生物学重复性,并且选择公母羊两类群体作为研究对象,探究性别差异差异对瘤胃微生物的影响。Zhang等[14]在湖羊饲料效率的研究中采用单栏饲喂的方式,而这无疑忽略了动物运动对机体造成的影响。研究发现,肉牛身体活动量与RFI 表型相关系数高达0.32,其中10%的RFI 变异由身体活动所导致,这些活动包括采食、反刍以及物理性运动[15]。在动物不同生长阶段,性别对生物发育速度具有较大的影响,动物生长初期雌性动物的生长发育速度较快,而在生物生长发育后期,雄性动物的生长发育速度则较快[16-17],因此动物性别也可能会对饲料效率有影响。
3.1 性别差异对微生物菌群的影响
近年来发现性别是影响机体代谢的重要因素。Verschuren 等[18]报道了性别是影响不同饲料效率猪肠道微生物的重要影响因素。Ahmed 等[3]报道了安格斯牛性别差异对饲料效率的影响,研究发现性别对不同RFI 动物生长性能和瘤胃微生物无显著影响,但对瘤胃上皮基因表达差异显著。在本研究中,极低组母羊和公羊PD_whole_tree 差异明显,而其他公母滩羊性别对不同RFI 瘤胃微生物菌群α 多样性无显著影响。通过不同性别RFI 滩羊PCoA 和NMDS 图分析发现,各组别差异较小,β 多样性差异不显著。α 和β 多样性指数是反映样本微生物丰富度和均匀度的两个重要指标。这表明性别对微生物生长影响较小。从微生物组成来看,在门水平上,不同性别组别间优势菌群种类一致,极高组公母羊拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)和变形菌门(Proteobacteria)差异显著。在属水平上,公母羊间第1 优势菌属发生变化,Olsenella菌属为母羊第1 菌属,普雷沃氏菌属(Prevotella)为公羊第1 优势菌属。极高组公母羊Olsenella、普雷沃氏菌属和韦荣球菌属UGG-002 (Erysipelotrichaceae_UCG_002)差异显著。极低组公母羊月单胞菌属(selenomonas)差异显著。韩学平等[19]研究了不同性别间牦牛瘤胃微生物多样性,发现公母牦牛间微生物组成在种属间组成一致,而在本研究中发现,不同RFI 母滩羊在属水平下优势种属发生了变化。因此Olsenella菌属可能与母羊RFI 有关,不同RFI 公母羊具有不同的优势,可能影响瘤胃微生物的不同发酵方式。
3.2 不同RFI 微生物菌群差异
瘤胃是反刍动物的主要消化器官,栖居大量的微生物,并具有复杂的微生物生态系统。研究发现,微生物菌群的变化直接影响动物瘤胃发酵和营养物质的吸收,进而导致不同饲料效率的差异[20-21]。为此,对不同RFI 滩羊瘤胃微生物进行了研究。本研究选择16S V3-V4,341F-806R 区域,分析瘤胃内微生物多样性。
本研究中,序列有效读取率为98%,数据具有较高的完整性。其中极低RFI 公羊具有较高的OTUs 数目,公羊极高组(1 812)极低组(1 610) OTUs数目相差较大,极低组高于极高组,而母羊极高极低组OTUs 数目差异不显著,极高组高于极低组。马万浩等[22]报道发现,不同RFI 湖羊公羊中OTUs数目高RFI 组(394)高于低RFI 组(353)。造成这种差异的因素有很多,例如精粗比,随着精料上升,菌群数量下降,也可能是动物品种和饲喂环境的原因。α 和 β 多样性指数是研究微生物多样性的两个重要参数。本研究发现,α 多样性中物种数、Chao1、ACE 值、Shannon 指数、Simpson 指数和覆盖率等参数在4 个群体间没有差异,其中只有PD_whole_tree在极高和极低RFI 公羊中差异显著,通过柱状图、PCoA 图和UPGMA 等没有聚类和分离。这与马万浩等[22]研究结果一致,同样与Myer 等[23]通过肉牛平均日采食量和动物体重来评估饲料效率研究结果一致,所有样本和类群发现观察到的OTUs、丰富度(Chao1)或多样性没有差异。这表明每个类群内微生物群落之间的相似性。
在门分类学水平上,4 个组别具有相同的优势菌门,分别是拟杆菌门(Bacteroidetes)和厚壁菌门(Firmicutes),拟杆菌门差异显著,这与众多研究结果一致[24-26],其优势菌门差异的变化主要发生在厚壁菌 门 (Ruminococcus、Lachnospira、Lachnospira_NK3A20、Erysipelotrichaceae_UCG-002、Quinella、Selenomonas菌属中),Myer 等[23]恰好证明厚壁菌门的增加与ADFI 较高的动物有关,这可能与饲料效率有关。Erysipelotrichaceae_UCG-002 和Selenomonas主要参与发酵代谢类型,参与丙酮酸、乳酸发酵和葡萄糖发酵主要产生乙酸和丙酸,为机体供能。在本研究结果中极高和极低母羊组中具有较高的放线 菌 门 中 的 菌 属 (Olsenella、Bifidobacterium),Bifidobacterium可以对葡萄糖进行异型乳酸发酵,生成L ( + )乳酸和乙酸(摩尔比为3 ∶ 2),不产CO2,经常作为益生菌的调节剂[27]。Perea 等[28]的研究表明,不同RFI 的阉羊在瘤胃内纤维杆菌、瘤胃球菌、糖酵母菌、双歧杆菌、变形杆菌存在显著差异。占今舜等[29]报道,随着精料比例增加瘤胃中放线菌门数量增加。研究报道,拟杆菌门丰度的高低可能与高纤维饲粮有关,拟杆菌门又是降解碳水化合物的组要微生物群[30]。本研究中,极高组公羊与极高组母羊拟杆菌门差异显著,高RFI 公羊具有较高的拟杆菌门,因此高RFI 公羊具有较高消化纤维类饲料能力。在公羊不同RFI 表型中广古菌门(Euryarchaeota)中的甲烷短杆菌(Methanobrevibacter)和变形菌门差异显著。广古菌门在动物胃肠道中主要以产甲烷菌的形式存在,Methanobrevibacter通常将无机或有机化合物厌氧发酵转化成甲烷和二氧化碳排放,造成巨大的能量损失和环境污染。众多研究发现,甲烷排放与饲料效率有关[31-33],Ellison 等[34]发现采用青贮料饲喂羔羊时,Methanobrevibactr在低RFI 羊瘤胃中丰度更高,这与本研究结果一致。然而,也有研究报道,瘤胃内产甲烷菌的丰度在高RFI 动物中更高,这似乎更符合理论事实[35]。造成这种原因的结果可能与其他菌群相互作用有关,董利锋等[36]报道,Geobacterspp的存在能够直接通过电子转移与产甲烷菌形成协同作用,减少有机物发酵产物的积累而促进产甲烷菌的定植。Geobacterspp是属于变形菌门Geobacteraceae 科的微生物,恰好在本研究结果中发现, 两种变形菌门菌属Olsenella、Bifidobacterium,并 且Olsenella和Bifidobacterium在极高RFI 母羊中的丰度均高于极低RFI 母羊,因此Olsenella和Bifidobacterium具 有 与Geobacterspp类似的作用。
4 结论
性别对极高或极低RFI 个体瘤胃微生物有显著影响,Olsenella菌属为母羊第1 菌属,普雷沃氏菌属(Prevotella)为公羊第1 优势菌属异,但不同RFI滩羊群体间瘤胃菌群多样化和丰富度相似,瘤胃微生态环境相对稳定。优势菌群的种类和丰度在不同RFI 组滩羊瘤胃微生物区系中基本一致,厚壁菌门菌群数量的增加可能与RFI 有关;不同RFI 公羊组广古菌门中的Methanobrevibacter 菌群在低RFI 羊瘤胃中丰富度更高,不同RFI 母羊组微生物多样性差异不显著。

