中间偃麦草全基因组的选择
郭 鹏, 曹 晟
(天津农学院计算机与信息工程学院,天津 300384)
中间偃麦草[(Host) Nevski]原产于东欧,是一种根系发达、抗寒性强、耐旱、耐盐、适应性广、再生性好的优良牧草。作为多年生牧草,中间偃麦草产量高,已经在畜牧业发达的国家得到了广泛应用。中间偃麦草的果实可食用,其谷物制成的食品在美国某些市场上已有销售。此外,中间偃麦草对小麦锈病、白粉病等病害免疫,对黑穗病、叶枯病和根腐病等病害具有高抗性,能与小麦杂交,已经成为小麦遗传改良中具有重要利用价值的野生亲本之一。李振声院士获得了偃麦草与小麦的杂交种子,并将其广泛用作与小麦远缘杂交的野生亲本材料,通过杂交将其优良基因转移进小麦,已经成为小麦遗传改良的有效途径。
黑龙江省分子细胞遗传与遗传育种重点实验室研究团队对中间偃麦草种质资源在直接驯化改良、远缘杂交改良等方面进行了研究,并对利用基因组学育种技术进行中间偃麦草遗传育种研究的重点和选育策略进行了展望。全基因组选择使用单核苷酸多态性(single nucleotide polymorphism,SNP)标记信息和表型特征值估计候选个体育种值,根据育种值的大小对候选个体进行筛选。作为育种领域的新兴技术,与传统的仅使用个体表型的估计育种值相比,全基因组选择具有准确度更高、世代间隔短、育种效率高等优点,目前已经被越来越多地应用在农作物的育种领域。
在国外,Zhang等利用基因组选择技术构建了基因标记数为3 883个、大小为1 126个的育种群体,并用不同的基因组选择方法进行了研究,证明了使用基因组选择技术进行中间偃麦草遗传育种的可行性。Bajgain等使用全基因组关联分析寻找中间偃麦草表型发育相关的基因座,利用显著位点作为固定效应预测育种值,使用rrBLUP在SNP标记数为8 899个的数据集中进行育种值的估计,结果表明,该方法可使预测能力提高14%。考虑到环境因素对表型发育的影响,Bajgain等此后使用GBLUP、贝叶斯方法研究加性、显性效应以及环境效应对中间偃麦草性状发育的影响,在标记数为 8 899 个、群体大小为451个的基因数据集中进行了穗质量、穗长、每个花序小穗数、产量等9种性状的全基因组选择研究。Crain等研究了中间偃麦草 3 658 个个体的46个性状,对18 357个SNP标记的基因组数据使用GBLUP方法进行了研究,发现预测能力随着训练群体规模、标记数量的增加而提高。
本研究对标记数为23 495个、研究群体大小为5 521个的中间偃麦草数据采用随机方式抽取个体构建训练群体和校验群体,使用交叉验证的方式计算候选育种个体全基因组估计育种值和育种准确度,对比GBLUP、BayesA、BayesB、BayesCπ法的基因组育种值估计结果,以期找到适合中间偃麦草性状育种分析的全基因组选择方法来提高育种效率,为中间偃麦草全基因组育种提供参考。
1 材料与方法
本研究所用数据是从美国国立生物技术信息中心(NCBI)网站(https://www.ncbi.nlm.nih.gov/bioproject/)上下载的美国堪萨斯州萨利纳市土地研究所的中间偃麦草的公共数据。在试验中选择中间偃麦草的自由脱粒率、穗产量、株高、种子质量、每个花序小穗数、穗长、落粒性等7种性状进行研究,研究群体包含5 521个个体,基因组数据包括23 495个SNP标记位点。
生物育种中的遗传力也称遗传率,表示遗传因素对表型发育所起作用的大小。由于表型数据中存在表型值缺失或无效的情况,因此需要对数据进行筛选,经过筛选后具有有效表型数据的7种性状数据值的平均值、标准差、最大值和最小值的统计结果及对应的遗传力如表1所示,其中自由脱粒、穗产量、株高、种子质量、每个花序小穗数、穗长、落粒性具有有效表型数据和SNP标记数据的群体中的个体数分别为5 019、5 230、5 488、4 703、5 013、4 512、4 694个。
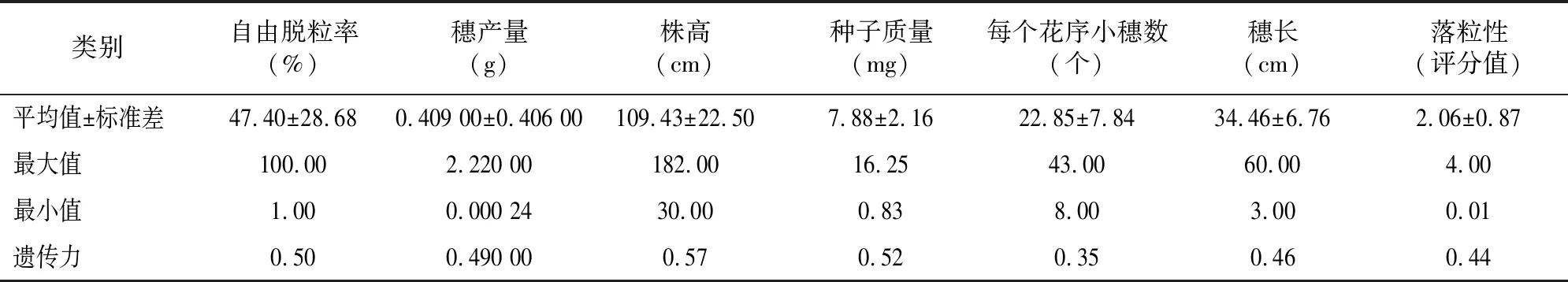
表1 表型数据及遗传力
1.1 统计模型
本研究选用的统计模型如下所示,

式中:为个体的性状表型值;为标记的数量;为平均表型值;为第个分子标记的效应值;为个体第个标记的基因型编码(编码符号为0、1或2);为个体的随机残差效应值。
1.2 BayesA
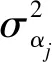
1.3 BayesB
BayesB假设只有部分位点效应能够影响到遗传效果,在所有位点的遗传方差分布中,只有少数位点具有遗传方差。因此,在BayesB中使用参数控制位点是否具有标记效应。
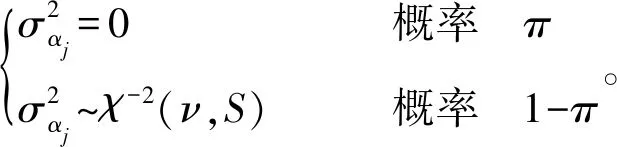
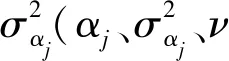
1.4 BayesCπ
BayesCπ假设所有位点具有1个公共方差,而不是每个位点都有自己的方差,并且假设控制标记效应为0的参数()未知。公共方差符合自由度=42、缩放因子为的尺度逆卡方分布,其中的推导过程与BayesB中缩放因子()的推导过程相同。概率()未知,并且假设符合(0,1)间的均匀分布,符合概率为(1-)的SNP效应由多元分布的混合模型计算得到,标记效应值也是使用正态分布取样的方式进行估计的。
春秋末期,宗法制逐步被破坏,导致天子与各诸侯国的关系也发生了重大变化。天子作为天下共主的地位和权威进一步丧失。“春秋时犹严祭祀,重聘享,而七国则无其事矣;春秋时,犹论宗姓氏族,而七国则无一言及之矣;春秋时,犹宴会赋诗,而七国则不闻矣;春秋时犹赴告策书,而七国则无有矣。邦无定交,土无定主。”[23]P715宗法、会盟、祭祀、等诸多方面都发生了重大变化。
1.5 GBLUP
在GBLUP中,基于系谱的亲缘关系矩阵由遗传关系矩阵(G阵)代替,然后使用最佳线性无偏估计计算育种值。G矩阵的公式如下:
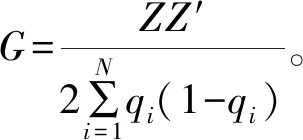
式中:为标记的数量;为标记的等位基因频率;为SNP标记效应的中心关联矩阵。
1.6 GEBV计算
全基因组估计育种值(genomic estimated breeding value,Gebv)使用如下公式计算:

式中:为个体的基因组估计育种值。
1.7 交叉验证
本研究选用随机掩蔽交叉验证方法将中间偃麦草的表型数据、基因型数据分成校验集合和训练集合,使用5倍交叉验证方法对群体进行分析。每次随机抽取约1/5个体构成校验集合,剩余的个体构成训练集合,具体分组见表2。对于每种性状,重复基因组育种值估计10次,以10次估计结果的平均准确度作为全基因组估计的准确度。

表2 训练群体和校验群体分组情况
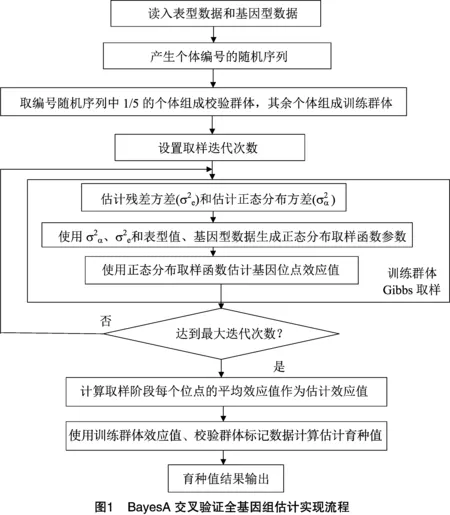
数据分组后,使用训练群体中的表型数据、基因型数据估计位点效应值,以BayesA为例,交叉验证全基因组估计实现流程如图1所示。验证阶段使用估计的效应值,校验群体中的基因型编码计算校验群体中个体的育种值。
1.8 估计准确性标准
将全基因组估计所得育种值与表型数据求相关系数,作为评判标准,计算公式:
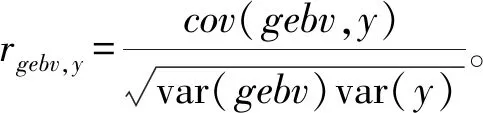
2 结果与分析
2.1 试验环境与试验相关参数
本研究的环境:AMD RyZen 5 1600六核(12线程)中央处理器(CPU),32 G DDR4 2 666 MHz内存,希捷2 TB(1万转)硬盘;中文版64位Windows 7操作系统。
在试验中,BayesA、BayesB、BayesCπ中的burnin和总取样运行次数分别设置成400 0、20 000。=4012、=0.002 0作为BayesA中的先验分布参数;=4.2作为BayesCπ中的先验分布参数,=4234、=0.042 9作为BayesB中的先验分布参数,在BayesB、BayesCπ算法中,有效基因位占比()=0.01。
2.2 估计准确度
本试验采用5倍交叉验证,经过10次运行后,计算每次运行产生的估计准确度。由于训练群体和校验群体的构建采用随机抽取的方式选择个体,不同个体的SNP标记数据和表型数据存在差异,使得每次最终估计的育种值准确度存在差异,因此选用育种值估计准确度的“均值±标准偏差”作为最终估计结果。由7种中间偃麦草全基因组育种值估计的准确度(表3)可以看出,GBLUP预测的准确度最低,标准偏差也最低,说明GBLUP的性能最稳定。在用BayesB方法得出的结果中,4个最优,1个次优;在用BayesCπ方法得出的结果中,2个最优,2个次优;在用BayesA方法得出的结果中,1个最优,3个次优;在用GBLUP方法得出的结果中,1个次优。总体而言,BayesB方法的估计准确度表现最优异。
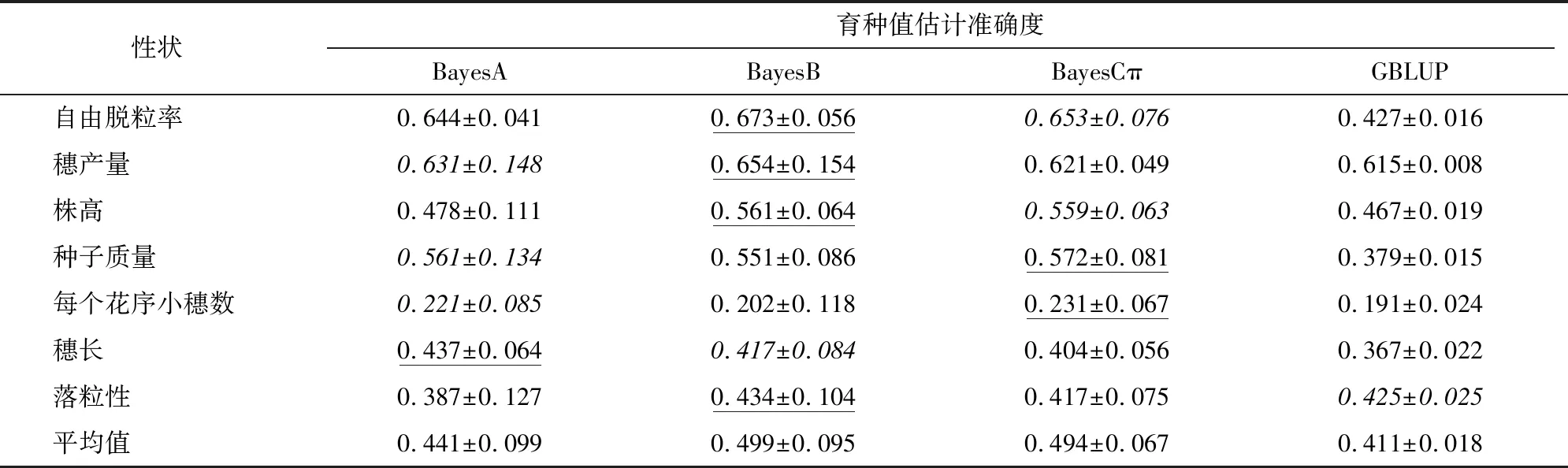
表3 全基因组育种值估计的准确度
2.3 运行时间
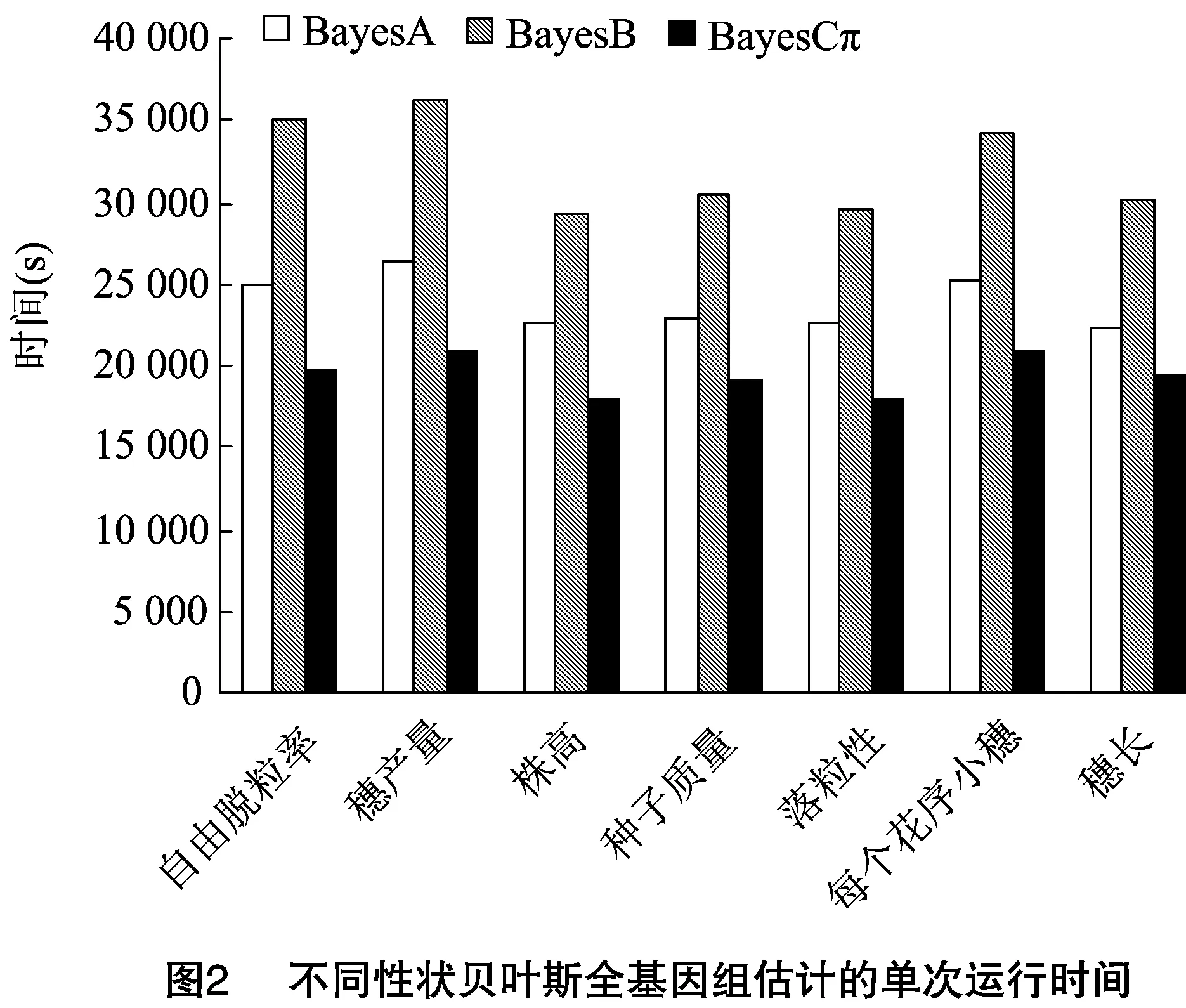
在10次交叉验证运行后,产生的10次运行时间如表4所示。可以看出,同一性状的3种贝叶斯方法的运行时间均高于GBLUP法的运行时间,而且3种贝叶斯方法的运行时间也有差异,排序为BayesB>BayesA>BayesCπ。

表4 不同方法的运行时间
在分析单次贝叶斯全基因组估计的运行时间时,选择7种性状的第1次运行时间进行比较。如图2所示,BayesA法的运行时间大约是BayesB法的75%,BayesCπ法的运行时间大约是BayesB法的60%。
2.4 中间偃麦草性状全基因组估计育种值
由于不同全基因组选择方法估计的中间偃麦草育种值不同,因此选择10个不同个体,使用对各性状估计精度最高的方法计算全基因组估计育种值,结果见表5。在育种实践中,不同性状的表型值间存在较大差异,使得估计所得育种值在数量级上的差异较大。因此在育种过程中可以针对某一种性状进行个体的选择,也可以针对所有性状对候选个体进行取舍。在对所有研究性状的估计育种值进行选择时,考虑到不同育种值间数量级的差异,通常情况下对不同性状赋予不同的权重,采用加权求和的方式或者根据综合公式计算综合育种值,再根据综合育种值筛选候选个体。
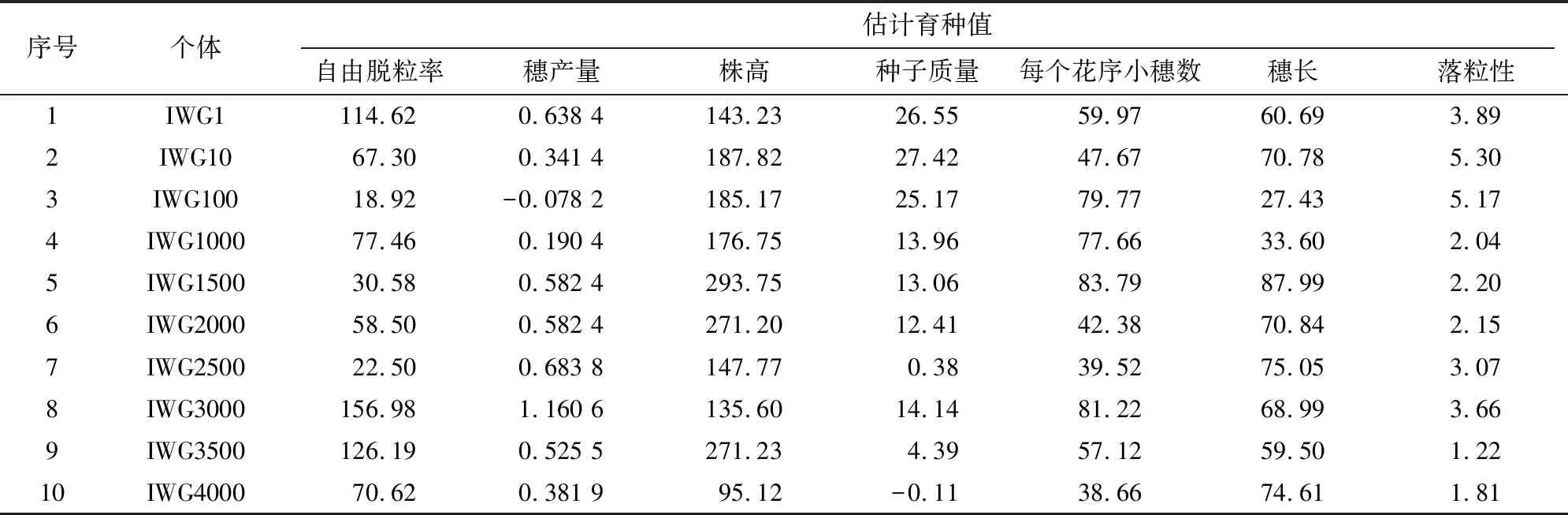
表5 中间偃麦草不同性状全基因组的估计育种值
3 讨论
育种过程的关键环节是估计育种值,作为新兴的育种技术,全基因组选择在育种值估计准确度、缩短育种世代间隔、遗传力低的性状的选育中表现出了优异性能。中间偃麦草作为一种优良的牧草,对小麦病害具有高抗性,甚至具有免疫性,是杂交小麦重要的野生亲本,进行中间偃麦草的全基因组育种研究既能促进优良牧草的改良,又能为杂交小麦育种提供重要的遗传亲本,具有重要的实用价值。
本试验对中间偃麦草自由脱粒率、穗产量、株高、种子质量、每个花序小穗数、穗长、落粒性7种性状的全基因组进行了选择性分析。结果显示,从育种值估计准确度上看,BayesB法对自由脱粒率、穗产量、株高、落粒性4种性状的估计准确度最高;BayesCπ法对种子质量、每个花序小穗数的估计准确度最高;BayesA法对穗长性状的估计准确度最高。究其原因,全基因组选择育种值估计的准确度受到群体大小、全基因组选择方法、遗传力、种群数据等因素的影响,很少有某种方法能够保证在所有性状育种值的估计中都能获得最优的准确度。
GBLUP使用遗传关系矩阵代替亲缘关系矩阵,利用最佳线性无偏估计方法估计育种值,相校于传统的基于表型的BLUP方法,更能反映个体发育过程中性状的遗传性能,在保持BLUP较短运行时间的情况下,准确性更高,是当前动植物育种领域全基因组选择的重要工具。GBLUP假设所有位点都具有遗传效应,并未考虑单个SNP标记效应,在估计过程中不会出现由于随机取样产生的偶然性因素,因此性能稳定,中间偃麦草7种性状的全基因组估计结果的偏差最小。
贝叶斯方法在基因组选择过程中求解每个SNP位点的效应值,更能反映基因位点对遗传影响的真实情况。BayesA假设所有位点对性状发育产生遗传效应,BayesB、BayesCπ假设只有少量基因位点可以影响性状的发育,其余的位点效应可以忽略不计,从目前全基因组关联分析的结果可以看出,少量显著位点效应影响性状的发育更能反映遗传效果。BayesB假设每个位点都具有自己的方差,BayesCπ假设所有位点都有1个公共的方差,BayesB更能反映SNP效应的真实情况,因此在中间偃麦草7种性状全基因组育种值的估计精度中,BayesB法得出的最优结果最多。
就运行时间而言,使用SNP标记数据计算遗传关系矩阵占据了GBLUP绝大部分运行时间,在计算机运行环境相同的情况下,全基因组选择群体大小、个体SNP标记数量决定了基因型数据,进而主导着GBLUP的运行时间。BayesA、BayesB、BayesCπ全基因组选择方法假设SNP标记效应值符合正态分布,该正态分布的参数符合尺度逆卡方分布,通过随机取样的方式从尺度逆卡方分布中产生正态分布函数的参数,然后使用马尔科夫链蒙特卡罗(Markov chain Monte Carlo,MCMC)取样技术(BayesA使用Gibbs取样,BayesB、BayesCπ使用Metropolis Hastings取样),利用正态分布取样函数产生标记效应值。MCMC的运行过程包括burn-in训练阶段和sampling取样阶段,为了消除取样偶然性的影响,burn-in阶段、sampling阶段的迭代次数通常设成较大的值,贝叶斯全基因组选择方法属于高度密集型计算任务,往往需要较长的运行时间。BayesCπ假设所有位点具有1个公共的方差,BayesB、BayesA假设每个位点具有自己的方差,需要为每个位点估计正态分布方差,而且解决相同的问题,Metropolis Hastings取样运行时间比Gibbs 取样运行时间更长,因此在中间偃麦草群基因组选择中,GBLUP的运行时间最短。在3种贝叶斯方法中,BayesB的运行时间最长,BayesCπ的运行时间最短。贝叶斯方法的全基因组选择准确度高,但漫长的运行时间是育种实践应用过程中的不利因素,使用更高主频的高性能处理器结合并行计算技术缩短运行时间,将有助于提高贝叶斯方法全基因组选择技术的实用性。
4 结论
本研究使用GBLUP、BayesA、BayesB和BayesCπ进行中间燕麦草的自由脱粒率、穗产量、株高、种子质量、每个花序小穗数、穗长、落粒性7种性状的全基因组选择,经过5倍的交叉验证发现,在4种方法育种值的估计结果中,BayesB的准确度最优,运行时间最长。进行中间偃麦草全基因组的选择需要针对不同性状选择最优的全基因组选择方法。在育种实践中,综合考虑不同方法育种性状估计准确度和算法运行时间才能在中间偃麦草育种中高效地利用全基因组选择技术,提高育种效率。

