玉米NRL基因家族鉴定与逆境表达分析
赵长江,都梦翔,宋巨奇,徐尚缘, 贺 琳,徐晶宇,杨克军,李佐同
(1.黑龙江八一农垦大学 农学院,黑龙江 大庆 163319;2.农业农村部东北平原农业绿色低碳重点实验室,黑龙江 大庆 163319; 3.黑龙江省秸秆资源化利用工程技术研究中心,黑龙江 大庆 163319;4.黑龙江省现代农业栽培技术 与作物种质改良重点实验室,黑龙江 大庆 163319)
NRL(NPH3/RPT2-Like)是植物所特有一类的蛋白家族,一般包含3个典型结构域:N端的BTB结构域、C端的螺旋-螺旋结构域和居中NPH3结构域[1],所有成员都具有NPH3结构域[2]。其中NPH3(NONPHOTOTROPIC HYPOCOTYL3)和 RPT2(ROOT PHOTOTROPISM2)于1999年首先被报道参与拟南芥(Arabidopsisthaliana)幼苗对蓝光的向光性调节[3]。陆续有研究指出,NPH3在基于横向生长素梯度产生的向光弯曲及叶片定位和伸展调控中发挥决定作用,RPT2作为向光反应转换器通过改变NPH3的定位和磷酸化状态来调节向光反应[4-5]。NRL家族成员可与向光素(Phototropin)蓝光受体协调实现对植物向光性、叶绿体积累运动、叶片定位和伸展等生物学功能的调控[6-7]。根据NRL家族成员在向光素介导光调控生理活动的参与程度,可将其分为两类:NRL依赖和NRL独立的光信号途径[6]。其中,向光性、叶绿体积累、叶柄定位和叶片伸展是NRL依赖的,而叶绿体回避运动和气孔开放是NRL独立的。上述研究表明,NRL家族蛋白作为向光素受体信号通路的重要组分,其作用功能应该具有多样性,或是具有明确分工。
通过对拟南芥NRL家族蛋白功能的研究发现,该家族成员除参与的蓝光生理调控外,还在植物生长发育调控、植物抗病性方面发挥重要作用[6]。其中,生长发育调控主要涉及花粉萌发、花粉管生长[8]、叶脉结构图案形成[9]以及根重力应答[10]等。不同寄主植物中该家族成员免疫表现不同,例如AtNRL31与钙信号通路转录因子AtSR1(SIGNAL RESPONSIVE1)互作导致其泛素化降解参与寄主防卫反应调控,其中nrl31突变体拟南芥对丁香假单胞菌(Pseudomonassyringae)敏感[11]。同时,马铃薯(Solanumtuberosum)StNRL1作为致病疫霉菌(Phytophthorainfestans)致病效应子的靶标蛋白,该基因诱导沉默烟草降低了病菌定植及其引起的细胞死亡,增强了寄主的抗病性[12]。当然,NRL基因抗病表现的不同可能与植物或是植物-病原物特异系统不同有关,或是与借助光反应调节不同有关,仍有待深入研究。
目前,NRL基因家族在拟南芥[6]、水稻(Oryzasativa)[13]和番茄(Lycopersiconesculentum)[14]等模式植物中被鉴定出来,但高光效作物玉米NRL基因家族的研究尚未见报道。玉米(Zeamays)作为世界上种植最多的谷物作物,其籽粒和秸秆都具有良好的经济价值[15]。对玉米NRL家族基因研究具有重要的理论和实践意义。本研究采用生物信息学方法在玉米全基因组水平鉴定NRL家族成员并对其进化关系和组织表达进行分析,同时采用实时荧光定量PCR对其逆境表达进行分析,旨在为揭示玉米NRL基因功能提供理论依据。
1 材料和方法
1.1 试验材料
选用玉米自交系B73为试验材料,用10%次氯酸钠对精选的种子消毒30 min,用蒸馏水清洗至无味,然后浸泡6~8 h,将种子放在22 ℃培养箱中暗培养24 h催芽,挑选萌发1.5 cm左右根的种子置于漂浮泡沫板孔洞中,使用1/2 Hoagland营养液培养,并在人工气候室(22 ℃,16 h光/8 h暗)中对培养至两叶一心期的玉米幼苗进行处理。其中,200 mmol/L NaCl和20% PEG6000通过营养液添加实现,35 ℃高温通过光照培养箱调温实现,通过牙签嵌入法实现立枯丝核菌AG1-IA(四川农业大学,郑爱萍教授惠赠)接种处理。上述处理均于处理48 h取样,液氮速冻后存放于-80 ℃冰箱。每个处理3次生物学重复。
1.2 基因家族成员鉴定
从phytozome(https://phytozome.jgi.doe.gov/pz/portal.html)中下载玉米fasta和gff3文件,使用TBtools[16]对fasta和gff3文件分别进行转化,生成cds文件和pep文件。使用拟南芥和水稻的NRL家族基因[6,13]与玉米蛋白序列在TBtools上进行比对,用NCBI(https://blast.ncbi.nlm.nih.gov/Blast.cgi)、SMART[17](http://smart.embl-heidelberg.de/)以及Pfam[18-19]进行结构域的确认,去除预测结构域长度小于1/2个NPH3结构域或 BTB 结构域长度的基因。
1.3 蛋白相关分析
利用在线工具ExPASy(https://web.expasy.org)分析蛋白质的理化性质,利用WoLF PSORT(https://wolfpsort.hgc.jp/)进行亚细胞定位预测。分别从Tair(https://www.arabidopsis.org/)和RGAP(http://rice.plantbiology.msu.edu/)下载已知拟南芥和水稻的蛋白序列,从phytozome中下载高粱的蛋白序列,从茄科基因组库(https://solgenomics.net/)中下载番茄的蛋白序列,用于构建系统进化树。使用MEGAX软件[20]最大似然法(Maximum likelihood)建树,选择最适模型(JTT+G+I+F),Bootstrap 设置为1 000。
使用MEME(https://meme-suite.org/meme/tools/meme)分析蛋白保守Motif,供试参数分别为:最大Motif 数设置为10,位点分布选择任何重复数分布,最佳Motif宽度设置为≥6 和≤50,并使用TBtools[16]绘制玉米基因结构和保守基序图。采用SWISS-MODEL(https://swissmodel.expasy.org/interactive)预测蛋白三级结构。
1.4 基因相关分析
利用TBtools[16]绘制基因在染色体上的分布图,使用MCScanX软件[21]对基因复制事件进行分析,使用Circle Gene View绘制出串联重复基因图,并利用Ka/Ks Calculator 功能计算基因的 Ka/Ks 值。使用TBtools软件分析基因内含子-外显子结构,同时取编码区上游2 000 bp的序列作为基因启动子,并提交PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)鉴定顺式作用元件,分析统计顺式作用元件类型、数量和分布。
1.5 全生育期组织表达谱及共表达网络分析
从GEO数据库中下载GSE50191转录组表达谱数据,选取23个组织部位的表达数据,使用TBtools[16]的HeatMap功能进行热图绘制。使用OneStep WGCNA工具进行加权共表达网络构建,对样本数据进行筛选,保留含有8个及以上样本且FPKM值>1的基因数据,计算合适的加权系数β值,构建无尺度网络与划分相关模块。使用Pannzer 2(http://ekhidna2.biocenter.helsinki.fi/sanspanz/)对玉米基因组进行GO注释,使用TBtools对关键模块进行GO富集分析,以玉米基因组为参考数据库,并使用Enrichment Bar Plot对GO富集结果可视化。
1.6 RNA提取与实时荧光定量PCR
称取0.1 g玉米叶片,液氮研磨后用TRIzol试剂(Invitrogen)提取总RNA。使用1%琼脂糖凝胶电泳进行验证,同时使用NanoDrop one(Thermo ScientificTM)测定RNA浓度。使用反转录试剂盒 FSK-101(TOYOBO)对RNA进行反转录,使用Talent qPCR PreMix(SYBR Green)(天根)进行qRT-PCR反应。使用β-Actin作为内参基因,扩增基因引物序列信息见表1,基因引物由Invitrogen公司合成。使用CFX96荧光定量PCR仪(Bio-Rad)进行扩增,每个反应重复3次,表达数据使用2-ΔΔCt方法[22]进行数据分析。

表1 qRT-PCR引物序列信息Tab.1 Primers for qRT-PCR
2 结果与分析
2.1 玉米NRL家族成员鉴定
经Pfam和Smart结构域鉴定,在玉米全基因组水平上共鉴定31个ZmNRL基因(表2),不均匀地分布在9条染色体上,根据在染色体上的位置依次命名为ZmNRL1~ZmNRL31。其中,2号染色体分布最多,有7个基因。玉米NRL家族基因的CDS序列长度在1 395~2 250 bp,编码的氨基酸序列长度在 464~749个,蛋白质的相对分子质量在48.64~80.02 ku,理论等电点在4.97~10.04。其中12个玉米NRL蛋白理论等电点小于7,为酸性蛋白。玉米NRL蛋白被预测具有叶绿体、细胞核和细胞质等亚细胞定位。

表2 ZmNRL编码蛋白序列理化性质分析Tab.2 Physiochemical property of ZmNRLs gene and their coding protein
2.2 玉米NRL家族成员进化及物种间共线性分析
对包含玉米在内的5个物种144个NRL基因进行系统进化分析(图1),包括拟南芥33个、番茄27个、水稻27个和高粱26个基因。蛋白进化分析可将NRL家族成员划分为4组(Ⅰ~Ⅳ),分别有23,29,30,62个成员。其中Ⅰ组4个(ZmNRL7、ZmNRL13、ZmNRL25、ZmNRL26)成员,Ⅱ组6个(ZmNRL4、ZmNRL6、 ZmNRL8、ZmNRL16、ZmNRL18、ZmNRL22)成员,Ⅲ组5个(ZmNRL1、ZmNRL11、ZmNRL28、ZmNRL30、ZmNRL31)成员,Ⅳ组16个(ZmNRL10、ZmNRL14、ZmNRL15、ZmNRL20、ZmNRL2、ZmNRL3、ZmNRL5、ZmNRL9、ZmNRL12、ZmNRL17、ZmNRL19、ZmNRL21、ZmNRL23、ZmNRL24、ZmNRL27、ZmNRL29)成员。在各个组中可以发现单子叶植物多聚合在同一个分支,而双子叶植物常聚合在一起;而且4个亚组中都同时包含有单子叶植物和双子叶植物,推断在单子叶与双子叶植物分离之前,就出现了NRL基因的共同祖先。
31个玉米NRL基因不均匀地分布在其9条染色体上(图2)。玉米种内有4对片段重复基因和1对串联重复基因。4对重复基因分别为ZmNRL5—ZmNRL19、ZmNRL9—ZmNRL21、ZmNRL13—ZmNRL25和ZmNRL17—ZmNRL21,而且它们的Ka/Ks值均小于1,说明它们受到纯化选择;串联基因为ZmNRL30—ZmNRL31。玉米与高粱、玉米与拟南芥之间分别存在36,5对共线性基因,而拟南芥与高粱之间也存在9对共线性基因。

Ⅰ~Ⅳ代表基因所属亚族;玉米、高粱和水稻分别用圆形、菱形和正方形来表示;拟南芥和番茄分别用倒三角形和三角形表示。 Ⅰ—Ⅳ represents the gene subgroup;Zea mays,Sorghum bicolor,and Oryza sativa are represented by circles,diamonds, and squares;Arabidopsis thaliana and Lycopersicon esculentum are represented by inverted triangles and triangles.
2.3 玉米NRL基因结构和蛋白保守基序分析
玉米NRL家族基因结构具有一定的保守性,属于同一个进化分支的成员大多具有相似的基因结构(图3-A)。第Ⅰ和Ⅱ组多数成员(除ZmNRL10和ZmNRL20外)含有4个外显子;第Ⅲ和Ⅳ组分别含有2~3和3~5个外显子。除处在同一进化分支5个(ZmNRL7、ZmNRL13、ZmNRL14、ZmNRL15和ZmNRL25)成员有保守基序的缺失外,多数玉米NRL家族成员拥有10个保守基序(图3-B)。其中,保守基序4、2、7、9和5组成BTB结构域,而保守基序10、6、8、3、1则对应的是NPH3结构域,并对所有成员都具有的NPH3结构域的5个保守基序进行展示(图3-C)。而且,处于同一进化分支上的NRL蛋白三级结构相似。

图中Chr和chr以及数字1~5分别表示玉米、高粱和拟南芥的染色体;红色线条为玉米物种内的片段重复基因对;灰色线条为玉米和高粱的片段重复基因对;蓝色线条为玉米和拟南芥物种间的片段重复基因对;绿色线条为拟南芥和高粱物种间的片段重复基因对;玉米物种内的串联重复基因用红色菱形来标识。
2.4 玉米NRL全生育期表达分析及共表达网络构建
玉米NRL基因(ZmNRL10除外)组织表达模式大致可以分为4类,分别为生育期高表达、中高表达、中低表达和低表达(图4)。其中ZmNRL2、ZmNRL4、ZmNRL24和ZmNRL29在多数组织部位中高表达。而且多数基因在叶片生长、叶片气孔以及叶片对称等组织部位表达较高,说明玉米NRL基因可能参与叶片的生长发育等过程。进而,对生长发育组织部位相关10个样本18 000个基因用于加权基因共表达网络的构建(图5),共得到12个模块,其中MEturquoise模块基因数目最多,为9 118个,占总基因数的1/2以上。
通过质量性状与模块关联分析发现(图6),18个玉米NRL基因可能参与共表达调控,其中MEbrown模块中的NRL基因数目多达6个,分别为ZmNRL4、ZmNRL6、ZmNRL13、ZmNRL16、ZmNRL23和ZmNRL25,且该模块与叶片生长性状的相关性值为0.83。选择含NRL基因数目最多且与叶片生长相关的关键模块MEbrown模块进行后续功能富集分析(图7),图中只显示富集程度最多的前30个GO term。主要涉及质体生物发生和组装(GO:0009657)、叶绿体生物发生和组装(GO:0009658)等生物过程,在叶绿体、质体等细胞中发挥核糖体RNA结合(GO:0019843)分子功能参与对叶片生长发育的调控。
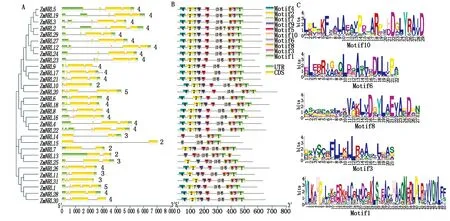
A中数字代表内含子的数量,下方比例尺表示基因碱基数;B下方比例尺表示蛋白长度,CDS为编码区,UTR为非编码区。 The number in A represents intron number,and the lower scale represents gene bases the number;the lower scale of B represents the length of the protein,CDS is the coding region,and UTR is the non-coding region.

图例表示标准化的FPKM值,红色表示高表达水平,蓝色表示低表达水平。 The legend shows the standardized FPKM value, the red shows the high expression level, and the blue shows the low expression level.
2.5 玉米NRL基因启动子顺式作用元件分析
对玉米NRL基因启动子区域的上游 2 000 bp的顺式作用元件进行分析,结果显示,基因启动子主要存在22种已知功能的元件序列,涉及植物激素、胁迫、光响应和生长发育相关的响应元件(图8-A)。其中茉莉酸甲酯响应元件(CGTCA-motif)、脱落酸响应元件(ABRE)、光响应元件(G-box、Sp1)、抗氧化反应元件(ARE)的数量较多,分别为142,115,112,60,54个。ZmNRL25启动子元件数量最多,有41个,ZmNRL7的元件数量最少,仅有12个。结果表明,该家族基因除参与光调控外,可能受茉莉酸和脱落酸等激素调控,甚至与抗氧化等逆境调控密切相关。启动子顺式作用元件分析,推测家族基因可能与脱落酸介导的高温、盐和干旱等非生物胁迫以及茉莉酸介导的立枯丝核菌接种的生物胁迫密切相关。
2.6 玉米NRL基因逆境表达分析
权衡系统进化、组织表达和作用元件情况,选定同为第Ⅳ组组织中低表达的ZmNRL5和ZmNRL12,以及与ZmNRL5重复的组织中高表达ZmNRL19和第Ⅰ组中ZmNRL7(元件数量最少和组织中高表达)
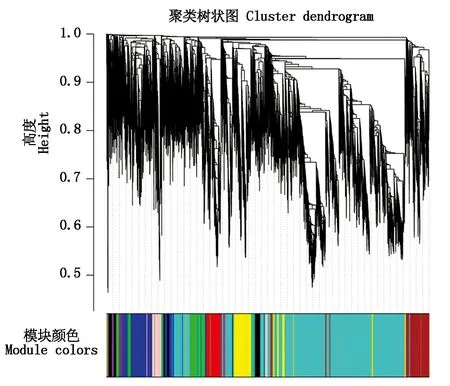
图5 WGCNA聚类树状图Fig.5 WGCNA clustering dendrogram
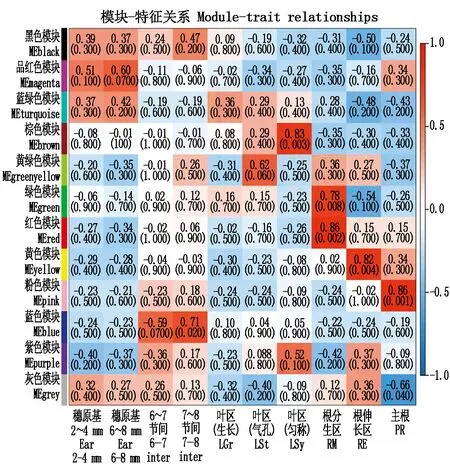
图6 WGCNA模块-特征关系图Fig.6 WGCNA module-feature relationship diagram

图7 MEbrown模块GO富集分析Fig.7 GO enrichment analysis of the MEbrown module
等4个代表基因进行逆境表达分析(图8-B)。供试4个基因中,高温处理下ZmNRL5、ZmNRL12和ZmNRL19上调表达, 同时ZmNRL12在盐胁迫下也表现出上调趋势。其余供试基因在盐、干旱和立枯丝核菌接种均表现为下调。其中,干旱胁迫下供试基因全部下调表达,盐胁迫下ZmNRL5、ZmNRL7和ZmNRL19下调表达,立枯丝核菌接种的生物胁迫下ZmNRL5、ZmNRL12和ZmNRL19下调近5倍,可能与病原物对叶片光合或叶绿体影响比较大有关。

A为顺式作用元件分布图;B为4种胁迫(高温、NaCl、PEG干旱、AG1-IA立枯丝核菌)下基因表达谱。 图中小写字母表示基因在0.05水平差异显著。 A is the distribution map of cis-acting elements;B is the gene expression profile under 4 stresses(high temperature, NaCl,PEG drought, Rhizoctonia solani AG1-IA).Lowercase letters present the significant difference at 0.05 level.
3 结论与讨论
本研究通过生物信息学手段结合实时荧光定量PCR技术,在玉米基因组水平鉴定出31个NRL基因,研究表明,该家族基因不仅具有明显的组织表达特异性,而且可参与生物和非生物逆境应答。本研究中,玉米NRL家族基因数介于拟南芥(33个)[6]和水稻(27个)[13]之间,三者数量较为接近,表明玉米NRL家族基因相比于前二者没有发生较大的扩增。而且,种间基因共线性分析发现,玉米与高粱和拟南芥之间分别存在36,5对共线性关系基因,说明同为C4作物的玉米与高粱之间的关系比拟南芥更近。基因复制是进化的驱动力之一,染色体片段复制和串联复制事件可以扩大基因组中基因家族成员的数量,这些复制促进了新基因的形成和功能分化[23-24]。在共线性分析中,玉米种内NRL家族基因存在4对片段重复基因,复制基因对之间受到纯化选择,表明复制是早期发生的事件。其中,ZmNRL5和ZmNRL19这对片段重复基因在蛋白进化中二者为同一分支,它们的基因结构和蛋白保守结构基本相同,但是它们的生育期组织表达模式不尽相同,可能与复制时的片段位置有关,因为它们的启动子区顺式作用元件也不同,或是与亚细胞定位不同有关;同时这对基因在供试的逆境表达分析中,其表达模式基本一致,启动子区顺式作用元件无法解释这种情况。推测该类基因在生长发育调控中进化基本成熟,逆境应答仍处在早期未分化阶段;或是基因启动子顺式作用元件对基因表达模式决定分析超出笔者的分析范畴,当然不能排除一对基因样本量小代表性不好的原因。
尽管玉米NRL家族基因生育期表达模式类型不同,但是多数基因在叶片生长、叶片气孔以及叶片对称等组织部位表达较高,说明玉米NRL家族基因可能参与叶片的生长发育等过程。例如ZmNRL2、ZmNRL4、ZmNRL24和ZmNRL29为多数组织部位中高表达的一类,可能与它们(ZmNRL2、ZmNRL24和ZmNRL29)处于同一个进化分支有关。这4个基因预测为细胞质或细胞核定位,而不是叶绿体定位。同时,基于生育期组织共表达数据及性状模块功能基因富集分析发现,玉米NRL家族基因参与的叶片生长发育调控主要涉及质体和叶绿体生物发生和组装等生物学过程。推测可能与调控叶绿体积累、叶柄定位和叶片伸展功能的NRL依赖光信号途径密切相关[6]。
通过对玉米NRL家族基因启动子顺式作用元件进行分析发现,光响应元件、茉莉酸和脱落酸反应元件数量较多,推测该家族基因除参与光调控外,可能受茉莉酸和脱落酸等激素调控,或是参与两类激素相关的生物和非生物逆境调控。启动子区域的顺式作用元件的类型、数量及分布基因表达产生不同的调节作用[25]。进而通过qRT-PCR分析ZmNRL5、ZmNRL7、ZmNRL12和ZmNRL19等4个基因在不同逆境下的表达,发现高温处理下仅有1个基因下调表达,除ZmNRL12外,盐、干旱和立枯丝核菌接种每个处理均引起供试3个基因的下调表达,可能与这3种逆境对叶片光合作用或叶绿体影响比较大有关。本研究发现的玉米NRL基因参与对死体营养型病原物的防卫反应,与拟南芥[11]和马铃薯[12]中的研究结果一致,表明NRL基因不仅可参与对不同病原物的广谱抗性反应,而且参与对不同逆境的广谱抗/耐性调节。

