杧果NFU家族基因的克隆、鉴定及其对非生物胁迫的响应分析
李又欢,鲍中奇*,高太平,黄建峰,王力敏,张洪霞,宋志忠,3**
杧果NFU家族基因的克隆、鉴定及其对非生物胁迫的响应分析
李又欢1,鲍中奇1*,高太平2,黄建峰2,王力敏1,张洪霞1,宋志忠1,3**
1. 鲁东大学农林工程研究院/山东省高校作物高产抗逆分子模块育种重点实验室,山东烟台 264025;2. 中国热带农业科学院热带作物品种资源研究所,海南海口 571101;3. 剑桥大学植物系,英国剑桥 CB2 3EA
从二倍体杧果‘桂热82’中克隆和鉴定了4个Fe-S簇装配所需的NFU支架蛋白编码基因,命名为~。MiNFU2和MiNFU3拥有全部3个NFU蛋白典型的Motif基序,MiNFU1缺失Motif 3而MiNFU4缺失Motif 1和Motif 2;MiNFU1~MiNFU3拥有相似的三级结构,而MiNFU4的三级结构与其差异较大,极为独特;13种不同科属植物之间的NFU家族成员数目略有差异,拟南芥、盐芥和短柄草3种一年生草本植物基因组中含有更多的支架蛋白,而非功能性支架蛋白在杧果、草莓、桃、苹果、柑橘和葡萄等多年生果树作物中更易发生丢失;植物NFU同源蛋白在系统发育树上可以分为2个亚家族,且在遗传进化关系上差异较大,同为十字花科、禾本科或蔷薇科植物的NFU同源蛋白分别倾向于紧密聚在一起,杧果NFU蛋白与柑橘和番茄相应的同源蛋白紧密聚在一起;杧果NFU家族基因在不同组织/器官中的表达水平差异显著,在所有检测组织中的整体表达表达水平最高,其次是和,而整体表达水平最低,杧果家族基因均在幼苗叶片中的表达量最高,其次是韧皮部和盛开期花朵,在果实(幼果和熟果)中的表达水平相对较低;杧果NFU家族基因在转录水平对缺铁、高铁毒害、NaCl、PEG和低温处理的响应差异明显,虽然的整体表达最低,但却不受5种非生物胁迫的影响,和对高铁处理最为敏感,其表达水平在‘桂热82’幼苗全身组织均受高铁处理的调控而显著增加。此外,NaCl处理显著增加在根部的表达量,缺铁和PEG处理倾向于降低根部响应的NFU基因的表达水平,而低温处理倾向于降低叶片中响应的NFU基因的表达水平。本研究为明确杧果Fe-S簇装配分子机制提供基因资源,并为解析热带作物果树铁素营养和铁代谢奠定理论基础。
杧果;铁代谢;Fe-S簇装配机制;NFU支架蛋白;非生物胁迫
铁(Fe)作为铁硫蛋白和细胞色素的组成成分,参与多种代谢途径和生命活动,在植物生长、发育、果实品质和产量方面发挥重要作用[1-3]。铁硫蛋白(Fe-S protein)存在于植物质体、线粒体、细胞质和细胞核等亚细胞器官中,参与光合作用、呼吸作用、氮和硫同化、氨基酸和嘌呤代谢、植物激素合成和DNA修复等多种重要的代谢途径和生命过程[4-11]。铁硫簇(Fe-S cluster)是铁硫蛋白的活性部位,其组装过程需要多种功能性蛋白参与[4, 8-9, 11]。
前人研究表明:植物Fe-S簇装配机制是一个高度复杂且协调有序的过程,是植物铁代谢和铁素营养的核心环节,在植物生命过程中不可或缺。一个典型的Fe-S簇装配机制包括2个阶段:第一阶段,铁和硫在支架蛋白上特异结合形成Fe-S簇,第二阶段,Fe-S簇被转移到靶蛋白上,整个装配过程涉及到铁供体、硫供体、支架蛋白及转运蛋白等数十个功能蛋白参与[4-12]。NFU家族基因编码典型的支架蛋白,在其作用下可将硫供体提供的S和铁供体提供的Fe形成Fe-S簇,在拟南芥()中研究较为透彻[4, 9, 13]。拟南芥中共含有5个NFU家族蛋白,其中,AtNFU1、AtNFU2和AtNFU3参与叶绿体Fe-S簇装配机制,而AtNFU4和AtNFU5在线粒体Fe-S簇装配中发挥作用[9, 13];基因被敲除后导致植株矮化,叶片褪绿,Fe-S蛋白活性降低[14-15];拟南芥AtNFU2通过重组可形成一个二聚体并结合一个2Fe–2S簇,该簇在体外可转移到载脂蛋白铁氧还蛋白[16]。
近几年,国内外学者围绕植物Fe-S簇装配机制开展的研究主要集中在拟南芥[4, 9]、水稻()[17]和大豆()[18]等一年生模式植物,果树学中Fe-S簇装配的分子机制尚未清晰,仅在桃[19-20]和葡萄[21]中鉴定了Fe-S簇装配机制相关的基因。然而,热带作物中有关铁素营养和代谢的分子基础研究仍为空白。
杧果()是全球知名的热带果树作物之一,在印度、中国和泰国等国家栽培面积大,深受消费者喜爱[22]。本研究以二倍体‘桂热82’杧果为材料,克隆杧果NFU家族基因并鉴定其生物信息学特征,明确杧果NFU家族基因在杧果不同组织/器官的组织特异性表达特征及其对缺铁、高铁毒害、NaCl、PEG和低温等胁迫的响应差异,为研究杧果Fe-S簇装配和热带作物铁素营养与代谢的分子机制提供基因资源和理论依据。
1 材料与方法
1.1 材料
供试材料为五年生‘桂热82’杧果和一年生嫁接幼苗,均由中国热带农业科学院热带作物品种资源研究所提供。分别采集五年生‘桂热82’杧果一年生新叶(采集时间:2019年4月1日)、新生韧皮部(采集时间:2019年4月1日)、新生根(采集时间:2019年4月1日)、盛开期花朵(采集时间:2019年4月1日,分为花瓣、花粉、花柱和花托)、幼果(采集时间:2019年5月1日)和成熟果实(采集时间:2019年8月1日)等材料,液氮冷冻后用于组织特异性表达特征分析。利用一年生‘桂热82’嫁接幼苗,按照LIANG等[17]和SONG等[19]的方法,以1/2 MS液体培养基为对照,分别进行缺铁、50 mmol/L高铁胁迫、4℃低温胁迫、200 mmol/L NaCl和10% PEG6000(w/V)胁迫,处理1周后分别采集叶片、茎部和根部材料,用于转录水平的表达差异分析。
1.2 方法
1.2.1 杧果NFU家族基因克隆 以拟南芥5个NFU家族基因的氨基酸序列为参考,在WANG等[22]报道的杧果基因组数据中检索杧果NFU家族基因,下载编码区CDS(coding sequence)序列和氨基酸序列,检索结果在Pfam(http://pfam. xfam.org/search)在线软件验证功能结构域。根据CDS序列设计上下游引物,提取‘桂热82’叶片RNA,通过PrimeScriptTMRT reagent Kit 反转录试剂盒(TaKaRa,大连)合成第一链cDNA作为模板,利用Prime STARTMHS DNA聚合酶(TaKaRa,大连)扩增目的基因,送生工生物工程(上海)股份有限公司进行测序验证。
1.2.2 生物信息学分析 通过LIANG等[17]和GAO等[23]的方法,利用ProtParam在线软件(http://expasy.org/tools/protparam.html)分析杧果NFU蛋白的化学公式、分子量、理论等电点(pI)、不稳定性指数(insteadility index)、脂肪系数(aliphatic index)、总平均亲水性GRAVE(grand average of hydropathicity)等理化性质;运用SignalP4.0在线软件(http://www.cbs.dtu.dk/ services/SignalP-4.0/)预测杧果NFU蛋白的信号肽情况;利用Phyre2在线软件(http://www. sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index)分析杧果NFU蛋白的三级结构。
1.2.3 系统发育树分析 根据SONG等[19]和GAO等[23]的方法,利用ClustalX 2.0.13软件对杧果(漆树科)、白杨(,杨柳科)、番茄(,茄科)、葡萄(,葡萄科)、柑橘(,芸香科)、拟南芥(十字花科)、盐芥(,十字花科)、大豆(禾本科)、水稻(禾本科)、短柄草(,禾本科)、桃(,蔷薇科)、苹果(,蔷薇科)和草莓(,蔷薇科)13种植物NFU和ISU同源蛋白分别进行氨基酸序列比对,利用软件MEGA 13.0中的邻接法(Neighbor-joining, NJ)构建系统进化树,分析遗传进化关系。
1.2.4 实时荧光定量PCR分析 利用NCBI/ Primer-BLAST在线软件,设计杧果NFU家族基因的特异性表达引物(表1),以杧果为内参,通过ABI 7500实时荧光定量PCR仪检测杧果NFU家族基因的组织特异性表达特征及其对胁迫处理的响应差异,每个反应进行3次生物学重复。使用SYBR Green(TaKaRa,大连)荧光染料进行PCR反应,程序设定为:95℃预变性30 s;95℃变性5 s,58℃退火34 s,40个循环;最后72℃延伸10 s。获得相应的Ct值,经内参基因均一化处理,采用2-ΔΔCT法计算相对表达量,通过Log2法计算胁迫处理前后表达倍数的变化,以对照条件的表达值设定为1,若胁迫条件下的相对表达值<1,表示基因表达水平降低,若相对表达值>1,则表示基因表达水平上升,并通过HemI软件绘制表达倍数变化的热图[17-20, 23]。

表1 实时荧光定量PCR所用的特异性引物
1.3 数据处理
利用软件SPSS 13.0(SPSS Chicago,美国)对数据进行显著性分析,杧果幼苗在胁迫处理与对照条件2个独立样品间进行-检验。
2 结果与分析
2.1 杧果NFU家族基因的鉴定与克隆
通过BLAST同源比对法,在杧果数据库里检索到4个潜在的NFU家族蛋白,Pfam注释为NifU-like功能域或NFU Scaffold protein,GO分子功能注释为Fe-S簇结合(GO:0051536),证实均为Fe-S簇装配机制中的NFU家族基因,命名为杧果。分别下载各基因电子CDS序列,设计上下游引物进行PCR扩增,测序验证后提交NCBI获得Genbank ID(表2)。杧果NFU家族基因的登陆号、基因位置、染色体分布、CDS长度和编码蛋白数量等信息详见表2,4个杧果NFU家族基因分布在4条不同的染色体上,基因CDS最长,最短。

表2 杧果NFU家族基因信息
与已报道的拟南芥[4, 9]、桃[19]、水稻[17]和葡萄[21]基因组中NFU家族成员数量相比较,杧果NFU家族中缺失了NFU5支架蛋白,进一步比对杧果、拟南芥、盐芥、短柄草、番茄、大豆、水稻、白杨、草莓、桃、苹果、葡萄和柑橘13种不同科属植物基因组中NFU同源蛋白的存在情况(表3),结果发现拟南芥、盐芥和短柄草3种一年生草本植物基因组中含有所有的NFU成员,其他10种植物在基因组中均缺失NFU5蛋白。此外,进一步比对上述13种植物中同为支架蛋白的ISU家族成员存在情况,结果发现拟南芥、短柄草和盐芥3种草本植物基因组中含有所有的ISU蛋白成员,而水稻、番茄和白杨在其基因组中缺失ISU3蛋白,杧果、草莓、苹果、桃、葡萄和柑橘6种代表性果树基因组中均缺失了ISU2和ISU3(表3)。

表3 13种植物NFU和ISU同源家族基因比较
2.2 杧果NFU家族蛋白的理化性质与高级结构分析
蛋白理化性质分析结果可知(表4),杧果NFU家族蛋白均含有不同数量的C、H、N、O和S五大元素;除MiNFU2蛋白的等电点pI>7.00,其他3个成员的等电点pI<7.00,表明杧果NFU家族蛋白含有的酸性氨基酸较多;MiNFU1和MiNFU3的脂肪系数大于100、且GRAVY亲水指数大于0,表明这2个NFU成员为疏水蛋白,其他2个成员均为亲水蛋白(脂肪系数小于100,GRAVY亲水指数小于0);信号肽分析表明杧果NFU家族蛋白均不具有信号肽。此外,稳定性分析表明杧果NFU蛋白的不稳定性指数大于40,均为不稳定蛋白(表4)。
Motif分析结果表明杧果NFU家族蛋白含有3个典型的Motif基序(图1),Motif1~Motif3,其中,MiNFU2和MiNFU3拥有全部3个Motif基序,MiNFU1缺失Motif3基序而MiNFU4缺失Motif1和Motif2两个基序(图1A);Motif1~ Motif3分别含有41、23和50个特征氨基酸数目(图1B)。
蛋白质高级结构预测结果表明杧果Group I亚家族的3个NFU蛋白成员(MiNFU1~MiNFU3)拥有三级结构极为相似,而属于Group II亚家族的MiNFU4蛋白具有独特的三级结构,与Group I亚家族成员的三级结构差异显著(图2)。
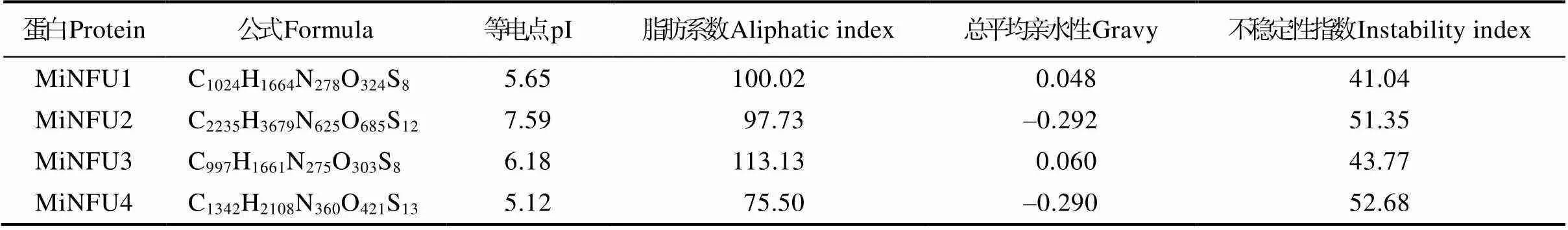
表4 杧果NFU家族蛋白理化性质分析

图1 杧果NFU家族蛋白Motif基序预测

图2 杧果NFU家族蛋白三级结构预测
2.3 植物NFU同源蛋白系统发育树分析
构建杧果、拟南芥、盐芥、短柄草、番茄、大豆、水稻、白杨、草莓、桃、苹果、葡萄和柑橘13种植物NFU家族同源蛋白的系统发育树(图3),结果表明,13种植物NFU同源蛋白在系统发育树中可以分为2个亚家族,其中NFU1~ NFU13同源蛋白属于Group I,而NFU4同源蛋白属于Group II,且在遗传进化关系上存在差异,水稻和短柄草2种禾本科单子叶植物NFU同源蛋白在遗传距离上是最近的,其NFU1~NFU4同源蛋白分别两两紧密地聚在一起;其他11种双子叶植物NFU同源蛋白倾向于聚在一起,其中,杧果MiNFU2和MiNFU3分别与柑橘相应的同源蛋白两两紧密地聚在一起,杧果MiNFU4和番茄SlNFU4紧密聚在一起,拟南芥和盐芥2种十字花科植物的NFU同源蛋白在遗传距离上更为相近,其NFU1~NFU4同源蛋白分别紧密地两两聚在一起,草莓、桃和苹果同属于蔷薇科植物,其NFU同源蛋白紧密的聚在一起,其NFU同源蛋白倾向于聚在一起,而大豆作为一年生草本植物,其Group I家族3个成员蛋白却倾向于与上述多种木本植物NFU同源蛋白聚在一起。
2.4 杧果NFU家族基因组织特异性表达分析
qRT-PCR分析结果表明杧果NFU家族基因在五年生‘桂热82’新生叶片、新生韧皮部、盛开期花朵、幼果、熟果和一年生嫁接幼苗根、茎和叶等多种组织/器官中均有表达,且相对表达量差异较大(图4);其中,在所有检测组织中的整体表达水平都是最高的,其次是和,而的整体表达水平最低;特别地,杧果NFU家族基因在所有检测组织中的表达趋势相似,均在幼苗叶片中的表达水平最高,远高于其他检测组织中的表达量,其次是韧皮部和盛开期花朵,在果实(幼果和熟果)中的表达水平相对较低。

图3 13种植物NFU同源蛋白系统发育树

图4 杧果NFU家族基因组织特异性表达分析
2.5 幼苗中杧果NFU家族基因对不同非生物胁迫的差异响应
以‘桂热82’杧果组培幼苗为材料,通过qRT-PCR分析杧果NFU家族基因在转录水平对不同非生物胁迫(包括缺铁、高铁毒害、低温、PEG和NaCl处理)的响应特征。结果表明(图5),基因在转录水平的表达量最易受外界非生物胁迫影响,至少在1个检测组织部位(叶片、茎部或根部)对所有处理均有响应,而的表达最为稳定,均不受5种非生物胁迫的影响。具体地,杧果NFU家族基因对高铁毒害处理最为敏感,除外,其他3个杧果基因至少在1个检测组织部位(叶片、茎部或根部)对高铁处理有响应,其中,和对高铁处理最为敏感,其表达水平在‘桂热82’幼苗全身组织中均受高铁处理影响而显著增加。在根部的表达水平也受高铁处理的影响而显著增强,且上升倍数(2.9倍)最为显著。、和基因在根部的表达水平受缺铁处理抑制而降低,在根部及在茎部和根部的表达水平受PEG处理抑制而降低,和基因在叶片的表达水平受低温处理抑制而降低,而在根部的表达量受NaCl处理上调而增加。
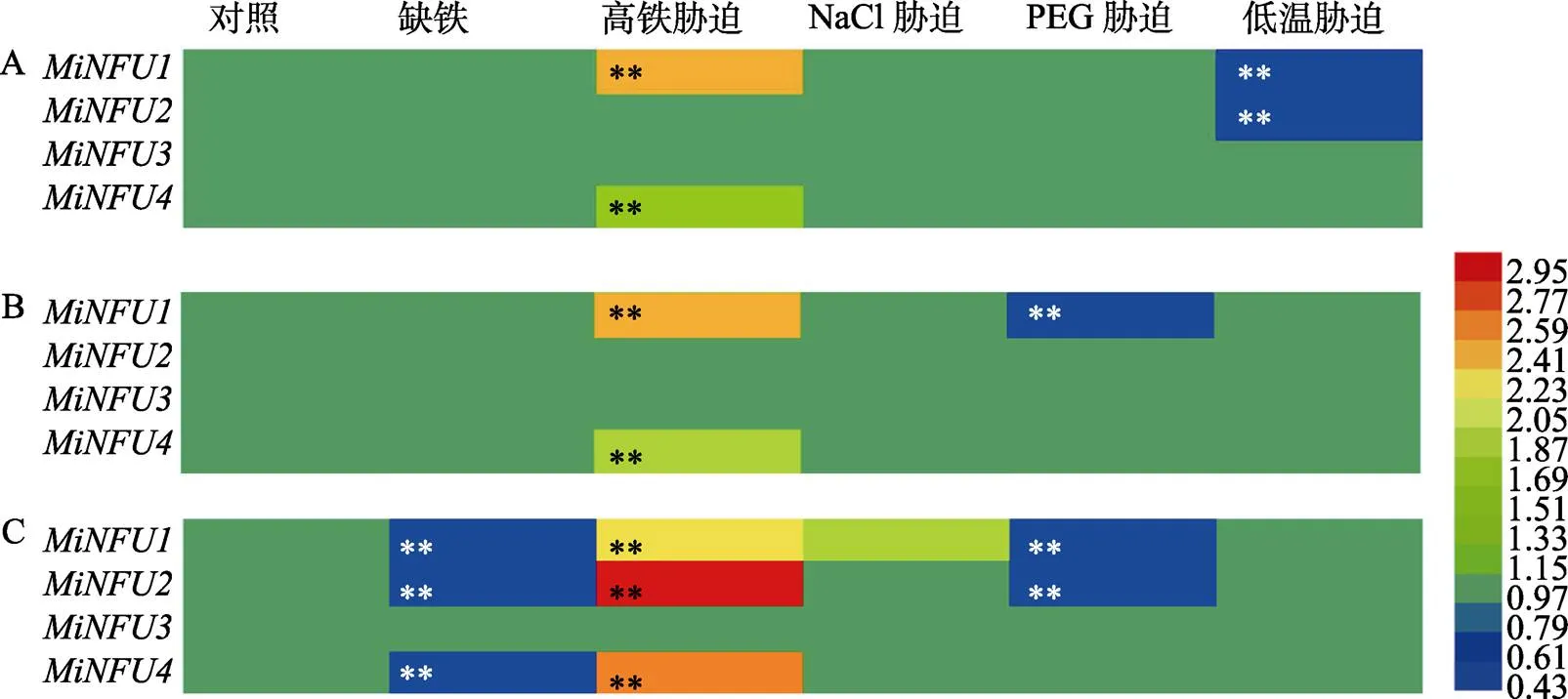
A:幼苗叶片;B:幼苗茎部;C:幼苗根部;**表示差异极显著。
3 讨论
铁是果树生长发育最为重要的微量元素之一,与果树生长、发育、果实品质和产量密切相关[1-3, 24-26]。然而,相关研究集中在生理生化层面,有关果树铁素营养和铁代谢的分子机制研究较少,杧果Fe-S簇装配的分子机制依然未知。
植物Fe-S簇装配机制是高度复杂但却非常保守和稳定的代谢过程,牵扯到数十个基因,分别在质体、线粒体、细胞质和细胞核中发挥作用[4, 8-9]。前人研究表明拟南芥存在5个NFU家族成员[4, 9],然而,杧果中仅检索到4个NFU家族成员,发生丢失(表2),此外,在水稻[17]、大豆[18]和桃[19]基因组中亦发生缺失。已有研究表明拟南芥同样编码支架蛋白参与Fe-S簇装配过程,其在水稻[17]、桃[19]和葡萄[21]Fe-S簇装配机制相关基因中的的表达水平都是最高的,暗示ISU1是植物Fe-S簇装配机制必不可少的支架蛋白,拟南芥、盐芥、短柄草和大豆等4种一年生植物中拥有3个ISU家族成员,水稻、番茄和白杨中缺失成员,然而,草莓和多种木本果树作物(杧果、苹果、柑橘和桃)仅仅含有1个成员。特别地,LEON等[13]发现拟南芥中和基因的表达水平很低,推断该基因可能没有生物学功能或是假基因。为了验证这一推断,本研究进一步比对13种高等植物基因组中ISU和NFU同源蛋白的存在情况,结果发现相较于拟南芥、盐芥和短柄草等3种草本植物,蔷薇科(草莓、苹果和桃)和芸香科(柑橘)等多年生果树作物均缺失ISU3、NFU4和NFU5成员(表3),这些发现再次暗示植物Fe-S簇装配机制中的非功能性支架蛋白在长期进化过程中更容易发生丢失,多年生木本植物铁素营养代谢过程中可能利用更少数量的功能性支架蛋白。
尽管上述13种植物NFU同源蛋白在部分结构域区段的氨基酸序列是高度一致的,但在系统发育树遗传进化关系上的差异较大,共属于同一科属(蔷薇科、禾本科或十字花科)植物的NFU同源蛋白更倾向于聚集在一起,而杧果NFU家族蛋白分别和柑橘或番茄相对应的同源蛋白紧密地聚在一起(图3),暗示这些同源蛋白的遗传距离更近,在长期进化过程中更倾向于拥有相同或相近的生物学功能。因此,研究模式作物番茄NFU功能可能为柑橘和杧果等热带或亚热带果树作物NFU同源蛋白的生物学功能提供理论依据和技术支撑。
生物学中,基因组织表达特征及其蛋白质高级结构均与基因的生物学功能密切相关。本研究发现杧果Group I与Group II亚家族成员蛋白拥有的Motif基序和三级结构差异都很明显(图1、图2),且杧果NFU家族基因的组织特异性表达特征差异较大(图4),且在转录水平对缺铁等非生物胁迫的响应情况差异明显(图5),这些结果充分表明杧果NFU家族基因在杧果不同组织或器官Fe-S簇装配过程中可能发挥的生物学功能差异较大,仍需进一步的分子生物学实验证实。
此外,本研究表明杧果NFU家族基因在转录水平的表达水平易受铁素供应条件的影响,特别是对高量铁素的毒害处理较为敏感,不同组织部位中NFU响应基因的表达水平倾向于受高铁处理诱导而增强,而根中NFU响应基因的表达水平受缺铁抑制而显著降低(图5)。的整体表达水平最高,其在幼苗叶片、茎部和根部中的表达量分别约为其他基因的2~10倍,且在根部受高铁毒害处理的诱导显著增加(约2.9倍),受缺铁处理的抑制而降低(约2.4),暗示MiNFU2是Fe-S簇装配过程中具有较强活性的支架蛋白,其活性直接受外界铁素水平的调控,这些结果与水稻[17]和大豆[13]中有关NFU基因响应铁素水平的报道相似。SONG等[19]发现桃中NFU家族基因在转录水平的表达情况不受铁素供应的影响,虽然在杧果不同组织中的整体表达水平极低,但却非常稳定,其在转录水平不受本研究设定的5种非生物胁迫的影响,暗示该支架蛋白可能持续稳地的发挥自身活性,进而可有效地参与杧果Fe-S簇装配过程。此外,值得注意的是,杧果是典型的热带果树作物,其在温带和寒冷地区的生长适应性相对很差,本研究发现和在叶片中的表达水平受4℃低温处理的抑制而显著降低,说明低温环境可能抑制叶片中这些NFU支架蛋白的活性,一定程度上影响了叶片中Fe-S簇装配过程,进而引起一些必需铁的代谢途径或生命活动发生变化,以适应低温环境的胁迫。
[1] BARTON L L, ABADIA J. Iron nutrition in plants and rhizospheric microorganisms[M]. New York: Springer-Verlag, 2006: 85-101.
[2] KOBAYASHI T, NISHIZAWA N K. Iron uptake, translocation, and regulation in higher plants[J]. The Annual Review of Plant Biology, 2012, 63: 131-152.
[3] 李俊成, 于 慧, 杨素欣, 冯献忠. 植物对铁元素吸收的分子调控机制研究进展[J]. 植物生理学报, 2016, 52(6): 835-842.
LI J C, YU H, YANG S X, FENG X Z. Research progress of molecular regulation of iron uptake in plants[J]. Plant Physiology Joumal, 2016, 52(6): 835-842. (in Chinese)
[4] BALK J, LOBRÉAUX S. Biogenesis of iron–sulfur proteins in plants[J]. Trends in Plant Science, 2005, 10(7): 324-331.
[5] JOHNSON D C, DEAN D R, SMITH A D, JOHNSON M K. Structure, function, and formation of biological iron-sulfur clusters[J]. Annual Review of Biochemistry, 2005, 74(1): 247-281.
[6] LILL R, MÜHLENHOFF U. Iron-sulfur protein biogenesis in eukaryotes: components and mechanisms[J]. Annual Review of Cell & Developmental Biology, 2006, 22(1): 457-486.
[7] ROUAULT T A, TONG W H. Iron-sulfur cluster biogenesis and human disease[J]. Trends in Genetics, 2008, 24(8): 398-407.
[8] LILL R. Function and biogenesis of iron-sulphur proteins[J]. Nature, 2009, 460(7257): 831.
[9] BALK J, PILON M. Ancient and essential: the assembly of iron-sulfur clusters in plants[J]. Trends in Plant Science, 2011, 16: 218-226.
[10] COUTURIER J, TOURAINE B, BRIAT J F, GAYMARD F, ROUHIER N. The iron-sulfur cluster assembly machineries in plants: current knowledge and open questions[J]. Frontiers in Plant Science, 2013, 4: 259.
[11] NETZ D J A, MASCARENHAS J, STEHLING O, PIERIK A J, LILL R. Maturation of cytosolic and nuclear iron-sulfur proteins[J]. Trends in Cell Biology, 2013, 24(5): 303-312.
[12] BERNARD D G, NETZ D J A, LAGNY T J, PIERIK A J, BALK J. Requirements of the cytosolic iron-sulfur cluster assembly pathway in[J]. Philosophical Transactions of the Royal Society B: Biological Sciences, 2013, 368: 20120259.
[13] LÉON S, TOURAINE B, RIBOT C, BRIAT J F, LOBRÉAUX S. Iron-sulphur cluster assembly in plants: distinct NFU proteins in mitochondria and plastids from[J]. Biochemical Journal, 2003, 371: 823-830.
[14] TOURAINE B, BOUTIN J P, MARION-POLL A, BRIAT J F, PELTIER G, LOBRÉAUX S. Nfu2: a scaffold protein required for [4Fe-4S] and ferredoxin iron-sulfur cluster assembly inchloroplasts[J]. Plant Journal, 2004, 40: 101-111.
[15] YABE T, MORIMOTO K, KIKUCHI S, NISHIO K, TERASHIMA I, NAKAI M. Thechloroplastic NifU-like protein CnfU, which can act as an iron-sulfur cluster scaffold protein, is required for biogenesis of ferredoxin and photosystem I[J]. Plant Cell, 2004, 16: 993-1007.
[16] YABE T, YAMASHITA E, KIKUCHI A, MORIMOTO K, NAKAGAWA A, TSUKIHARA T, NAKAI M. Structural analysis ofCnfU protein: an iron-sulfur cluster biosynthetic scaffold in chloroplasts[J]. Journal of Molecular Biology, 2008, 381: 160-173.
[17] LIANG X J, QIN L, LIU P W, WANG W H, Ye H. Genes for iron-sulphur cluster assembly are targets of abiotic stress in rice[J]. Plant, Cell & Environment, 2013, 37(37): 780-794.
[18] QIN L, WANG M H, CHEN L Y, LIANG X J, WU Z G, LIN Z H, ZUO J, FENG X Y, ZHAO J, LIAO H, YE H. Soybean Fe-S cluster biosynthesis regulated by external iron or phosphate fluctuation[J]. Plant Cell Reports, 2015, 3(34): 411-424.
[19] SONG Z Z, YANG Y, XU J L, MA R J, YU M L. Physiological and transcriptional responses in the iron-sulphur cluster assembly pathway under abiotic stress in peach (L.) seedlings[J]. Plant Cell Tissue & Organ Culture, 2014, 117(3): 419-430.
[20] SONG Z Z, MA R J, ZHANG B B, GUO S L, YU M L, KORIR N K. Differential expression of iron-sulfur cluster biosynthesis genesduring peach fruit development and ripening, and their response toiron compound spraying[J]. Scientia Horticulturae, 2016, 207: 73-81.
[21] 张 璐, 宗亚奇, 徐维华, 韩 蕾, 孙浈育, 陈朝晖, 陈松利, 张 凯, 程杰山, 唐美玲, 张洪霞, 宋志忠. 葡萄Fe-S簇装配基因的鉴定、克隆和表达特征分析[J]. 中国农业科学, 2021, 54(23): 5068-5082.
ZHANG L, ZONG Y Q, XU W H, HAN L, SUN Z Y, CHEN Z H, CHEN S L, ZHANG K, CHENG J S, TANG M L, ZHANG H X, SONG Z Z. Identification, cloning, and expression characteristics analysis of Fe-S cluster assembly genes in grape[J]. Scientia Agricultura Sinica, 2021, 54(23): 5068-5082. (in Chinese)
[22] WANG P, LUO Y, HUANG J F, GAO S H, ZHU G P, DANG Z G, GAI J T, YANG M, ZHU M, ZHANG H K, YE X X, GAO A P, TAN X Y, WANG S, WU S Y, CAHOON E B, BAI B B, ZHAO Z C, LI Q, WEI J Y, CHEN H R, LUO R X, GONG D Y, TANG K X, ZHANG B, NI Z G, HUANG G D, HU S N, CHEN Y Y. The genome evolution and domestication of tropical fruit mango[J]. Genome Biology, 2020, 21: 60.
[23] GAO Y C, YU C Y, ZHANG K, ZHANG H X, ZHANG S Y, SONG Z Z. Identification and characterization of the strawberry KT/HAK/KUP transporter gene family in response to K+deficiency[J]. Acta Physiologiae Plantarum, 2021, 43: 1.
[24] CHENY, BARAK P. Iron nutrition of pants in calcareous soils[J]. Advances in Agronomy, 1982, 35: 217-240.
[25] JIMÉNEZ S, GOGORCENA Y, HÉVIN C, ROMBOLÀ A D, OLLAT N. Nitrogen nutrition influences some biochemical responses to iron deficiency in tolerant and sensitive genotypes of vitis[J]. Plant and Soil, 2007, 290(1/2): 343-355.
[26] PESTANA M, BEJA P, CORREIA P J, DE VARENNES A, FARIA E A. Relationships between nutrient composition of flowers and fruit quality in orange trees grown in calcareous soil[J]. Tree Physiology, 2005, 25(6): 761-767.
Cloning and Identification of NFU Family Genes in Mango and Responsive Analysis to Abiotic Stresses
LI Youhuan1, BAO Zhongqi1*, GAO Taiping2, HUANG Jianfeng2, WANG Limin1, ZHANG Hongxia1, SONG Zhizhong1,3**
1. Engineering Research Institute of Agriculture and Forestry, Ludong University / Key Laboratory of Molecular Module-based Breeding of High Yield and Abiotic Resistant Plants in Universities of Shandong, Yantai, Shandong 264025, China; 2. Tropical Crops Genetic Resources Institute, Chinese Academy of Tropical Agricultural Sciences, Haikou, Hainan 571101, China; 3. Department of Plant Science, University of Cambridge, Cambridge, UK CB2 3EA
Four NFU family genes that involved in Fe-S cluster assembly were isolated and identified from diploid mango ‘Guire82’, which were entitledto, respectively. MiNFU2 and MiNFU3 proteins had all three typical motifs of NFU family proteins, while MiNFU1 lacks motif 3 and MiNFU4 lacks motif 1 and motif 2. MiNFU1 to MiNFU3 had a similar tertiary structure, while the tertiary structure of MiNFU4 was quite unique and different. There are slight differences in NFU family members among13 different plant families and genera. Three annual herbs of,, andcontain more scaffold proteins, while non-functional scaffold proteins are more likely to be lost in perennial fruit crops, such as mango, strawberry, peach, apple, citrus and grape. According to the phylogenetic tree, plant NFU homologous proteins can be divided into two subfamilies, which have differences in genetic and evolutionary relationships. Among them, NFU homologous proteins from,ortend to be closely clustered, respectively, and mango NFU proteins are closely clustered with the corresponding homologs of citrus and tomato. In addition, there were significant differences in the expression levels offamily genes in different tissues / organs of mango. The overall expression level ofwas the highest in all tested tissues, followed by4 and1, while the overall expression level of3 was the lowest. The expression levels offamily genes in seedling leaves were the highest, followed by phloem and full blooming flowers, while the expression levels offamily genes in fruits (young and mature fruits) was relatively low. Notably, the transcriptional responses ofNFU family genes in mango seedlings to iron depletion, high iron toxicity, NaCl, PEG and low temperature treatments were significantly different. Although the overall expression of3 was the lowest, it was not affected by all tested abiotic stresses.andwere more sensitive to high iron treatment, whose expression levels were significantly increased throughout the whole plant under high iron treatment. In addition, NaCl treatment significantly increased the expression ofin roots. Iron deficiency and PEG treatment tended to reduce the expression levels of responsiveNFUgenes in roots, while low temperature treatment were prone to reduce the expression levels ofresponsiveNFU genes in leaves. This study provides gene resources to elucidate the molecular mechanisms of Fe-S cluster assembly in mango, and lays a theoretical foundation to reveal Fe nutrition and metabolism in tropicalfruit crops.
mango; Fe metabolism; Fe-S cluster assembly machinery; NFU Scaffold protein; abiotic stress
S667.7
A
10.3969/j.issn.1000-2561.2022.07.001
2022-01-25;
2022-02-24
国家重点研究计划子课题(No. 2019YFD1000504);国家自然科学基金项目(No. 31501743,No. 31601819)。
李又欢(2000—),男,本科生,研究方向:果树分子生物学;*同等贡献作者:鲍中齐(2001—),男,本科生,研究方向:果树分子生物学。**通信作者(Corresponding author):宋志忠(SONG Zhizhong),E-mail:szhzh2000@163.com。

