大黄花虾脊兰内生真菌及土壤真菌的群落特征研究
黄敏, 江标, 高大中, 谢冰, 韦姝羽, 邢韶华
大黄花虾脊兰内生真菌及土壤真菌的群落特征研究
黄敏, 江标, 高大中, 谢冰, 韦姝羽, 邢韶华*
北京林业大学生态与自然保护学院, 北京 100083
大黄花虾脊兰是列入我国《全国极小种群野生植物拯救保护工程规划》中亟待拯救的极小种群物种之一。内生真菌及土壤微生物是影响兰科植物生长繁育的重要因素, 为摸清大黄花虾脊兰内生真菌及土壤微生物的群落特征, 促进其种群扩繁, 对采自安徽泾县大黄花虾脊兰开花期和果期的根、根际土、根围土进行了高通量测序, 对所得序列进行聚类分析、α多样性分析、β多样性分析和LEfSe分析, 探究不同时期下大黄花虾脊兰的内生真菌和土壤真菌的组成及多样性变化。结果: 测序得到1127529条有效真菌序列, 通过聚类分析将其划分为761个OTUs, 分属于8门、93科、121属; Venn图分析发现根和土壤中有24个属的菌类属于共有真菌, 植物根存在特有的菌类; 在不同时期大黄花虾脊兰的优势菌群不同, 花期和果期的优势内生真菌分别为Tulasnellaceae(15.68%)和(56.75%), 优势土壤真菌分别为(12.19%)和(15.93%); 优势内生真菌在根、根际土、根围土的三个不同的水平距离上, 真菌丰度呈梯度性递减趋势; 大黄花虾脊兰的微生境真菌群落在群落结构和组成上有明显的季节变化特征。
大黄花虾脊兰; 内生真菌; 土壤真菌; 群落结构; 高通量测序
0 前言
大黄花虾脊兰(Decne.)是兰科虾脊兰属植物, 花的形态美观, 在对12种兰科虾脊兰属植物的分析与评价中, 其观赏性得分最高, 生长适宜性仅次于三褶虾脊兰()和银带虾脊兰()[1], 极具观赏价值和经济价值。大黄花虾脊兰对生境要求苛刻, 种群数量极少, 属于亟待拯救的极小种群物种[2]。然而该如何拯救?影响该物种种群扩繁的关键因素又是什么?目前尚未有明确的答案。早在1903年, Bernard就证实了在自然环境下的兰科植物种子只有感染了合适的真菌后才能正常萌发生长[3]。近年来, 国内外对兰科植物的研究日渐增多, 关于兰科植物内生真菌的研究更是研究热点。不过, 目前针对大黄花虾脊兰的研究仅涉及生态学、组培学[4–5], 而关于其内生真菌的研究还少有报道。最新的研究成果解决了大黄花虾脊兰的无菌播种技术问题[5], 但是组培苗往往存在移栽成活率低、生长缓慢、开花延迟甚至不开花等问题[6], 而内生真菌可以显著提高组培苗的移栽成活率以及鲜重、株高等植株品质[7]。例如, 天麻种子与紫萁小菇等萌发菌伴播技术的研发极大推动了天麻的量产, 提高了天麻的质量[8]。内生真菌还能提高宿主植物对病虫害的抗胁迫性, 目前已成功应用到多种植物病害的防治研究中。刘艳明等从铁皮石斛中筛选出34株具有拮抗炭疽菌的内生真菌, 它们对致病菌的抑制率高达85%—95%[9]。此外, 兰科植物的内生真菌群落并不是一成不变的, 且内生真菌群落结构的变化很可能与寄主植物的物候相关。侯天文等发现兰科菌根的真菌群落多样性在生长期达到顶峰, 到果期时则大幅下降[10]。为了较为全面地了解濒危植物大黄花虾脊兰的内生真菌, 本研究通过高通量测序技术, 获取植物根和土壤的大部分真菌序列, 研究大黄花虾脊兰的根部内生真菌和土壤真菌的群落结构和多样性特点, 对比分析真菌群落在两个重要生殖生长期之间的变化, 以及优势内生真菌在水平距离上的分布规律, 对了解大黄花虾脊兰与其内生真菌的共生关系具有重要意义, 为该种群的高效扩繁、原地保护和种群再引入提供参考数据。
1 材料与方法
1.1 实验材料采集及预处理
于2019年4月(花期)和9月(果期), 在安徽省宣城泾县, 取同一地点大黄花虾脊兰的根围土、根际土、根三类样品, 考虑到大黄花虾脊兰是极小种群物种, 因此在相邻不超过0.5 m的3个植株上每类样品各取1份, 每次取样9份, 共计18份样品。
大黄花虾脊兰所在生境的土层薄, 约10—15 cm。清除土壤表面的枯枝落叶, 挖土壤剖面(从土壤表面挖到岩石表面), 每株植物取3根完整的植株根, 清除根表面明显的枯落物和大土块, 置于无菌自封袋中, 放入干冰桶内带回。本研究将距离根系表面0—1 cm的土壤视为根围土, 采用抖落的方法, 经2 mm土壤标准筛过筛后存于无菌自封袋中。将距离根表面0.1 mm范围内的土壤视为根际土, 采用超声波震荡的方法将土粒从根表面剥离, 存于无菌自封袋中。土壤样本中每株植物都尽可能地多取, 每份土壤样品>5g即可。选取新鲜健康的营养根, 用自来水将根冲洗干净, 将根剪成2—3 cm长的小段, 再用蒸馏水清洗, 随后放入0.1%的升汞溶液中浸泡5—8 min, 取出后用蒸馏水冲洗1—2次, 再放入75%的酒精中浸泡20 s左右, 最后用蒸馏水冲4—6次, 晾干后放入无菌EP管。经初步处理后, 将以上样品移至-80 ℃冰箱中保存。
1.2 真菌基因组DNA提取、扩增及测序
利用DNA提取试剂盒(MN NucleoSpin 96 Soi)对样品中的DNA进行提取。根据张志东等的研究, 内生真菌多样性分析中采用ITS1-5F引物对, 可获得更全面的内生真菌的多样性数据[11]。因此, PCR扩增采用真菌通用引物ITS1-5F引物对(ITS5: 5’-GGAAGTAAAAGTCGTAACAAGG; ITS2: 5’-GCTGCGTTCTTCATCGATGC-3’)对样品中真菌的ITS1区域进行扩增, 引物合成和测序由北京百迈客生物科技有限公司进行。PCR反应体系为10 μL体系, 即1 μL DNA模板, 3 μM上游引物, 3 μM下游引物, 5 μL KOD FX Neo Buffer, 4 mM dNTP, 0.2 μL KOD FX Neo, 用ddH2O补足至10 μL。PCR扩增程序为: 95 ℃预变性5 min; 95 ℃变性30 s, 50 ℃退火30 s, 72 ℃延伸40 s, 循环35次; 72 ℃延伸7 min, 最后于4 ℃保温。在1.8%的琼脂糖凝胶, 电压120 V的条件下通过 Solexa PCR(20 μL体系)运行40 min。根据电泳定量结果, 将产物按照质量比1: 1进行混样。混样后, 采用OMEGA DNA 纯化柱进行过柱纯化。在1.8%的琼脂糖凝胶中, 120 V电压下电泳40 min后, 切目的片段, 并用Monarch DNA 胶回收试剂盒进行回收, 使用Illumina Hiseq2500 PE250进行双向测序。
1.3 序列分析
对原始数据进行拼接(FLASH, v1.2.11), 将拼接得到的序列进行质量过滤(Trimmomatic, v0.33), 并去除嵌合体(UCHIME, v8.1), 得到高质量的Tags序列。使用USEARCH(v10.0)软件对Tags在97%的相似度水平下进行聚类、获得OTU, 以测序所得的所有序列数的0.005%作为阈值过滤OTU[12], 并基于UNITE(Release8.0)分类学数据库, 在置信度阈值为0.8的水平下, 通过RDP Classifier(v2.2)软件对OTU进行分类学注释。由于本次测序所产生的数据量不一致, 因此需要在随机抽样的基础上进行序列拉平处理。
在Mothur(v.1.30)中使用summary.single指令对各个样品进行α多样性指数评估[13], 包括ACE丰富度指数, Chao1丰富度指数, Shannon多样性指数, 以及Simpson多样性指数。Chao1和Ace指数分别用于衡量样品和群落中的真菌种类的多寡。Shannon和Simpson指数用于衡量样品的真菌多样性。真菌丰度一定时, 群落中各物种的均匀度越大, 则Shannon指数值越大, Simpson指数值越小, 群落的多样性越大。相关计算公式如下[14–17]:
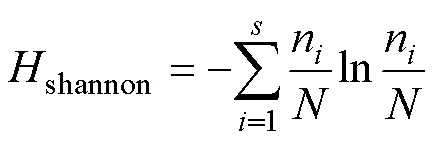
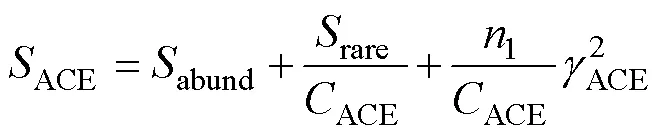
其中,是指实际测量出的OTU数目;n指含有条序列的OTU数目;1指仅包含1条序列的OTU数目;2, 仅包含2条序列的OTU数目;abund指丰富物种数(丰度阈值大于);rare指稀有物种数(丰度阈值小于或等于); γ2ACE指稀有物种变异系数的估算值, 具体计算公式请参考文献[17]。
基于Binary Jaccard和Bray Curtis距离算法, 通过QIIME软件中的beta_diversity_through_plots.py计算β多样性距离矩阵, 利用R语言中的vegan包中的metaMDS()函数和adonis()函数分别进行NMDS分析[18]和PERMANOVA分析, 使用python绘图展现样本之间的差异。NMDS分析通过函数将样品之间的差异程度量化为点与点之间的距离来展现样品间的差异。PERMANOVA分析的本质是基于统计的方差分析, 依据距离矩阵对总方差进行分解的非参数多元方差分析方法, 可体现不同分组因素对样品间差异的解释度, 并使用置换检验进行显著性统计, 以此来寻找造成显著差异的关键因素。
基于QIIME软件的sumarize_taxa.py实现样本分类信息的统计, 借助LEfSe在线分析工具实现LEfSe分析, LEfSe分析在样本内真菌的种属关系的基础上结合具有统计学意义的标准检验(Kruskal- Wallis检验和两两Wilcoxon检验)和线性判别分析的方法进行特征选择, 寻找在所有组间具有显著性差异的物种[19]。
2 结果与分析
根据优质序列的鉴定结果, 我们剔除了数据中的非真菌序列和生物学重复表现不理想的样本, 仅保留真菌序列与没有鉴定结果的序列, 最终获得1127529条优质序列, 聚类分析获得761个OTUs。其中, 来自根部的真菌序列有453194(占40.19%), 来自土壤的真菌序列有674335(占59.81%)。经分析, 所有样本中的序列长度主要分布在100 bp—460 bp之间。通过分类学注释, 得到70%以上的真菌序列在各个分类水平上的分类地位, 包括8门20纲51目93科121属103种。在所有序列中, 属于子囊菌门(Ascomycota)真菌的序列占所有序列的49.31%, 担子菌门(Basidiomycota)真菌则占了19.25%, 其余菌类的分类鉴定结果如图1, 另有25.65%的序列没有明确的鉴定结果。
2.1 内生真菌群落特征
在大黄花虾脊兰的开花期, 于其根内获取223194条优质序列。其中, 属于子囊菌门的真菌序列占12.57%, 属于担子菌门的序列占22.32%, 有高达64.81%的序列没有明确的鉴定结果。在所有序列中, 有15.68%属于Tulasnellaceae, 1.95%属于Hydnodontaceae, 1.82%属于Herpotrichiellaceae, 1.5%属于, 1.37%属于Meruliaceae, 其余真菌类群所占比例均小于1%。在这些内生真菌中, Tulasnellaceae的丰度最大, 为该生长时期的优势内生真菌, 其余的内生真菌占比少。
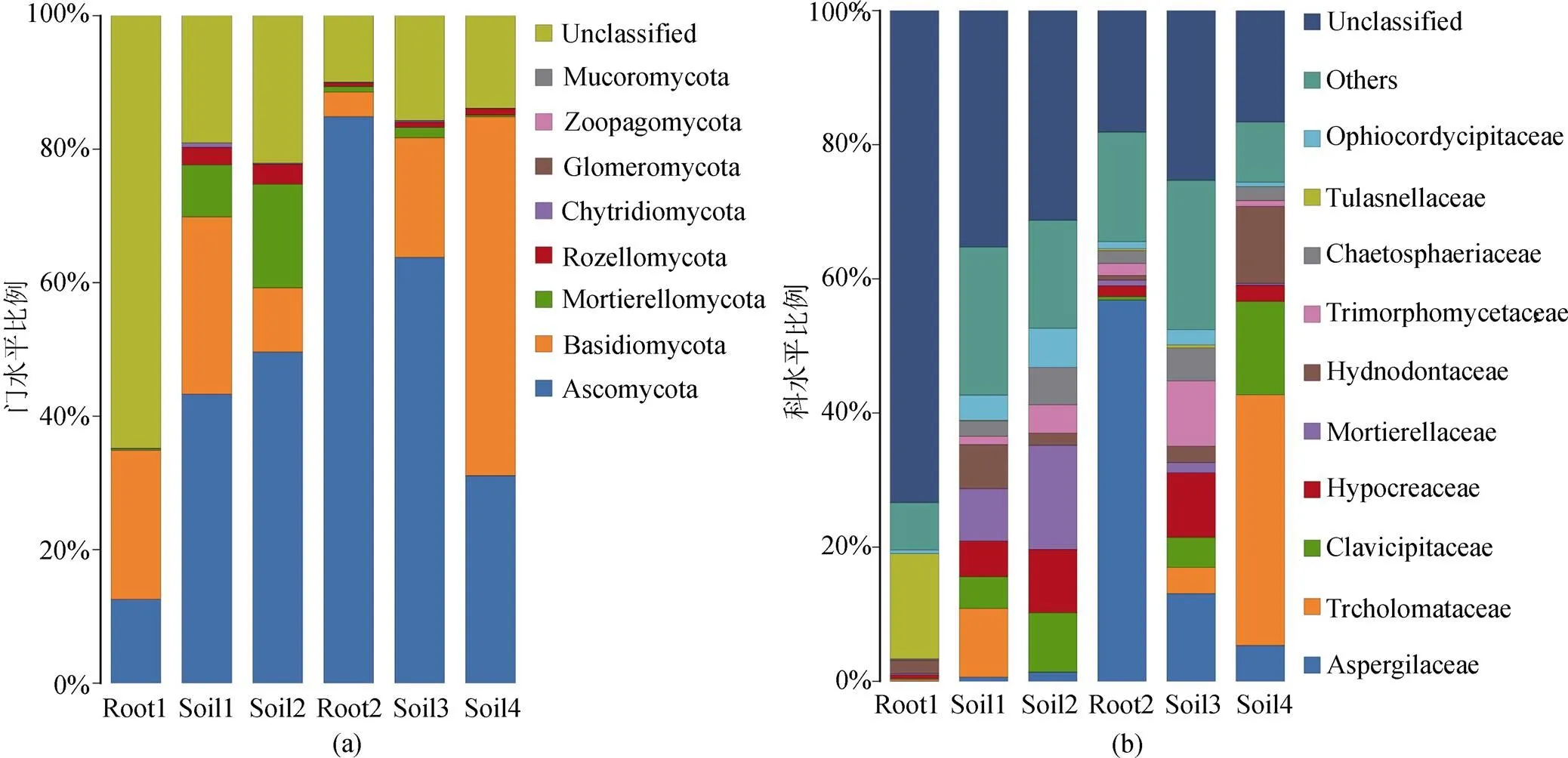
注: 为使视图效果最佳, 只显示丰度水平前十的物种; Root1: 花期根; Root2: 果期根; Soil1: 花期根际土; Soil2: 花期根围土; Soil3: 果期根际土; Soil4: 果期根围土。
Figure 1 Distribution of species on the level of the Phylum(a) and Family(b)
在大黄花虾脊兰的果期, 于其根内获取1060648条优质序列。为了让开花和结果时期的数据具有更好的可比性, 从上述序列中随机抽取230000条序列作为代表序列进行后续分析。在所有序列中, 属于子囊菌门的真菌序列占84.88%, 担子菌门的真菌序列占3.66%, 有9.94%的序列没有明确的鉴定结果。在所有序列中, 有56.75%属于, 4.53%属于Didymellaceae, 2.83%属于Helotiaceae, 1.95%属于 Chaetosphaeriaceae, 1.78%属于, 1.59%属于, 1.11%属于Leotiaceae, 1.07%Ophiocordycipitaceae, 其余真菌类群所占比例均小于1%。在这些内生真菌中,真菌占所有序列的一半以上, 说明真菌在果期的大黄花虾脊兰中占有绝对优势, 属于该生长时期的优势内生真菌。
在logarithmic LDA score为4的基础上, 通过LEfSe分析, 发现和Tulasnellaceae在两个生殖生长期的内生真菌和土壤真菌的群落之间具有显著性差异, 说明这两类真菌很可能是影响大黄花虾脊兰生长发育的关键因子之一。
2.2 土壤真菌群落特征
在花期, 从根际土壤中获得152577条真菌序列。在所有序列中, 有43.29%属于子囊菌门真菌, 26.57%属于担子菌门真菌, 19.04%没有明确的鉴定结果; 主要真菌类群有(10.21%),(7.81%), Hydnodontaceae(6.57%), Didymellaceae(5.87%),(5.3%), 其余真菌类群所占比例均小于5%。从根围土壤中获取201041条真菌序列。在所有序列中, 有49.63%的序列属于子囊菌门真菌, 15.51%的序列属于Mortierellomycota, 22.09%的序列没有明确的鉴定结果; 主要的真菌类群有(15.51%),(9.43%), Clavicipitaceae(8.85%), Chaetomiaceae(8.4%), Ophiocordycipitaceae(5.88%), Chaetosphaeriaceae(5.53%), 其余真菌类群所占比例均小于5%。
在果期, 从根际土壤中获得205341条真菌序列。在所有序列中, 有66.78%属于子囊菌门真菌, 17.95%属于担子菌门真菌, 15.69%没有明确的鉴定结果; 主要的真菌类群为Aspergillaceae(13.07%),(9.75%),(9.59%), Helotiaceae (8.72%), 其余真菌类群所占比例均小于5%。从根围土壤中获取115376条真菌序列。在所有序列中, 有31.1%属于子囊菌门真菌, 53.76%属于担子菌门真菌, 13.87%没有明确的鉴定结果; 主要的真菌类群为(37.36%), Clavicipitaceae(13.92%), Hydnodontaceae(11.49%), Aspergillaceae(5.35%), 其余真菌类群所占比例均小于5%。
2.3 不同时期真菌群落结构的比较
不同生长季节的优势内生真菌不同: 在大黄花虾脊兰的根内, 花期和果期的优势内生真菌分别为Tulasnellaceae和; 在土壤中, 花期和果期的优势真菌分别为。此外, 在属的分类水平上,等24个属的菌类在所有样品中均存在, 属于共有真菌;等7个属的菌类仅存于果期的植物根及其根围土中;真菌仅存在花期的植物根及其周围土壤中;真菌在花期和果期的植物根内均有发现, 但在土壤中没有发现该真菌, 如图2所示, Venn图中颜色越深代表所含菌类的种类越多。
以上结果表明, 受宿主植物的物候影响, 优势真菌会发生一定程度的改变。
在两个生殖生长期的真菌群落中, 优势内生真菌Tulasnellaceae和在根、根际土、根围土的三个不同的距离上, 真菌丰度呈梯度性递减的趋势, 即随着离大黄花虾脊兰距离的增加, 真菌丰度减少, 如图3。说明根、根际土和根围土之间的优势内生真菌在不同的水平距离上存在梯度性差异。
在OTU水平上, 计算各样品真菌群落的α多样性指数, 通过独立样本T检验, 发现两个生殖生长期的大黄花虾脊兰的内生真菌群落的Ace指数、Chao1指数和Shannon指数均显著低于其土壤真菌群落, 说明内生真菌群落的真菌多样性和真菌丰富度远小于土壤真菌群落; 在同一时期的所有样品中, 根际土的真菌群落的Ace指数、Chao1指数和Shannon指数最大, 说明大黄花虾脊兰根表面的真菌种类丰富, 根表面的真菌丰富度要比周围土壤中的真菌丰富度大, 如表1所示。
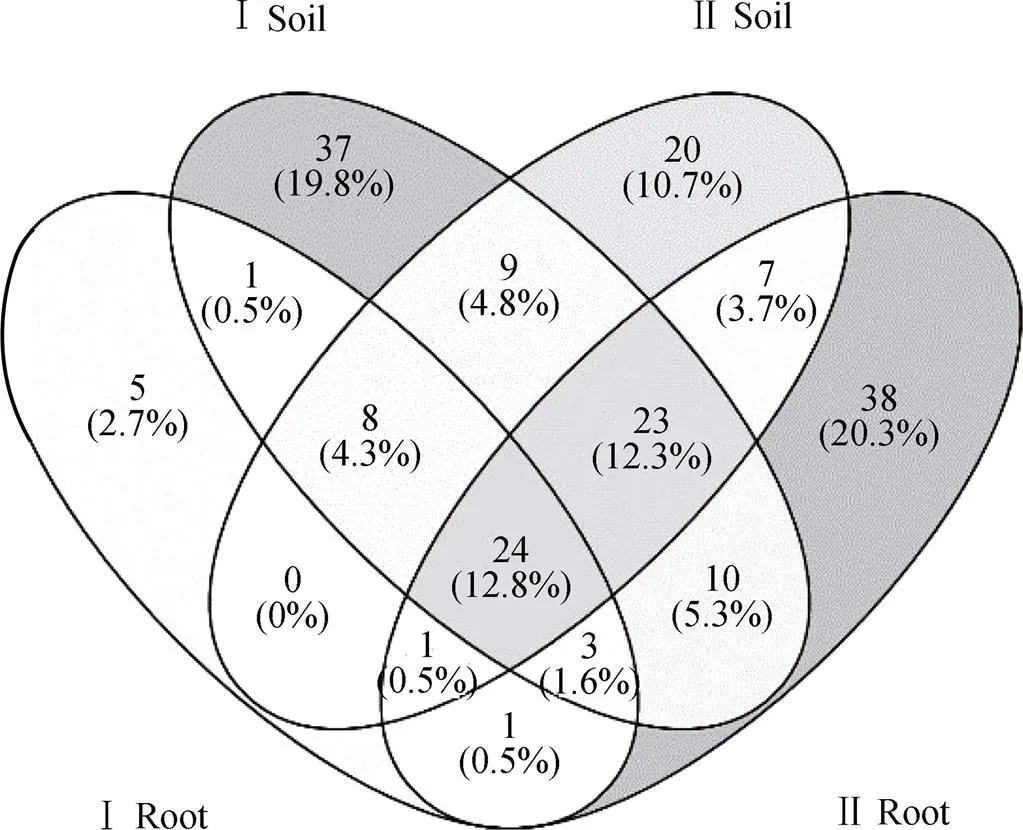
注: ⅠRoot: 花期根; ⅡRoot: 果期根; ⅠSoil: 花期土; Ⅱ Soil: 果期土。
Figure 2 Venn diagram showing endophytic fungi and soil fungi in different growing seasons
在OTU水平上, 通过Bray Curtis(考虑真菌丰度和物种有无)距离算法和Binary Jaccard(仅考虑物种有无)距离算法, 对花期和果期的大黄花虾脊兰的根、根际土、根围土的真菌群落进行NMDS分析和PERMANOVA分析, 结果如图4、5。结果显示, 每类样品的真菌群落各自聚在一起, 不同类别样品的真菌群落互相分开, 且存在一定距离, 表明各类样品之间的真菌群落结构有一定的差异, 如图4(a)。除了花期的根际土和根围土在真菌群落组成上没有明显差别之外, 其余样品的真菌群落组成均有一定差异, 如图4(b)。其中, 内生真菌之间的距离比土壤真菌之间的距离远, 说明花期和果期之间内生真菌群落的差异最大, 且果期内生真菌的多样性远大于花期。这进一步证明, 大黄花虾脊兰的内生真菌群落在一定时间内发生了一定程度的变化, 而这种变化远大于土壤真菌群落的变化。在两个生殖生长期中, 大黄花虾脊兰的微生境真菌在真菌群落结构(2= 0.223,=0.001)和真菌群落组成(2= 0.287,=0.001)上存在明显差异, 且这种差异大于生殖生长期内土壤真菌和内生真菌群落之间的差异, 如图5。这说明大黄花虾脊兰的微生境真菌群落在群落结构和组成上存在明显的季节变化特征。
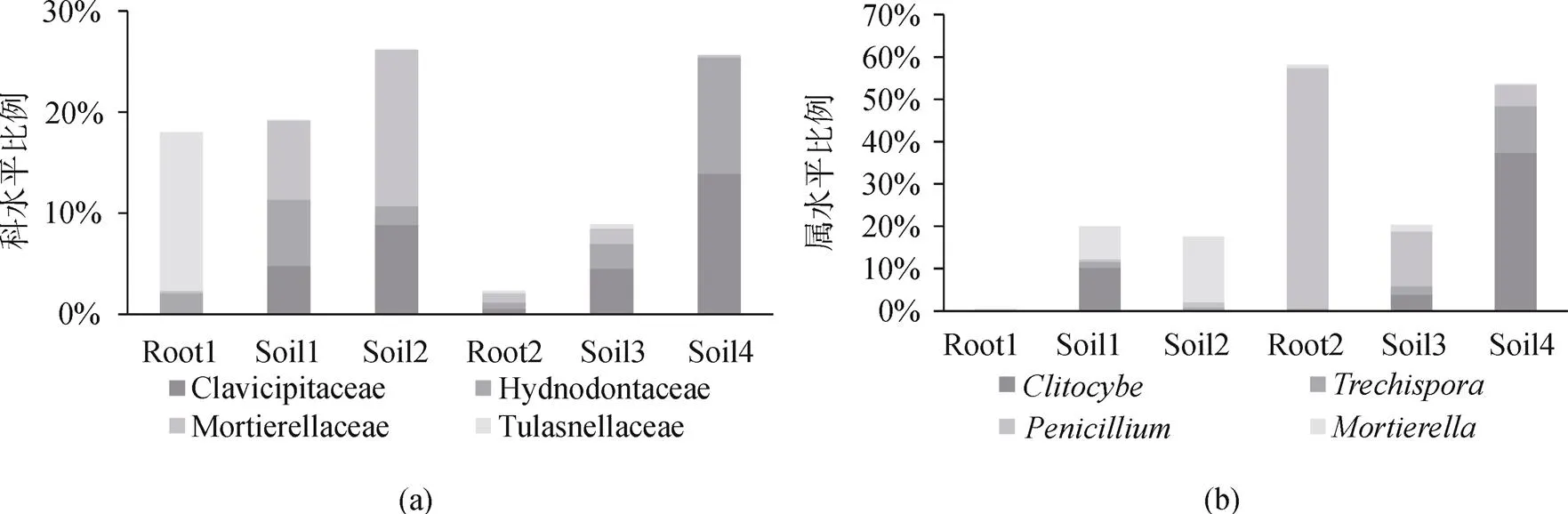
注: 为使视图效果最佳, 只显示相对丰度>10%的物种; Root1: 花期根; Root2: 果期根; Soil1: 花期根际土; Soil2: 花期根围土; Soil3: 果期根际土; Soil4: 果期根围土。
Figure 3 The dominant fungi of relative abundance of mycorrhizal fungi at different sites of the orchids

表1 大黄花虾脊兰内生真菌和土壤真菌的α多样性分析
注: 表中数据为数字±标准误; 同一列中均值统一采用独立样本t检验进行比较, 同列相同字母表示两组样品间没有显著性差异(>0.05); Root1: 花期根; Root2: 果期根;Soil1: 花期根际土;Soil2: 花期根围土;Soil3: 果期根际土; Soil4: 果期根围土。
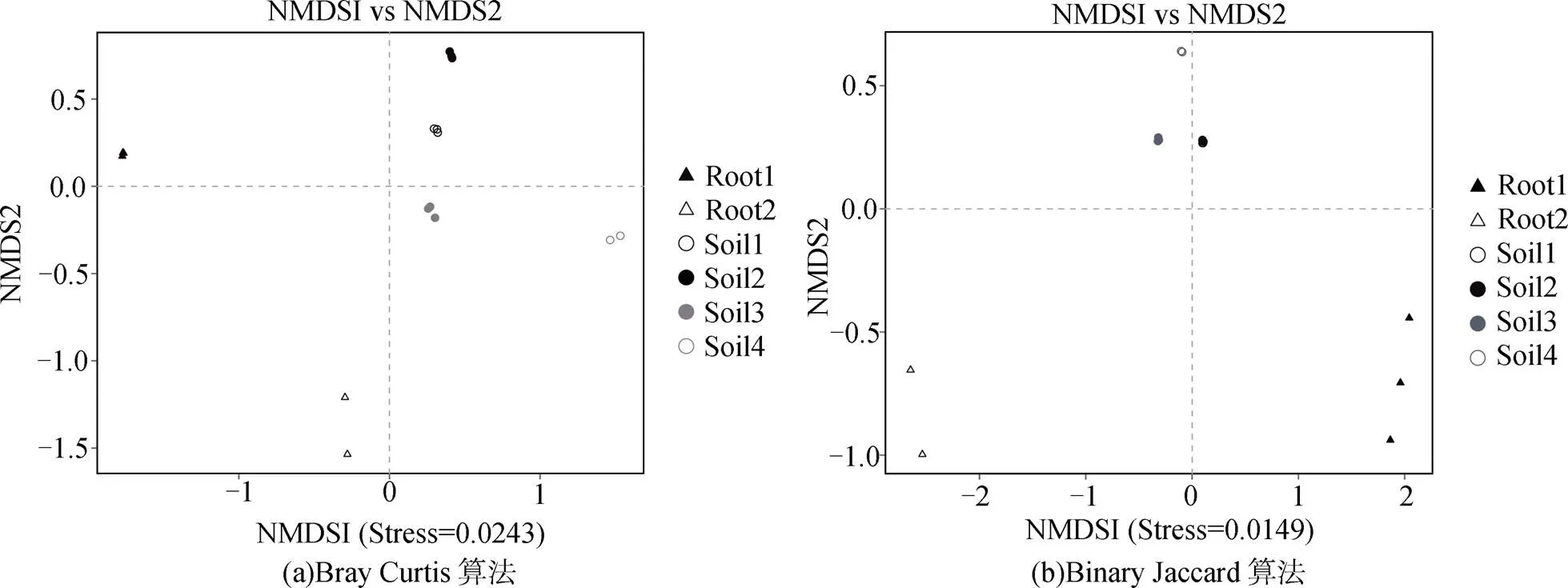
注: Root1: 花期根; Root2: 果期根; Soil1: 花期根际土; Soil2: 花期根围土; Soil3: 果期根际土; Soil4: 果期根围土。
Figure 4 The NMDS analysis of fungi in soil and the roots of the orchids
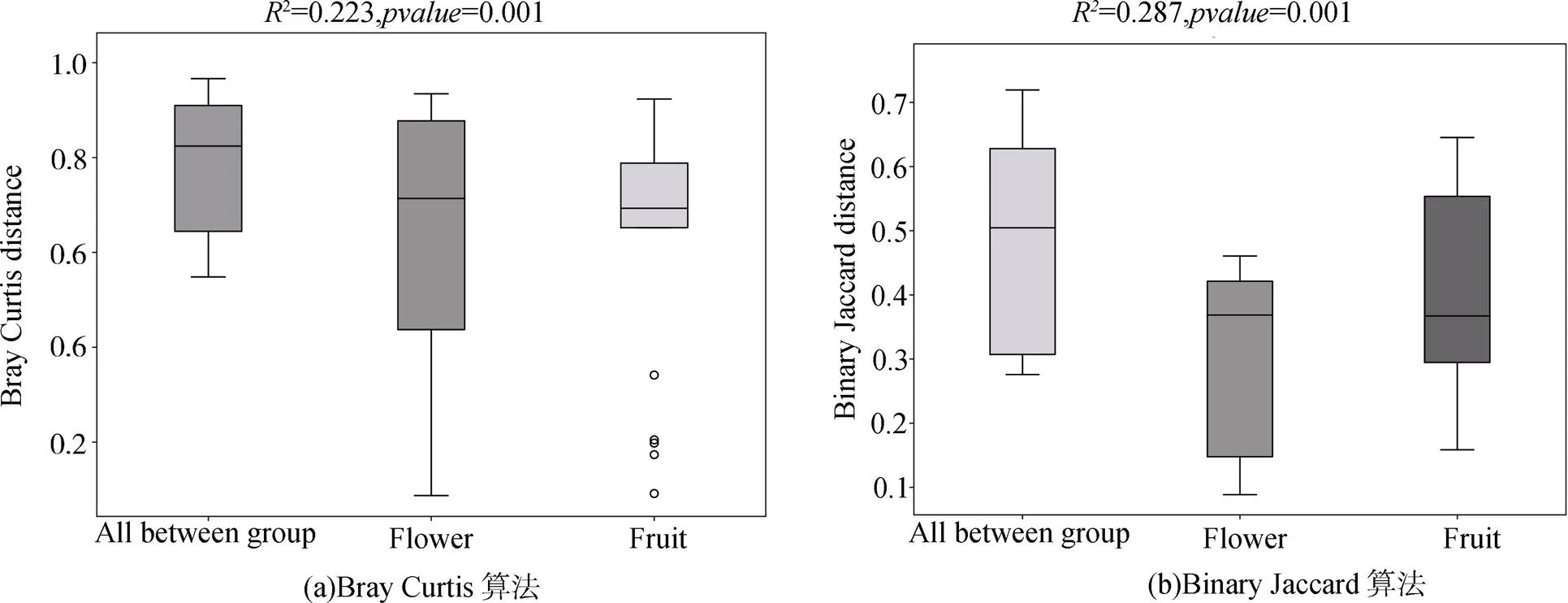
图5 在开花期和结果期的PERMANOVA分析
Figure 5 PERMANOVA analysis of fungi in flowering and fruiting
3 讨论
在花期, 大黄花虾脊兰的内生真菌Tulasnellaceae占优势, 而果期的内生真菌占优势, 其绝对丰度和相对丰度远大于花期的优势内生真菌。这和预期结果一致, 大黄花虾脊兰的优势真菌类群具有阶段性变化的特点, 至少在花期和果期这两个生殖生长期之间, 优势内生真菌类群发生彻底的转变。造成这种转变可能是外在环境因素和植物内在生理生化作用共同促成的结果, 但哪个因素作为主导仍需进一步研究验证。许国琪等研究表明土壤电导率、空气CO2浓度以及土壤温度等环境因子与大部分优势菌显著相关[20]。兰科植物幼苗可与多种真菌共生萌发, 但是进一步发育成成熟植株则会选择只留下与之共生效益最佳的真菌[21]。这在一定程度上解释了优势内生真菌改变的内在原因: 在不同生长时期, 大黄花虾脊兰会允许与其共生效益最佳的内生真菌大量增殖, 逐渐替代别的菌类成为优势种。
根据FUNGuild数据库[22], Tulasnellaceae是被普遍承认的兰科菌根真菌, 而作为兰科菌根真菌则少有报道。且这两类真菌在两个生殖生长期之间存在显著性差异, 推测这两种菌类是大黄花虾脊兰的菌根真菌, 对大黄花虾脊兰的生长繁殖起主要作用。但要确定这些真菌是否是大黄花虾脊兰的菌根真菌, 还需要确认其在植物根内的形态是否为“菌丝结”, 或进行真菌的分离纯化, 再进行真菌回接试验, 确定其是否对大黄花虾脊兰种子的萌发、生长起促进作用。有的植物内生真菌的生态状态极不稳定, 随着生活史进程会在不同的功能营养型之间转换, 采取不同的生存策略来适应环境的变化[23]。相对于内生真菌, 动植物病原菌和木质腐生菌是真菌更常见的生态型[22]。这可能与真菌具有复杂的生活史有关, 当其作为植物内生真菌时能提高植株对各种矿物质元素的吸收进而增大植株生长量, 当植物组织衰败其作为腐生菌时, 以分解死亡有机物为生。
大黄花虾脊兰的内生真菌与土壤真菌共享了至少24个属(12.8%)的真菌类群, 说明有一部分内生真菌可能来自根际土壤。这可能由于土壤真菌长期与植物根紧密接触而有机会突破根的表皮进入植物根内定植[24], 但不是所有的真菌都能突破根的表皮进入植物体, 与植物互利共生成为内生真菌, 这可能是本研究中内生真菌多样性比根际土真菌多样性低的原因。在花期和果期,作为只存在于植物根内而不存在土壤中的真菌, 可能以菌丝或孢子的形式通过花粉或传粉昆虫等载体进入植物体内, 且已有研究表明作为内生真菌对植物的生长有促进作用[25–26], 不排除其作为兰科植物的内生真菌也具有促进植物生长的能力的可能性, 但目前该菌类作为兰属植物的内生真菌还少有报道。
在大黄花虾脊兰中, 优势内生真菌Tulasnellaceae和在根、根际土、根围土3个水平距离上, 真菌丰度呈梯度性递减的趋势。这可能是植物通过分泌某种真菌自身无法合成的化学物质吸引真菌富集在植物根附近, 这些根际真菌与根内真菌连接形成真菌网络作为营养输送“通道”, 由此扩大兰科植物在土壤中吸收营养的范围[27]。此外, 还有人发现兰科植物的菌根真菌主要分布在植物体内及其周围土壤, 其菌根真菌丰度随距兰科植物地理距离增加而下降[28–29], 与本文关于优势内生真菌在不同距离上呈梯度性变化的观点一致, 不同的是Mccormick等和Waud等均将距离植株50 cm的范围作为实验度量, 表明该梯度规律适用于一定的尺度距离范围。
在花期和果期之间, 大黄花虾脊兰的微生境真菌群落在群落结构和组成上存在明显差异, 果期的内生真菌多样性大于花期, 与吕立新等关于内生真菌群落季节动态规律的观点一致[30]。受不同季节的土壤理化性质的影响, 土壤真菌多样性和群落组成产生了明显的变化[31]。内生真菌受温度、降水等环境因子的影响, 随着茅苍术发育进程发生动态变化[32]。推测造成大黄花虾脊兰的微生境真菌群落结构发生显著变化的原因除了植物物候期对应的土壤、温度和降水等环境因子的改变, 还有不同生殖生长期之间植物内在生理生化的变化。菌根真菌在植物内成功定殖之前, 有一个反复被植物消解而让兰科植物获取营养的过程[33]。在花期的植物根内占优势的Tulasnellaceae在果期的植物根内仍存在, 但丰度极小。这可能是该真菌在花期定殖之后被兰科植物反复消化吸收而数量减少, 而后来者提供的营养物质更符合果期兰科植物的需求, 因而大量定殖。至少在对碳的需求上, 果期的兰科植物由叶片向生殖器官运输的14C同化物约为花期的2倍[34]。相对于花期, 果期的兰科植物需要吸收更多的营养物质用于产生种子, 而花期的优势内生真菌不能完全满足这种需求逐渐被植物消化而丰度减小, 果期的优势内生真菌能产生植物激素, 如赤霉素等促进植物生长, 或进一步扩大植物吸收氮源的范围, 满足植物繁育后代的物质需求而大量定殖, 真菌丰度增大[6]。
4 结论
采用生物分子技术对濒危植物大黄花虾脊兰的内生真菌和土壤真菌进行检测, 较为全面地反映了其微生境真菌的群落组成、结构及分布规律, 为兰科植物就地保护和迁地保护中涉及的生境评估提供参考。通过分析大黄花虾脊兰开花期和果期的根、根际土、根围土的真菌群落发现受不同生殖生长期的植物生理影响, 大黄花虾脊兰的优势内生真菌随发育进程发生相应转变, 再加上外在环境因子的综合作用, 使得微生境的真菌群落结构和组成发生显著变化。Tulasnellaceae和分别作为花期和果期的大黄花虾脊兰的优势内生真菌, 在水平距离上, 真菌丰度呈梯度性递减的特点。与以往的研究相比, 本研究更关注不同生殖生长期对微生境真菌群落动态变化的影响, 以找出对大黄花虾脊兰生长发育起关键作用的菌类, 为大黄花虾脊兰的保护提供更合理的依据。但仍存在一定不足, 如本研究的大黄花虾脊兰种群仅采自安徽泾县同一个地点, 范围未涉及不同生境下的植物, 不同生境下大黄花虾脊兰的真菌群落结构与分布是否有相似特点和规律有待进一步探讨。
致谢:感谢北京林业大学公共分析测试中心的张峻琦老师在实验过程中的指导。
[1] 黄元贞, 刘海平, 马良, 等. 基于层次分析法评价12种虾脊兰属植物的观赏性和生长适应性[J]. 福建农林大学学报: 自然科学版, 2018, 47(6): 667–672.
[2] 谢丹, 张成, 张梦华, 等. 湖北单子叶植物新记录[J]. 西北植物学报, 2017, 37(4): 815–819.
[3] BERNARD N. Recherches expérimentales sur les orchidées[J]. Revue Générale de Botanique, 1904, 16: 405–451.
[4] 张孝然, 蒲真, 黄治昊, 等. 大黄花虾脊兰生境特征及生存群落物种生态位分析[J]. 植物科学学报, 2017, 35(6): 799–806.
[5] 刘海平, 陈秀萍, 李珺, 等. 大黄花虾脊兰种子特性及无菌播种[J]. 福建农林大学学报: 自然科学版, 2020, 49(1): 29–34.
[6] 伍建榕, 马焕成. 地生兰与菌根真菌共生关系研究[M]. 北京: 科学出版社, 2016.
[7] 王亚妮. 兰科石斛属植物根部内生真菌多样性研究及应用[D]. 北京: 北京林业大学, 2013.
[8] 徐锦堂. 我国天麻栽培50年研究历史的回顾[J]. 食药用菌, 2013, 21(1): 58–63.
[9] 刘艳明, 黄雅丽, 李婷, 等. 野生与人工栽培铁皮石斛内生真菌生物多样性比较及抗炭疽病活性菌株的筛选[J]. 华南农业大学学报, 2020, 41(3): 47–55.
[10] 侯天文, 金辉, 刘红霞, 等. 四川黄龙沟优势兰科植物菌根真菌多样性及其季节变化[J]. 生态学报, 2010, 30(13): 3424–3432.
[11] 张志东, 朱静, 唐琦勇, 等. 不同扩增引物对高通量测序分析盐角草内生真菌多样性的影响[J]. 微生物学通报, 2020, 47(2): 411–419.
[12] WANGYu, SHENG Huafang, HE Yan, et al. Comparison of the levels of bacterial diversity in freshwater, intertidal wetland, and marine sediments by using millions of illumina tags[J]. Applied and Environmental Microbiology, 2012, 78(23): 8264–8271.
[13] KOZICH J J, WESTCOTT S L, BAXTER N T, et al. Development of a dual-index sequencing strategy and curation pipeline for analyzing amplicon sequence data on the MiSeq Illumina sequencing platform[J]. Applied and Environmental Microbiology, 2013, 79(17): 5112–5120.
[14] SHANNON C E. A mathematical theory of communication[J]. The Bell System Technical Journal, 1948, 27(3): 379–423.
[15] SIMPSON E H. Measurement of Diversity[J]. Nature, 1949, 163: 688.
[16] CHAO A. Nonparametric estimation of the number of classes in a population[J]. Scandinavian Journal of Statistics, 1984, 11(4): 265–270.
[17] CHAO A, YANG M C K. Stopping rules and estimation for recapture debugging with unequal failure rates[J]. Biometrika, 1993, 80(1): 193–201.
[18] LOOFT T, JOHNSON T A, ALLEN H K, et al. In-feed antibiotic effects on the swine intestinal microbiome[J]. Proceedings of the National Academy of Sciences, 2012, 109(5): 1691–1696.
[19] SEGATA N, IZARD J, WALDRON L, et al. Metagenomic biomarker discovery and explanation[J]. Genome Biology, 2011, 12(6), doi: 10.1186/gb-2011-12-6-r60.
[20] 许国琪, 刘怡萱, 曹鹏熙, 等. 青藏高原不同生态环境下冰川棘豆()内生细菌多样性研究[J]. 高原科学研究, 2020, 4(1): 20–29.
[21] RASMUSSEN H N. Recent developments in the study of orchid mycorrhiza[J]. Plant and Soil, 2002, 244(1/2): 149–163.
[22] NGUYEN N H, SONG ZW, BATES S T, et al. FUNGuild: an open annotation tool for parsing fungal community datasets by ecological guild[J]. Fungal Ecology, 2016, 20: 241–248.
[23] ARNOLD A E, LUTZONI F. Diversity and host range of foliar fungal endophytes: Are tropical leaves biodiversity hotspots? [J]. Ecology, 2007, 88(3): 541–549.
[24] 周婕, 苗一方, 方楷, 等. 紫茎泽兰内生真菌及其根际土壤真菌的多样性研究[J]. 生态科学, 2019, 38(5): 1–7.
[25] 杜疏炀, 杨自强, 韩婷, 等. 促进拟南芥生长的西红花内生真菌的筛选和研究[J]. 中药材, 2018, 41(6): 1271–1275.
[26] 张娜莎. 红景天内生真菌及其对宿主生长发育影响的初步研究[D]. 太原: 山西大学, 2014: 35–43.
[27] 盖雪鸽, 邢晓科, 郭顺星. 兰科菌根的生态学研究进展[J]. 菌物学报, 2014, 33(04): 753–767.
[28] MCCORMICK M K, TAYLOR D L, WHIGHAM D F, et al. Germination patterns in three terrestrial orchids relate to abundance of mycorrhizal fungi[J]. Journal of Ecology, 2016, 104(3): 744–754.
[29] WAUD M, BUSSCHAERT P, LIEVENS B, et al. Specificity and localized distribution of mycorrhizal fungi in the soil may contribute to co-existence of orchid species[J]. Fungal Ecology, 2016, 20: 155–165.
[30] 吕立新, 王宏伟, 梁雪飞, 等. 不同化学型和季节变化对茅苍术内生真菌群落多样性的影响[J]. 生态学报, 2014, 34(24): 7300–7310.
[31] 任玉连, 范方喜, 彭淑娴, 等. 纳帕海沼泽化草甸不同季节土壤真菌群落结构与理化性质的关系[J]. 中国农学通报, 2018, 34(29): 69–75.
[32] 曹益鸣, 陶金华, 江曙, 等. 茅苍术内生真菌生物多样性与生态分布研究[J]. 南京中医药大学学报, 2010, 26(2): 137–139.
[33] 范黎, 郭顺星, 肖培根. 密花石斛等六种兰科植物菌根的显微结构研究[J]. 植物学通报, 2000, 17(1): 73–79.
[34] 潘瑞炽, 叶庆生. 国兰生理[M]. 北京: 科学出版社, 2006.
Community characteristics of endophytic fungi and soil fungi in the
HUANG Min, JIANG Biao, GAO Dazhong, XIE Bing, WEI Shuyu, XING Shaohua*
School of Ecology and Nature Conservation, Beijing Forestry University, Beijing 100083, China
is one of the rare and endangered plant species which has been identified as an extremely small population in the national-level. Endophytic fungi and soil microorganisms are important factors affecting the growth and breeding of Orchidaceae. In order to find out the community characteristics of endophytic fungi and soil microorganisms associated with, and to promote its breeding and propagation, the high-throughput sequencing was performed on the roots, rhizoplane soil and rhizosphere soil ofcollected from Jingxian County, Anhui Province in flowering and fruiting. We performed Cluster analysis, Alpha diversity analysis, Beta diversity analysis and LEfSe analysis on the obtained sequences to explore the composition and diversity of endophytic fungi and soil fungi in different periods. The results showed that a total of 1127529 valid sequences of fungi were obtained by high-throughput sequencing, which divided into 761 OTUs by cluster analysis, belonging to 8 Phyla, 93 families and 121 genera. Venn diagram analysis showed that 24 genera of fungi in the root and soil were common fungi, whileonly in the root of plants. The fungi ofhad different dominant groups in different periods, with Tulasnellaceae (15.68%) and(56.75%) being dominant in the flowering and fruiting period; the dominant soil fungi were(12.19%) and(15.93%) respectively. The abundance of the dominant endophytic fungi showed a ladder-type decreasing tendency at three different horizontal distance from the root to rhizoplane soil and to rhizosphere soil. The community structure and composition of the microflora fungi community ofhad noticeable seasonal variation in characteristics.
; endophytic fungi; soil fungi; community structure; high throughput sequencing
10.14108/j.cnki.1008-8873.2022.04.014
Q939.5
A
1008-8873(2022)04-111-09
2020-08-03;
2020-09-02
国家林业和草原局野生动植物保护与自然保护区管理司委托项目; 北京市大学生科学研究与创业行动计划项目(S201910022098)
黄敏(1998—), 女, 广西梧州人, 硕士研究生, 主要研究生物多样性保护与利用, E-mail: 623629892@qq.com
通信作者:邢韶华, 男, 博士, 副教授, 主要从事生物多样性保护与利用和自然保护区规划研究, E-mail: steelboy78@163.com
黄敏, 江标, 高大中,等. 大黄花虾脊兰内生真菌及土壤真菌的群落特征研究[J]. 生态科学, 2022, 41(4): 111–119.
HUANG Min, JIANG Biao, GAO Dazhong, et al. Community characteristics of endophytic fungi and soil fungi in the[J]. Ecological Science, 2022, 41(4): 112–119.

