化肥配施有机肥对花生根际细菌群落结构及共存网络的影响①
李桂龙,李朋发,吴 萌,李忠佩*
化肥配施有机肥对花生根际细菌群落结构及共存网络的影响①
李桂龙1,2,李朋发1,2,吴 萌1,2,李忠佩1,2*
(1 土壤与农业可持续发展国家重点实验室(中国科学院南京土壤研究所),南京 210008;2 中国科学院大学,北京 100049)
为评估化肥配施有机肥对花生根际细菌群落的影响,田间试验条件下研究了不施肥(CK)、施化学氮磷钾肥(NPK) 和化学氮磷钾肥+有机肥配施(NPKM) 对花生根际细菌群落多样性、结构和共存网络的影响。结果表明:NPKM处理下花生根际土养分状况明显改善,特别是有效磷的含量,较CK和NPK处理分别提高了5.31倍 ~ 12.16倍和3.24倍 ~ 6.50倍;而NPK处理下花生根际土养分状况并没有明显提升;SourceTracker 分析显示,NPKM处理下根际细菌群落中只有2.1% ~ 5.5% 的物种来源于有机肥自身,但其花生根际细菌群落多样性在生育期前期要明显高于CK和NPK处理,而CK和NPK处理下根际细菌群落多样性差异不大;限制性主成分分析(CAP) 显示,施肥措施和生育期均对根际细菌群落结构产生了显著影响,NPKM处理下花生根际土中富集了属于根瘤菌目(Rhizobiales)、梭菌目(Clostridiales)和芽孢杆菌目(Bacillales)这一类根际促生菌,根际细菌共存网络结构更加复杂,且网络中重要连接点的比例要也明显高于CK和NPK处理网络。可见,化肥配施有机肥有助于构建一个健康稳定的根际细菌群落。
化肥配施有机肥;根际微生物;细菌共存网络
为了满足不断增长的人口对粮食的需求,在过去的50年里,全世界的化肥使用量增加了700%[1]。然而,过量施用化肥会产生一系列环境问题,包括土壤质量退化、地下水污染及温室气体排放增加等[2]。因此,其他替代性农艺措施如化肥配施有机肥已被广泛采用,以缓解上述因过量使用化肥所导致的环境问题[3-4]。化肥配施有机肥的目标是在维持环境可持续的同时,保证农业生态系统的服务能力,而土壤微生物对农业生态系统的服务能力至关重要[5]。
根际微生物群落被称为植物的“扩展基因组”或“第二基因组”,在决定植物生长发育方面起着重要作用[6]。以往的研究表明,施肥措施会影响土壤和植物相关微生物群落,进而影响农田生态系统服务能力[7]。因此,在越来越多地采用化肥配施有机肥的情况下也需要评估其对微生物群落的影响。以往比较常规施肥和化肥配施有机肥措施对土壤微生物的影响的研究大多集中在土壤微生物群落上。例如,刘佳等[8]发现有机无机配施后显著增加了旱地红壤细菌群落的多样性;Bi等[9]发现化肥配施有机肥通过调控土壤解磷菌群落提升了稻田土壤磷的可利用性。尽管有关化肥配施有机肥对根际微生物群落的影响已有相关报道[10-11],但这些研究多集中于某一生育期下施肥措施对根际微生物群落的影响。由于多种因素的影响,根际微生物群落在不同的生育期具有明显的差异性。因此,有必要了解化肥配施有机肥措施下根际微生物群落随生育期的动态变化,这对于评估化肥配施有机肥对根际微生物的影响具有重要意义。
微生物群落的结构对其功能有重要影响。然而,由于微生物群落众多成员之间的相互关系十分复杂,这使得研究微生物群落的结构成为一项艰巨的任务。微生物共存网络分析可以揭示这种复杂的相互关系,能够为群落结构的研究提供新的见解[12-13]。目前,网络分析已被用于探索不同环境中不同微生物类群之间的相互作用[14-15]。网络分析还可以识别对微生物群落结构和潜在功能影响最大的关键物种或其他重要微生物[13],而这些往往很难用微生物生态学中广泛使用的标准α或β多样性指标来揭示。Fan等[10-11]通过网络分析发现,化肥配施有机肥能够提升根际生态网络中关键模块的微生物群落稳定性及关键微生物菌群的多样性,进而提高小麦产量。可见,利用网络分析探究施肥措施对根际微生物群落相互作用的影响,可能会帮助人们向预期方向调节植物微生物群,提高农业生态系统的资源利用效率。
花生是我国重要的油料作物和经济作物,然而由于不合理的农业管理措施,造成了花生产量和品质的持续下降[16]。提高花生产量和品质的一个潜在方法是通过合理的农艺措施来调控支持植物健康的微生物群落以发挥其促生功能,特别是那些在根际的微生物群落。为此,本研究通过田间试验,以花生这一典型作物为研究对象,研究化肥配施有机肥对花生根际细菌群落结构和共存网络的影响,以期为管理丰富的植物微生物组提供科学依据。
1 材料与方法
1.1 试验地概况
田间试验设置于江西省农业科学院赣东北红壤综合试验区,该试验区位于江西省抚州市东乡县境内(116°35′11″E、28°10′59″N),属亚热带湿润气候区,过去50年平均年降水量为1 750 mm,降水主要集中在4—6月。试验地土壤类型为第四纪红黏土母质发育而来的红壤,表层土壤(0 ~ 20 cm)的基础养分特性为:pH 4.49,有机质13.82 g/kg,全氮1.11 g/kg,全磷0.46 g/kg,全钾11.75 g/kg,碱解氮90.0 mg/kg,有效磷18.8 mg/kg,速效钾130.4 mg/kg。
1.2 试验设计与样品采集
田间试验始于2014年,试验设3个处理:①不施肥(CK),②施化学氮磷钾肥(NPK),③化学氮磷钾肥配施有机肥(NPKM)。肥料年施用量为:N 135 kg/hm2、P2O581 kg/hm2和K2O 135 kg/hm2。在NPKM处理中,50% 的 N 来源于化肥,50% 的 N 来源于有机肥,即保证总施氮量不变。氮肥使用尿素(N,460 g/kg),磷肥使用过磷酸钙(P2O5,120 g/kg),钾肥使用氯化钾(K2O,600 g/kg),有机肥使用猪粪(每年施用前测定其N含量)。所有肥料在作物种植前一次性基施,肥料基施方式为株间穴施。试验设3次重复,随机区组排列,小区面积为27 m2(6 m × 4.5 m)。供试花生品种为粤油256,于4月中旬条播,行距40 cm,穴距20 cm,每穴播种2粒,种植密度约为12.5 万穴/hm2。
于2020年在花生苗期(5月18日,T1)、花针期(6月17日,T2)、结荚期(7月16日,T3)各取样1次。采样时每小区分作2个半区进行,每个半区取长势相对一致具有代表性的花生样品5株,将花生完全取出后,轻轻抖动附着在根上的土壤,然后用无菌毛刷轻轻刷下紧密结合在根表的那部分土壤,即为根际土,彻底混匀后作为1个样品,即每处理获得6个重复样品。土样采集后尽快带回室内,拣除杂质后过2 mm筛,而后将每个样品分为两份:一份风干用于测定土壤化学性质,一份保存在–80℃的冰箱中用于提取土壤DNA。另外,在花生成熟期时统计各处理小区内所有花生产量。
1.3 土壤化学性质测定
土壤化学性质的测定依照鲁如坤[17]的方法进行:采用电位法测定土壤pH(水︰土=2.5︰1,/),采用重铬酸钾氧化–容量法测定土壤有机碳(SOC),采用凯氏定氮法测土壤全氮(TN),采用碳酸钠熔融法测定土壤全磷(TP),采用碱解扩散法测定土壤碱解氮(AN),采用碳酸氢钠浸提–钼锑抗比色法测定土壤有效磷(AP)。
1.4 土壤DNA提取和16S rRNA基因高通量测序
称取500 mg土壤样品用FastDNA ® SPIN Kit for Soil试剂盒(MP Biomedicals,美国)提取DNA,具体方法和步骤依据试剂盒说明书进行。有机粪肥的DNA提取也采用上述方法进行。采用通用引物515F/907R 扩增细菌16S rRNA 基因片段[18]。将PCR扩增后的产物用2% 琼脂糖凝胶电泳检测,纯化后建库,采用Illumina MiSeq系统进行双向高通量测序(上海美吉)。
1.5 高通量测序数据分析
基于QIIME1分析平台进行高通量数据分析。序列经质控、拼接、比对后在97% 相似性水平划分操作分类单元(operational taxonomic unit,OTU),通过与RDP数据库(http://rdp.cme.msu.edu/classifier/ classifier.jsp)比较确定细菌序列的系统学分类。
1.6 数据处理
花生产量、土壤化学性质、细菌多样性指标采用Duncan多重比较来判断差异显著性(<0.05)。以NPKM处理的样品为对照,以有机粪肥样品为来源,采用SourceTracker2[19]来预测NPKM处理下样品中细菌物种来源于有机粪肥中细菌物种的比例。采用R软件vegan包的capscale()函数进行限制性主成分分析(partial canonical analysis of principal coordinates,CAP),以探究施肥及生育期对细菌群落结构的影响。Mantel分析利用R软件vegan包中的mantel()函数执行。采用R软件edgeR包的decideTestsDGE()函数分析不同施肥处理之间的差异物种。基于Pearson相关性矩阵的共存网络分析利用R软件WGCNA包中的corAndPvalue()函数计算相关性系数。首先,剔除出现频次少的OTU(保留在1/3及以上的样品中出现的OTU)。值经过BH矫正,之后删除值小于0.85且值大于0.0001的值。网络参数平均度、连通性、平均路径长度分别利用R软igraph包中的degree()、edge_connectivity()、average.path.length()函数进行计算。通过计算节点的值(within-module connectivity,模块内连通性)和值(among-module connectivity,模块间连通性)来对网络中的节点进行分类[20]。将≤2.5且≤0.62的节点定义为边缘节点(peripherals),这类节点在整个网络中与其他节点联系较少;将>2.5且≤0.62的节点定义为模块中心节点(module hubs),这类节点在网络模块内部连接性较高;将≤2.5且>0.62的节点定义为重要连接点(connectors),这类节点为联系模块之间的枢纽;将>2.5且>0.62的节点定义为网络中心节点(network hubs),这类节点为在整个网络和模块中连接性都较高的节点。网络图在Gephi(https://gephi.org/)软件中绘制;和弦图用R软件circlize包绘制;其他图均用R软件ggplot2包绘制完成。
2 结果
2.1 不同施肥措施下花生产量
花生的产量统计结果(表1)显示,NPKM处理可以显著提高花生产量,较CK处理提升了0.8倍;而NPK处理下花生产量与CK相比并没有明显提升。
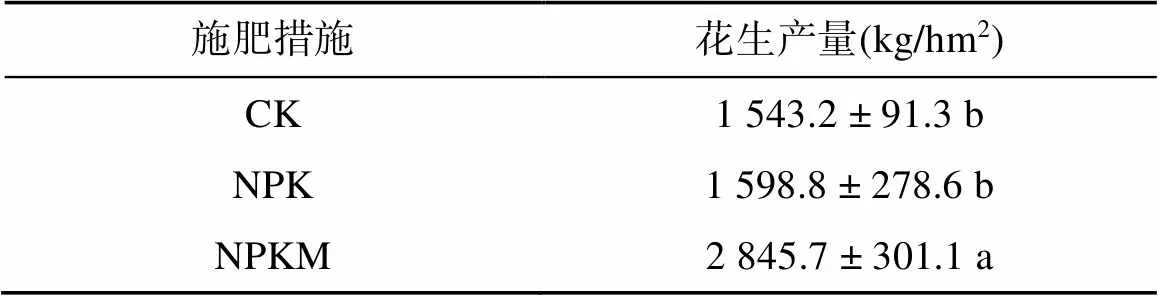
表1 不同施肥措施下花生产量
注:表中同列不同小写字母表示处理间差异显著(<0.05)。
2.2 不同施肥措施下土壤化学性质
不同施肥处理下,花生根际土的化学性质产生了明显的变化(表2)。CK和NPK处理下根际土的化学性质差异不显著(>0.05),仅在T2时期观察到NPK处理下根际土的TP含量显著高于CK处理(<0.05)。与CK和NPK处理相比,NPKM处理下根际土的pH较高,在T2时期显著高于其他两个处理(<0.05);其余指标,NPKM处理下均显著提高(<0.05),特别是AP含量,较CK和NPK处理分别提高了5.31倍 ~ 12.16倍和3.24倍 ~ 6.50倍。
2.3 有机肥细菌群落的分类组成及其向土壤的潜在传递
有机肥中细菌的群落与NPKM处理下花生根际细菌群落组成有很大不同(图1A)。有机肥中细菌群落中拟杆菌门(Bacteroidetes)、γ-变形杆菌(Gammaproteobacteria)、α-杆菌(Alphaproteobacteria)和厚壁菌门(Firmicutes)细菌门类占据主导优势;而NPKM处理下花生根际细菌群落以α-杆菌、放线菌门(Actinobacteria)、酸杆菌门(Acidobacteria)、β-变形杆菌(Betaproteobacteria)和绿弯菌(Chloroflexi)为主。此外,有机肥中细菌群落的多样性明显小于NPKM处理下花生根际细菌群落多样性(图1B)。SourceTracker的结果显示,NPKM处理下根际细菌群落中只有2.1% ~ 5.5%(T1时期:5.5%;T2时期:3.7%;T3时期:2.1%)的物种可能来自于有机肥自身所携带的细菌物种(图1C)。以上结果说明,有机肥中的细菌组成对花生根际细菌种群结构产生的影响甚小。

表2 不同施肥措施对根际土壤化学性质的影响
注:同列不同小写字母表示同一时期不同处理间差异显著(<0.05)。

(M代表有机粪肥;NPKM_T1、NPKM_T2、NPKM_T3代表T1 ~ T3时期下NPKM处理下花生根际土样品;图B中不同小写字母表示处理间差异显著(P<0.05))
2.4 不同施肥措施下花生根际微生物群落特征
与CK和NPK处理相比,NPKM处理在生育期前期显著提高了花生根际细菌多样性;而在T3时期各处理间细菌多样性没有明显差异。CK和NPK处理下根际细菌多样性差异不大,仅在T2时期NPK处理下细菌多样性相对于CK显著增加,但增加的幅度要明显小于NPKM处理(图2)。
CAP分析表明,施肥和生育期均对根际细菌群落结构产生了显著影响,分别解释了整个群落变异的13.1%(施肥:=0.001)和16.7%(生育期:=0.001)(图3)。CK和NPK处理沿着第二轴分开,而NPKM处理与CK和NPK处理沿着第一轴分开,说明NPKM处理与CK(NPK)处理细菌群落的差异要大于CK与NPK处理细菌群落的差异(图2A)。T2和T3时期细菌群落沿着第二轴分开,而T1时期细菌群落与T2和T3时期细菌群落沿着第一轴分开,说明T1与T2(T3)时期细菌群落的差异要大于T2与T3时期细菌群落的差异(图2B)。Mantel检验表明,各土壤化学指标均显著影响细菌群落结构;其中AP对细菌群落结构的影响最大(表3)。
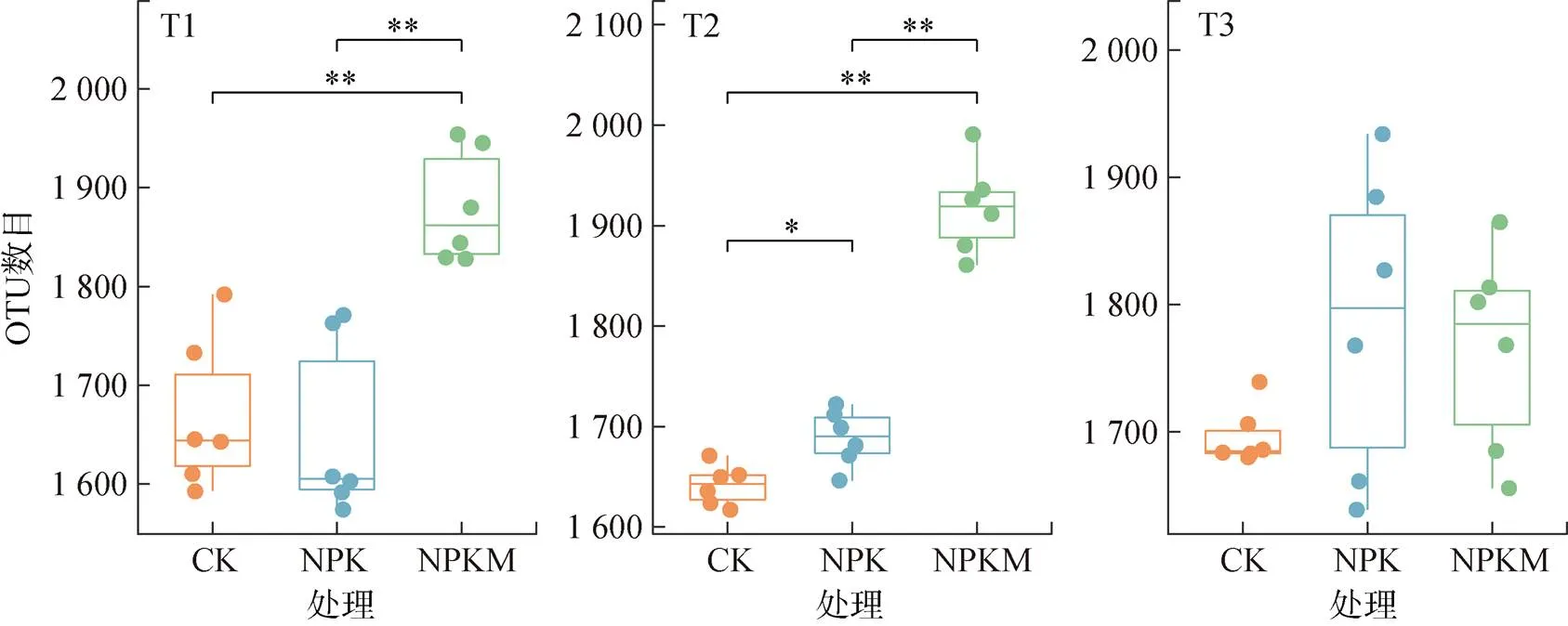
(图中*表示在P<0.05 水平上差异显著,**表示在P<0.01水平上差异显著)

(不同变量(施肥措施和生育期)对整个根际细菌群落变异的解释率标注于图的上方)
2.5 不同施肥措施下土壤细菌差异OTU
在T1时期,CK处理富集了9个OTU,主要归属于根瘤菌目(Rhizobiales)和厌氧绳菌目(Anaerolineales);NPK处理富集了20个OTU,主要归属于纤线杆菌目(Ktedonobacterales)和变形杆菌(Proteobacteria)下一个未鉴别的目;NPKM处理富集了53个OUT,大部分归属于根瘤菌目(Rhizobiales)、鞘脂单胞菌目(Sphingomonadales)、芽孢杆菌目(Bacillales)和梭菌目(Clostridiales)(图4A,4D)。在T2时期,有8个OTU在CK处理中富集,主要隶属于鞘氨醇杆菌目( Chitinophagales);只有4个OTU在NPK处理下富集;而NPKM处理下富集了70个OTU,主要隶属于根瘤菌目、梭菌目、红螺菌目(Rhodospirillales)、鞘脂单胞菌目(Sphingomonadales)、芽孢杆菌目和弗兰克氏目(Frankiales)(图4B,4E)。同样,在T3时期NPKM处理下富集了最多数目的OTU,主要归属于根瘤菌目、梭菌目、酸杆菌暂定目(Candidatus Solibacter)、红螺菌目和鞘脂单胞菌目(图4C,4F)。总体上看,与CK和NPK处理相比,NPKM处理下花生根际属于根瘤菌目、梭菌目和芽孢杆菌目的物种丰度显著增加。
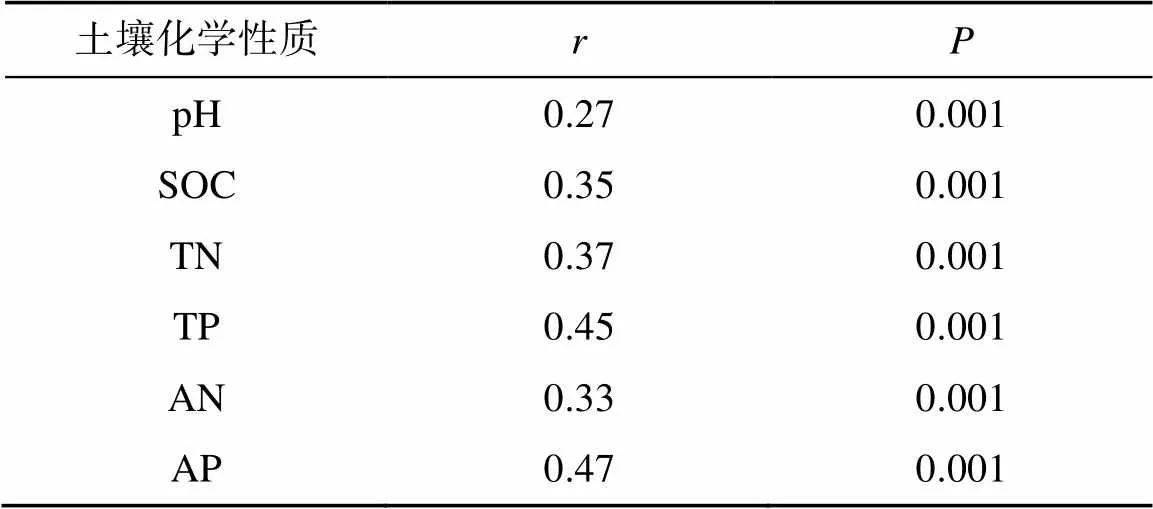
表3 根际细菌群落组成与土壤化学性质的Mantel分析
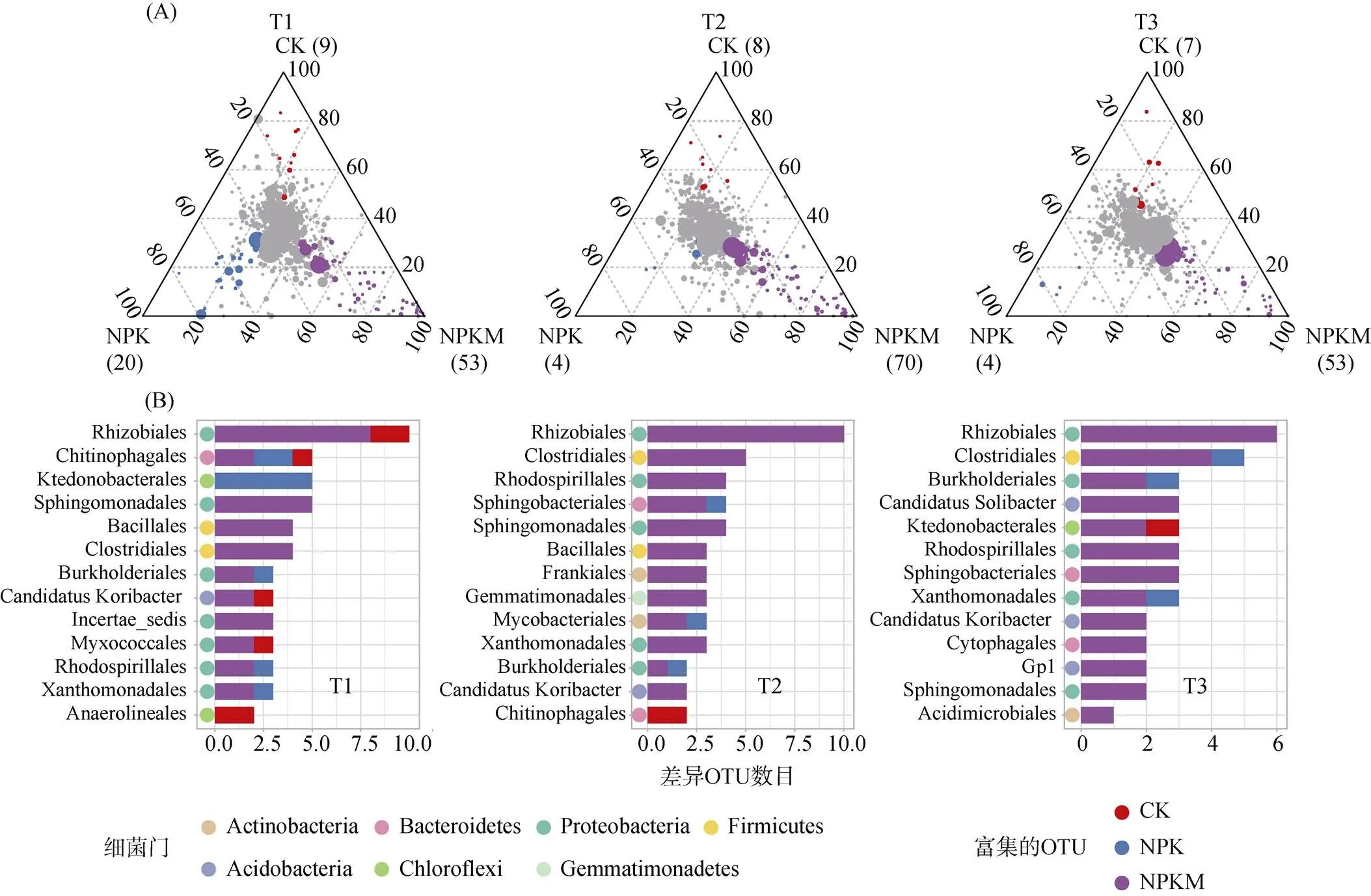
(图A中每一个点代表一个OTU,点的大小代表每个OTU在所有样品中的平均相对丰度,点的位置代表该OTU的相对丰度在所有处理中的相对比例,彩色的点表示与其他两组处理比较,只在该组中显著富集的OTU(橙色代表只在CK处理中显著富集,蓝色代表只在NPK处理中显著富集,紫色代表只在NPKM处理中显著富集),灰色的点代表3组间两两比较无显著差异的OTU,括号中的数字代表每个处理下富集的OTU数目;图B中每条柱子左边彩色的点代表该分类单元所属的门)
2.6 不同施肥措施下花生根际细菌群落的共存网络特征
对不同施肥处理下的花生根际细菌群落进行共存网络分析,以探索根际细菌之间的相互作用(图5A)。CK处理下的细菌网络包含144个节点,159条边;NPK处理下的细菌网络包含237个节点,323条边;NPKM处理下细菌网络包含288个节点,620条边。NPKM处理下根际细菌网络与其他处理下的根际细菌网络相比具有更高的连通性(connectivity)和平均度(average degree),表明NPKM处理下花生根际细菌之间的相互作用要比其他处理下复杂。而NPKM处理下细菌网络中平均路径长度(average path length)却低于其他处理,表明NPKM处理下细菌网络中物种间的信息传递效率更高。
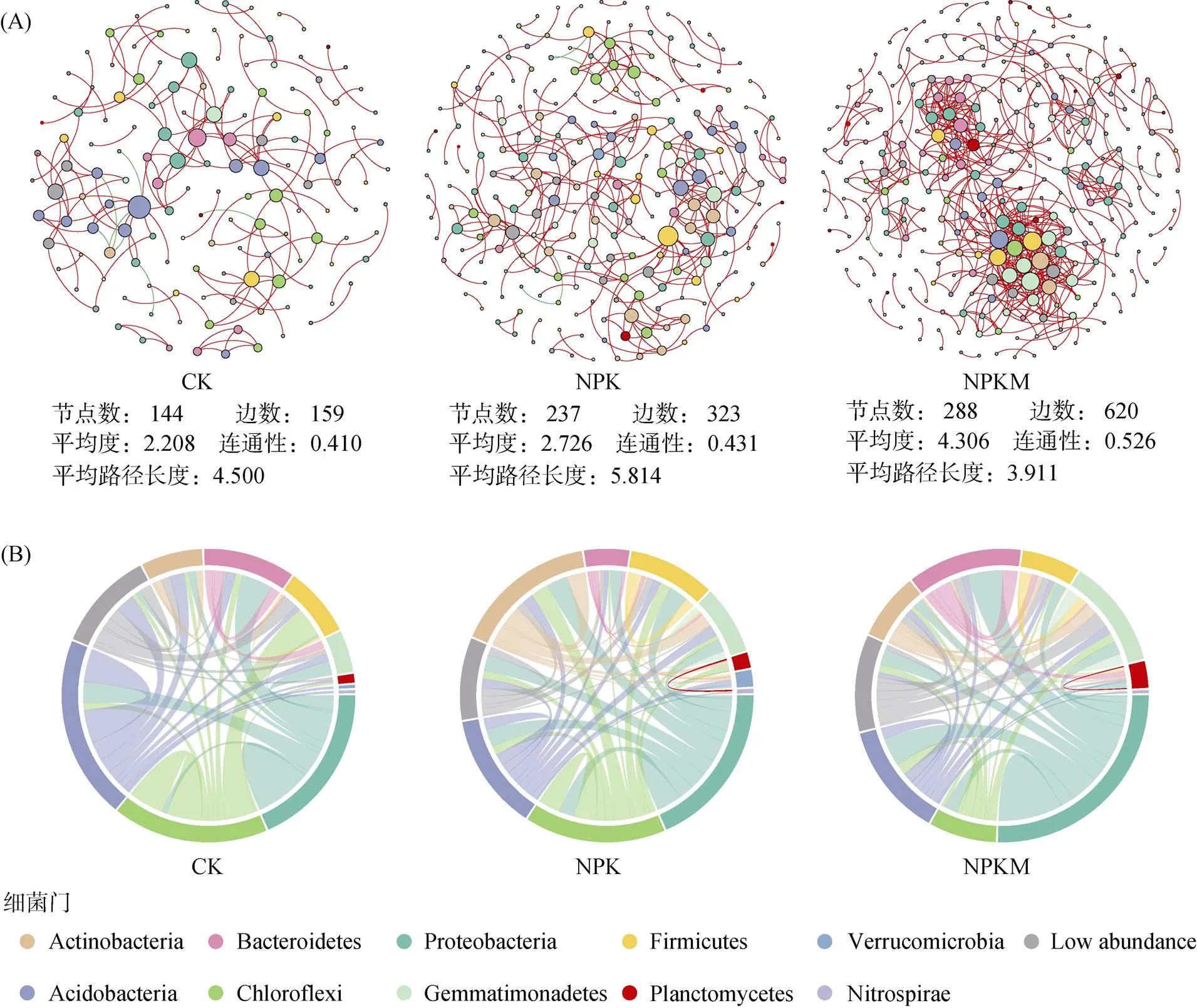
(A图中每个节点代表一个OTU,点的大小代表节点的度,边的颜色代表正负相关性(红色正相关,绿色负相关),根据节点所属的门对其着色;B图中两个扇形之间的条带代表两个门之间的连接数,每个条带的宽度代表其对应的连接数占整个网络连接数的比例,扇形依据其所属的门进行着色)
各施肥处理下网络中细菌间的相互作用(边)在门水平上的分布模式明显不同(图5B)。在NPK和NPKM处理网络中,对细菌相互作用贡献最大的是变形杆菌门(Proteobacteria);而在CK处理网络中,贡献最大的是酸杆菌门(Acidobacteria)。拟杆菌门(Bacteroidetes)在NPK处理网络中占有的连接数的比例(9.29%)要远远小于其在CK和NPKM处理网络中(CK:18.24%;NPKM:21.77%);而放线菌门(Actinobacteria)在NPK处理网络中所占有的边的比例(27.24%)要远高于其在CK和NPKM处理网络中(CK:11.94%;NPKM:14.35%)。此外,绿湾菌门(Chloroflexi)在CK和NPK处理网络中对细菌相互作用的贡献(CK:25.79%;NPK:26%)要大于其在NPKM处理网络中(14.19%);而芽单胞菌门(Gemmatimonadetes)在NPKM处理网络中所占有的边的比例(21.94%)要远高于其在CK和NPK处理网络中(CK:9.43%;NPK:13.93%)。
各处理下的网络中均没有观察到网络中心节点;CK和NPK处理网络中各检测到一个模块中心节点,而NPKM处理网络中没有检测到此类节点(图6)。值得注意的是,NPKM处理网络中重要连接点的比例要明显高于CK和NPK处理网络,表明NPKM处理下花生根际细菌群落之间的相互作用更加密切。
3 讨论
化肥配施有机肥料能够有效提升土壤肥力、改善土壤生态环境[21]。本研究中,NPKM处理下花生根际土的AP含量显著提高,这与Chen等[22]的田间试验结果相似。一方面,有机肥自身所含有的大量有机磷通过矿化转变为无机磷,提高了根际土壤的AP含量。另一方面,有机碳的添加提升了土壤微生物利用难溶性磷的能力,从而活化出更多的可利用性磷[23]。其他土壤养分指标,如SOC、TN、TP和AN在NPKM处理下含量也明显升高。同时,与CK相比,NPK处理下花生根际的养分状况并没有得到明显改善。刘佳等[8]的研究也表明,旱地红壤经过长期施肥,NPK处理对土壤肥力的提升效果远不如NPKM处理。本研究田间产量数据也表明,经过长达7年的连续施用化学氮磷钾肥 (NPK处理),相对于CK,花生的产量并没有明显提升,而化肥配施有机肥措施下(NPKM处理)花生产量较CK提高了0.8倍。这说明在红壤旱地花生生产中,化肥有机肥配施要优于单施化学氮磷钾肥。

(图中百分数代表各类节点占整个网络中节点总数的比例,图中垂直虚线代表横坐标值为0.62,水平虚线代表纵坐标值为2.5)
土壤微生物多样性在支持多种生态系统功能中起着至关重要的作用[24]。本研究中,尽管在最后一个时期各处理间根际细菌群落多样性差异不大,但在前期NPKM处理下花生根际细菌群落多样性明显提高。有研究表明,有机肥的添加为土壤微生物提供了充足的底物,有利于微生物在根际定殖,导致根际微生物多样性增加[25]。此外,外源添加的有机粪肥自身携带的大量微生物也是造成NPKM处理下根际细菌群落多样性提高的一个重要原因[26]。本研究通过比较有机粪肥与NPKM处理样品中细菌群落组成与多样性的差异,发现有机粪肥自身携带的细菌物种与NPKM处理样品中细菌群落组成明显不同,其群落多样性远小于NPKM处理;Source Tracker分析也表明,NPKM处理样品中细菌物种只有很少的比例来自于有机粪肥自身携带的细菌物种。说明NPKM处理下花生根际细菌群落多样性提高主要是由有机粪肥中充足的养分造成的。根际微生物在植物生产力、养分吸收和抗病能力方面发挥着关键作用[6]。本研究表明,NPKM处理下花生根际属于根瘤菌目(Rhizobiales)、梭菌目(Clostridiales)和芽孢杆菌目(Bacillales)的物种的相对丰度明显增加。以往的研究表明,属于梭菌目和芽孢杆菌目的菌属具有促生作用,对植物的健康生长至关重要[27-28];属于根瘤菌目(Rhizobiales)的根瘤菌能够与豆科植物互作,行使共生固氮作用,对陆地氮循环具有重要意义[29];且豆科植物根际属于根瘤菌目的根瘤菌的丰度和多样性对于根瘤的形成起着至关重要的作用[30]。这在一定程度上也表明,配施有机肥能够通过调控根际有益菌属进而促进花生结瘤固氮及生长,对于农业生产具有指导意义。
共存网络分析为认识土壤微生物群落之间复杂的相互关系提供了新的途径[13]。越来越多的研究表明,共存网络的特性可能代表微生物之间的相互作用,可以影响微生物群落对外界扰动的响应[31-32]。本研究中,共存网络分析表明,NPKM处理下根际细菌网络的拓扑学特性如节点数、连接数、平均度以及连通性明显提高,表明NPKM处理下花生根际细菌群落形成了一个更为复杂的网络。前人的研究表明,土壤养分在调节微生物群落之间相互作用的强度方面起着关键作用[33]。NPKM处理下根际土壤养分状况明显改善,而土壤养分的提升能够为微生物提供更多的资源生态位,因此能够塑造更加复杂的群落结构和互作关系,形成更复杂的共存网络。Santolini和Barabási[34]认为,具有较高连通性的复杂网络比具有较低连通性的简单网络对环境扰动的抵抗性更强。从这一点来说,NPKM处理网络的复杂性可能表明,在化肥配施有机肥管理措施下,花生根际微生物对环境胁迫具有更强的弹性。重要连接点对于整个网络的稳定性和连接性至关重要[35]。本研究发现,NPKM处理网络中未观测到模块中心节点,但重要连接点的比例明显增加。这意味着与其他处理相比,NPKM处理下花生根际细菌群落能够维持相对稳定的状态,进一步表明化肥配施有机肥有助于花生根际细菌群落形成更稳定的网络结构以增强根际微生物对外界干扰的抵抗力。
4 结论
化肥配施有机肥能够有效提升花生根际的养分水平并提高根际细菌群落的多样性。化肥配施有机肥改善了花生根际细菌群落结构,富集了有益于花生结瘤固氮和生长的物种。化肥配施有机肥有助于根际细菌群落形成复杂的共生网络,同时增加了网络中重要节点的比例,有利于根际细菌群落应对复杂多变的根际环境。
[1] Tilman D, Cassman K G, Matson P A, et al. Agricultural sustainability and intensive production practices[J]. Nature, 2002, 418(6898): 671–677.
[2] Bender S F, Wagg C, van der Heijden M G A. An underground revolution: Biodiversity and soil ecological engineering for agricultural sustainability[J]. Trends in Ecology & Evolution, 2016, 31(6): 440–452.
[3] Liang Q, Chen H Q, Gong Y S, et al. Effects of 15years of manure and inorganic fertilizers on soil organic carbon fractions in a wheat-maize system in the North China Plain[J]. Nutrient Cycling in Agroecosystems, 2012, 92(1): 21–33.
[4] Chivenge P, Vanlauwe B, Six J. Does the combined application of organic and mineral nutrient sources influence maize productivity? A meta-analysis[J]. Plant and Soil, 2011, 342(1/2): 1–30.
[5] Delgado-Baquerizo M, Maestre F T, Reich P B, et al. Microbial diversity drives multifunctionality in terrestrial ecosystems"[J]. Nature Communications, 2016, 7: 10541.
[6] Compant S, Samad A, Faist H, et al. A review on the plant microbiome: Ecology, functions, and emerging trends in microbial application[J]. Journal of Advanced Research, 2019, 19: 29–37.
[7] Gu S S, Hu Q L, Cheng Y Q, et al. Application of organic fertilizer improves microbial community diversity and alters microbial network structure in tea () plantation soils[J]. Soil and Tillage Research, 2019, 195: 104356.
[8] 刘佳, 陈晓芬, 刘明, 等. 长期施肥对旱地红壤细菌群落的影响[J]. 土壤学报, 2020, 57(2): 468–478.
[9] Bi Q F, Li K J, Zheng B X, et al. Partial replacement of inorganic phosphorus (P) by organic manure reshapes phosphate mobilizing bacterial community and promotes P bioavailability in a paddy soil[J]. Science of the Total Environment, 2020, 703: 134977.
[10] Fan K K, Delgado-Baquerizo M, Guo X S, et al. Microbial resistance promotes plant production in a four-decade nutrient fertilization experiment[J]. Soil Biology and Biochemistry, 2020, 141: 107679.
[11] Fan K, Delgado-Baquerizo M, Guo X, et al. Biodiversity of key-stone phylotypes determines crop production in a 4-decade fertilization experiment[J]. The ISME Journal, 2021, 15(2): 550–561.
[12] Barberán A, Bates S T, Casamayor E O, et al. Using network analysis to explore co-occurrence patterns in soil microbial communities[J]. The ISME Journal, 2012, 6(2): 343–351.
[13] Deng Y, Jiang Y H, Yang Y F, et al. Molecular ecological network analyses[J]. BMC Bioinformatics, 2012, 13: 113.
[14] Li D, Ni H W, Jiao S, et al. Coexistence patterns of soil methanogens are closely tied to methane generation and community assembly in rice paddies[J]. Microbiome, 2021, 9(1): 20.
[15] Yuan M M, Guo X, Wu L, et al. Climate warming enhances microbial network complexity and stability">[J]. Nature Climate Change, 2021, 11(4): 343–348.
[16] Liu W X, Wang Q L, Wang B Z, et al. Changes in the abundance and structure of bacterial communities under long-term fertilization treatments in a peanut monocropping system[J]. Plant and Soil, 2015, 395(1/2): 415–427.
[17] 鲁如坤. 土壤农业化学分析方法[M]. 北京: 中国农业科技出版社, 2000.
[18] Biddle J F, Fitz-Gibbon S, Schuster S C, et al. Metagenomic signatures of theMargin subseafloor biosphere show a genetically distinct environment[J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(30): 10583–10588.
[19] Knights D, Kuczynski J, Charlson E S, et al. Bayesian community-wide culture-independent microbial source tracking[J]. Nature Methods, 2011, 8(9): 761–763.
[20] Poudel R, Jumpponen A, Schlatter D C, et al. Microbiome networks: A systems framework for identifying candidate microbial assemblages for disease management[J]. Phytopathology®, 2016, 106(10): 1083–1096.
[21] Diacono M, Montemurro F. Long-term effects of organic amendments on soil fertility. A review[J]. Agronomy for Sustainable Development, 2010, 30(2): 401–422.
[22] Chen Y, Sun R B, Sun T T, et al. Organic amendments shift the phosphorus-correlated microbial co-occurrence pattern in the peanut rhizosphere network during long-term fertilization regimes[J]. Applied Soil Ecology, 2018, 124: 229–239.
[23] Liu E K, Yan C R, Mei X R, et al. Long-term effect of chemical fertilizer, straw, and manure on soil chemical and biological properties in northwest China[J]. Geoderma, 2010, 158(3/4): 173–180.
[24] Cardinale B J, Matulich K L, Hooper D U, et al. The functional role of producer diversity in ecosystems[J]. American Journal of Botany, 2011, 98(3): 572–592.
[25] Tang H, Xu Y, Sun J, et al. Soil enzyme activities and soil microbe population as influenced by long-term fertilizer management under an intensive cropping system[J]. Journal of Pure and Applied Microbiology, 2014, 8: 15–23.
[26] Watts D B, Torbert H A, Feng Y C, et al. Soil microbial community dynamics as influenced by composted dairy manure, soil properties, and landscape position[J]. Soil Science, 2010, 175(10): 474–486.
[27] Niu D D, Liu H X, Jiang C H, et al. The plant growth-promoting rhizobacteriumAR156 induces systemic resistance inby simultaneously activating salicylate- and jasmonate/ ethylene-dependent signaling pathways[J]. Molecular Plant-Microbe Interactions: MPMI, 2011, 24(5): 533–542.
[28] Polianskaia L M, Vedina O T, Lysak L V, et al. The growth-promoting effect ofandsp. cultures on some agricultural crops[J]. Mikrobiologiia, 2002, 71(1): 123–129.
[29] Coskun D, Britto D T, Shi W M, et al. How plant root exudates shape the nitrogen cycle[J]. Trends in Plant Science, 2017, 22(8): 661–673.
[30] Suzaki T, Kawaguchi M. Root nodulation: A developmental program involving cell fate conversion triggered by symbiotic bacterial infection[J]. Current Opinion in Plant Biology, 2014, 21: 16–22.
[31] de Vries F T, Griffiths R I, Bailey M, et al. Soil bacterial networks are less stable under drought than fungal networks[J]. Nature Communications, 2018, 9: 3033.
[32] Wei Z, Gu Y A, Friman V P, et al. Initial soil microbiome composition and functioning predetermine future plant health[J]. Science Advances, 2019, 5(9): eaaw0759.
[33] Fernandez C W, Kennedy P G. Revisiting the 'Gadgil effect': Do interguild fungal interactions control carbon cycling in forest soils? [J]. The New Phytologist, 2016, 209(4): 1382–1394.
[34] Santolini M, Barabási A L. Predicting perturbation patterns from the topology of biological networks[J]. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(27): E6375–E6383.
[35] Guimerà R, Nunes Amaral L A". Functional cartography of complex metabolic networks[J]. Nature, 2005, 433(7028): 895–900.
Effects of Chemical Fertilizer Combined with Organic Manure on Peanut Rhizosphere Bacterial Community Structure and Co-occurrence Network
LI Guilong1, 2, LI Pengfa1, 2, WU Meng1, 2, LI Zhongpei1, 2*
(1 State Key Laboratory of Soil and Sustainable Agriculture, Institute of Soil Science, Chinese Academy of Sciences, Nanjing 210008, China; 2 University of Chinese Academy of Sciences, Beijing 100049, China)
To evaluate the effects of different fertilization on peanut rhizosphere bacterial community, three fertilization treatments were conducted in the field plot experiments. The treatments include no fertilizer (CK), nitrogen, phosphorus and potassium fertilizer (NPK), and application of NPK plus manure (NPKM). The diversity, community composition, and co-occurrence network of the rhizospheric bacteria were studied. The results showed that NPKM significantly improved the nutrient status in peanut rhizosphere soil, especially for the content of available phosphorus, which was 5.31–12.16 and 3.24–6.50 times higher than those of CK and NPK, respectively. However, NPK did not significantly improve the nutrient status of peanut rhizosphere soil. The results of SourceTracker showed that only 2.1%–5.5% of the bacteria community in NPKM potentially originated from organic manure. In the early growth stage, NPKM had a significantly higher bacterial community diversity as compared with CK or NPK. However, no significant differences of bacterial community diversity were detected between the CK and NPK. Partial canonical analysis of principal coordinates (CAP) corroborated that both growth stages and fertilization regimes had significant effects on the assembly of rhizosphere bacterial communities. A number of beneficial bacterial OTUs classified to Rhizobiales, Clostridiales, and Bacillales, were significantly enriched under NPKM. Rhizosphere bacterial co-occurrence network was most complex under NPKM, and the proportion of connectors was also highest under NPKM. The results indicate that chemical fertilizer combined with organic manure could help to build a healthy and stable rhizosphere bacterial community.
Chemical fertilizer combined with organic manure; Rhizosphere bacteria; Bacteria co-occurrence network
S154.3
A
10.13758/j.cnki.tr.2022.03.009
李桂龙, 李朋发, 吴萌, 等. 化肥配施有机肥对花生根际细菌群落结构及共存网络的影响. 土壤, 2022, 54(3): 498–507.
国家自然科学基金项目(41771298)和江西省重点研发计划一般项目(20203BBF63039)资助。
(zhpli@issas.ac.cn)
李桂龙(1988—),男,山西大同人,博士研究生,主要从事花生根际微生物与其结瘤固氮效率关系的研究。E-mail:glli@issas.ac.cn

