傅立叶变换红外光谱技术对常见食源性致病菌和真菌快速分类鉴别
龚 方,刘小菁,康秋燕,汪 颖,李兆杰,
(1.中共青岛西海岸新区工委军民融合办,山东青岛 266000;2.威海职业学院,山东威海 264210;3.青岛农业大学食品科学与工程学院,山东青岛 266000)
食源性致病菌和真菌毒素都是引起食源性疾病的主要风险因素[1],Hermann 等[2]整理了2015 到2018 年的食品安全综述文章,发现关于致病菌和真菌毒素的综述研究位居常见风险因素排行的前两位,在食品安全研究中处在首要位置,也是各国食品安全管理部门重点关注和监管的检测项目,因此,非常有必要建立有效、准确的食源性致病菌和真菌的分类识别方法以实现快速检测,减少其带来的风险[3]。
近年来,光谱技术越来越多地应用于食品检测的多个领域[4],包括傅立叶变换红外光谱(FT-IR)[5]、拉曼光谱(Raman spectroscopy)[6]、傅立叶变换近红外光谱(Fourier tranform near infrared spectroscopy FT-NIR)[7]等,其中,FT-IR 技术应用较早,研究也更为广泛。早在1984 年,Naumanan 等[8]提出了该技术应用在微生物种类鉴定方面的可能,并研究了种间水平的鉴定方法[9]。FT-IR 技术主要是以完整细胞的FT-IR 光谱特殊指纹区为依据,反映不同菌种蛋白质、核酸等物质特征上的区别[10],采集形成由多种标准菌株光谱构建成的光谱数据库,结合化学计量分析方法,建立有效的聚类模型,实现微生物种间的分类鉴定,一般包括样品制备、光谱采集、谱图处理、数据分析、方法验证等步骤[11]。FT-IR 常见应用于不同细菌的分类研究:代群威等[12]通过谱图分析,验证了FT-IR 技术在人体细菌与土壤细菌鉴定中的应用,慈云祥等[13]对酵母菌、细菌等微生物的分类进行了初步光谱分析,王若男等[14]研究了脂环酸芽孢杆菌等不同杆菌的种间分类鉴定,Lefier 等[15]探索了FT-IR 结合CVA 分析对单核细胞增生李斯特菌进行了亚种分类的研究,并分析了处理方法、培养时间、温度等不同因素对鉴定结果的影响,相对来说,针对真菌分类的研究并不多见。
本文创新性地利用FT-IR 技术构建易造成食品污染的13 种常见致病菌和12 种常见真菌导数谱数据库,结合数据分析模型,建立基于FT-IR 技术的菌种鉴定分类方法,对傅立叶红外光谱技术在微生物鉴定领域的应用是一个新的扩展,也为微生物食品安全风险的快速检测提供新的方法支撑。
1 材料与方法
1.1 材料与仪器
1.1.1 标准菌株 蜡样芽孢杆菌(Bacillus cereus)ATCC 12826、大肠埃希氏菌O157:H7(Escherichia coliO157:H7)ATCC 43895、金黄色葡萄球菌(Staphylococcus aureus)ATCC 12600、表皮葡萄球菌(Staphylococcus epidermidis)ATCC 12228、大肠杆菌(Escherichia coli)ATCC 10536、阪崎肠杆菌(Enterobacter sakazakii)ATCC 51329、宋内氏志贺氏菌(Shigella sonnei)ATCC 25931、单核细胞增生李斯特菌(Listeria monocytogenes)ATCC 19114、弗劳地柠檬酸杆菌(Citrobacter freundii)ATCC 10787、鼠伤寒沙门氏菌(Salmonella typhimurium)ATCC 14028、产气肠杆菌(Enterobacter aerogenes)ATCC 49701、阴沟肠杆菌(Enterobacter cloacae)ATCC 13047 美国模式培养物集存库(ATCC);肠炎沙门氏菌(Salmonella enteritidis)CICC 21482 工业微生物菌种保藏中心(CICC);禾谷镰孢霉(Fusarium graminearum,F.graminearum)CGMCC 3.4733、栖土曲霉(Aspergillus terricola,A.terricola)CGMCC 3.3557、黄暗青 霉(Penicillium citreonigrum,P.citreonigrum)CGMCC 3.5694、黄曲霉(Aspergillus flavus,A.flavus)CGMCC 3.6434、寄生曲霉(Aspergillus parasiticus,A.parasiticus)CGMCC 3.6156、桔灰青霉(Penicillium aurantiogriseum,P.aurantiogriseum)CGMCC 3.5691、拟康氏木霉(Trichoderma pseudokoningii,T.pseudokoningii)CGMCC 3.3002、土生链孢霉(Alternaria humicola,A.humicola)CGMCC 3.3907、鲜绿青 霉(Penicillium viridicatum,P.viridicatum)CGMCC 3.5690、烟曲霉(Aspergillus funigatus,A.funigatus)CGMCC 3.6452、杂色曲霉(Aspergillus versicolor,A.versicolor)CGMCC 3.6410 中国普通微生物菌种保藏管理中心(CGMCC);茄病镰刀菌(Fusarium solani,F.solani)CPCC 460008 中国药用微生物菌种保藏中心(CPCC)。
1.1.2 试剂与仪器 冷冻鲐鲅鱼1.5 kg、辣椒粉1 kg市场购买;营养肉汤、EMB 平板、BS 平板、XLD平板、Baird-Parker 琼脂和血平板、PDA 平板 北京陆桥生物科技有限公司,按说明书配制;无水乙醇分析纯,国药集团化学试剂有限公司;超纯水 电阻18.2 MΩ,实验室自制。
VERTEX70 傅立叶变换红外光谱仪 德国布鲁克公司;UV1800A 核酸蛋白分析仪 Bio-rad 公司;CR22G III 离心机 日本日立公司;IH250 恒温恒湿培养箱、干燥箱 IRM 公司;ZnSe 窗片(透过波长7800~440 cm−1、透过率大于68%、直径25 mm、厚度2 mm) 筱晓(上海)光子。
1.2 实验方法
1.2.1 样品制备
1.2.1.1 细菌样品制备 对蜡样芽孢杆菌(Bacillus cereus)ATCC 12826 等13 种致病菌,从−70 ℃各挑取一环分别接种于营养肉汤中,(36±1)℃静置培养18~24 h,备用。
各菌挑取活化肉汤培养物一环,接种于100 mL营养肉汤中,(36±1)℃、180 r/min 培养18~24 h,用核酸蛋白分析仪测定并记录菌液浓度[16],分别吸取培养物1 mL,5000×g 离心5 min,无菌生理盐水和超纯水悬浮洗涤各两次,50 μL 超纯水悬浮混匀。吸取不同稀释度的菌悬液10 μL 于ZnSe 窗片中心,置于45 ℃干燥箱中烘干,得到干燥菌斑。每个菌株进行10 次试验,每次3 个重复。
1.2.1.2 真菌样品制备 对禾谷镰孢霉(Fusarium graminearum,F.graminearum)CGMCC 3.4733 等12 种真菌,将菌种接种到马铃薯葡萄糖琼脂培养基(PDA)上进行活化,28 ℃培养3~5 d,备用。
挑取各菌活化培养物转到新的PDA 平板上,(28±1)℃静置培养5 d。待菌丝充足,刮下菌丝置于1 mL 无菌生理盐水中,5000×g 离心5 min 弃上清,无菌生理盐水和超纯水悬浮洗涤各两次,50 μL纯水悬浮混匀。吸取不同稀释度的菌悬液10 μL 于ZnSe 窗片置于45 ℃干燥箱得到干燥菌斑[17]。每个菌株进行6 次试验,每次3 个重复。
1.2.2 光谱采集 制作好的ZnSe 窗片置于光谱仪上采集光谱信息,参数设置为:波段范围4000~500 cm−1[18],分辨率4 cm−1,自动扣除大气背景,扫描次数为64 次,累计求平均[19]。
1.2.3 加标验证
1.2.3.1 细菌加标验证
a 供试样品制备:取冷冻鲐鲅鱼平均分为3 份,每份500 g,(121±1)℃高温灭菌15 min,得到无菌鲐鲅鱼,剪碎均质后向其中分别添加5×102CFU/mLE.coliATCC 10536、S.enteritidisCICC 21482、S.aureusATCC 12600 标准菌株溶液各10 mL,搅拌混匀,备用。
b 细菌培养:供试样品每份称取25 g,分别按照大肠杆菌、沙门氏菌、金黄色葡萄球菌选择培养标准进行增菌培养。增菌后的培养液浓度尽量控制E.coli的OD600在0.85 左右,S.enteritidi的OD600在0.7左右,S.aureus的OD600在0.9 左右。
c 硒化锌(ZnSe)窗片的制作:方法同1.2.1.1。
d 光谱采集:分别采集各菌FT-IR 光谱,光谱采集参数同1.2.2。
1.2.3.2 真菌加标验证
a 供试样品制备:取辣椒粉平均分为2 份,每份500 g,(121±1)℃高温灭菌15 min,得到无菌辣椒粉,用无菌小铁勺分别将培养好的A.flavusCGMCC 3.6434 和F.graminearumCGMCC 3.4733 菌丝从培养基上刮下,制作菌悬液添加至无菌辣椒粉中,搅拌混匀,冷冻备用。
b 真菌培养:称取供试辣椒粉样品25 g 均质于225 mL 无菌超纯水中,分别吸取1 mL 样品匀液于马铃薯葡萄糖琼脂平板中,倒置(28±1)℃培养5 d。
c 硒化锌(ZnSe)窗片的制作:方法同1.2.1.2。
d 光谱采集:分别采集各菌FT-IR 光谱,光谱采集参数同1.2.2。
1.3 数据处理
1.3.1 标准菌株光谱处理和数据分析 用VERTEX70自带的扫描分析软件OPUS 6.5 软件进行透过率-吸光度转化、基线校正[20],对波数范围光谱一阶导数(平滑点选择17 点)运算得到导数谱后做矢量归一处理,将归一化后的导数谱,转化为excel 数据格式。
将1.2.1.1 处理后的13 种细菌977.9~1805.3 cm−1波数范围光谱数据分别导入Matlab 6.5 和Statistica 6.0 软件,分别得到13 种致病菌导数谱的HCA 分布图和PCA 分布图。
将1.2.1.2 处理后的12 种真菌900~1800 cm−1和2800~3700 cm−1波数范围光谱数据分别导入Matlab 6.5 和Statistica 6.0 软件,分别得到12 种真菌导数谱的HCA 分布图和PCA 分布图。
1.3.2 加标验证数据分析 将可疑待测菌E.coli、S.enteritidi、S.aureus977.9~1805.3 cm−1波数范围光谱数据与1.3.1 构建的13 种致病菌光谱信息数据库合并,选择HCA 分析方法进行聚类分析,得到待测菌的聚类结果。
将可疑待测菌A.flavus和F.graminearum900~1800 cm−1和2800~3700 cm−1波数范围光谱数据分别与1.3.1 构建的12 种真菌的光谱信息数据库合并,选择HCA 分析方法进行聚类分析,得到待测菌的聚类结果。
2 结果与分析
2.1 细菌浓度
培养18~24 h 后,根据核酸蛋白仪测定的结果,各菌种的吸光值及浓度见表1。

表1 细菌OD600 和浓度Table 1 Value of OD600 and concentration of bacteria
2.2 光谱导数谱数据库的建立
2.2.1 细菌光谱数据库的建立 对各菌种的导数谱取平均,得到不同菌种的平均光谱,见图1。在图示波数范围内,各菌的峰形、峰位以及峰的数量差距不大,仅凭谱图无法区别各菌种之间的差异,需建立光谱数据库借助数据分析和化学计量学运算进行分析。
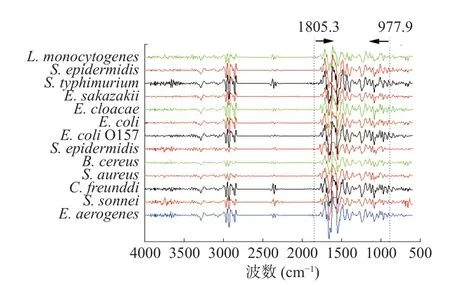
图1 13 种致病菌平均导数谱Fig.1 Average FT-IR spectral derivatives of 13 species of pathogenic bacteria
13 种常见致病菌FT-IR 光谱数据库的建立:根据试验反复验证结果,本文确定的光谱采集波数范围为4000~500 cm−1,用于数据分析的波数范围为977.9~1805.3 cm−1。应用OPUS 6.5 软件对原始光谱进行预处理得到导数谱,又对导数谱进行了适量归一化,建立了B.cereus、E.coliO157、S.aureus、S.epidermidis、E.sakazakii、S.sonnei、L.monocytogenes、C.freunddi、S.typhimurium、S.enteritidis、E.aerogenes、E.cloacae等13 种菌的FT-IR 导数谱数据库。
2.2.2 真菌光谱数据库的建立 对采集并处理后的各菌种的FT-IR 光谱取平均,得到各菌种的平均光谱,见图2。由图可见,经基线校正和矢量归一化后,光谱间的一些差异可用肉眼区分。在选择的900~1800 cm−1和2800~3700 cm−1波数范围内,根据12 个菌株FT-IR 光谱图中吸收峰所表征的意义,可将其分为5 个具有差别的光谱特征区域分别是3700~2996 cm−1处、2996~2800 cm−1处、1800~1485 cm−1处、1485~1185 cm−1处、1185~900 cm−1处,这些不同的特征区域分别反映了真菌细胞不同化学物质的伸缩振动带,菌种不同,化学物质含量和区域分布便会不同,能够区别一定的菌种差异。尽管光谱的一些差异可通过肉眼鉴别,为了建立的方法更系统直观,需要建立光谱数据库进行数据分析。

图2 12 种真菌平均导数谱Fig.2 Average FT-IR spectral derivatives of 12 species of fungi
12 种常见真菌FT-IR 光谱数据库的建立:应用OPUS6.5 软件对得到的原始光谱进行转化和基线校正,对12 种菌4000~600 cm−1波数范围光谱进行矢量归一化处理,即建立了F.graminearum、A.terricola、P.citreonigrum、A.flavus、A.parasiticus、P.aurantiogriseum、T.pseudokoningii、A.humicola、P.viridicatum、A.funigatus、A.uersicolor、F.solani等12 种真菌的FT-IR 光谱数据库。
2.3 PCA 和HCA 分析结果
2.3.1 PCA 分析结果 通过对13 种致病菌和12 种真菌的光谱数据进行PCA 分析,发现不管用2 种主成分还是3 种主成分为聚类依据,均不能对菌种进行较好聚类。分析其原因,理论上PCA 分析可以在不降低光谱差异的前提下,减少数据维数实现直观呈现,但减少维数也使得对在多维空间能较好分类的数据点产生交叉或重叠,导致降维后不能准确分类[21−23],也说明PCA 方法在处理多维数据时并不具有优势。
2.3.2 HCA 分类结果 利用皮尔森积矩相关系数算法描绘树状分支图,对13 种致病菌和12 种真菌的光谱数据进行HCA 分析。如图3 所示,13 种菌的样本按照菌种准确聚类,样本间没有出现交叉和重叠,证明该分析方法能够将图中各菌种较好聚类,实现种间分类的目的。图4 是12 种真菌的光谱数据树状聚类分布图,每个菌种的样本均能够归为一类,实现种间的准确分类。证明该方法可以适用于致病菌和真菌的菌种分类。

图3 13 种常见致病菌 HCA 聚类树状图Fig.3 HCA cluster results of FT-IR spectra of 13 species of pathogenic bacteria
同时,从图4 可以看出,虽然每个真菌菌株红外光谱的6 次重复都可正确地归为一类,但真菌分类系统的层次等级不能在聚类分析图中体现出来。例如,同属于曲霉属的5 个种黄曲霉、烟曲霉、栖土曲霉、杂色曲霉、寄生曲霉在第二层次“属”的归类中,未能同时归于一类中。这一现象与光谱识别方法与传统真菌分类方法依据不同有一定关系,传统生物学方法依据的是形态学、细胞结构等,而光谱方法主要通过识别细胞生物大分子的组成。

图4 12 种真菌HCA 聚类树状图Fig.4 HCA cluster results of FT-IR spectra of 12 species of fungi
2.4 加标验证结果
分别向无菌鲐鲅鱼样品中添加可疑待测菌,验证所建立的13 种常见食源性致病菌的FT-IR 光谱数据库及种间分类鉴定方法的准确性,结果见图5。由图可见,样品中的E.coli、S.enteritidi、S.aureus分别准确聚类到相应的菌种中,实现了对可疑待测菌的准确鉴定。说明本文建立的光谱数据库给可疑待测菌的种类鉴定提供了准确的归类依据,也说明FTIR 技术结合HCA 分析建立的致病菌菌种分类鉴定方法是可行的。

图5 致病菌加标验证结果Fig.5 The result of adding standard to verify pathogenic bacteria
通过向无菌辣椒粉样品中添加可疑待测真菌,验证所建立的12 种真菌的FT-IR 光谱数据库及种间分类鉴定方法的准确性,结果见图6。由图可见,可疑待测真菌A.flavus和F.graminearum分别准确聚类到数据库中对应的菌种,验证了方法的准确性,说明本文建立的方法能够实现可疑待测真菌种类的鉴定,为常见真菌的菌种检测提供了新的方法支撑。

图6 真菌加标验证结果Fig.6 The result of adding standard to verify suspected fungi
3 结论
FT-IR 技术简单、快速、高效等优点,使其在微生物分类鉴定方面的作用越来越得到重视,在成分检测、性能比对、临床医学等领域中的快速鉴定也得到了较多的研究[24−26]。传统菌种鉴定方法操作繁琐,需要进行生化实验和血清学实验,检测周期约需5~7 d甚至更长。相比于传统生物学鉴定方法,本文建立的方法具有易操作、周期短、成本低等特点。从可疑菌落到检测结果只需要几分钟,整个检测过程仅需要2~3 d,检测所用的试剂更少,主要耗材ZnSe 窗片可重复利用。本文使用的主成分分析(PCA)和聚类分析(HCA)是对光谱数据进行描述的方法,都属于化学计量学统计方法中的无监督型法,即在未知确切分类的情况下进行的数据分析[27]。其中,PCA 是在不降低差异同时减少维数,而后将综合变量进行比较后画出散点图[28],HCA 是根据数据本身所具有的特征进行归类和内在结构分析,得到光谱簇间距离[29]。两种不同方法的聚类结果各有优势,也受到采集参数、制备条件、种间差异等因素影响[30],同时,一些研究也发现,应用光谱方法对菌种进行鉴定,其重复性受到包括培养条件、实验操作标准化、分析方法等因素的影响[31],因此应确保菌种培养参数、样品制备以及光谱分析等与建立光谱数据库的条件保持一致[32]。
本研究确定了常见食源性致病菌的977.9~1805.3 cm−1光谱分析灵敏区,真菌的3700~2996 cm−1、2996~2800 cm−1、1800~1485 cm−1、1485~1185 cm−1与1185~900 cm−1五个光谱分析灵敏区,获得了分辨率高、重现性好的红外谱图,构建光谱数据库,通过HCA 分析将13 种细菌和12 种真菌直观清楚地区分开来。从验证试验的结果来看,无论致病菌还是真菌,可疑待测菌均能聚类到各自对应的菌种光谱数据库中,验证了FT-IR 技术在微生物种间水平鉴定上的准确性,拓展了FT-IR 技术结合化学计量学分析方法在微生物分类鉴定方面的应用,对于开展食品中致病菌和真菌的快速检测也具有实际应用价值。
研究中也发现了一些问题,例如,真菌的分类在属间水平与传统方法不同,鉴定结果的稳定性还需要进一步检验。下一步,应继续开展将光谱识别应用于微生物属间、菌株间的方法研究,增加对未知待测菌株的鉴定研究,并与传统检测方法进行全面比较分析,进一步检验方法的准确性和稳定性,使FT-IR 结合化学计量学应用于微生物分类鉴定的方法研究更加系统完善。

