基于生物信息学方法筛选PTCL-NOS关键基因及治疗药物
张洪禄,滕 悦,汤唯艳,汤依群,吴剑秋
(1.江苏省肿瘤医院内科,江苏 南京 210000;2.中国药科大学基础医学与临床药学学院,江苏 南京 210000)
非特指性外周T 细胞淋巴瘤(PTCL-NOS)是外周T 细胞淋巴瘤(PTCL)中最常见的类型,超过30%的PTCL 患者被归类于PTCL-NOS[1]。由于PTCL-NOS 具有高度异质性和耐药性,目前所采用的一线方案是沿袭B 细胞淋巴瘤治疗方案而来,绝大部分治疗方案缺乏高水平的证据或生物学基础[2]。虽然近年来不断有针对PTCL-NOS 的新药面世,但此类患者与B 细胞淋巴瘤患者相比仍存在预后差、复发率高的问题,因此阐明PTCL-NOS 的分子机制和寻找有效药物对该病的治疗尤为重要。近年来,生物信息学技术在确定疾病潜在的关键基因靶点、信号途径,疾病诊断和疾病发生的预测等多方面发挥着越来越重要的作用。本研究使用R 语言软件与在线生物信息学分析工具,筛选影响PTCL-NOS 的关键信号通路和差异表达基因,并对可能对PTCL-NOS 产生治疗作用的药物进行筛选,以期为PTCL-NOS 的研究和治疗提供新的思路。
1 资料与方法
1.1 GEO 基因芯片的获取和分析 登录美国国立生物中心基因数据库(NCBI-GEO)网站(https://www.ncbi.nlm.nih.gov/geo/),将筛选研究类型设置为expressionprofilingbyarray,筛选种属为homospiens,检索词为expression data from PTCL 后,检索到GSE132550 芯片数据集,GSE132550 芯片数据集中包含了68 个PTCL-NOS 患者的样本,18 个Lennert淋巴瘤患者样本和16 个来自于健康志愿者T 细胞的样本。下载GSE132550 的表达谱(.txt 格式文件)和相关临床信息(.soft 格式文件)。应用GPL18281平台对其进行表达谱注释分析。选取GSE132550 芯片数据集中PTCL-NOS 和健康志愿者的样本进行进一步研究。
1.2 差异基因表达分析 通过R 语言对芯片数据进行质量分析和标准化处理。为提高差异基因分析的准确性,将差异基因分析阈值设定为|Log2FC(foldchange)|>4,P<0.01。在R 语言中加载附加模块Limma 包和affyPLM 包,对PTCL-NOS 样本和健康T 细胞样本进行差异基因的表达分析[3]。
1.3 基因本体论与KEGG 途径富集分析 将之前所获得的差异基因上传至生物信息学微阵列分析数据库DAVID(https://david.ncifcrf.gov/tools.jsp)以获得基因本体论(GO)和京都基因和基因组百科全书(KEGG)通路的分析结果。GO 的结果包括生物学的3 个方面:细胞构成(cell components,CC)、分子功能(molecular functions,MF)和生物学过程(biological processes,BP)。
1.4 差异基因的蛋白质交互网络富集分析 STRING(11.0 版)(https://string-db.org/) 是一个在线数据库,包含已知和预测的蛋白质-蛋白质相交互用(PPI)[4]。将所有差异基因上传到STRING 中,并将最小交互分数设置为0.9(最高条件)以上作为显著阈值。将所获得的PPI(.TSV 格式文件)上传到Cytoscape 软件以构建PPI 网络[5]。随后,采用Cytoscape 软件内的分子复合物检测(MCODE)-app技术来识别关键基因间的连接网络。MCODE 中的所有参数设置为default。
1.5 药物-基因相互作用分析 DGIdb(https://www.dgidb.org)是一个基于数十个可信数据库的,用于筛选分析药物-基因相互作用的在线数据库[6]。将MCODE 获得的关键基因上传到DGIdb 中,以搜索现有的药物-基因相互作用,并利用与药物匹配的关键基因进行功能富集分析。
1.6 统计学分析 所有的统计分析均由R 软件(4.0.2)进行,整个分析流程见图1。
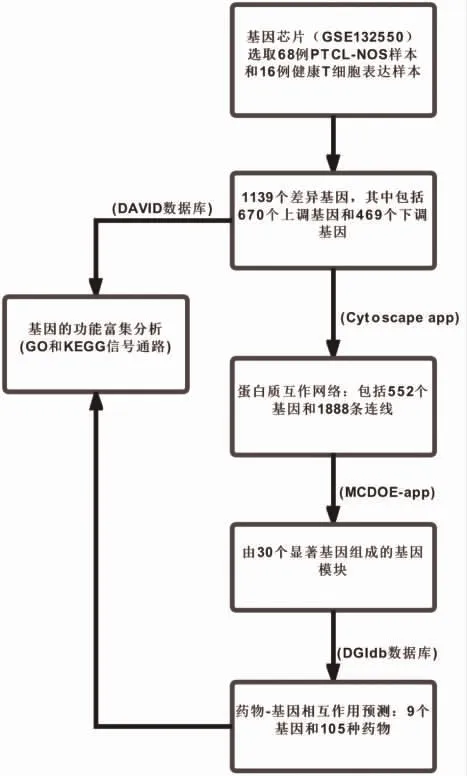
图1 数据分析流程
2 结果
2.1 差异表达基因的获取 用R 软件分析GSE132550 数据集中PTCL-NOS 样本和健康T 细胞样本,根据|Log2FC|>4,P<0.01 的筛选标准,共获得1139 个存在差异表达的基因,其中包括670 个上调基因和469 个下调基因,见图2。
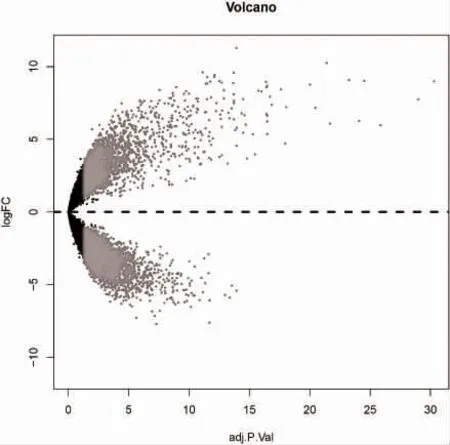
图2 1139 个差异基因的火山图
2.2 GO 与KEGG 信号通路的富集分析 将所有的差异基因上传到DAVID 数据库进行GO 富集分析,得到差异基因的主要富集项目,包括KEGG 信号通路、BP、CC 和MF。在KEGG 信号通路中差异基因主要富集于PI3K-AKT 信号通路,细胞外基质受体相互作用通路和阿米巴病相关信号通路,见图3;BP注释中差异基因主要存在于细胞外基质组织、细胞外结构组织和细胞连接组件,见图4;CC 注释中差异基因主要涉及胶原蛋白-细胞外间质、基底膜和胶原三聚体的组成成分,见图5;MF 注释中差异基因与细胞外基质结构成分、生长因子结合和胶原蛋白结合密切相关,见图6。
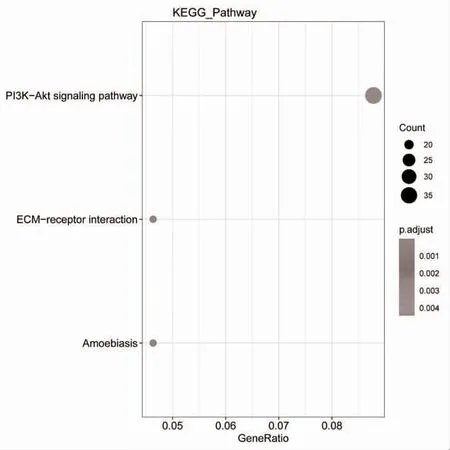
图3 KEGG 信号通路富集结果
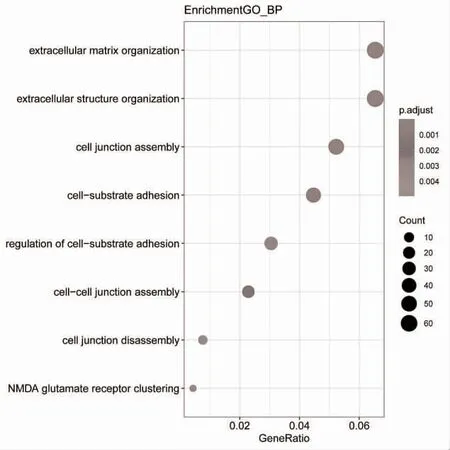
图4 BP 富集结果
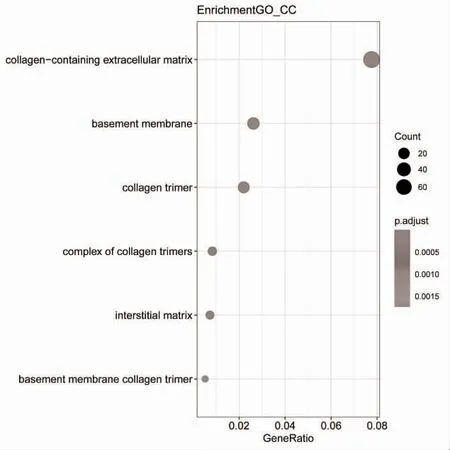
图5 CC 富集结果
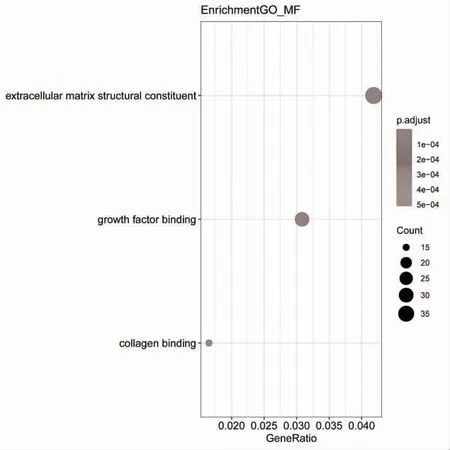
图6 MF 富集结果
2.3 PPI 富集分析 将1139 个差异基因上传到STRING 数据库进行PPI 富集分析,得到包含552 个基因节点和1888 条连线的PPI 网络,使用Cytoscape软件中的MCODE-app 筛选出包含显著差异基因的模块,交互模块(分数:15.655)涉及30 个基因节点和227 条连线,模块中的差异基因主要包含在G 蛋白偶联受体信号通路、胞外区和质膜中,见图7。
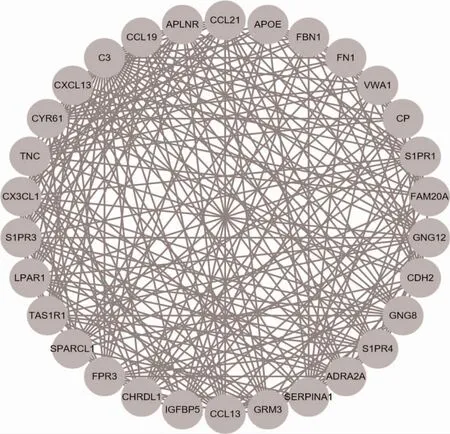
图7 30 个显著基因组成的蛋白交互网络
2.4 药物与基因的相交互用及潜在基因功能富集分析 将2.3 中模块1 的30 个MCODE 基因上传到DGIdb 数据库中进行药物-基因相交互用分析,在筛选出与基因存在特定交互关系的药物后,发现了105 个药物靶向9 个特定基因(其中包括90 种针对ADRA2A 的药物),所有的药物都有其初始适应证,见表1;这些基因主要富集在G 蛋白偶联受体信号通路(BP)、质膜(CC)和神经活性配体-受体相交互用(KEGG 通路),见图8、表2。
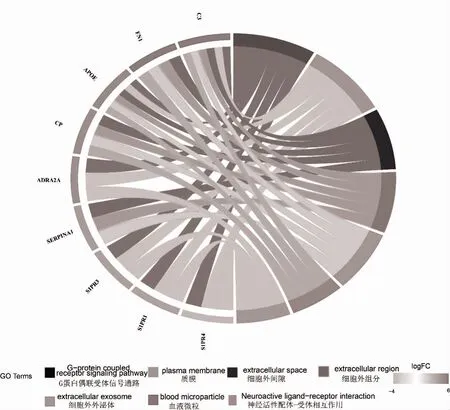
图8 9 个存在药物-基因相交互用的基因富集弦图
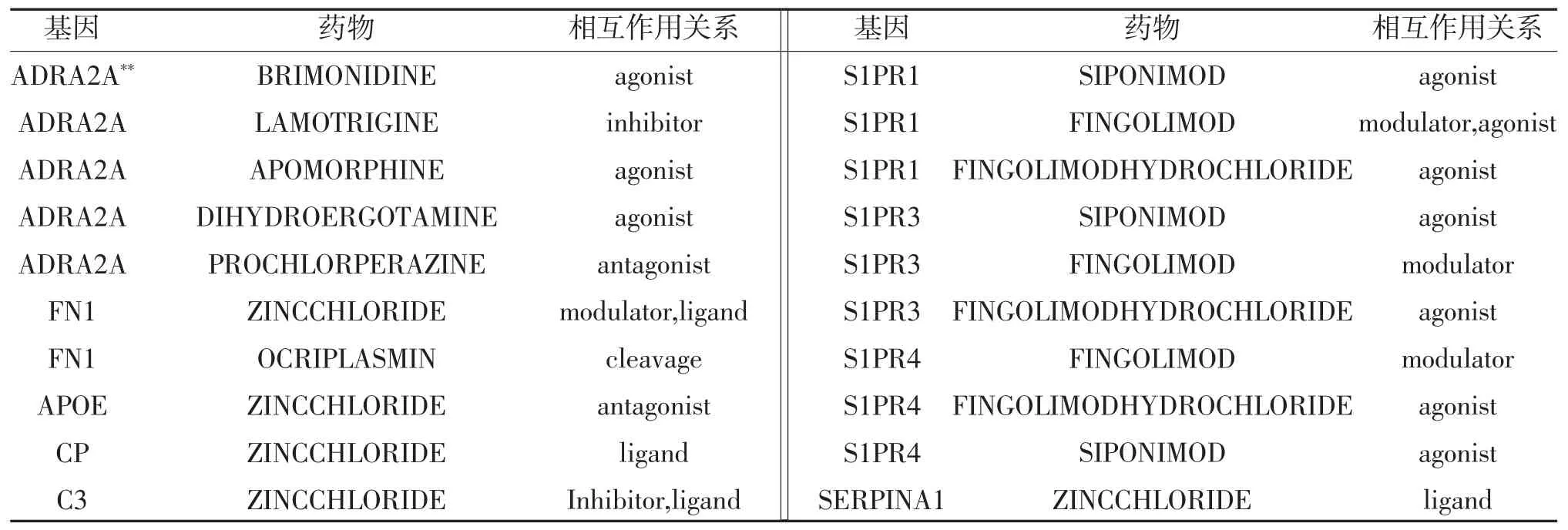
表1 基因-药物相互作用
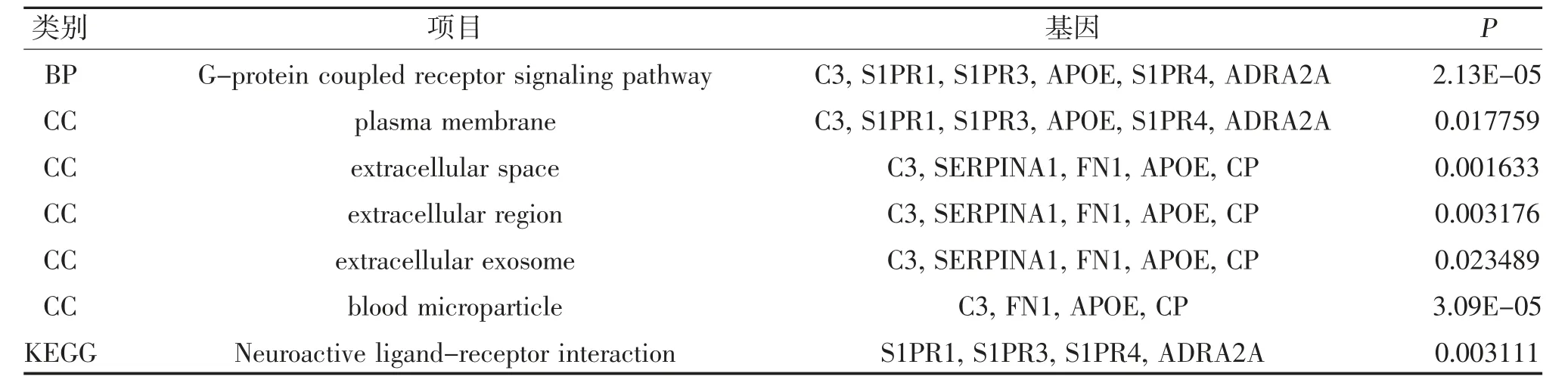
表2 具有基因-药物交互结果的9 个基因富集分析
3 讨论
PTCL-NOS 是PTCL 最常见的亚型,通常表现出侵袭的临床过程且分子机制尚未完全阐明[7]。几乎所有PTCL-NOS 的治疗方案都缺乏高水平的证据、分子或生物学基础[8]。目前,其一线治疗方案主要来源于侵袭性B 细胞淋巴瘤的治疗方案,存在复发率高、远期疗效差等问题[9]。近年来,随着转录组学的发展,许多疾病的分子特征和靶向药物的发现,为个体化治疗提供了可能。本研究旨在通过对PTCL-NOS 基因组学和转录组学的分析,采用生物信息学方法寻找TCL-NOS 关键基因和靶向药物。
本研究发现,PTCL-NOS 患者的部分KEGG 信号通路活动发生了明显改变,其中PI3K-Akt 信号通路的改变最为显著,共有36 个包含在该通路内的基因发生了表达改变。磷脂酰肌醇-3 激酶(PI3K)/蛋白激酶B(AKTe)/雷帕霉素(mTOR)信号通路的哺乳动物靶点在包括代谢、凋亡、血管生成和细胞增殖在内的生物调节中起着重要作用,也是人类癌症中最常见的失调信号通路之一[10]。PI3K 是磷脂酰肌醇-3,4,5-三磷酸(PIP3)的脂类激酶。PIP3 是AKT的对接位点,通过激活下游效应子mTOR 信号通路促进细胞生长、蛋白质合成、细胞运动和存活[11]。PTEN 是PI3K 的负性调节因子,能抑制PI3K 抑制肿瘤生长。在癌症环境下,PTEN 的突变使其失去对PI3K 的调节能力,进而失调下游通路[12]。总之,在癌症中,PI3K/AKT/mTOR 通路的过度激活增强了营养转运蛋白和代谢酶的活性,导致细胞代谢改变,从而为肿瘤细胞的生长提供了合成代谢需求,同时也增加了肿瘤细胞的耐药性[13]。
根据DGIdb 数据库的基因-药物相交互用分析,共有105 种治疗PTCL-NOS 的潜在药物(其中有90 种药物针对ADRA2A),靶向9 个基因(ADRA2A、S1PR1、S1PR3、S1PR4、CP、FN1、APOE、SERPINA1、C3)。ADRA2A 被认为是多种肿瘤细胞系的潜在调节因子[14]。SERPINA1 过表达显著改善了癌细胞迁移、集落形成和对细胞凋亡的抵抗力[15]。APOE 可通过与膜受体结合介导循环脂蛋白和组织之间的脂质转移[16]。APOE 可通过LR8 受体直接抑制循环髓源性抑制细胞的存活,并在激活免疫系统中发挥抗肿瘤作用[17]。FN1 在多种癌症中上调,沉默FN1 可促进肿瘤生长和转移[18,19]。S1P-S1PRs 信号通路诱导免疫细胞从淋巴器官流入血液,从而影响抗肿瘤免疫反应[20,21]。西尼莫得是一种高效的S1PRs拮抗剂,已被批准用于治疗多发性硬化症,其安全性和有效性已被证实。另一种S1PRs 抑制剂芬戈莫德也证明了其治疗癌症的潜力[20]。
综上所述,本研究发现9 个与PTCL-NOS 有关的关键基因(ADRA2A、S1PR1、S1PR3、S1PR4、CP、FN1、APOE、SERPINA1、C3)和105 个FDA 批准的药物,如芬戈莫德、西尼莫得、氯化锌、丙氯拉嗪等。另外PI3K-AKT 信号通路对于PTCL-NOS 疾病的作用也值得关注。但由于本研究获得的关键基因和药物是基于软件统计处理和数据库分析,它们是否能在PTCL-NOS 中发挥作用仍需进一步的分子生物学实验来验证。

