茶树CsbHLH024和CsbHLH133转录因子功能鉴定
刘任坚,王玉源,刘少群,舒灿伟,孙彬妹,郑鹏*
茶树和转录因子功能鉴定
刘任坚1,王玉源1,刘少群1,舒灿伟2,孙彬妹1,郑鹏1*
1. 华南农业大学园艺学院,广东 广州 510642;2. 华南农业大学植物保护学院/广东省微生物信号与作物病害防控重点实验室/群体微生物研究中心,广东 广州 510642
茶树叶片毛状体含有多种次生代谢产物,在茶叶外观质量以及茶树响应生物和非生物胁迫方面起着重要作用。通过双荧光分子互补(BiFC)试验、GUS活性染色试验以及过表达试验对茶树叶片毛状体相关候选基因和的功能进行鉴定。结果表明,CsbHLH024/CsbHLH133和CsTTG1蛋白在植物中能够相互作用,并且它们的启动子能够在叶片组织中驱动下游基因的表达。进一步将它们分别过表达到野生型拟南芥Col和对应的拟南芥纯合突变体中,发现它们能够影响拟南芥叶片毛状体的形成,恢复突变体的表型,并引起毛状体相关基因表达水平的变化。本研究为进一步揭示茶树叶片毛状体形成的分子调控机制提供理论依据。
茶树;毛状体形成;;;分子调控
植物毛状体是从表皮细胞发育而来,主要由单细胞或多细胞组成[1-5]。毛状体主要分布在植物的叶片、茎、叶柄、花瓣、种皮等组织[6]。在不同植物中,毛状体的形态和功能各不相同。毛状体按照形态和功能,可分为有分支毛状体和无分支毛状体、有腺毛状体和无腺毛状体,后者是用来区分毛状体的常用标准[1]。植物毛状体一般与叶片的水分和湿润性有关[7-8],在抵抗生物和非生物胁迫方面起着重要作用。其中,有腺毛状体通过产生、储存和分泌多种次生代谢产物,如生物碱、尼古丁、萜烯类物质等,增强植物的抗虫性从而抵抗生物胁迫[9-11];而无腺毛状体能够增强植物对极端温度、干旱、紫外线辐射等非生物胁迫的抗性[12],保障植物在逆境条件下仍能正常生长发育。此外,植物有腺毛状体分泌出来的次生代谢产物在食品和制药行业中均得到应用[13-14]。植物毛状体在形成过程中受到一系列相关基因的调控,且分泌腺毛状体和非分泌腺毛状体在形成调控上具有显著差异。在番茄和青蒿中,分泌腺毛状体形成的调控机制相似。其中,茉莉酸(Jasmonic acid,JA)可以通过降解茉莉酸信号传导抑制子,释放HD-Zip IV型转录因子,从而促进有腺毛状体的形成;并且MYB和HD-Zip转录因子在有腺毛状体形成的过程中起着主要调控作用[15]。拟南芥的毛状体属于单细胞非分泌腺毛状体,其形成的分子调控机制已经十分清晰,该调控网络主要通过一系列正向调控因子[MYB23/GLABRA1(GL1)、ENHANCER OF GLABRA3/GLABRA3(EGL3/GL3)和TRANSPARENT TESTA GLABRA 1(TTG1)]结合形成复合蛋白,激活诱导表皮细胞中()基因的表达,从而促进拟南芥毛状体的形成[16]。棉花毛状体的发育过程和基因调控模式与拟南芥的相似,其中,、、()、(Knotted related homeobox)和(Teosinte branched1/cycloidea/)等基因能够参与棉花毛状体发育的起始和伸长阶段[17-19]。黄瓜毛状体属于多细胞非分泌腺毛状体,目前已有一些与毛状体形成相关的基因被克隆,如、和的突变体均表现为无毛[20-21];Liu等[22]发现CsGL3、TRIL、MICT、TBH和CsGL1等编码HD-Zip蛋白的基因可能是黄瓜表皮细胞分化和毛状体发育的主要调控因子。此外,水稻表皮毛相关的基因也曾被报道。和(拟南芥的同源基因)可以抑制拟南芥毛状体的发育,但无法影响水稻表皮毛的形成[23];并且克隆出来的是否与水稻表皮毛形成相关还不清楚[24]。
茶树[(L.) O. Kuntze]是多年生常绿木本植物,主要包括中国种(var.)和阿萨姆种(var.)两个栽培品种[25]。茶树毛状体主要分布在叶片、嫩茎和子房中,并且作为茶树叶片的主要特征物之一,毛状体是茶组植物系统分类的重要依据[26]。茶树叶片上的毛状体又称为“毫”,由单细胞组成,属于不分支非分泌腺毛状体,其体内含有茶氨酸、茶多酚、咖啡碱、萜烯类等次生代谢物[27]。这些次生代谢产物均与茶叶的品质和风味相关[28-30]。然而,Cao等[31]通过代谢组学和转录组学联合分析发现,毛状体对茶叶的生化品质没有明显的贡献,或许仅有助于提高茶树对生物和非生物胁迫的抗性和茶叶外观质量。茶树叶片上的毛状体不仅可以通过减少叶片水分流失,降低叶片表面温度和反射外界光,降低茶树遭受高温、干旱和过度太阳辐射的风险[32-34],还可以阻碍昆虫在茶树叶片上产卵、附着和取食[35-37]。此外,最近研究发现,一些与疾病相关的信号基因以及抗草食动物和抗非生物胁迫的多肽在茶树叶片毛状体中特异转录,这也表明毛状体可能具有增强茶树物理和化学防御的功能[27]。近年来,也有报道有关茶树叶片毛状体形成的研究。Yue等[38]对有毛状体和无毛状体茶树品种的叶片进行转录组测序分析,筛选出21 599个差异表达基因,其中发现参与植物信号传导和纤维素合成的相关基因可能对茶树毛状体的形成起着重要作用。Sun等[39]研究发现,茶树的表达水平与毛状体密度相关,并且在拟南芥中过表达该基因,可以增加拟南芥叶片毛状体的密度。茶树叶片毛状体的形成十分复杂,其分子调控机制仍需进一步研究。
在植物中,Basic helix-loop-helix(bHLH)转录因子在叶片毛状体的形成和生长发育过程中起着重要作用。课题组前期对茶树bHLH转录因子家族进行全面和系统的鉴定与分析,依据系统发育进化树和基因表达模式分析结果,初步筛选出与毛状体形成相关的候选基因和[40]。本研究拟通过双荧光分子互补(BiFC)试验、GUS活性染色和基因过表达试验对候选基因的功能进行鉴定,为研究茶树叶片毛状体形成的分子调控机理提供理论基础。
1 材料与方法
1.1 试验材料
试验所用本氏烟草()和野生型拟南芥Col()种子为实验室保存,拟南芥突变体材料[salk_019114C()和salk_118201C()]从AraShare平台(https://www.arashare.cn/index)购买。
1.2 载体构建
依据(ID号:TEA008168.1)、(ID号:TEA033721.1)和(ID号:TEA000080.1)的CDS序列以及载体酶切位点,使用CE Design V1软件设计同源重组引物(表1),下划线部分为载体的酶切位点。以白叶单丛的cDNA为模板,使用PrimeSTAR酶进行PCR扩增。PCR产物经过琼脂糖凝胶电泳后回收目的条带,连接到对应的线性化载体上,转化至DH5后进行鉴定并测序,将阳性克隆提取质粒,用于转化农杆菌。
1.3 BiFC试验
将和的CDS序列(去除终止密码子)分别构建到pSPYNE-35S/pUC-SPYNE(YNE)载体上,将的CDS序列(去除终止密码子)构建到pSPYCE-35S/pUC-SPYCE(YCE)载体上,以空白载体为对照,使用热激法转化农杆菌感受态GV3101。用侵染液(10 mmol·L-1MgCl2,10 mmol·L-1MES,200 μmol·L-1AS,pH为5.6,现配现用)将收集后的菌体重悬至OD值为0.4~0.6,活化2 h后,将YNE/YNE- CsbHLH024/YNE-CsbHLH133、YCE/YCE-CsTTG1和细胞核marker(35S::NLS::Dsred)按体积比1∶1∶1混合,使用无针头注射器将菌液从叶片下表皮注射到本氏烟草中,黑暗保湿培养24 h,再正常培养48 h后,用生物显微镜BX53(OLYMPUS,日本)进行观察和拍照。
表1 载体引物序列
1.4 花序浸渍法侵染拟南芥花序
将和的CDS序列(去除终止密码子)构建到PBI121-GFP载体上,将和的启动子片段构建到CP013-GUS载体上,使用热激法转化农杆菌感受态GV3101。用侵染液(2.1 g·L-1MS,50 g·L-1蔗糖,0.5 g·L-1MES,100 μL·L-1Silwet L-77,pH为5.7,现配现用)将收集后的菌体重悬至OD值为0.6~1.0,活化2 h后,将拟南芥的花序(去除已成熟的角果)完全浸泡在侵染液中,侵染1~3 min后,再将植株水平放置在黑色托盘中,置于22℃黑暗条件下保湿1 d,然后转移至正常环境下培养,直至种子完全成熟后收集T0代种子。将T0代种子播种在1/2 MS筛选培养基(卡那霉素筛选质量浓度50 μg·mL-1)上,筛选两周后,将绿色植株移栽到土中,并提取植株叶片基因组DNA进行PCR扩增,将能够扩增出目的基因片段的植株培养,并收集T1代种子,将T1代种子再次播种在1/2 MS筛选培养基上,筛选两周后,将绿色植株移栽到土中,培养收集T2代种子备用。
1.5 GUS活性染色试验
将T2代种子播种在土中生长,期间采集以下样品:第一片真叶、子叶期植株、莲座叶期植株和抽薹期植株,使用北京酷来搏科技有限公司的GUS染色试剂盒(CODE:SL7160)对植株不同组织进行染色,在显微镜下观察染色情况。
1.6 实时荧光定量PCR(qRT-PCR)
1.7 数据统计与分析
使用Adobe Illustrator 2020软件对图片进行优化,使用IBM SPSS Statistics 22.0软件对数据进行方差分析。
2 结果与分析
2.1 CsbHLH024/CsbHLH133与CsTTG1之间的蛋白互作
bHLH转录因子通常与其他转录因子相互作用共同调控植物的生长发育以及次生代谢产物的合成。课题组前期研究发现,CsbHLH024/CsbHLH133和CsTTG1在酵母系统中能够相互作用;并且在不同发育叶片组织中的表达模式与和一致[40]。BiFC试验结果显示,共转CsbHLH024-nYFP+cYFP-CsTTG1和CsbHLH133-nYFP+cYFP-CsTTG1的本氏烟草叶片中的细胞核会激发出强烈的黄色荧光,而单独转入其中任一基因的本氏烟草叶片中的细胞核均无法激发出黄色荧光(图1)。表明CsbHLH024/CsbHLH133和CsTTG1蛋白在植物中能够相互作用。
2.2 CsbHLH024和CsbHLH133启动子驱动表达分析
为进一步确定和的表达部位,将候选基因启动子构建到CP013载体上,并且将重组载体转入农杆菌GV3101中,利用花序浸渍法侵染拟南芥,通过抗生素筛选以及PCR扩增鉴定出转基因株系(图2-A),并对这些株系部分T2代植株进行GUS活性染色试验。结果显示,pro-CsbHLH024和pro-CsbHLH133能够在拟南芥第一片真叶和莲座叶上驱动的表达,将叶片染成蓝色。其中,在子叶期,pro-CsbHLH024能在拟南芥下胚轴驱动的表达,将下胚轴染成蓝色,pro-CsbHLH133能在拟南芥子叶和下胚轴驱动的表达,将子叶和下胚轴染成蓝色;在抽薹期,pro-CsbHLH024和pro-CsbHLH133能够在拟南芥莲座叶和茎生叶上驱动的表达,将叶片染成蓝色(图2-B)。这表明启动子pro-CsbHLH024和pro-CsbHLH133可能是在叶片组织中启动和的表达。
表2 定量引物序列

注:比例尺=50 μm。Bright:明场通道;DsRed:红色荧光通道;YFP:黄色荧光通道;Merged:融合通道
2.3 异源过表达CsbHLH024和CsbHLH133
为了进一步确定基因功能,在野生型拟南芥Col上进行了过表达试验。其中,筛选出23株OE-和13株OE-转基因植株(图3-A)。测定和观察3个OE-(OE--4,OE--7和OE--9)和3个OE-(OE--5,OE--19和OE--25)转基因株系T2代植株的相关基因表达量和毛状体表型。qRT-PCR结果显示,过表达基因在转基因株系中的表达量显著高于野生型对照(图3-B)。相对野生型植株,OE-和OE-转基因株系第一、二片真叶中间部位毛状体分布较少(图3-C),统计数据显示,转基因株系第一、二片真叶上的毛状体数量均显著少于野生型植株(图3-D)。进一步测定转基因株系中毛状体相关基因的表达量,发现相对于野生型植株,除了和外,其他毛状体相关基因的表达量在转基因株系中均显著降低。其中,表达量在OE--4和OE--7转基因株系中显著上升;在OE--4和OE--7转基因株系中显著降低;的表达量在3个OE-株系中均显著上调(图3-E)。上述结果表明,在野生型在OE--9转基因株系中,表达量降低,表达量升高;而和拟南芥中分别过表达和,会导致大部分与毛状体形成相关基因的表达下调,转基因拟南芥植株上的叶片毛状体数量减少。
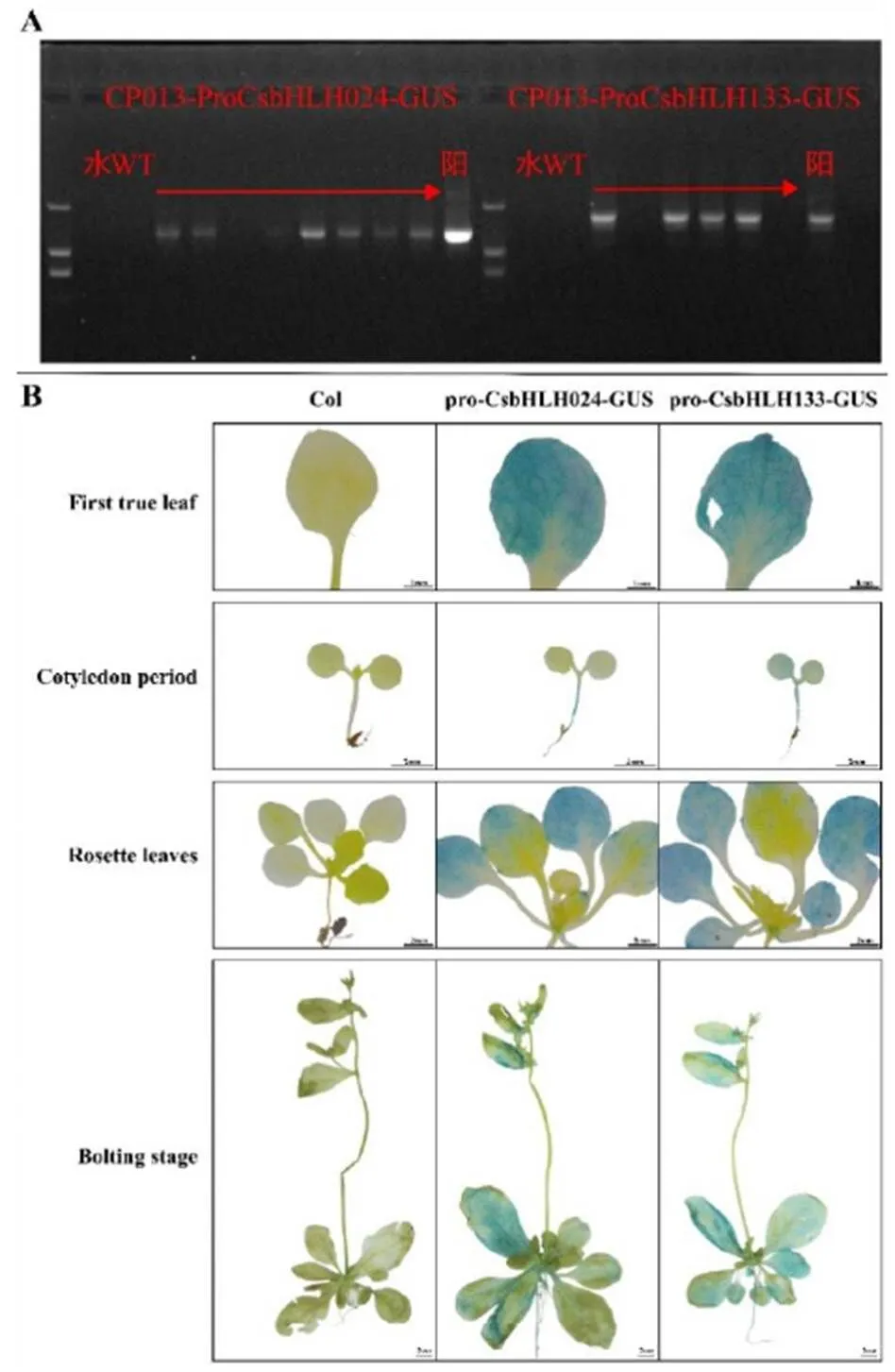
注:A. 转基因株系PCR检测。本试验使用的是2 000 bp DNA Maker,水作为空白对照,Wild type(WT)-Col作为阴性对照,阳性对照为CP013-Pro-CsbHLH024/Pro-CsbHLH133-GUS质粒。B. GUS活性染色结果。First true leaf:第一片真叶;Cotyledon:子叶期;Rosette leaf:莲座叶;Bolting stage:抽薹期
2.4 CsbHLH024和CsbHLH133互补拟南芥突变体
互补拟南芥突变体也是验证异源基因功能的重要试验。在和突变体纯合材料中进行基因互补试验,筛选出12株-35S:和16株-35S:转基因植株(图4-A)。测定和观察3个-OE-(-OE--4,-OE--10和-OE--11)和3个-OE-(-OE--13,-OE--15和-OE--16)转基因株系的基因表达量和毛状体表型。qRT-PCR结果显示,过表达基因在对应转基因株系中的表达量均显著高于原突变体纯合子材料(图4-B)。相对原突变体植株,转基因株系第一、二片真叶中间部位毛状体分布增多(图4-C);统计数据显示,转基因株系第一二片真叶上毛状体的数量均显著多于原突变体植株(图4-D)。进一步测定转基因株系中、毛状体相关基因的表达量,发现相对于原突变体植株,-OE-和-OE-转基因株系中毛状体相关基因的表达变化并没有明显的规律性。其中,在3个-OE-转基因株系中,几乎所有的毛状体相关基因的表达量都显著降低;而在3个-OE-转基因株系中,负调控基因的表达量显著下调,正调控基因的表达量显著升高(图4-E)。结果表明,在和中分别过表达和,其表型得到恢复,植株第一、二片真叶上毛状体数量增多,并且毛状体相关基因的表达量也受到影响。

注:A. 转基因株系PCR检测。本试验使用5 000 bp DNA Maker,水作为空白对照,WT-Col作为阴性对照,阳性对照为PBI121-CsbHLH024/CsbHLH133-GFP质粒。B. T2代转基因植株目的基因表达情况。C. 毛状体表型观察。观察部位是第一二片真叶。D. 毛状体数量统计。数据是用平均值±标准差(n=24)来表示,采用Student’s test与阴性对照Col进行显著性分析(**,P<0.01)。E. 拟南芥毛状体相关基因在T2代转基因植株中的表达情况。基因表达量数据是用平均值±标准差来表示,采用Student’s test与阴性对照Col进行显著性分析(**,P<0.01)
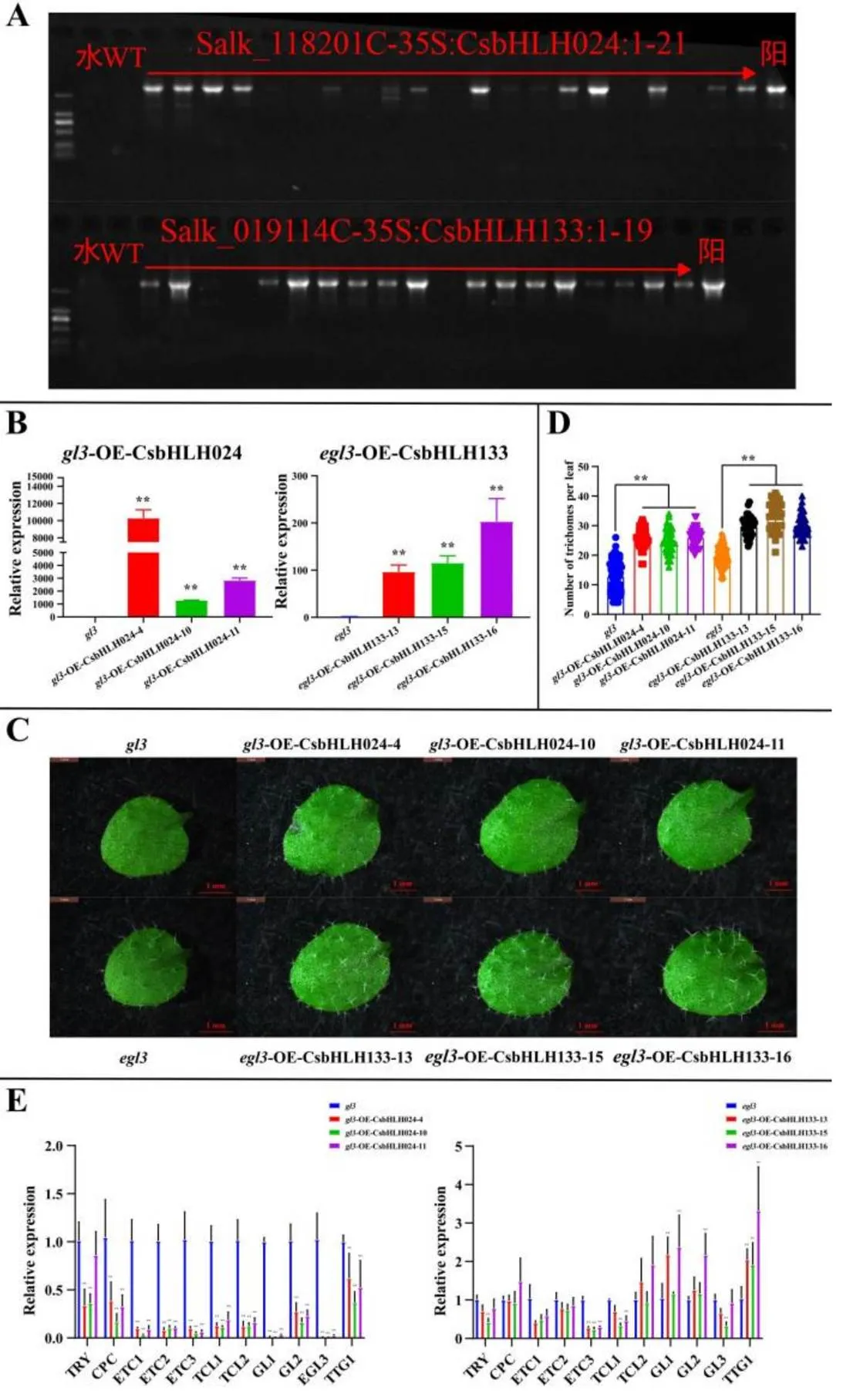
注:A. 转基因株系PCR检测。本试验使用2 000 bp DNA Maker,水作为空白对照,WT(Salk_118201C和Salk_019114C)作为阴性对照,阳性对照为PBI121- CsbHLH024/CsbHLH133-GFP质粒。B. T2代转基因植株目的基因表达情况。C. 毛状体表型观察。观察部位是第一、二片真叶。D. 毛状体数量统计。数据是用平均值±标准差(n=36)来表示,采用Student’s test与阴性对照gl3和egl3进行显著性分析(**,P<0.01)。E. 拟南芥毛状体相关基因在T2代转基因植株中的表达情况。基因表达量数据是用平均值±标准差来表示,采用Student’s test与阴性对照gl3和egl3进行显著性分析(**,P<0.01)
3 讨论
茶树叶片毛状体能够提高茶叶的外观质量,增强茶树抵抗病虫害的抗性。前人研究表明,bHLH转录因子在毛状体的形成过程中起到重要作用[42-46],然而bHLH转录因子是否参与调控茶树叶片毛状体的形成还尚未清晰。本研究通过一系列的分子生物学手段对毛状体形成相关候选基因和的功能进行鉴定。
bHLH转录因子通常与其他转录因子相互作用,共同调控植物生长发育过程。课题组前期对茶树基因的功能进行研究,发现过表达该基因可提高拟南芥毛状体的密度[39];在4个不同发育阶段叶片组织中的表达模式与和的一致,并且在酵母系统中,CsbHLH024/CsbHLH133可以与CsTTG1相互作用[40]。本研究通过BiFC试验进一步验证它们之间的互作关系。结果显示,CsbHLH024/CsbHLH133在植物中也可以与CsTTG1相互作用(图1)。在拟南芥中,AtTTG1也能够与AtEGL3或AtGL3相互作用[45-46]。这表明CsbHLH024和CsbHLH133可能是通过与CsTTG1相互作用,共同参与调控茶树叶片毛状体的形成。茶树毛状体主要分布在叶片、嫩茎和子房中。本研究通过GUS活性染色试验初步鉴定出候选基因的表达部位。结果显示,Pro-CsbHLH024和Pro-CsbHLH133主要是在叶片组织中驱动下游的表达(图2)。这表明和可能是在叶片组织中转录翻译,并发挥功能。
茶树是多年生木本植物,它的外植体离体再生率较低,并且茶树组织中富含茶多酚,多酚类物质能够抑制农杆菌的活性,导致茶树遗传转化效率很低[47]。目前很难在茶树本体上进行转基因试验进而直接验证基因的功能。因此,本研究在野生型拟南芥Col中进行了基因过表达试验,对候选基因的功能进行鉴定。结果显示,在野生型拟南芥分别过表达和,其后代转基因株系的第一、二片真叶表面的毛状体数量显著少于野生型植株,并且这些转基因株系中大部分毛状体相关基因的表达量下降。Szymanski等[48]在拟南芥中过表达,也发现其在一定程度上导致叶片毛状体数量的减少。这一结果可能是由于拟南芥毛状体调控途径中存在着负反馈调节机制;并且在异源物种中进行基因过表达试验,由于物种间基因调控机制存在差异,一些基因往往无法发挥其应有的功能。此外,也有部分研究发现,过表达株系表型与拟南芥突变体表型一致,过表达株系没有导致功能获得,而是功能缺失[49-50]。因此,本研究还在对应的拟南芥纯合突变体上进行了基因互补试验。结果显示,在和突变体材料中过表达和,其表型得到恢复,转基因株系第一、二片真叶上毛状体数量增多,但毛状体相关基因表达水平的变化并没有明显的规律性,这可能是由于拟南芥毛状体的形成是由多个调控因子共同调控的,基因之间存在功能冗余[16]。上述结果表明,和能够影响叶片毛状体的形成、数量以及相关基因的表达水平。但是,具体的调控机制还需要进一步探究。
[1] Werker E. Trichome diversity and development [J]. Adv Bot Res, 2000, 31: 1-35.
[2] Serna L, Martin C. Trichomes: different regulatory networks lead to convergent structures [J]. Trends Plant Sci, 2006, 11(6): 274-280.
[3] Schilmiller A, Last R, Pichersky E. Harnessing plant trichome biochemistry for the production of useful compounds [J]. Plant J, 2008, 54(4): 702-711.
[4] Ishida T, Kurata T, Okada K, et al. A genetic regulatory network in the development of trichomes and root hairs [J]. Annu Rev Plant Biol, 2008, 59: 365-386.
[5] 曹敏, 张璐, 高新梅, 等. 植物表皮毛发育分子调控机制的研究进展[J]. 安徽农业科学, 2013, 41(10): 4231-4235.
Cao M, Zhang L, Gao X M, et al. Advances in molecular regulation of plant trichome development [J]. Journal of Anhui Agricultural Sciences, 2013, 41(10): 4231-4235.
[6] Wagner G, Wang E, Shepherd R. New approaches for studying and exploiting an old protuberance, the plant trichome [J]. Ann Bot, 2004, 93(1): 3-11.
[7] Fernandez V, Sancho-Knapik D, Guzman P, et al. Wettability, polarity, and water absorption of holm oak leaves: effect of leaf side and age [J]. Plant Physiol, 2014, 166(1): 168-180.
[8] Svetlikova P, Hajek T, Tesitel J. Hydathodetrichomes actively secreting water from leaves play a key role in the physiology and evolution of root-parasitic rhinanthoid Orobanchaceae [J]. Ann Bot, 2015, 116(1): 61-68.
[9] Dayan F, Duke S. Trichomes and root hairs: natural pesticide factories [J]. Pesticide Outlook, 2003, 14(4): 175. doi:10.1039/b308491b.
[10] Bleeker P, Mirabella R, Diergaarde P, et al. Improved herbivore resistance in cultivated tomato with the sesquiterpene biosynthetic pathway from a wild relative [J]. Proc Natl Acad Sci U S A, 2012, 109(49): 20124-20129.
[11] Luu V, Weinhold A, Ullah C, et al. O-acyl sugars protect a wild tobacco from both native fungal pathogens and a specialist herbivore [J]. Plant physiology, 2017, 174(1): 370-386.
[12] Yamasaki S, Murakami Y. Continuous UV-B irradiation induces endoreduplicationand trichome formation in cotyledons, and reduces epidermal cell division and expansion in the first leaves of pumpkin seedlings (Duch.×.Duch.) [J]. Environmental Control in Biology, 2014, 52(4): 203-209.
[13] Weathers P, Arsenault P, Covello P, et al. Artemisinin production in: studies in planta and results of a novel delivery method for treating malaria and other neglected diseases [J]. Phytochem Rev, 2011, 10(2): 173-183.
[14] Mellon J, Zelaya C, Dowd M, et al. Inhibitory effects of gossypol, gossypolone, and apogossypolone on a collection of economically important filamentous fungi [J]. J Agric Food Chem, 2012, 60(10): 2740-2745.
[15] Chalvin C, Drevensek S, Dron M, et al. Genetic control of glandular trichome development [J]. Trends Plant Sci, 2020, 25(5): 477-487.
[16] Schellmann S, Hulskamp M. Epidermal differentiation: trichomes inas a model system [J]. Int J DevBiol, 2005, 49(5/6): 579-584.
[17] 杨君, 马峙英, 王省芬. 棉花纤维品质改良相关基因研究进展[J]. 中国农业科学, 2016, 49(22): 4310-4322.
Yang J, Ma S Y, Wang S F. Advances in studies on genes related to cotton fiber quality improvement [J]. Chinese Agricultural Sciences, 2016, 49(22): 4310-4322.
[18] 唐凯. 棉花磷脂酶D基因家族的分子特征及其GhPLDα1功能分析[D]. 北京: 清华大学, 2017.
Tang K. Molecular characterization of phospholipase D gene family and functional analysis of GhPLDα1 in cotton [D]. Beijing: Tsinghua University, 2017.
[19] Shi Y H, Zhu S W, Mao X Z, et al. Transcriptome profiling, molecular biological, and physiological studies reveal a major role for ethylene in cotton fiber cell elongation [J]. Plant Cell, 2006, 18(3): 651-664.
[20] Chen C, Liu M, Jiang L, et al. Transcriptome profiling reveals roles of meristem regulators and polarity genes during fruit trichome development in cucumber (L.) [J]. J Exp Bot, 2014, 65(17): 4943-4958.
[21] Pan Y, Bo K, Cheng Z, et al. The loss-of-functionmutation in cucumber is due to LTR-retrotransposon insertion in a class IV HD-ZIP transcription factor genethat is epistatic over[J]. BMC Plant Biology, 2015, 15: 302. doi: 10.1186/s12870-015-0693-0.
[22] Liu X, Bartholomew E, Cai Y, et al. Trichome-related mutants provide a new perspective on multicellular trichome initiation and development in cucumber (L) [J]. Front Plant Sci, 2016, 7: 1187. doi: 10.3389/fpls.2016.01187.
[23] Zheng K, Tian H, Hu Q, et al. Ectopic expression of R3 MYB transcription factor genein Arabidopsis, but not rice, affects trichome and root hair formation [J]. Sci Rep, 2016, 6: 19254. doi: 10.1038/srep19254.
[24] Qin B, Tang D, Huang J, et al. Rice OsGL1-1 is involved in leaf cuticular wax and cuticle membrane [J]. Mol Plant, 2011, 4(6): 985-995.
[25] Ming T, Bartholomew B. Theaceae [J]. Flora China, 2007, 12: 366-478.
[26] 陈亮, 虞富莲, 童启庆. 关于茶组植物分类与演化的讨论[J]. 茶叶科学, 2000, 20(2): 89-94.
Chen L, Yu F L, Tong Q Q. Discussion on the classification and evolution of tea plants [J]. Journal of Tea Science, 2000, 20(2): 89-94.
[27] Li P, Xu Y, Zhang Y, et al. Metabolite profiling and transcriptome analysis revealed the chemical contributions of tea trichomes to tea flavors and tea plant defenses [J]. J Agric Food Chem, 2020, 68(41): 11389-11401.
[28] Zhu M, Li N, Zhao M, et al. Metabolomic profiling delineate taste qualities of tea leaf pubescence [J]. Food Res Int, 2017, 94: 36-44.
[29] Wei C, Yang H, Wang S, et al. Draft genome sequence ofvar.provides insights into the evolution of the tea genome and tea quality [J]. PNAS, 2018, 115(18): E4151-E4158.
[30] Gilbert N. The science of tea's mood-altering magic [J]. Nature, 2019, 566(7742): S8-S9.
[31] Cao H, Li J, Ye Y, et al. Integrative transcriptomic and metabolic analyses provide insights into the role of trichomes in tea plant () [J]. Biomolecules, 2020, 10(2): 311. doi: 10.3390/biom10020311.
[32] Barman T, Baruah U, Saikia J. Seasonal changes in metabolic activities of drought tolerant and susceptible clones of tea (L.) [J]. Journal of Plantation Crops, 2008, 36(3): 259-264.
[33] Konrad W, Burkhardt J, Ebner M, et al. Leaf pubescence as a possibility to increase water use efficiency by promoting condensation [J]. Ecohydrology, 2015, 8(3): 480-492.
[34] Das S, Zaman A, Borchetia S, et al. Genetic relationship in tea germplasms with drought contrasting traits [J]. Plant Breeding and Biotechnology, 2016, 4(4): 484-494.
[35] 杨丽丽, 郑高云, 梁丽云, 等. 茶树抗病虫机制的研究进展[J]. 福建茶叶, 2008(2): 8-11.
Yang L L, Zheng G Y, Liang L Y, et al. Research progress on resistance mechanism to disease and insect in tea plant [J]. Tea in Fujian, 2008(2): 8-11.
[36] Dutta M. Morphological resistance of certain tea clones to red spider mite () in tea [J]. Journal of Entomology and Zoology Studies, 2015, 4(3): 454-457.
[37] Bandyopadhyay T, Gohain B, Bharalee R, et al. Molecular landscape of helopeltis theivora induced transcriptome and defense gene expression in tea [J]. Plant molecular biology reporter, 2015, 33(4): 1042-1057.
[38] Yue C, Cao H, Chen D, et al. Comparative transcriptome study of hairy and hairless tea plant () shoots [J]. J Plant Physiol, 2018, 229: 41-52.
[39] Sun B, Zhu Z, Liu R, et al.() regulates leaf trichome density in tea[J]. Nordic Journal of Botany, 2020, 38(1): 1-10.
[40] Liu R, Wang Y, Tang S, et al. Genome-wide identification of the tea plant bHLH transcription factor family and discovery of candidate regulators of trichome formation [J]. Sci Rep, 2021, 11(1): 10764. doi: 10.1038/s41598-021-90205-7.
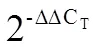
[42] Rerie W, Feldmann K, Marks M. Thegene encodes a homeo domain protein required for normal trichome development in[J]. Genes Dev, 1994, 8(12): 1388-1399.
[43] Luo D, Oppenheimer D. Genetic control of trichome branch number in: the roles of the FURCA loci [J]. Development, 1999, 126(24): 5547-5557.
[44] Payne C, Zhang F, Lloyd A. GL3 encodes a bHLH protein that regulates trichome development inthrough interaction with GL1 and TTG1 [J]. Genetics, 2000, 156(3): 1349-1362.
[45] Zhang F, Gonzalez A, Zhao M, et al. A network of redundant bHLH proteins functions in all TTG1-dependent pathways of[J]. Development, 2003, 130(20): 4859-4869.
[46] Zhao M, Morohashi K, Hatlestad G, et al. The TTG1-bHLH-MYB complex controls trichome cell fate and patterning through direct targeting of regulatory loci [J]. Development, 2008, 135(11): 1991-1999.
[47] Mondal T, Bhattacharya A, Laxmikumaran M, et al. Recent advances of tea () biotechnology [J]. Plant Cell, Tissue and Organ Culture, 2004, 76(3): 195-254.
[48] Szymanski D, Jilk R, Pollock S, et al. Control ofexpression inleaves and trichomes [J]. Development, 1998, 125(7): 1161-1171.
[49] Boeglin M, Fuglsang A, Luu D, et al. Reduced expression ofnucleoporin gene affects auxin response in[J]. BMC Plant Biol. 2016, 16: 2. doi: 10.1186/s12870-015-0695-y.
[50] 张晨光. 苹果核孔蛋白MdNup54/62调控开花和高温胁迫响应功能研究[D]. 杨凌: 西北农林科技大学, 2021.
Zhang C G. Study on the function of apple nuclear pore protein MDNUP54/62 in regulation of flowering and heat stress response [D]. Yangling: Northwest A&F University, 2021.
Functional Identification ofandTranscription Factors in Tea Plants
LIU Renjian1, WANG Yuyuan1, LIU Shaoqun1, SHU Canwei2, SUN Binmei1, ZHENG Peng1*
1. College of Horticulture, South China Agricultural University, Guangzhou 510642, China; 2. College of Agriculture, South China Agricultural University/Guangdong Province Key Laboratory of Microbial Signals and Disease Control/Integrative Microbiology Research Center, Guangzhou 510642, China
Tea plant leaf trichomes contain various secondary metabolites and play an important role in the tea appearance quality as well as the response of tea plants to biotic and abiotic stresses. In this study, the function of leaf trichome-related genesandwere analyzed using Bimolecular Fluorescent Complimentary (BiFC), GUS staining and overexpression experiments. The results show that CsbHLH024/CsbHLH133 and CsTTG1 could interact in plants, and their promoters could drive downstream gene expression in leaf tissues. They were further transformed into wild(Col) and corresponding homozygous mutants, respectively to get overexpression lines. Both genes could affect the leaf trichome formation in, restore the phenotype of the mutants, and induce the expression levels of trichome-related genes. This study provided a theoretical basis for further research on the molecular regulation mechanism of trichome formation in tea leaves.
tea plant, trichome formation,,, molecular regulation
S571.1
A
1000-369X(2022)03-347-11
2021-12-18
2022-01-03
广州市科技计划项目(202102020290)、广东省自然科学基金项目(2018A030313089、2021A1515012091)
刘任坚,男,硕士研究生,主要从事茶树遗传育种与分子生物学研究。*通信作者:zhengp@scau.edu.cn
(责任编辑:赵锋)

