基于酯酶和ISSR技术的平菇单核菌株遗传多样性分析
刘晓雪 王强 张彬彬 王春霞 郭金英 郑素月

摘要:应用酯酶同工酶和ISSR分子标记技术对69个野生平菇单核菌进行遗传多样性研究。结果表明,69个单核菌株的酯酶同工酶酶谱共检出6条酶带,多态性带型占83.3%;ISSR分子标记技术从7个ISSR引物中扩增出56条清晰的DNA片段,多态性位点占87.5%;2种方法的聚类结果显示,当GS值为0.67左右时,根据样本量的多少均可将69个单核菌株依此划分为A、B、C、D等4个类群;将交配型、酯酶同工酶和ISSR分子标记3种分类结果进行对比后发现,交配型为A2B2的P11、P12、P34、P59、P73、P85和P90這7个单核菌株均同属于一类,分类结果并没有严格按照交配型来进行归类。说明酯酶同工酶和ISSR分子标记技术可用于平菇单核菌株的遗传多样性研究,并可作为交配型鉴定的辅助手段,为平菇杂交育种过程中优良亲本单核体的选择提供理论依据。
关键词:平菇;单核菌株;酯酶同工酶;ISSR分子标记
中图分类号:S646.1+40.3 文献标志码: A
文章编号:1002-1302(2022)09-0027-06
平菇(Pleurotus ostreatus)是一种物美价廉、高蛋白低脂肪的健康食品,因其栽培技术简单、适应性强而成为担子菌中广泛栽培的品种之一[1-2]。研究表明,平菇是标准的四极性异宗结合菌,同一菌株产生的担孢子有4种交配型即AxBx、AyBy、AxBy、AyBx[3],而目前食用菌的交配型鉴定通常采用单核体两两交配后镜检观察锁状联合的方式进行,该方法工作量大、耗时长,且易产生人为误检。因此,寻找一种更快速、准确地挑选出优良的单核菌株并鉴定出其交配型因子类型的方法尤为必要。
酯酶同工酶是从蛋白质水平上研究生物群体遗传差异的重要方法之一[4-5],在食用菌种类遗传多样性分析方面应用广泛[6-10]。但同工酶谱的差异是由编码同工酶基因决定的,不能直接反映出菌种在DNA水平上的差别,另一方面,同工酶试验容易受到培养方式、时间等因素影响[11-12]。因此,在酯酶同工酶分析的基础上结合更加稳定的遗传标记方法去分析植株的遗传多样性是非常必要的。简单重复间序列(ISSR)是DNA分子标记方法之一,呈孟德尔式遗传,能从基因水平揭示菌株间的亲缘关系和遗传差异,具有良好的稳定性和多态性[13-14],其检测手段简单、高效,已经大量应用于不同品种和食用菌菌株间的遗传差异分析中[15-19]。宋小亚等以黑木耳单核菌株为试验材料分析了ISSR标记在其育种过程中的应用价值[20];努尔孜亚·亚力买买提等通过ISSR标记技术对15株野生平菇菌株的遗传多样性进行了分析,当遗传相似系数在0.60水平上时,将供试菌株分为了5个类群[21];陈小敏等利用ISSR技术鉴别了从全国年各地收集的75份香菇种质资源的遗传多样性[22];叶海丹研究了ISSR分子标记技术在桑黄、黑木耳遗传分析上的应用[23]。本研究在1个野生平菇单核菌株交配型已确定的基础上(已另文发表),通过酯酶同工酶和ISSR分子标记技术对该平菇的69个孢子单核菌株进行遗传分析和分类鉴定,探讨单核菌株的遗传差异和交配型之间的关系,以期为育种时亲本单核体的筛选提供重要的基础。
1 材料与方法
1.1 供试菌株
本试验于2021年3月5日至6月20日在河北工程大学食用菌研究室完成。试验所需野生平菇4种交配型孢子单核体(A 1B 1、A 2B 2、A 1B 2、A 2B 1),由河北工程大学食用菌研究室鉴定、保藏(表1)。
1.2 试验方法
1.2.1 菌丝体培养
将供试平菇单核菌株活化后,转接至铺有玻璃纸的培养基上隔膜培养,菌丝长满平板后刮取菌丝备用。
1.2.2 酯酶同工酶分析
取0.5 g样品用液氮研磨之后加入700 μL磷酸缓冲液(pH值=7.5),4 ℃ 12 000 r/min 离心10 min后取上清液备用。试验采用聚丙烯酰胺凝胶垂直电泳法[24-25],电泳设备为DYC2-24EN型双垂直电泳仪(北京六一生物科技有限公司),试验具体步骤参照文献[26]。
1.2.3 ISSR分子鉴定
采用植物基因组DNA提取试剂盒(天根生化科技有限公司)提取单核菌株DNA。采用1%琼脂糖凝胶检测DNA的质量和浓度。所用ISSR引物均由生工生物工程(上海)股份有限公司合成,ISSR扩增结合李辉平等的方法[27-28]筛选出最佳程序及电泳条件,ISSR电泳设备采用DYY-6C电泳仪(北京六一生物科技有限公司)。扩增体系(20 μL):2×Taq PCR Mix 10 μL,引物1 μL,DNA模板1 μL,ddH 2O 8 μL。
1.3 数据处理
电泳结果均采用凝胶成像分析系统(北京君意东方电泳设备有限公司)拍照记录并进行条带分析。采取0/1赋值记带,构建数据矩阵,用聚类分析软件NTSYSpc Version 2.10e计算菌株间的遗传相似系数,并根据结果采用SAHN程序,以不加权算数平均组对法(UPGMA)进行聚类分析。
2 结果与分析
2.1 酯酶同工酶结果
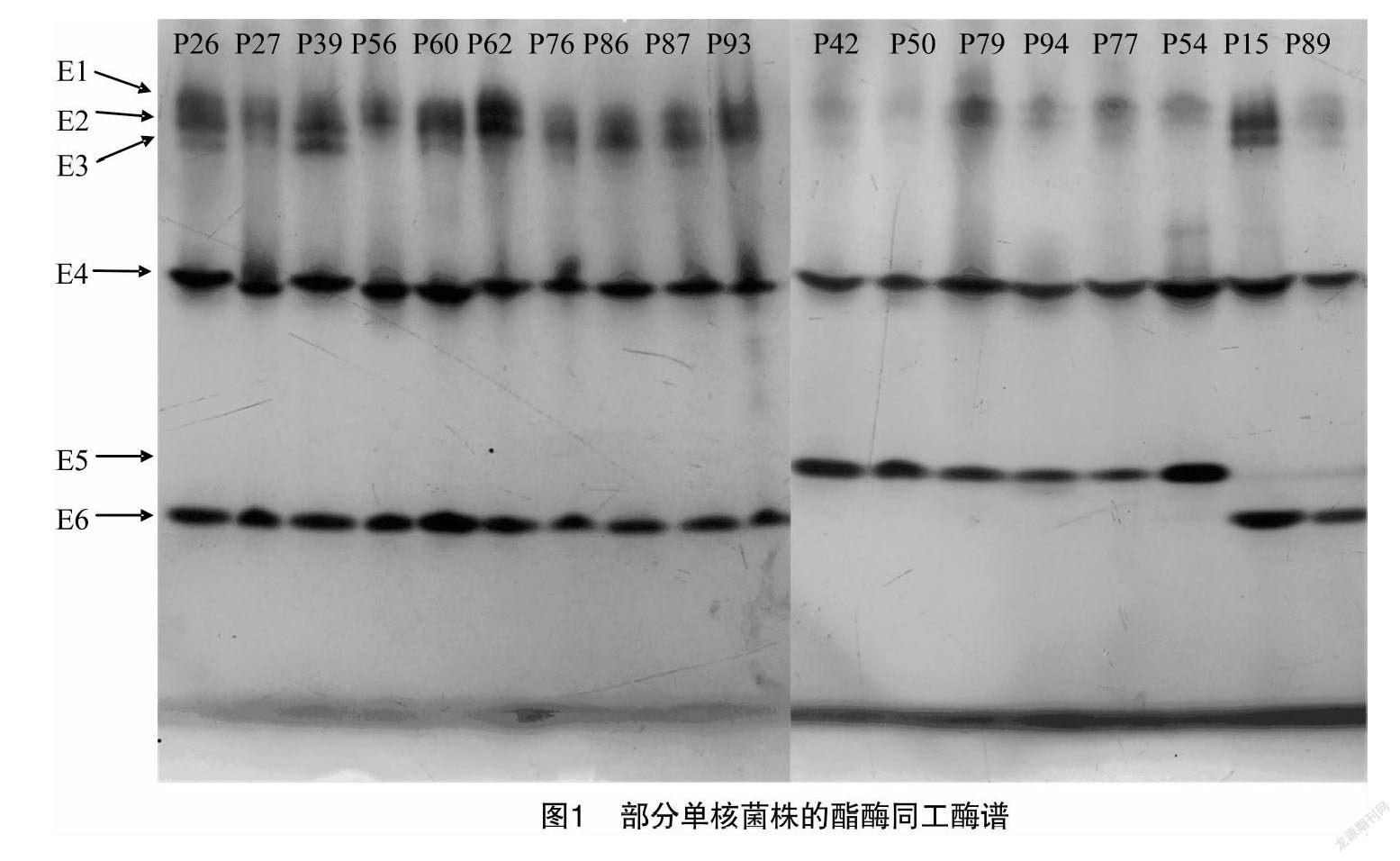
2.1.1 酯酶同工酶酶谱
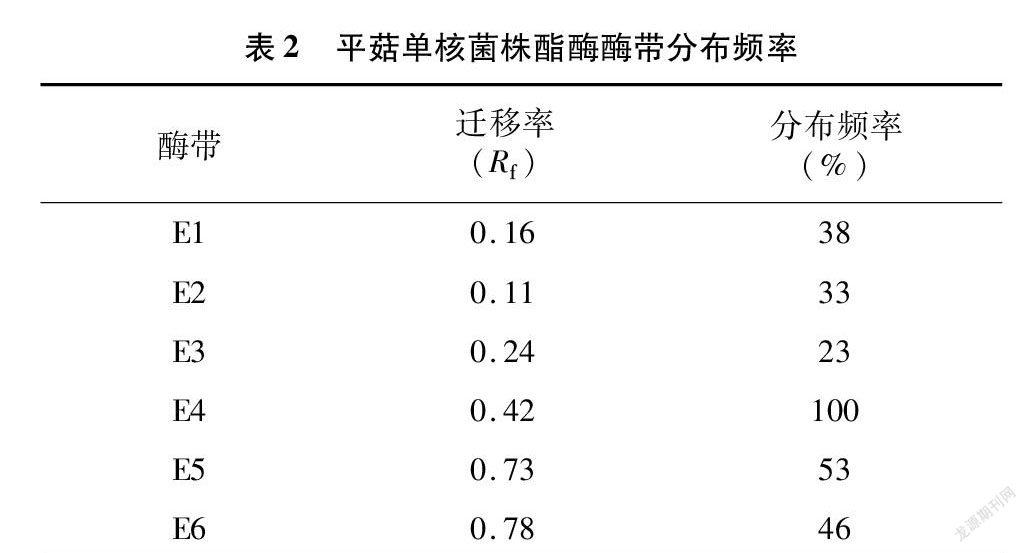
部分孢子单核菌株酯酶同工酶电泳图见图1。由图1可知,根据酯酶同工酶的电泳结果显示,69个平菇孢子单核菌株共检测到6条酶带;各菌株酶带数目为3~5条;迁移率(R f)在 0.16~0.78之间,多态性带型占83.3%(表2)。其中,E4、E5、E6颜色最深,最为明显,并且R f为0.42的E4酶带为69个单核菌株所共有,可认为是野生平菇单核体的特征谱带。
2.1.2 酯酶同工酶聚类分析
聚类分析树状图见图2。由图2可知,69个孢子单核菌株间的遗传相似系数(GS)变化范围为0.38~1.00。当GS值约为0.665时,可将菌株分为4组。根据组内样本量依此分为A、B、C、D等4个类群, A组为P1、P2等31个菌株;B组为P14、P15等21个菌株;C组为P17、P28等10个菌株;D组为P9、P38等7个菌株。DBF325F8-402B-4818-AB45-ED577340AA49
2.2 ISSR引物筛选及聚类分析
2.2.1 DNA质量检测
部分DNA电泳检测结果见图3。由图3可知,在凝胶成像仪下呈现的DNA条带清晰,杂质较少,纯度高,可用于后续PCR扩增。
2.2.2 ISSR扩增图谱
本试验从28个ISSR引物中筛选出7个对野生平菇孢子单核体扩增条带清晰、多态性丰富、重复性好的引物,分别是P2、P5、P7、P11、P19、P24、P26,部分引物扩增图谱见图4、图5。由图4、图5可知,7个引物共扩增出56条条带,条带扩增最多的是引物P2、P19和P26,均扩增出10条;最少的为引物P5和P11,均扩增出6条。平均每个ISSR引物扩增出8条,其中多态性位点49个,占87.5%。
此外,试验根据单核菌株的4种交配类型,每个类型随机抽取1个单核体进行ISSR扩增(图6-A、图6-B)中分别代表引物P7、P24对4种交配型的扩增图谱,可明显看出每个交配类型扩增出来的条带均不同,如图6-A中T 2扩增出的第3条ISSR条带是不同于T 1、T 3和T 4的特异性条带。因此,ISSR分子标记技术可作为该孢子单核体交配型鉴定的有效工具。
2.2.3 ISSR聚类分析
利用ISSR标记计算供试菌株之间的GS值矩阵,采用UPGMA法构建了野生平菇单核菌株的遗传关系聚类图(图7)。由图7可知,69个菌株间的GS值变化范围为0.60~0.88。其中,单核P8和P35、P59和P75间的GS值最大,遗传相似程度最高,遗传距离最近,GS值达到0.88。
由图7可知,当GS值约为0.648时,将可将菌株分为4类。再依据样本量依此划分为A、B、C、D 4个类群,其中,A组由P10、P11等35个菌株组成;B组由P1、P8等22个菌株组成;C组由P9、P21等8个菌株组成;D组由P2、P14等4个菌株组成。
3 结论与讨论
笔者所在课题组前期采集到1株野生平菇菌株,经过ITS鉴定和交配型分析后,将获得的69株可用孢子单核体的交配型进行了准确鉴定(已另文发表)。本试验在此基础上采用酯酶同工酶和ISSR分子标记技术对其进行进一步分析,探讨单核菌株的交配型和遗传差异之间的联系,以便更快捷、准确地挑选交配型相配的优良单核菌株用于育种工作。本试验单从聚类图的分类趋势看来, 这2种聚类图大致呈一种趋势,研究表明,酯酶同工酶试验在69个单核菌株中检测出6条酶带,多态性带型占83.3%,GS值为0.665时将其归为4个类群,A组为P1、P2等31个菌株,B组为P14、P15等21个菌株,C组为P17、P28等10个菌株,D组为P9、P38等7个菌株;ISSR分子標记技术用筛选出的7条引物对69个野生平菇单核菌株的DNA进行ISSR扩增,共扩增出56条条带,多态性位点占87.5%,当GS值为0.684时分为4个类群,A组由P10、P11等35个菌株组成;B组由P1、P8等22个菌株组成;C组由P9、P21等8个菌株组成;D组由P2、P14等4个菌株组成。所以本研究表明,酯酶同工酶和ISSR分子标记技术可用于平菇单核菌株的遗传多态性研究,并可作为交配型鉴定的辅助手段,为其优良菌株的筛选及交配型鉴定提供参考。
观察相同交配型间的单核菌株可发现,相同交配型间单核菌株遗传多态性较低。例如数量居多、占优势的交配型A2B2的37个单核菌株与酯酶聚类A组对比,有P8、P11、P12等16个菌株同属一类;与ISSR聚类A组对比,有P11、P12、P19等20个菌株同属一类。与酯酶同工酶和ISSR聚类结果同时对比后发现,有P11、P12、P34、P59、P73、P85、P90这7个交配型相同的单核菌株同属为一类[20],这与谭琦等对香菇同种交配型孢子单核体的研究结果[29]相符。宋小亚等以黑木耳单核体为材料,研究了ISSR分子标记技术在其遗传分析中的应用,试验中F 1代的33个孢子单核菌株中有24个菌株与T 1聚为一类,剩余9个与T 2聚为一类。徐凯等更是采用了SRAP、ISSR和RAPD等3种方法对37个灵芝的单核体多态性进行了研究,结果表明,试验菌株130-29、130-33等7株交配型为A 2B 2的单核体聚为一类,其余29个既有A 1B 1也有A 2B 2的单核体聚为一类[30]。
但以上结果均未严格按照交配型来进行归类,造成此现象原因极有可能与交配型的控制因子A、B有关。张美彦在对香菇135菌株的遗传规律研究中发现,特异SCAR标记与交配因子A和B均不存在连锁关系,标记是随机的,这是基因组内部减数分裂结果,并且交配型因子在其基因组中所占比例极小,所以即使其交配型因子相同,也不能代表其它核苷酸序列相同[31]。因此,若要准确鉴定某种食用菌的交配型还需与其他方法联合使用。
参考文献:
[1]El-Batal A I,Mosallam F M,Ghorab M M,et al. Factorial design-optimized and gamma irradiation-assisted fabrication of selenium nanoparticles by chitosan and Pleurotus ostreatus fermented fenugreek for a vigorous in vitro effect against carcinoma cells[J]. International Journal of Biological Macromolecules,2020,156:1584-1599.
[2]Kumar K. Nutraceutical potential and processing aspects of oyster mushrooms (Pleurotus Species)[J]. Current Nutrition & Food Science,2020,16(1):3-14.DBF325F8-402B-4818-AB45-ED577340AA49
[3]程 莉. 平菇交配型因子组合的准确鉴定[D]. 武汉:华中农业大学,2007:8.
[4]傅安涛,宋爱荣,田雪梅,等. 同工酶技术及其在我国食用菌研究中的应用[J]. 菌物研究,2006,4(4):57-61.
[5]孔祥会,姚方杰,王 鹏. 食用菌种质资源评价方法及在品种选育上的应用实践[J]. 中国食用菌,2019,38(12):8-10.
[6]隋玉龙,宋 慧,杜 金,等. 灵芝栽培菌株酯酶同工酶的酶谱多样性[J]. 菌物研究,2013,11(2):136-140.
[7]张欣倪,王鸿磊,丁 强,等. 国外引进双孢菇菌种与国内栽培菌种的RAPD和酯酶同工酶指纹图谱分析[J]. 江苏农业科学,2015,43(7):254-256.
[8]高 锋,何明霞,刘 静,等. 基于酯酶同工酶的暗褐网柄牛肝菌遗传多样性分析[J]. 中国食用菌,2021,40(2):47-53,61.
[9]钱雪婷,陈文强,彭 浩,等. 秦巴山区黑木耳菌株酯酶同工酶的分析[J]. 江苏农业科学,2016,44(6):269-272.
[10]Wang H C,Wang Z S. The prediction of strain characteristics of Agaricus bisporus by the application of isozyme electrophoresis[J]. Mushroom Science,1989,12(1):87-100.
[11]梁建光,杨立红,王晓洁,等. 不同条件对食用菌酯酶同工酶谱多态性的影响[J]. 西南农业大学学报(自然科学版),2005,27(4):500-504.
[12]李守勉,李 明,田景花. 不同培养时间对杏鲍菇酯酶同工酶酶谱的影响[J]. 北方园艺,2014(14):98-100
[13]Fang D Q,Roose M L. Identification of closely related Citrus cultivars with inter-simple sequence repeat markers[J]. Theoretical and Applied Genetics,1997,95(3):408-417.
[14]史灵燕,刘保卫,顾新颖,等. 生化和ISSR分子标记在黑木耳品种鉴定中的应用[J]. 北方园艺,2019(9):136-141.
[15]Sudupak M A.Inter and intra-species Inter Simple Sequence Repeat (ISSR) variations in the genus Cicer[J]. Euphytica,2004,135(2):229-238.
[16]孟 虎,孫国琴,睢 韡,等. ISSR技术在食用菌研究上的应用[J]. 北方园艺,2016(5):207-210.
[17]刘志曦,李志强,魏海莲,等. 平菇ISSR遗传多样性分析[J]. 北方园艺,2014(12):94-99.
[18]Nazrul M I,Bian Y B. ISSR as new markers for identification of homokaryotic protoclones of Agaricus bisporus[J]. Current Microbiology,2010,60(2):92-98.
[19]张金霞,黄晨阳,管桂萍,等. 白黄侧耳Pleurotus cornucopiae微卫星间区(ISSR)分析[J]. 菌物学报,2007,26(1):115-121.
[20]宋小亚,肖 扬,边银丙. ISSR标记在黑木耳单核体遗传分析中的应用[J]. 菌物学报,2007,26(4):528-533.
[21]努尔孜亚·亚力买买提,贾文捷,郝敬喆,等. 野生平菇菌株的培养特征及遗传多样性分析[J]. 新疆农业科学,2021,58(1):143-150.
[22]陈小敏,吴海冰,辜运富. 香菇种质资源遗传多样性研究[J]. 四川农业科技,2019(12):40-44.
[23]叶海丹. ISSR分子标记技术在桑黄、黑木耳遗传分析上的应用研究[D]. 杭州:浙江农林大学,2017:55.
[24]胡能书,万贤国. 同工酶技术及其应用[M]. 长沙:湖南科学技术出版社,1985:84-88.
[25]郭尧君. 蛋白质电泳试验技术[M]. 北京:科学出版社,2001:3.
[26]史灵燕. 不同黑木耳品种种质鉴定及优良菌株筛选研究[D]. 邯郸:河北工程大学,2019:21-23.
[27]李辉平. ISSR在食用菌遗传多样性研究中的应用[D]. 北京:中国农业科学院,2007:26-27.
[28]郑 伟. 平菇优良菌株的选育及评价[D]. 邯郸:河北工程大学,2017:14-16.
[29]谭 琦,杨建明,陈明杰,等. 香菇孢子单核体与原生质体单核体遗传差异分析[J]. 中国食用菌,2001,20(6):3-5,25.
[30]徐 凯,唐传红,王天娇,等. 基于SRAP、ISSR和RAPD分析灵芝G0130菌株单核体多态性[J]. 工业微生物,2014,44(4):39-45.
[31]张美彦. 香菇135菌株特异SCAR标记的遗传规律[D]. 南京:南京农业大学,2007:5-6,40.DBF325F8-402B-4818-AB45-ED577340AA49

