应用生物信息学鉴定急性STEMI患者通路及Hub基因
刘 铮,王治财,仓 彦,钱云云,刘 静,尹依恒
(1.安徽理工大学医学院,安徽 淮南 232001 ;2.同济大学附属上海市第十人民医院心内科,上海 200072)
急性心肌梗死(acute myocardial infarction,AMI)是一种给社会和患者带来极大负担的急性冠状动脉疾病[1]。AMI主要包括急性ST段抬高型心肌梗死(ST-segment elevation myocardial infarction,STEMI)和急性非ST段抬高型心肌梗死(non-ST-elevation MI,NSTEMI),其高死亡率是威胁人类健康的主要因素,尤其是STEMI[2]。近年来,心脏生物标志物的发现和评估快速发展,然而目前已广泛应用于临床诊断的生物标志物在急性心肌梗死前4h内血清浓度一般不会达到峰值[3]745。
心肌标志物不仅具有诊断价值,而且对患者的预后能力存在相关性[4-5]。有研究表明肌钙蛋白是很好的预后指标,心肌肌钙蛋白水平升高明显增加心血管事件复发的风险,与肌钙蛋白浓度正常的患者相比,肌钙蛋白升高与死亡或心肌梗死复发风险增加约4倍相关[6-8],即使是在生物标志物不能用于诊断目的STEMI患者中,入院时肌钙蛋白升高与更糟糕的预后相关[9-10]。目前研究表明心力衰竭、肾病、心肌炎等其他病因可造成检测到的肌钙蛋白水平升高[11-12]和检测的“假阳性”[13]。
早期心肌梗死中不能准确确定肌钙蛋白的水平,最近集中在寻找更敏感和特异性的替代标志物,以促进AMI患者早期适当的治疗,在入院早期排除“假阳性”患者,从而尽早阻断或延缓其进展。本研究旨在利用生物信息学分析GEO数据库数据集,从基因表达综合数据库中提取RNA表达谱数据集,进行了注释、合并、标准化去除批次效应,对差异表达基因(differentially expressed genes,DEGs)进行基因本体(Gene Ontology,GO)富集分析和京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)通路分析,蛋白质相互作用(protein-protein interaction,PPI)网络分析后用Cytoscape软件绘制PPI网络图并筛选Hub基因。为了验证模型的稳定性,本研究建立了基于Hub基因的随机森林模型和逻辑回归模型,以期为进一步阐明STEMI的分子机制、帮助STEMI患者早期诊断及个性化治疗提供理论依据。
1 材料和方法
(1)数据采集与预处理
为了确定早期4h内ST段抬高型心肌梗死的关键基因和信号路径,从GEO数据库(https://www.ncbi.nlm.nih.gov/geo/)[14]获得GSE 60993( GPL 6884)和GSE 61144(GPL 6106)的RNA表达谱数据[15]12 981, 研究了ST段抬高型心肌梗死患者和正常人血液样本的微阵列。数据集的纳入标准如下: ①微阵列表达数据; ②人类样本外周血全基因组表达谱; ③数据集包括ST段抬高型心肌梗死患者和正常对照组; ④急性胸痛发作后4h内就诊的STEMI患者的血液。14个样本来自GSE 60993,该样本基于GPL 6884 Illumina HumanWG-6v3.0表达芯片,包括7名ST段抬高型心肌梗死患者和7名健康对照组样本。17个样本来自GSE 61144,该样本基于GPL 6106 Sentrix Human-6v2 Expression BeadChip,包括7名STEMI患者和10名健康对照的样本[15]12 972。随后将数据集中探针中转换为基因符号,将两个表达数据集合并为一个数据集,并且使用R的“SVA”软件包进行标准化处理,以消除批次效应影响[16]。
(2)DEGs鉴定
“limma”包的经验“Bayes”方法用来识别DEGs[17]。火山图和热图通过R的“limma”和“pheatmap”软件绘制。本研究中DEGs的临界值选择标准是矫正后P<0.05,FC=1.8。
(3)GO与KEGG分析
R软件Bioconductor “org.Hs.eg.db”软件包被用来将基因符号转换成entrezID。然后利用R的“cluster profiler”软件包[18]和“pathview”软件包[19]对转化后的entrezID进行GO富集分析和KEGG通路分析(P<0.05和Q<0.05),R的“ggplot2”包用于对结果进行可视化。
(4)PPI构建与Hub基因选择
在STRING数据库(http://string-db.org)中寻找DEGs相互作用关系[20-21],置信度阈值为≥0.7。Cytoscape软件用于绘制蛋白质相互作用关系网络图[22],分析从STEMI患者和正常对照组获得的DEG编码蛋白质之间的关系,应用cytoHubba 插件筛选Hub基因。R的“ggplot2”包用于对筛选出30个分值最大的候选基因进行可视化。
(5)模型的建立
根据蛋白质相互作用网络对筛选出7个Hub基因建立了随机森林模型和逻辑回归模型,其中连续预测变量是7关键基因的表达,结果变量是样本类型(STEMI和正常)。分别采用随机森林R包和R的“glm”包构建随机森林模型和逻辑回归模型[23-24]。R“Caret”包用于在两个模型中的5倍交叉验证[25]。用受试者操作特征曲线评价模型的特异性和敏感性,用曲线下面积(area under the curve,AUC)评价模型的准确性。
2 结果与分析
(1)DEGs的鉴定
与健康对照组相比,在STEMI样本中鉴定出了300个DEGs (标准为矫正后P<0.05和FC=1.8),包括237个上调基因和63个下调基因,火山图和热图如图1(a)和图1(b)所示,图1(b)中每行代表1个DEG,每列代表1个样本;N代表正常,S代表STEMI,显示出显著差异。其中上调基因中前10个差异基因为ALOX5AP、CEBPD、PREX1、SLA、IL18R1、FKBP5、MANSC1、HMGB2、IRAK3、IRS2,下调基因中前10个差异基因为MAP4K1、PTPRCAP、ADA、PLEKHF1、SCAP1、EOMES、GIMAP5、LOC387841、CD6、LOC127295,这些基因中大部分参与炎症、免疫过程,表明炎症、免疫在心肌梗死发生发展过程中起重要作用。
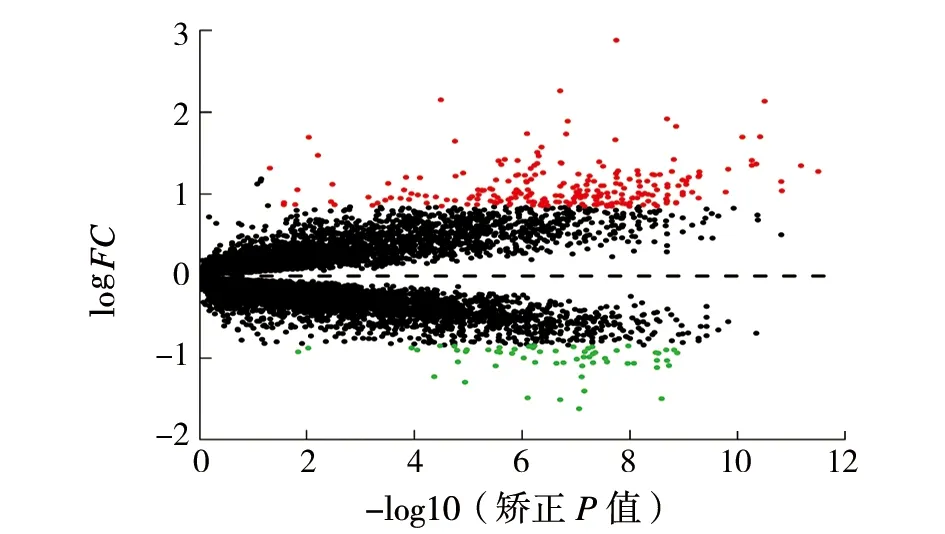
(a)DEGs的火山图
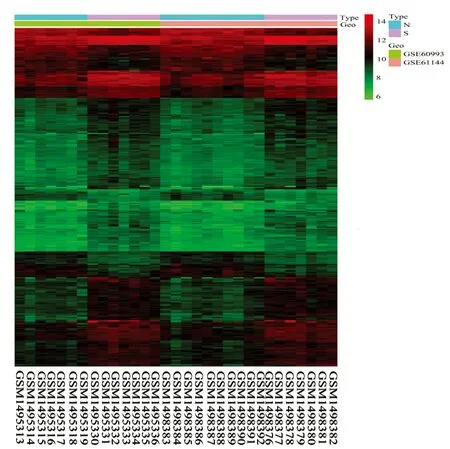
(b)DEGs的热图图1 火山图和热图
(2)GO和KEGG分析
为初步了解这些DEGs,根据已识别的DEGs进行GO和KEGG通路分析。如图2(a)和图2(b)所示,7个GO项和19条KEGG通路在分析中显著富集(P<0.05,校正后P<0.05)。根据GO富集分析表明,脂多糖结合、模式识别受体活性、RAGE受体结合、信号模式识别受体活性和水解酶活性(作用于碳氮,而不是肽键)是最显著富集的生物过程。此外,对于KEGG通路分析结果,利什曼病、造血细胞谱系和炎症性肠病是显著富集的信号途径,这些富集的通路在急性心肌梗死的发生发展中起着重要作用。KEGG途径富集分析表明,DEGs主要参与细胞粘附分子、吞噬体、金黄色葡萄球菌感染、人T细胞白血病病毒-1感染和病毒性心肌炎。

(a)GO分析
(3)PPI网络构建与Hub基因选择
通过筛选的DEGs构建了一个包含119个节点和416条边的PPI网络,并在Cytoscape中绘制了PPI网络图。如图3(a)所示,网络中间为7个Hub基因对应蛋白,所有节点的PPI分值由外向内依次升高,颜色由浅到深。通过R的ggplot2软件包对筛选出30个分值最高的基因进行可视化,如图3(b)所示,其中分值最高的7个基因FPR2、ITGAM、BST1、CEACAM8、MMP9、FPR1和ELANE被确定为Hub基因。

(a)PPI网络与Hub基因
(4)模型的构建
随机森林模型和逻辑回归模型基于PPI网络中选定的7个Hub基因,即FPR2、ITGAM、BST1、CEACAM8、MMP9、ELANE和FPR1,其中连续预测变量为7个Hub基因的表达,结果变量为样本类型(STEMI和正常)。5倍交叉验证随机森林模型和5倍交叉验证逻辑回归模型验证表明,如图4所示,两个模型的验证一致。两个模型关键基因对应样本分类特征表明,31个样本中包括14名STEMI患者和17名健康对照样本均能正确分类,分类准确率为100%,模型对STEMI的敏感性为100%、特异性为100%,ROC曲线下面积都等于1,表明两个模型都能准确地区分STEMI患者和健康对照组,从而验证了模型的准确性。

(a)采用5倍交叉验证法建立随机森林模型
3 讨论
经皮冠状动脉介入治疗是STEMI患者最突出的治疗方法之一。患者从急诊室到血管开通的时间应小于90min,因到达医院后再灌注时间的任何延迟都与住院死亡率的高风险相关[26]。然而,在实现这一目标方面存在重大障碍,涉及交通、医院设施、入院等候时间和诊断检测等问题。由于时间延迟导致了较差的结果,因此继续寻求诊断和治疗时间的改进,特别是在STEMI方面具有较高的死亡率,生物标志物作为重要的诊断标准之一。一般认为,在急性心肌梗死患者的功能失调效应发生之前,影响已经在细胞水平上出现。表明对暴露的血细胞中分子应激反应的分析可以产生关于每种作用模式和AMI不同发病机制的信息。由于分子应激反应是通过基因表达的变化来反映的,因此利用生物信息学方法对GEO数据集(GSE 60993和GSE 61144数据集)进行二次分析进一步阐明了STEMI患者分子机制。
急性心肌梗死的早期发现和适当治疗对于有效的治疗是非常重要的。传统生物标志物如肌钙蛋白(cTnI/T)和肌酸激酶-MB在STEMI的诊断中起着重要作用,但在4h内不能满足早期诊断的要求[3]745。本研究中通过对GSE 60993和GSE 61144数据集的系统深入分析,KEGG和GO分析证明,在STEMI的进展过程中,斑块中的炎症和免疫相关细胞被激活。因此,研究STEMI在心血管事件中的分子机制对其诊断和治疗具有重要意义。从数据集GSE 60993和GSE 61144中筛选出新的潜在ST段抬高型心肌梗死的生物标志物,用于进一步验证。本研究中FPR2、ITGAM、BST1、CEACAM8、MMP9、ELANE和FPR1被确定为STEMI的潜在生物标记物。
总之,通过分析STEMI关键基因来识别和选择出了新的生物标志物。基于这7个关键基因的随机森林模型和逻辑回归模型成功地将STEMI患者与健康对照组区分开来。因此,有必要进一步探讨这些基因在STEMI发病中的诊断意义。由于样本量的局限性,需要更多同质化人群、更大样本量和标准化方案的临床研究,以全面探索STEMI患者在这7个基因特征的预后潜力。

