三红蜜柚、琯溪蜜柚与水晶香柚的基因组序列多态性分析
陶星星 吴亚辉 刘 蕊 李国华 杜小珍 张志标
(1 广东省梅州市农林科学院果树研究所,梅州 514071;2 广东省梅州市农林科学院粮油研究所,梅州 514071)
基因突变指的是DNA 分子中发生碱基对的替换、增添和缺失而引起的基因结构的改变。基因突变主要包括单核苷酸多态性(SNP,Single nucleotide polymorphysim)、插 入 和 缺 失(InDel,Insertion and deletion)、结构变异(SV,Structural variation)、拷贝数变异(CNV,Copy-number variation)等几种[1]。SNP主要是指在基因组水平上由单个核苷酸的变异所引起的DNA 序列多态性,包括单个碱基的转换、颠换等。InDel 指小片段的插入和缺失。编码区或剪接位点处发生的插入缺失都可能会改变蛋白的翻译。移码变异,其插入或缺失的碱基串的长度为3 的非整数倍则可能导致整个阅读框的改变;移码变异与非移码变异相比较,前者对基因功能的影响更大,同时在群体中受到更大的筛选压力。SV 指基因组水平上大片段的插入(INS,Insertion)、缺失(DEL,Deletion)、倒置(INV,Inversion)、染色体内部迁移(ITX,Intra-chromosomal translocation)、染色体间的迁移(CTX,Inter-chromosomal translocation)[2]。CNV 指基因组片段的拷贝数增加或者减少,由基因组发生重排而导致,一般指长度为1Kb 以上的基因组大片段的拷贝数增加或者减少,主要表现为亚显微水平的缺失和重复,是基因组研究的主要多态性类型之一。
琯溪蜜柚果肉白色、味甜、微酸[3]。三红蜜柚是由琯溪蜜柚经过2 次芽变株系选育得到的优良品种,其果实外皮呈粉红色,果皮下的海绵层也是粉红色的,果肉呈红色,由此得名三红蜜柚[4]。水晶香柚是三红蜜柚芽变新品种,其果肉肉质爽脆、果味香气浓郁,成熟期比三红蜜柚早。三红蜜柚、琯溪蜜柚和水晶香柚3 者之间亲缘关系较近,在重要的农艺、产量性状上也存在较高的相似性,理论上来说除了个别基因变异之外,3 者大部分基因是相同的。
由于对3 者的亲缘关系及遗传相似性的认知仅限于表型性状描述和经验为主,为进一步挖掘3 个品种的亲缘关系和遗传多样性信息,通过对三红蜜柚、琯溪蜜柚和水晶香柚进行重测序,在全基因组水平系统解析了3 者的遗传组成差异,以期对今后蜜柚的亲本利用、重要性状基因区间追溯、新品种系谱分析、基因组水平上研究重要种质的遗传组成等方面提供重要参考。
1 材料与方法
1.1 材料试验材料取自广东省梅州市蕉岭县蕉城镇东山村柚果园,供试品种:琯溪蜜柚(编号为W3)、三红蜜柚(编号为R2)、水晶香柚(编号为S1)。植株生长健康、挂果正常,果园管理措施一致。
1.2 果实农艺性状测定每个品种随机选择10 个果实,果实完全成熟后采集。果实重量使用电子天平(精度:0.001g)测定,重复3 次,取平均值;果实成熟期、果实形状、果皮颜色与光滑度、种子数量、果肉颜色等采用直观观测;可食率=(单果重量-不可食部分重量)/单果重量×100%;可溶性固形物手持测糖仪型号:ATAGO(艾拓)通用型可溶性固体物浓度计PAL-1(NFC);参照GB 5009.86—2016《食品安全国家标准食品中抗坏血酸的测定》测定维生素C 含量[5];可溶性糖参考NY/T 2742—2015《水果及制品可溶性糖的测定3,5-二硝基水杨酸比色法》测定[6];可滴定酸参考GB/T 12293—1990《水果、蔬菜制品可滴定酸度的测定》测定[7]。
1.3 全基因组重测序与多态性位点鉴定用CTAB法分别提取3 个品种叶片的DNA。用琼脂糖凝胶电泳分析DNA 降解程度以及是否存在杂带、RNA及蛋白污染,并通过Nanodrop 检测DNA 的纯度(OD260/280 比值);然后通过Thermo Qubit 3.0 荧光计对DNA 浓度进行精确定量,选择OD 值在1.8~2.0之间,总量在1.5μg 以上的DNA 样品通过Covaris破碎机随机打断成长度为350bp 的片段后,采用Illumina NGS DNA Library Construction Kit 文库构建试剂盒进行建库。文库构建完成后,先使用Qubit 3.0 进行初步定量,稀释文库至1ng/μL,随后使用安捷伦Agilent 2100 生物分析仪对文库的Insert size 进行检测,Insert size 符合预期后,使用Q-PCR方法对文库的有效浓度进行准确定量(文库有效浓度>2nmol/L),以保证文库质量。库检完成后用Illumina 平台进行PE150 双端测序,建库和测序由北京诺禾致源科技股份有限公司完成。有效测序数据通过 Burrow-Wheeler Aligner(BWA)软件[6]比对到华中农业大学柚子(Citrus maxima)参考基因组[7](http://citrus.hzau.edu.cn/orange/download/HWB.chromosome.fa.tar.gz),比对结果经高通量测序数据处理软件SAMtools[8]去除重复,根据比对结果,进行SNP、InDel、SV 和CNV 的检测及注释。
1.4 SNP 检测及注释本研究采用SAMtools(参数:mpileup-m2-F 0.002-d1000)进行个体SNP 的检测。为了降低SNP 检测的错误率,选用SNP 的reads 支持数不低于4;比对质量值(MQ)不低于20为标准进行过滤。
1.5 InDel 检测及注释本研究利用SAMtools(参数:mpileup -m2-F0.002-d1000)检测长度小于50bp 的小片段插入与缺失(InDel),利用ANNOVAR软件对检测出的InDel 进行注释[9]。
1.6 SV 检测及注释本研究利用结构变异检测软件BreakDancer[9],基于Pair-end reads 方法将样品测序序列比对到参考基因组上,检测样品序列与参考基因组间的插入(INS)、缺失(DEL)、倒置(INV)、染色体内部迁移(ITX)、染色体间的迁移(CTX),过滤去掉Paired-end reads 支持数小于2 的SV 结果,并且对其中的INS、DEL、INV、ITX、CTX 通过ANNOVAR[10]进行变异注释。
1.7 拷贝数变异区间鉴定及比较分析通过CNVnator(参数:-call100)进行对供试材料相对于柚子参考基因组的CNV 变异区间检测,即通过基因组上不同的reads 覆盖深度,判断潜在的拷贝数减少(Deletion)和拷贝数增加(Duplication),并通过遗传变异位点注释软件ANNOVAR 进行变异注释[11]。
2 结果与分析
2.1 农艺性状比较田间调查发现3 个柚类品种植株树势基本相同,差异主要体现在果实和果肉外观上(图1、图2、表1)。在果实重量方面,水晶香柚平均单果重1480g,三红蜜柚平均单果重1775g,琯溪蜜柚平均单果重2100g;在果型方面,水晶香柚呈高扁圆型,三红蜜柚和琯溪蜜柚呈倒卵圆型;在果皮方面,琯溪蜜柚成熟期为黄绿色,三红蜜柚和水晶香柚成熟期为黄色但略有红色光泽;琯溪蜜柚果皮下的海绵层为白色,三红蜜柚和水晶香柚果皮下的海绵层为淡粉色;在果肉颜色方面,琯溪蜜柚果肉无色,三红蜜柚和水晶香柚果肉为红色;在种子数量方面,水晶香柚平均种子个数110 个,三红蜜柚和琯溪蜜柚一般无籽或种子发育不良,仅约芝麻大小;在成熟期方面,水晶香柚要比三红蜜柚和琯溪蜜柚早20d 左右;在可溶性固形物、可滴定酸、可溶性糖、维生素C 含量和可食率方面,3 者之间均差别不大;在果顶表现方面,水晶香柚果顶印圈明显完整,三红蜜柚和琯溪蜜柚无印圈或印圈很浅、不明显(图2)。
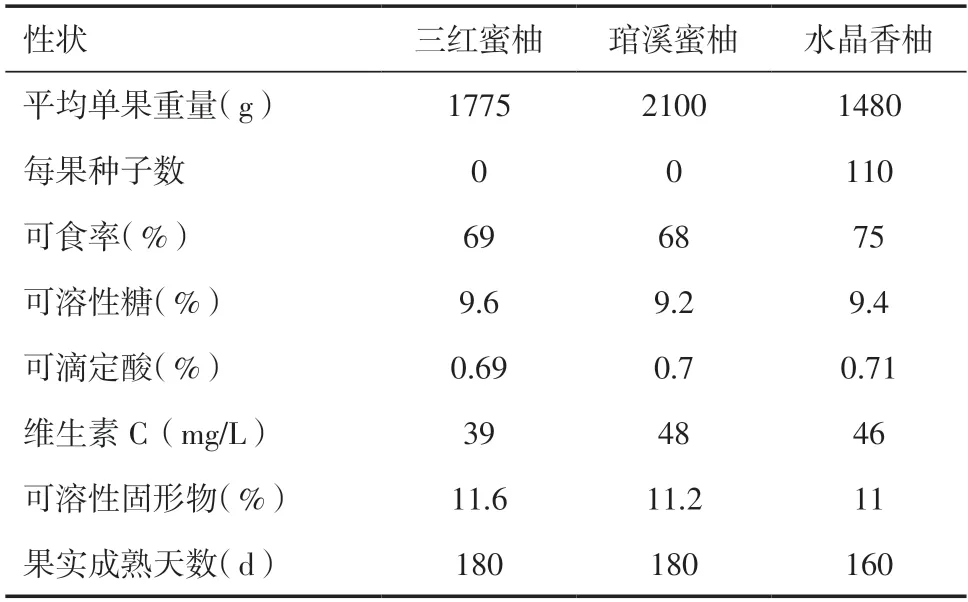
表1 3 个供试品种的农艺性状

图1 3 个供试品种果实果形及横剖面对比

图2 三红蜜柚和水晶香柚果顶印圈对比
2.2 全基因组多态性位点检测利用Illumina 平台对三红蜜柚、琯溪蜜柚、水晶香柚3 个品种进行全基因组测序,分别获得了12.1Gb、11.9Gb、12.4Gb 的重测序数据(表2)。参考基因组大小为345779982bp,所有样本重测序序列的比对率在98.83%~98.93%之间。通过和参考基因组序列进行测序数据的对比并除去重复序列,最终3 个品种重测序数据对参考基因组的平均覆盖深度在28.90×到29.89×之间。
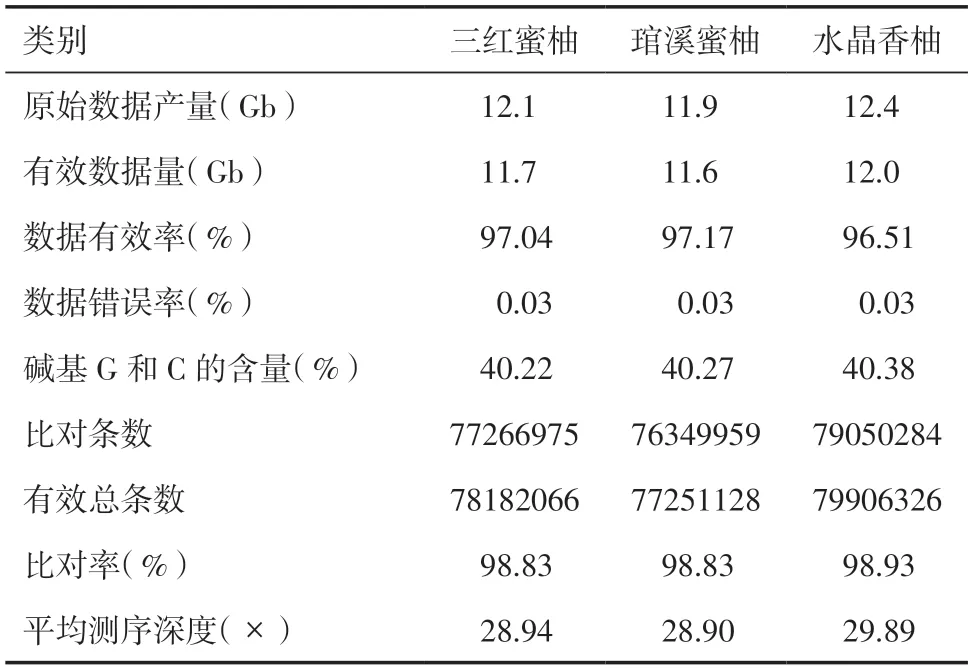
表2 重测序样品测序数据质量与参考基因组比对情况
2.3 SNP 检测及注释结果经过SAMtools 检测,与柚子参考基因组相比,三红蜜柚、琯溪蜜柚、水晶香柚分别鉴定出1672671、1654727、1561376 个SNP(表3),包括外显子区域变异(终止密码子获得变异、终止密码子失去变异、外显子同义变异、外显子非同义变异)、内含子区域变异、剪接位点变异、基因上游1Kb 区域变异、基因下游1Kb 区域变异、基因上下游1Kb 区域同时变异、基因间区变异等类型,其中基因间区变异最多,终止密码子失去变异最少。

表3 重测序样品测序数据SNP 检测及注释结果统计
2.4 InDel 检测及注释结果经过SAMtools 检测、ANNOVAR 软件进行变异注释,与柚子参考基因组相比,三红蜜柚、琯溪蜜柚、水晶香柚分别鉴定出247867 个、239948 个、236264 个InDel,包括基因上游1Kb 区域变异、外显子区域变异(终止密码子获得变异、终止密码子丢失变异、缺失导致蛋白质编码框改变变异、插入导致蛋白质编码框改变变异、缺失但蛋白质编码框未改变变异、插入但蛋白质编码框未改变变异)、内含子区域变异、剪接位点变异、基因下游1Kb 区域变异、基因上下游1Kb 区域同时变异、基因间区变异等类型,其中基因间区变异最多,终止密码子丢失变异最少(表4)。

表4 重测序样品测序数据InDel 检测及注释结果统计
2.5 SV 检测及注释结果经过BreakDancer 软件比对、ANNOVAR 软件进行变异注释,与柚子参考基因组相比,三红蜜柚、琯溪蜜柚、水晶香柚分别鉴定出23591 个、22816 个、22378 个SV,包括基因上游1Kb 区域变异、外显子区域变异、基因下游1Kb 区域变异、内含子区域变异、基因上下游1Kb区域同时变异、基因间区变异、剪接位点变异、片段插入变异、片段丢失变异、片段倒位变异、染色体内部重排变异、染色体间重排变异等类型,片段丢失变异SV(DEL)最多,片段插入变异SV(INS)最少,3个品种均没有片段插入变异(表5)。
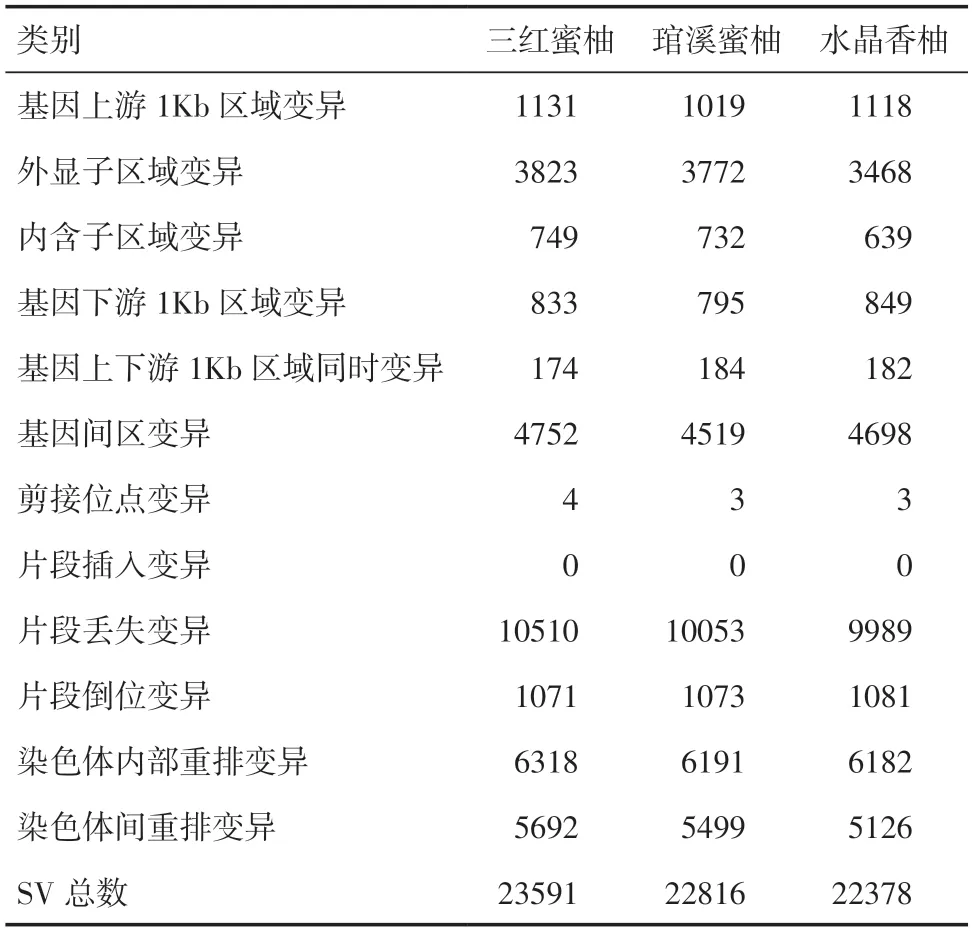
表5 重测序样品测序数据SV 检测及注释结果统计
2.6 CNV 检测及注 释 结果经过CNVnator 检测、ANNOVAR 软件进行变异注释,与柚子参考基因组相比,三红蜜柚、琯溪蜜柚、水晶香柚分别鉴定出5440 个、5445 个、5336 个CNV,包括基因上游1Kb 区域变异、外显子区域变异、内含子区域变异、基因下游1Kb 区域变异、基因上下游1Kb 区域同时变异、基因间区变异、拷贝数增加变异、拷贝数减少变异等类型,其中拷贝数减少变异CNV 最多,基因上下游1Kb 区域同时变异类CNV 最少(表6)。与柚子参考基因组相比,3 个品种基因组拷贝数减少长度在48335200~49301100bp 之间,拷贝数增加长度在14816900~18582000bp 之间。

表6 重测序样品测序数据CNV 检测及注释结果统计
3 结论与讨论
本研究对三红蜜柚、琯溪蜜柚、水晶香柚的农艺性状进行了调查和比较,3 个柚类品种差异主要体现在果实和果肉外观上,在可溶性固形物、可滴定酸、可溶性糖、维生素C 含量和可食率方面3 者之间相差不大,水晶香柚果实重量比三红蜜柚、琯溪蜜柚轻,品质与三红蜜柚、琯溪蜜柚不相上下,但成熟期更早。推广种植水晶香柚满足梅州柚种植户对早熟新品种的需求,能有效抢占市场先机,可以有效解决梅州柚成熟期较为集中的问题,优化梅州蜜柚品种结构。
本研究通过对三红蜜柚、琯溪蜜柚和水晶香柚3 个柑橘品种的重测序数据进行分析,系统研究了3 个品种在基因组水平上存在的CNV 区间和SNP 热点区间,比较了单核苷酸多态性(SNP)、插入/缺失(InDel)、结构变异(SV)、品种拷贝数变异(CNV)序列差异,在全基因组水平解析了3 者的遗传差异。与柚子参照基因组比较发现,3 个品种均有大量SNP、InDel、SV、CNV,变异类型总数SNP>InDel>SV>CNV,其中基因间区SNP 最多,终止密码子失去变异类SNP 最少;基因间区InDel 最多,终止密码子丢失InDel 最少;片段丢失变异SV(DEL)最多,片段插入变异SV(INS)最少;拷贝数减少变异CNV 最多,基因上下游1Kb 区域同时变异类CNV 最少;与柚子参照基因组相比,3 个品种基因组中各类变异的数量非常接近。本研究为进一步研究三红蜜柚、琯溪蜜柚和水晶香柚提供了重要的基因组变异数据,为今后柚子育种改良中更好地利用提供了重要遗传信息。本研究的方法为以后借助基因组测序方法快速定位和克隆候选基因提供了新的参考。

