茶树RAV基因家族鉴定与分析
田时雨, 张蓓林, 郑 吟, 吕立堂
(1.贵州农业职业学院, 贵阳 551400; 2.贵州大学茶学院, 贵阳 550025;3.贵州建设职业技术学院, 贵阳 551400)
作为植物中重要的抗逆性基因的一种,RAV基因家族对调控植物基因表达应对环境逆境胁迫起到了十分重要的作用[1],具有GmRAV3的转基因拟南芥植株提高了对高盐和干旱的抗性[2],拟南芥过表达AtRAV1的植株也增强了对高盐和干旱的耐受性[3],但同时也对植物生长有负调控作用,导致侧根生长受到抑制[4]和开花时间提前的情况[5]。在高盐和干旱的情况之下AtRAV1和AtRAV2的表达则受到了抑制[4],而将其转到棉花中则可提高棉花的耐干旱能力[6]。同时RAV基因家族在植物生长发育过程中也起到了至关重要的作用,可调控其对于植物激素的应答,如生长素[7]、ABA[8]、乙烯[9]和油菜素内酯[10]等。
RAV基因家族是具有B 3和AP 2两个结构域或只具有B 3结构域的基因家族[2],其中AP 2位于N-末端[11],B 3位于C-末端[12]。拟南芥中有13个RAV基因,其中有6个具有AP 2结构域,其他则只具有B 3结构域[13];大豆中同样有13个RAV基因,其中仅有5个具有AP 2结构域[14];番茄中则只有10个RAV基因,其中也只有3个有AP 2结构域[15]。可见不同物种之间RAV基因家族数量和具有AP 2结构域的基因数量有一定的变化。
茶树(CamelliasinensisL.)原产于我国西南地区,现广泛种植于南方各省,是一种极为重要的经济作物。研究茶树在逆境胁迫下的基因表达,提高其在逆境胁迫下的产量,对我国未来茶叶发展具有极为重要的意义。而RAV基因家族在植物抗逆和植物生长中起到了非常关键的作用。本研究通过茶树基因组数据[16-17],比对寻找茶树RAV基因,对其进行鉴定和分析,以期为进一步研究茶树RAV基因奠定基础。
1 材料与方法
1.1 茶树RAV基因家族的鉴定及筛选
茶树RAV基因组相关数据从茶树(http://tpia.teaplant.org/)数据库中进行下载,从而构建本地数据库,以拟南芥已经鉴定出的13个RAV蛋白序列为目标序列,在茶树蛋白库中进行BLAST对比,设定阈值为E<1e-5,进而得到假设的茶树RAV基因,继而使用鉴定得到的茶树RAV基因,再在NCBI在线软件(http://blast.Ncbi. nlm.nih.gov/Blast.cgi)上进行BLAST比对。将第二次所得的茶树RAV基因蛋白序列在NCBI-CDD(https://www.ncbi.nlm.nih.gov/Structre/cdd/wrpsb.cgi)进行保守结构域的鉴定,删除部分冗杂序列,最终得到茶树RAV基因。在Expasy(https://web.expasy.org/compute_pi/)上鉴定茶树RAV蛋白序列的等电点(PI)、分子质量和平均亲水系数。
1.2 茶树RAV基因的亚细胞定位预测
利用(http://www.csbio.sjtu.edu.cn/bioinf/plant-multi/)对茶树RAV蛋白进行亚细胞定位预测[18]。
1.3 系统进化树、基因结构和蛋白质保守基序分析
利用进化树分析软件MEGA 7使用muscle对拟南芥和茶树的RAV蛋白进行比对,继而采用临近法(neighborjoining,NJ)(Boostrap=1 000)进行系统进化树的构建。再将茶树RAV蛋白序列提交至MEME(http://meme-suite.org/)进行保守序列分析,设置为10个motif数量,motif宽度范围设置为6~50氨基酸。将茶树RAV基因的DNA序列在GSDS 2.0(http://gsds.cbi.pku.edu.cn/)进行基因结构分析[19]。
1.4 茶树RAV顺势作用元件分析
利用茶树基因组数据库,截取茶树RAV基因上游2 000 bp的基因序列,在PlantCare(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)中进行顺势作用元件的分析,最后利用GSDS 2.0软件进行可视化分析。
1.5 茶树不同组织及胁迫条件下RAV的表达分析
根据已鉴定的茶树RAV基因编号,在TPIA(http://tpia.teaplant.org)数据库上下载它们在茶树不同组织(包括顶芽,嫩叶,成熟叶,老叶,花,茎,根)、盐胁迫、寒冷胁迫、干旱胁迫和茉莉酸甲酯胁迫的转录组数据(TPM值),对数据进行标准化处理,最后再利用TBtools软件构建热图对其进行可视化处理。
2 结果与分析
2.1 CsRAV基因的鉴定及其理化性质
根据拟南芥13个RAV蛋白序列,在茶树基因组蛋白序列中通过两次BLAST比对,得到备选基因后,利用SMART、NCBI-CDD等在线网站对备选基因进行保守域结构的验证,删除部分冗杂序列,共得到10个CsRAV基因。为便于后续研究,将其命名为CsRAV1-CsRAV10。CsRAV基因编码蛋白质理化性质见表1。结果显示,CsRAV编码蛋白最短,为287个氨基酸(CsRAV 7),最长为500个氨基酸残基(CsRAV 5);CsRAV 1、CsRAV 6、CsRAV 8和CsRAV 10的等电点小于7,属于酸性蛋白质,CsRAV 2、CsRAV 3、CsRAV 4、CsRAV 6、CsRAV 7和CsRAV 9等电点大于7属于碱性蛋白质;分子量最大的是CsRAV 5(54 544.23),分子量最小的是CsRAV 7(31 732.14);所有的CsRAV蛋白的平均亲水系数都小于0,说明其均为亲水性蛋白;CsRAV蛋白的亚细胞定位除CsRAV 3位于细胞质外,其余均位于细胞核。
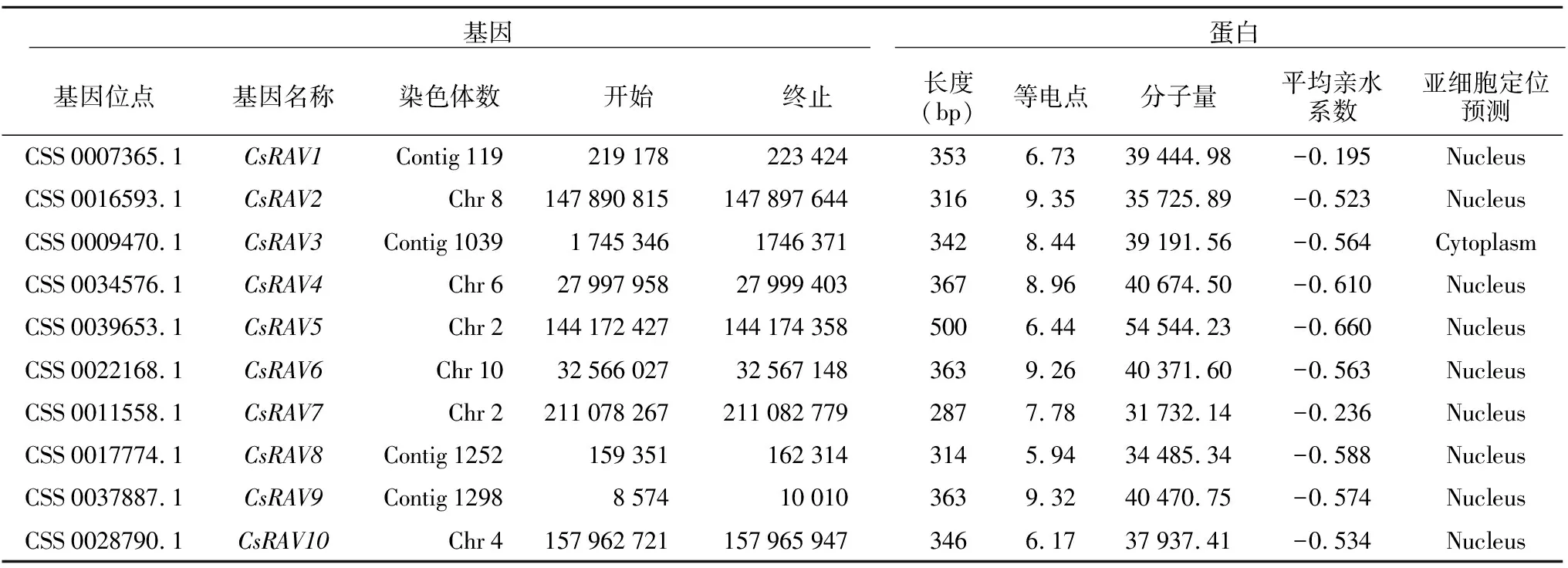
表1 CsRAV基因家族特性Tlabe 1 Characteristics of CsRAV gene family
2.2 CsRAV系统进化分析
将拟南芥的13个RAV(AtRAV)蛋白与茶树的10个RAV(CsRAV)蛋白进行多序列比对,并利用临近法(NJ)构建系统发育进化树,如图1,其中CsRAV 5、CsRAV 8和CsRAV 10划分入Ⅰ亚族;CsRAV 4、CsRAV 6和CsRAV 9划分入Ⅱ亚族;CsRAV 1、CsRAV 2、CsRAV 3和CsRAV 7划分入Ⅲ亚族。从系统进化树中不难发现AtRAV和CsRAV基因同源性相对较低,暗示了RAV基因的分化可能早于茶树与拟南芥之间的物种分化。
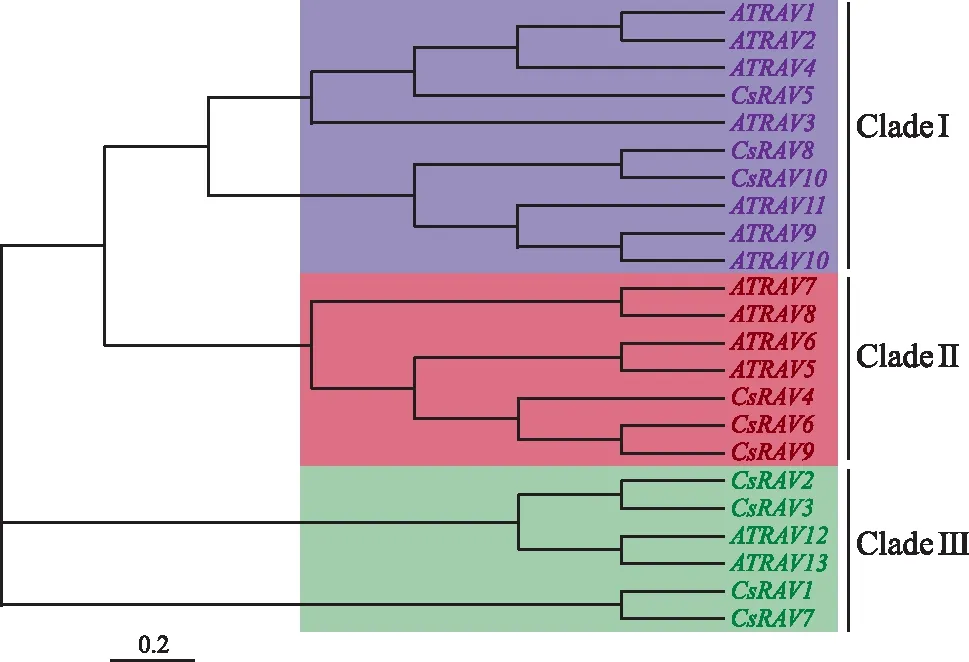
图1 RAV基因家族系统进化树Fig.1 Phylogenetic tree of RAV gene family
2.3 CsRAV基因家族染色体定位
根据茶树基因组信息,可将CsRAV基因定位于茶树染色体上(如图2)。从图2可以看出,2号染色体上有2个CsRAV基因,4、6、8、10号染色体上各有1个CsRAV基因,其他染色体上没有发现CsRAV基因。而CsRAV1、CsRAV3、CsRAV8和CsRAV9无法定位在茶树染色体上。可能是CsRAV基因在茶树进化过程中发生了进化或丢失,从而导致这种基因分布的发生。
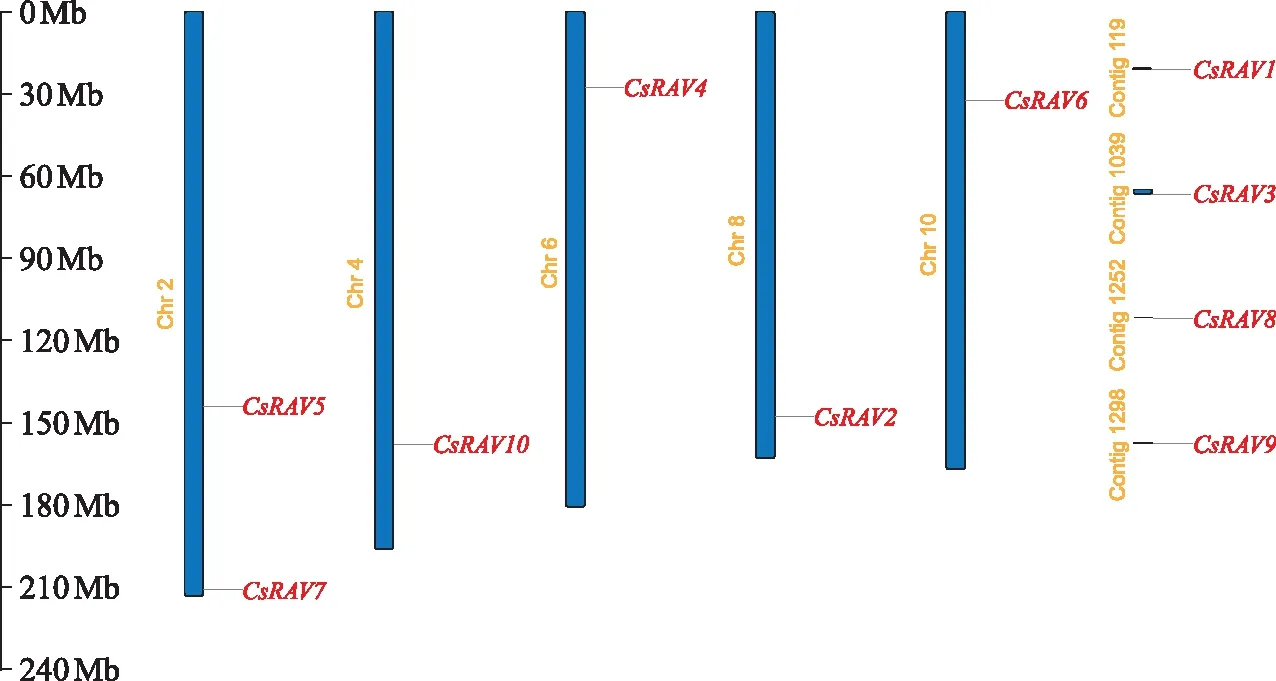
图2 CsRAV的染色体定位Fig.2 Chromosomal localization of CsRAV
2.4 CsRAV基因保守结构域的分析
CsRAV蛋白保守结构域及motif,其结果如图3所示,可以看出各亚族之中motif和保守结构域基本一致,其中motif 3和motif 4构成AP 2结构域,CsRAV3、CsRAV4、CsRAV6和CsRAV9具有AP 2保守结构域,其他CsRAV仅具有B 3结构域。
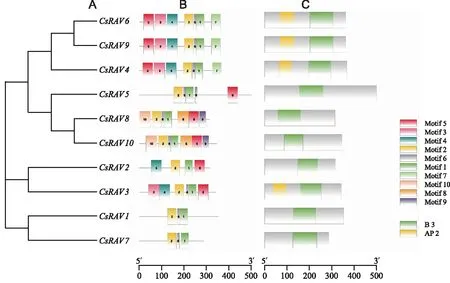
图3 CsRAV系统进化树(A)、保守基序(B)和保守结构域(C)Fig.3 Phylogenetic tree, conserved motifs and domains of CsRAV
2.5 CsRAV基因启动子顺势作用元件分析
提取CsRAV基因上游2 000 bp的启动子区域序列,在PlantCARE对其进行分析预测,其结果详见图4。结果(图4)显示,CsRAV基因启动子上主要元件有逆境胁迫相应相关元件,如干旱、高盐逆境胁迫相应元件:MYB、MBS、DRE和LTR;低温胁迫相应元件:MYC和TCA;抗伤害相应元件:TC-rich除逆境胁迫相应元件外,还有相当的植物激素相应元件,如茉莉酸甲酯相应元件:CGTCA-motif;赤霉素相应元件:GARE-motif;脱落酸相应元件:ABRE;生长素相应元件:AuxRR和TATC。进一步分析可知,逆境胁迫相应元件如MYB、CGTAC-motif、MYC和LTR等在CsRAV家族中大量富集,说明其在茶树逆境胁迫响应和调节茶树生长上发挥重要作用。
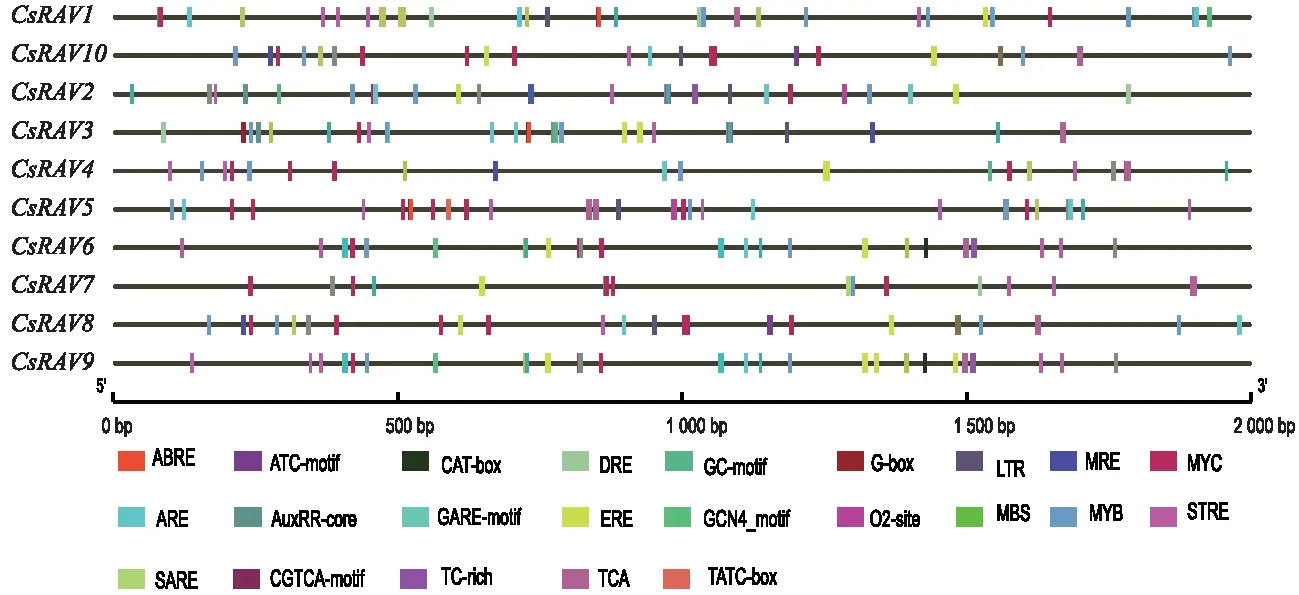
图4 CsRAV基因启动子顺势作用元件Fig.4 Homeopathic element of CsRAV gene promoter
2.6 CsRAV基因在茶树不同组织中表达情况
为进一步了解CsRAV在茶树不同组织的表达情况,从TPIA上下载了茶树不同组织转录组的TPM表达数据,对数据进行标准化处理,并对其结果构建热图进行分析研究,其结果详见图5。CsRAV在茶树不同组织的表达情况有很大差别,CsRAV2和CsRAV3(均来自Ⅰ亚族)在茶树各组织中均不表达;CsRAV6和CsRAV9(均来自Ⅱ亚族)表达量最高,但其在不同组织中表达情况不同,在老叶和果实中表达量最高,而在花中则表达量最低。表明不同的CsRAV基因可能参与调控茶树不同阶段的生长发育。
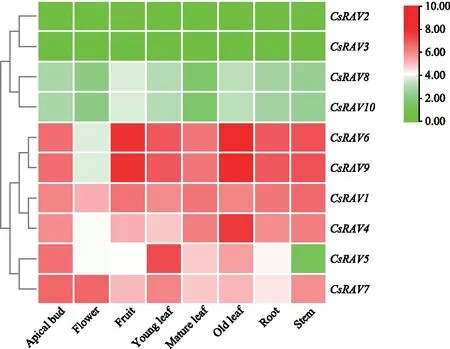
图5 CsRAV在茶树不同组织的表达模式Fig.5 Expression patterns of CsRAV in different tissues of tea plant
2.7 CsRAV基因在不同胁迫处理下的表达模式
利用TPIA下载的在不同胁迫条件下转录组的TMP值,对数据进行0-1标准化处理,构建热图,其结果如图6所示,在盐胁迫条件下(A),CsRAV8和CsRAV9其表达量持续上升,在处理72 h后达到最高;而CsRAV1、CsRAV5、CsRAV6、CsRAV7和CsRAV9的表达量则呈先上升后下降的整体趋势,在48 h后达到最高,而后呈下降趋势。
在干旱胁迫条件下(B),CsRAV1、CsRAV6和CsRAV9呈持续下降趋势,在处理72 h后达到最低;CsRAV8和CsRAV10表达量呈持续上升趋势;而CsRAV5表达量先上升后下降;CsRAV7的表达量则正相反先下降后上升。
在茉莉酸甲酯处理条件下(C),CsRAV7、CsRAV8和CsRAV10表达量呈持续下降,在处理48 h后达到最低,而CsRAV5、CsRAV6和CsRAV9的表达量呈先降后升的趋势,在处理48 h后达到最高;CsRAV1表达量则呈先降后升的整体趋势。
在寒冷处理条件下(D),在CA 1条件下CsRAV5、CsRAV7、CsRAV8和CsRAV10被诱导上调,CsRAV6和CsRAV9则被诱导下调,而CsRAV1则不受诱导影响;在CA 2条件下,CsRAV1、CsRAV5和CsRAV7被诱导上调,而CsRAV5、CsRAV9、CsRAV8和CsRAV10则被诱导下调。其余CsRAV2、CsRAV3和CsRAV4则完全不表达。
3 讨 论
本研究通过对茶树基因族的比对,共找到10个RAV基因,发现其数量与其他物种的RAV基因数量变化不大,如拟南芥13个[13],大豆13个[14],番茄10个[15],烟草6个[20],棉花10个[21],虽然各物种之间的RAV基因数量存在一定的差异,但变化不大,说明RAV基因家族在植物进化过程中相对保守。通过分析CsRAV基因的保守结构域和基序可知,共有4个CsRAV基因具有AP 2结构域,其余6个CsRAV基因仅具有B 3结构域。这有利于帮助确定多基因家族的进化关系[22],10个CsRAV基因可划分入3个亚族,其中Ⅰ亚族的3个基因均具有AP 2结构域,通过对基序的进一步部分析,可确定时motif 3和motif 4共同组成AP 2结构域。
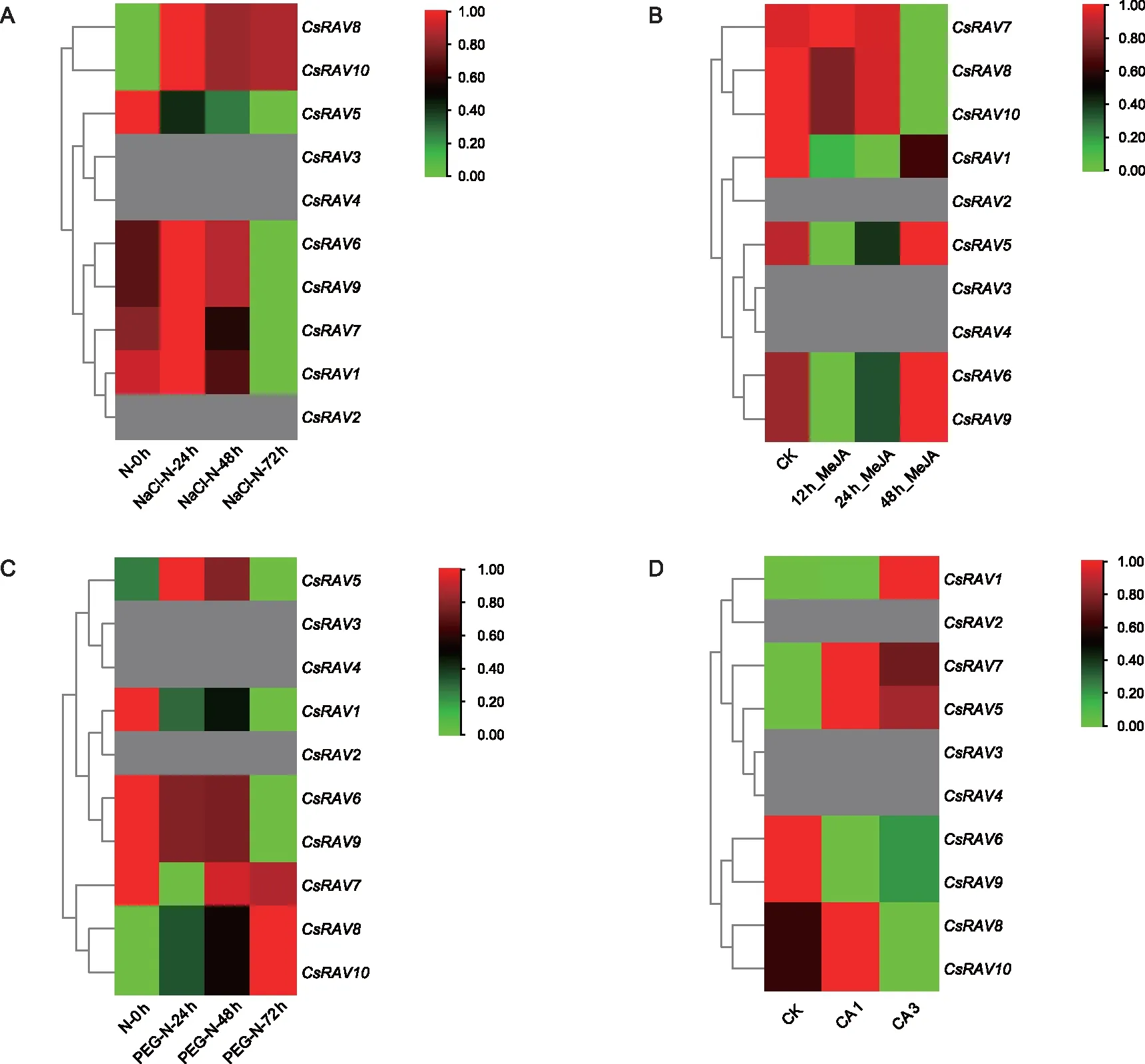
注:A为盐胁迫处理(0,24,48,72 h);B为干旱胁迫处理(0,24,48,72 h);C为茉莉酸甲酯处理(0,24,48 h);D为非驯化ck;最低点,完全驯化CA 1;平均温度达到10°以上,去驯化CA 2)。 图6 CsRAV基因在盐(A),干旱(B),茉莉酸甲酯(C)和寒冷冷(D)胁迫下的表达分析 Fig.6 Expression analysis of CsRAV gene under salt, drought, methyl jasmonate and cold stress
RAV基因对于植物抗逆[23]和调控植物生长发育有着非常重要的作用,通过对CsRAV基因的启动子分析可知,其顺势作用元件多与抗逆和调控植物生长相关[24]。其基因表达的分析也侧面印证这一结论,CsRAV基因在茶树不同组织表达情况存在很大差异可知,不同的CsRAV基因在茶树不同生长发育阶段所起到的作用有所不同,通过抗逆表达可知,在不同胁迫的条件下,各个CsRAV基因的表达变化趋势也有所区别,如CsRAV5具有相应寒冷逆境的顺势作用元件MYB[25],在寒冷胁迫下其表达量呈上升趋势,说明其可能具有调节茶树寒冷胁迫的能力。
4 结 论
茶树RAV家族作为一种重要的基因家族,对茶树生长发育和抗逆性起到重要作用,本研究通过鉴定分析茶树RAV基因家族共找到10个茶树RAV基因家族成员,通过保守结构域和基序确定其基因家族分支划分,并进一步研究其启动子上顺势作用元件以及在不同组织和逆境胁迫下的表达情况,发现不同RAV基因家族成员所起到的作用可能也有所不同,如CsRAV5基因就可在茶树抗寒中起到一定的作用,进而影响茶树抗逆性,本研究为下一步继续研究CsRAV基因提供理论依据,以进一步研究茶树的抗逆调控和生长发育。

