南瓜蚜传黄化病毒基因组遗传多样性分析
崔正秀,孙柳清,肖苗苗,王燕飞,罗永平,赫 娟,玉山江·麦麦提,陈 伟
(1. 山西师范大学生命科学学院,山西 临汾 041000;2. 新疆农业科学院,乌鲁木齐 830052)
【研究意义】新疆特殊的地理位置以及气候特征,使其成为国内重要的甜瓜生产基地。随着新疆甜瓜种植面积不断增大,甜瓜产量日益增加,已成为新疆农业生产的重要经济增长点。病毒病一直是影响新疆甜瓜种植的重要病害,受病毒病侵染的甜瓜植株会出现叶片畸形、花叶、黄化等多种症状,最终导致甜瓜果实品质严重下降、产量大大降低。因此明确CABYV的发生情况、种群结构和遗传多样性,对该病毒的有效预防和控制具有重要意义。【前人研究进展】国内许多学者对新疆侵染瓜类作物的病毒进行鉴定发现,侵染新疆甜瓜的病毒主要有西瓜花叶病毒(WMV)、黄瓜花叶病毒 (CMV)、烟草坏死病毒 (TNV)、甜瓜叶脉坏死病毒 (MVNV)和小西葫芦黄化花叶病毒 (ZYMV)等[1-2]。有研究证实在新疆喀什地区大面积暴发的甜瓜黄叶病病原主要为南瓜蚜传黄化病毒[2]。 南瓜蚜传黄化病毒(Cucurbit aphid-borne yellows virus, CABYV)是侵染葫芦科作物的重要的病毒,寄主包括甜菜、莴苣、蚕豆等[3-5]。在1992年的法国,南瓜蚜虫黄化病毒在南瓜和黄瓜植物中第一次被发现[6],后来在西班牙、突尼斯和意大利等国也报道了 CABYV的发生。该病毒为正义单链病毒(+ssRNA),属于黄症病毒科(Luteoviridae)马铃薯卷叶病毒属(Polerovirus),具有直径为25~30 nm的正二十面体球形病毒粒子,全长约5.7 kb,被约200 nt的非编码内部区域(IR)分成两部分[7]。开放阅读框0、1和2由基因组RNA表达,分别编码P0 蛋白、P1蛋白和P1-P2融合蛋白,其中,P1-P2融合蛋白是ORF1在翻译过程中C末端发生核糖体移码的结果。而其它3个阅读框(ORF3、4和5)由亚基因组RNA表达,ORF3编码P3外壳蛋白,该蛋白参与病毒在植物体内的传播和病毒组装,ORF4和ORF5编码运动蛋白P4和蚜虫传播病毒所需的通读蛋白[8]。CABYV局限在寄主植物的韧皮部组织,主要通过蚜虫(棉蚜和桃蚜)以循环、非繁殖的方式传播,可以引起叶片的黄化和增厚,包括最初的褪绿损伤,随后全叶变黄和老叶增厚,到生长季结束时,多达100%的植物出现黄变症状[9],造成田间植株约减产50%。【本研究切入点】CABYV在新疆甜瓜叶片上可引起严重的病害症状,对该病毒进行检测及明确其与不同分离物的遗传多样性有重要意义。【拟解决的关键问题】对在新疆阿克苏市随机采集的疑似感染CABYV的120份甜瓜叶片采用RT-PCR进行检测,明确其发生率并克隆CABYV基因组,并用生物信息技术对从不同国家和宿主中分离到的CABYV序列进行分子变异分析,为明确该病毒病在田间发生的流行规律和制定长期可持续防控策略提供理论基础。
1 材料与方法
1.1 材料
2019年,对在新疆阿克苏市采集的120份新鲜甜瓜叶片进行RT-PCR鉴定。
1.2 引物设计
从NCBI网站中获得从甜瓜中分离到的CABYV全基因组序列(登录号为KR231963.1),运用Primer 5.0进行引物设计,引物序列、产物大小如表1所示。

表1 扩增CABYV的引物
1.3 RNA提取和RT-PCR扩增与测序
根据王燕飞等[10]的方法进行总RNA提取、RT-PCR和转化大肠杆菌并筛选阳性克隆测序。
1.4 系统进化树分析
通过NCBI 网站进行 CABYV的全基因组序列的 BLAST比对,获得 23条与本研究相似的序列(来自不同的国家和寄主),23个CABYV分离物的完整核苷酸数据如表2所示。使用DNAMAN进行核苷酸序列比对,采用MEGA 7.0中的邻接法构建CABYV全基因组的系统进化树,自展值为1000,去掉低于50%的进化树分支。
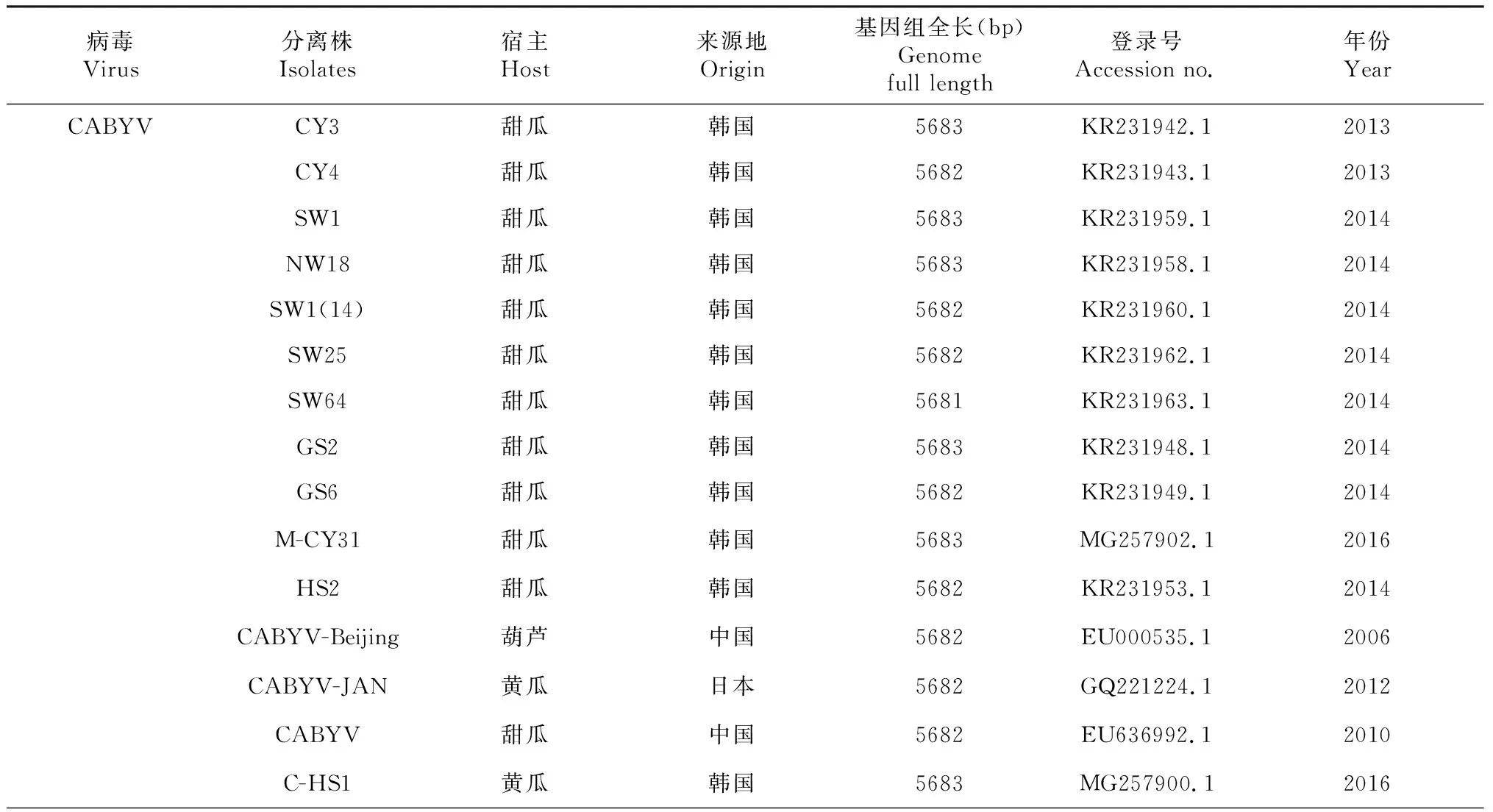
表2 23个CABYV完整的核苷酸数据
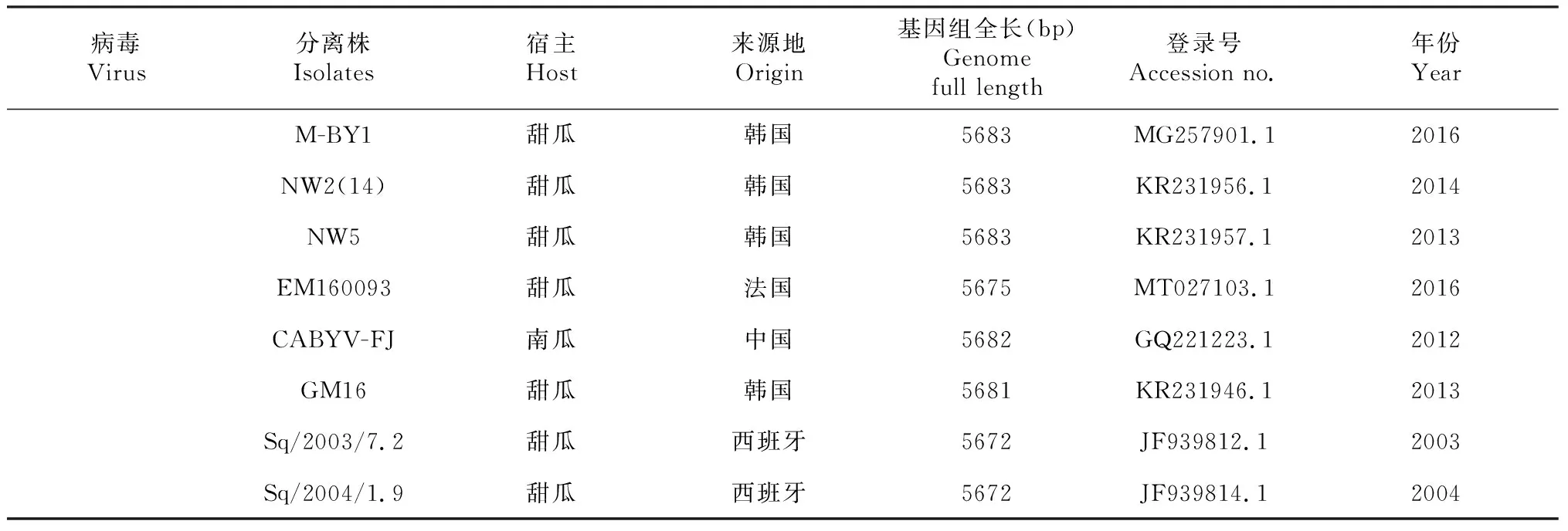
续表2 Continued table 2
1.5 遗传多样性分析
运用DnaSP 软件计算平均成对核苷酸多样性(π),分离位点的数量(S),非同义突变频率(Ka),同义突变频率(Ks),以及非同义突变与同义突变的比率(Ka/Ks)。若Ka/Ks>1,则认为有正选择效应。若Ka/Ks=1,则认为存在中性选择。如果Ka/Ks<1,则认为有纯化选择作用。进一步运用该软件进行Tajima’s D,Fu & Li’s D和Fu & Li’s F的中性检测来判定种群所面对的多样性。其中,Tajima’s D测验主要是在多态位点上对比例进行测定;Fu & Li’s D是在单位点突变的基础上检测数量差异和总体位点突变的数量差异;Fu & Li’s F是计算单位点突变与两两序列差异造成的平均值。
1.6 重组分析
采用重组检测程序RDP v.4.31软件对24个CABYV分离物进行重组分析,使用方法包括RDP、GENECONV、BootScan、MaxChi、Chimaera、SiScan和3Seq。其中接受至少5种不同方法检测到的P<10-6的重组事件为可靠结果。
2 结果与分析
2.1 CABYV的鉴定情况
新疆阿克苏市采集的120份哈密瓜叶片经RT-PCR检测有38份样品为 CABYV阳性。38份CABYV阳性样品测序后通过BLAST(https://www.ncbi.nlm.nih.gov/)在线分析发现,37 个CABYV分离物序列同在线CABYV的核苷酸和氨基酸一致率均为100%,有1个分离物(命名为CABYV-2)与已报道的CABYV分离物在核苷酸和氨基酸水平上均有差异,因此确定CABYV-2是1个新的CABYV分离株(表2)。
2.2 CABYV的基因组结构
在38个CABYV阳性样品中分离到1个新的分离株(CABYV-2),该分离物的全基因组长5497 bp,编码6个开放阅读框,非编码的内部区域(199 nt)被分为近5’端和近3’端两个区域(图1)。其中5’和3’非翻译区分别长为20 和165 nt,近5’端的开放阅读框ORF0编码239个氨基酸的P0蛋白,该蛋白是南瓜蚜传黄化病毒基因组编码的基因沉默抑制子,可能参与了植物防御机制PTGS的抑制[4]。ORF1编码P1蛋白,其氨基酸序列与丝氨酸蛋白酶的氨基酸序列相似。ORF2编码P1-P2功能蛋白,并具有RNA依赖性RNA聚合酶(RdRP)的典型氨基酸基序。近3’端的开放阅读框(ORF3~ORF5)分别编码P3外壳蛋白,P4运动蛋白和P3-P5通读蛋白。
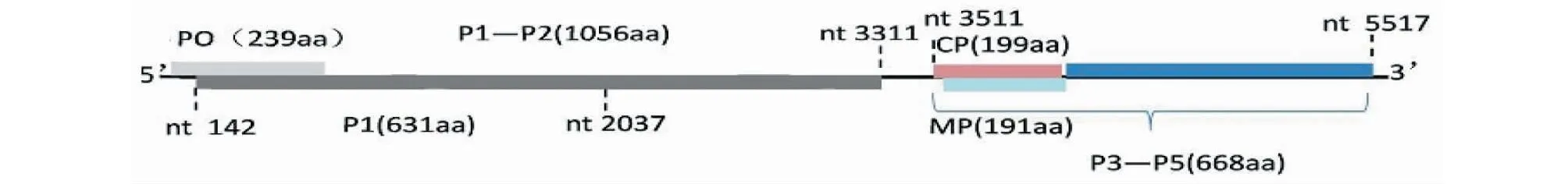
图1 CABYV的全基因组结构Fig.1 The genome structure of CABYV
2.3 系统进化树分析
系统进化树分析结果表明24个分离株被分为 2个分支(图2)。分支1含有21个分离株,这些分离株均来自亚洲,其中17个来自韩国,3个来自中国,1个来自日本。21个分离物株中,从黄瓜、葫芦、南瓜得到的分离株均有1个,其余序列全部从甜瓜中分离得到。本研究中的分离株CABYV-2与分离株CY3(KR231942.1)聚在一起,属于分支1。分支2包括3个分离株,这3个分离株来自于欧洲(西班牙和法国),且都从甜瓜中分离得到。这一结果初步表明遗传变异与地理分布有关,CABYV分离株可能是由于地理因素的不同而分为2个聚集的群,进而造成了遗传多样性的不同。
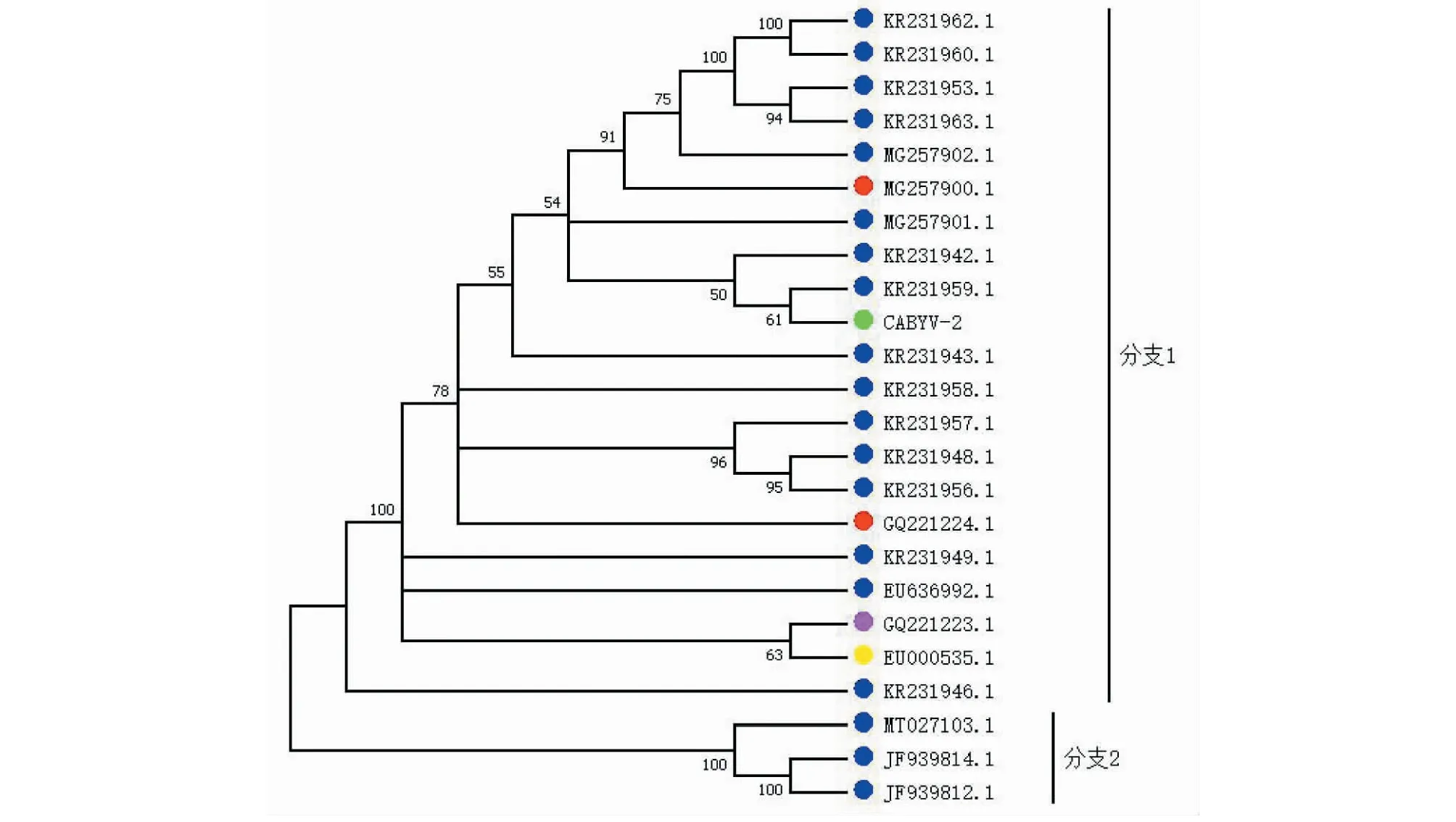
进化树上的彩色圆圈分别代表不同寄主的分离株,深蓝色、黄色、紫色、红色和绿色分别代表甜瓜、葫芦、南瓜、黄瓜和本研究的分离株The color circles on the evolutionary tree represent the isolated strain from different hosts, dark blue, yellow, purple, red and green represent melon, gourd, pumpkin, cucumber and the isolated strain from this study, respectively图2 基于24个CABYV全基因组的系统进化树Fig.2 Phylogenetic tree based on 24 CABYV genomes
为进一步分析这些分离株的多样性,本试验采用MEGA 7.0软件计算这24个分离株的遗传距离,分支1和分支2内的遗传距离分别为 0.043 和0.090,分支1与分支2间的遗传距离为2.024,组间遗传距离明显大于组内。
2.4 序列相似性分析
24 个CABYV分离株的核苷酸同一性范围为89.72%~99.44%(图3-A),氨基酸一致率范围为 81.46%~98.70%(图3-B),呈现出较高的一致性。本研究获得的分离株CABYV-2与序列KR231942.1的核苷酸和氨基酸的同源性都是最高的,其中核苷酸一致性为99.44%,氨基酸一致性为98.70%,与JF939812.1的核酸同源性最低,为89.72%。
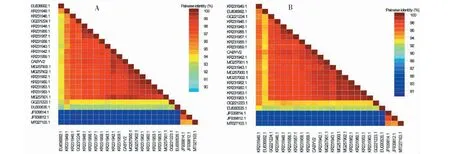
图3 24个CABYV全基因组核苷酸序列(A)和氨基酸序列(B)一致性的二维分布Fig.3 Two dimensional distributions of the consistency of nucleotide sequence (A) and amino acid sequence (B) of 24 CABYV genomes
2.5 遗传多样性分析
24个基因的Ka值明显小于Ks值,即ω均小于1(表3),说明CABYV分离物的各个基因均受到纯化选择的影响。此外,不同基因的单体型多态性值均接近1,π值在0.078~0.156,π值最高的基因是P1基因,说明该基因的变异性更大。通过Tajima’s D、 Fu&Li’s D和Fu&Li’ s F 3种检验,P1、P3和P4基因的检测值为负数 (表3),说明CABYV的种群处于扩张阶段。
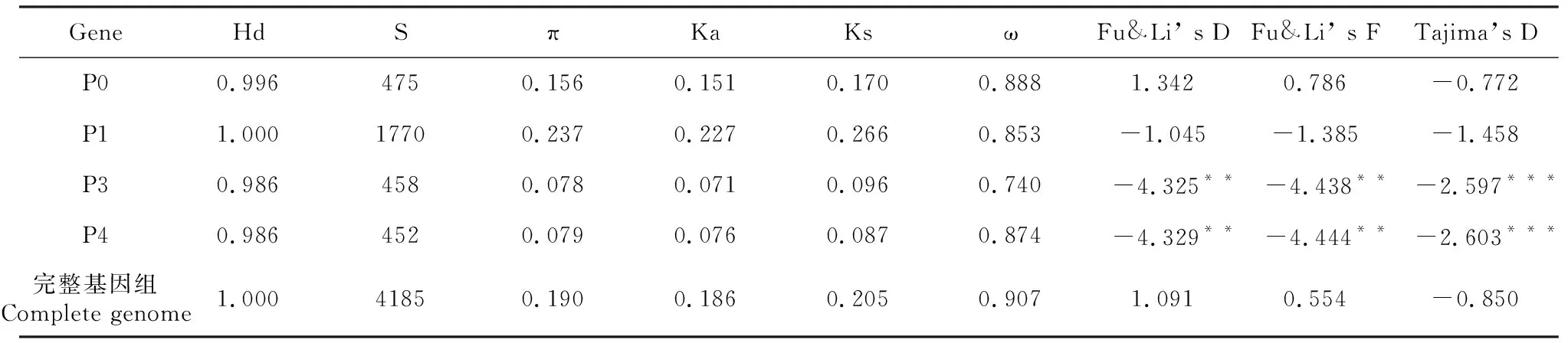
表3 24个不同CABYV分离株的遗传多样性

表4 CABYV种群差异检验
2.6 重组分析
为了检测CABYV群体中是否发生了重组事件,运用RDP软件中的7种方法分析了24个CABYV分离株的核苷酸序列。表明,在24个CABYV(序列或分离株)中有2个重组事件(表5),且这2个重组事件 GS6 (登录号:KR231949.1)和CABYV(登录号:EU636992.1)均位于分支1。来自新疆的分离株EU636992.1、本研究分离株CABYV-2以及GQ221223.1都参与了重组事件的发生,且EU636992.1重组事件是在甜瓜分离株CABYV-2和南瓜分离株GQ221223.1之间发生的,这说明CABYV的重组是可以在不同的寄主之间发生的。
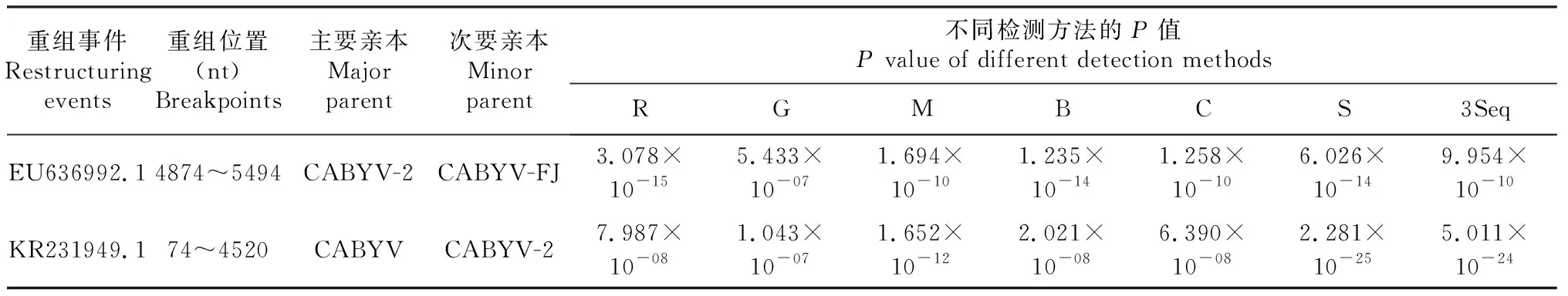
表5 2个CABYV重组事件
3 讨 论
CABYV是导致葫芦科作物发黄症状的重要病原体,在我国河北、河南、山西和陕西等地区的9种葫芦科作物上均检测到了CABYV病毒[11],随后该病毒在黑龙江、辽宁、新疆、北京、山东、浙江、安徽、湖南、云南、广西和海南等我国25个省市的葫芦科作物上被检测到[12]。在韩国,已经确认CABYV感染了黄瓜和东方甜瓜[13],此外,该病毒可与其它感染葫芦科的病毒一起造成共同感染,增加重组病毒出现的几率[14-16]。近年来,在我国的葫芦科作物上也检测到了该病毒,导致西瓜、黄瓜、南瓜等的产量和品质显著下降[17]。本试验的24个CABYV的不同分离株由于地理因素的差异分为2组,且两个组之间存在较大的遗传差异。对不同CABYV分离株的各个基因进行遗传多样性分析,结果表明,P1基因的核苷酸多样性值最大,为0.237,说明P1基因的变异性更大,P3基因的π值最小,说明其遗传稳定性高,具有高度的保守性,这可能与其功能有关[18]。中性检验结果大多数为负值,说明CABYVA种群处于扩张状态。
地理隔离可能是CABYV存在高度变异的重要原因。基于24个分离株的进化树与核苷酸一致性分析结果,由于地理因素的差异,不同CABYV分离株分为2个分支,分支1包括从中国、韩国和日本分离到的21个分离株,本研究中分离得到的CABYV-2与CY3(KR231942.1)聚在一起,属于分支1,分支2包括3个从欧洲不同国家得到的分离株,分支1和分支2的CABYV不同分离株亲缘关系较远,这可能是由于CABYV主要通过蚜虫传播,很难实现远距离传播[2]。尚巧霞等[17]将CABYV湖北和云南分离株与国内外不同分离株进行分析后得到CABYV分子变异与地理分布有关,与本研究结果一致。进一步对分支1和分支2两个种群进行种群差异检验,结果显示2个种群间的遗传分化指数Fst值为0.913,Kst*为0.131,Snn和Z*值分别为1.00和4.403,分支1与分支2间的遗传距离为2.024,说明分支1与分支2之间存在较大的遗传分化。该结果与系统发育分析结果共同说明可能是由于地理因素的不同和遗传距离的差异造成了CABYV的遗传多样性的不同。
重组加剧了CABYV的变异。对于单链RNA病毒来说,病毒间重组是一个主要的进化方式,会对病毒种群产生直接的影响,可以扩大寄主范围,适应新的环境,是引起病毒变异的重要因素[19]。据先前报道,重组CABYV株来自菲律宾、台湾[20]和泰国[21]等东南亚国家,本试验重组分析结果表明在24个分离株中有EU636992.1和KR231949.1两个重组事件(表5),分别来自中国和韩国,并且甜瓜分离物和南瓜分离物之间的CABYV的重组说明该病毒可能在进化的过程中侵染力越来越强,应该引起足够的重视。
4 结 论
CABYV在我国分布广泛且种群不断扩展,成为瓜类植物生产的限制因素。CABYV新疆阿克苏市分离株与韩国分离株KR231942.1亲缘关系最近,与来自欧洲国家的分离株亲缘关系最远,分支1和分支2的分离株之间存在较大的遗传差异,CABYV在不同宿主间的重组加剧了CABYV的变异,重组和负选择可能是影响CABYV遗传变异的重要原因。

