受损腺嘌呤与氨基酸残基堆积复合物的ABEEM极化力场
刘 翠, 王 洋, 郑明杰, 尚 尉, 郑 利
(辽宁师范大学 化学化工学院,辽宁 大连 116029)
细胞基因组常会因为水解和暴露于细胞内环境而导致正常碱基氧化和烷基化,发生突变,使DNA不能进行正常的复制和转录工作.损伤碱基数量积累过多将无法与生物体相容,除非它们被移除,否则会引发疾病,严重时会引发癌症[1].此种内源性损伤必须通过DNA修复途径中的碱基切除修复(BER)才能有效纠正,使基因组恢复正常.
碱基切除修复过程中涉及多种酶,其中用于特异性清除损伤碱基的酶称为DNA糖基化酶[2],它去除受损碱基的作用机理是切割核苷酸的糖苷键.在这一过程中,蛋白质与碱基间除了氢键作用外,堆积作用也至关重要.堆积作用是一种重要的非键作用[3],它主要发生在π-π共轭体系中,对于增强体系的稳定性具有重大意义.π-π堆积作用的研究非常广泛,包括了氨基酸与阳离子堆叠、糖-π堆叠、氨基酸-碳水化合物堆叠以及氨基酸-碱基的堆叠[4-6].例如Ebrahimi等人利用标准法和梯度平衡校正法优化尿嘧啶U与苯丙氨酸PHE、Na+和PHE之间的π相互作用;Gattani等人提出了一个平衡预测器——StackCBPred,基于从进化驱动的序列轮廓中提取的特征,预测蛋白质-碳水化合物结合位点.
碱基与芳香环氨基酸之间的π-π堆积作用近年来一直受到国内外研究学者的关注.例如:Baker等人[7]对氨基酸与碱基之间的相互作用进行研究,对比了蛋白质与核酸复合物的堆积作用的强弱,为修复酶识别碱基提供理论依据;Rutledge等人[8]通过扫描势能面,研究了腺嘌呤、损伤腺嘌呤(3-甲基腺嘌呤)与氨基酸残基之间的堆积作用,得出烷基化可增强修复酶活性部位的堆积相互作用,并给出了这种增强程度的度量.
目前为止,关于π-π堆积作用的研究包括化学实验法和理论计算方法两大类,而单纯的实验方法(如NMR和X射线晶体)虽然可以揭示DNA-蛋白质相互作用的结构,但是不能明确给出二体的相互作用强度信息.计算化学可以提供相关作用细节.Maria等人[9]在MP2/cc-pVDZ理论水平上,利用XMCQDPT2/sa2-CASSCF(12/12)方法对HBDI(4-羟基亚苄基-1,2-二甲基咪唑啉酮)与芳香族化合物之间的π-π堆积作用进行研究,并证明了变色位移与偶极矩的相关性.Stefan等人[10]利用ONIOM(QM:MM)方法,揭示了AAG(烷基腺嘌呤DNA糖基化酶)的活性位点结合不同的底物对脱糖基化的显著影响.
量子力学方法计算精准,但计算的体系大小有限.对于复杂的生物大分子体系,必须要考虑内部复杂的环境、极化效应、电荷转移效应、氢键作用及堆叠作用.常用力场,如AMBER[11]、OPLS[12]和AMOEBA[13]等,不能精准处理极化效应和电荷转移效应.因此发展一种快速、准确的极化力场,模拟修复酶与损伤碱基的堆积作用十分必要.
Yang等人将基于密度泛函理论(DFT)和电负性均衡原理建立的ABEEM电荷模型与分子力场相结合,建立ABEEM极化力场[14].此力场通过多位点模型,及位点电荷受环境和结构变化而变化,体现生物分子中的极化效应和电荷转移效应,而且计算速度快,适合于复杂生物大分子体系的模拟.
本工作通过对比从头算的基准结果,调节π位点电荷和堆叠作用函数,发展了适用于堆叠作用的腺嘌呤和氨基酸残基的ABEEM极化力场,在ABEEM极化力场中模拟4种受损腺嘌呤(次黄嘌呤(HYP)、1-N6-乙烯基腺嘌呤(εA)、和3,9-二甲基腺嘌呤(3,9MeA)、3-甲基腺嘌呤(3MeA))与4种芳香族氨基酸残基(组氨酸(HIS)、色氨酸(TRP)、苯丙氨酸(PHE)、酪氨酸(TYR))的堆积作用.获得有关DNA碱基与氨基酸残基之间堆积作用几何结构、电荷分布和作用强度的信息.由于这些相互作用在生命体系中扮演着无可替代的作用,因此该研究课题能为酶识别损伤腺嘌呤的机理提供理论指导.
1 模型分子
在研究氨基酸与碱基堆叠二聚体时,如果考虑蛋白质骨架或DNA骨架,会使得二聚体的相互作用强度发生变化,为了准确计算π-π堆积相互作用能,磷酸糖主链和氨基酸的蛋白质主链都用氢原子代替,各损伤腺嘌呤结构如图1.
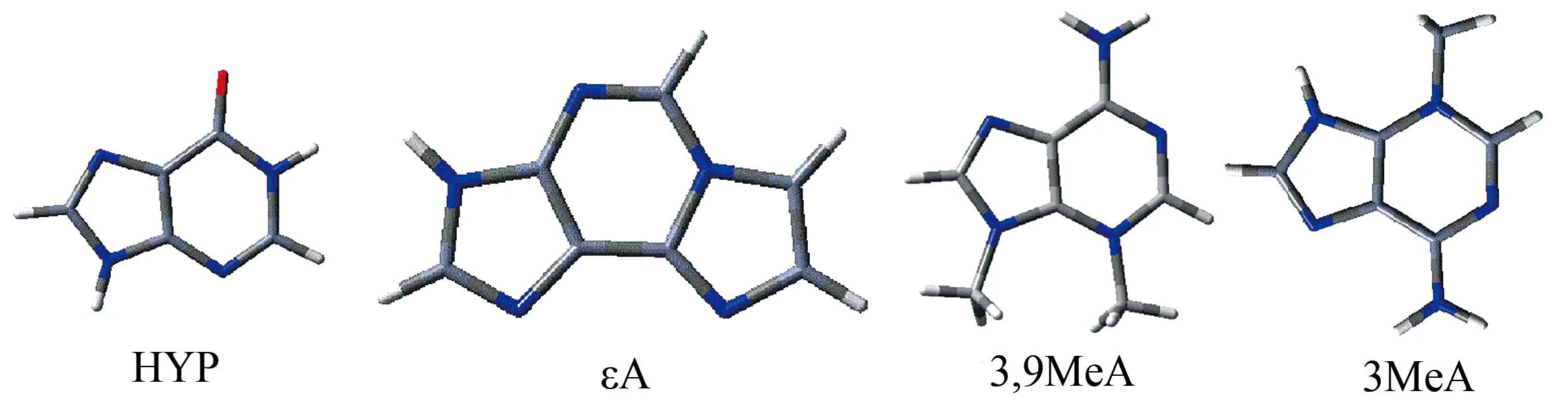
图1 4种损伤腺嘌呤模型图
氨基酸残基与受损腺嘌呤之间的π-π堆积作用会受到4种因素的影响,分别是垂直位移R1、旋转角α、水平位移R2和倾斜角θ[15],这4个变量如图2所示.以3,9MeA:PHE二聚体为例说明4个参数,其中,水平位移R2的方向按照图3虚线标明的角度进行移动.氨基酸残基结构如图3.选用可以形成二聚体稳定体系的4个变量值进行模型构建.结合文献中4个变量的值,4种损伤腺嘌呤与氨基酸残基的二聚体结构如图4所示.
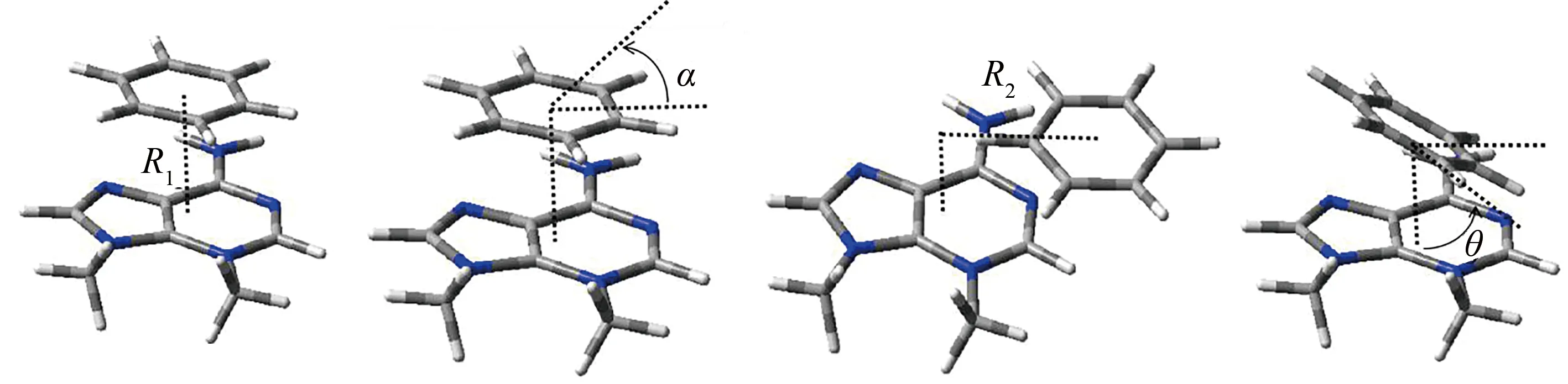
图2 垂直位移R1、旋转角α、水平位移R2和倾斜角θ变化示意图
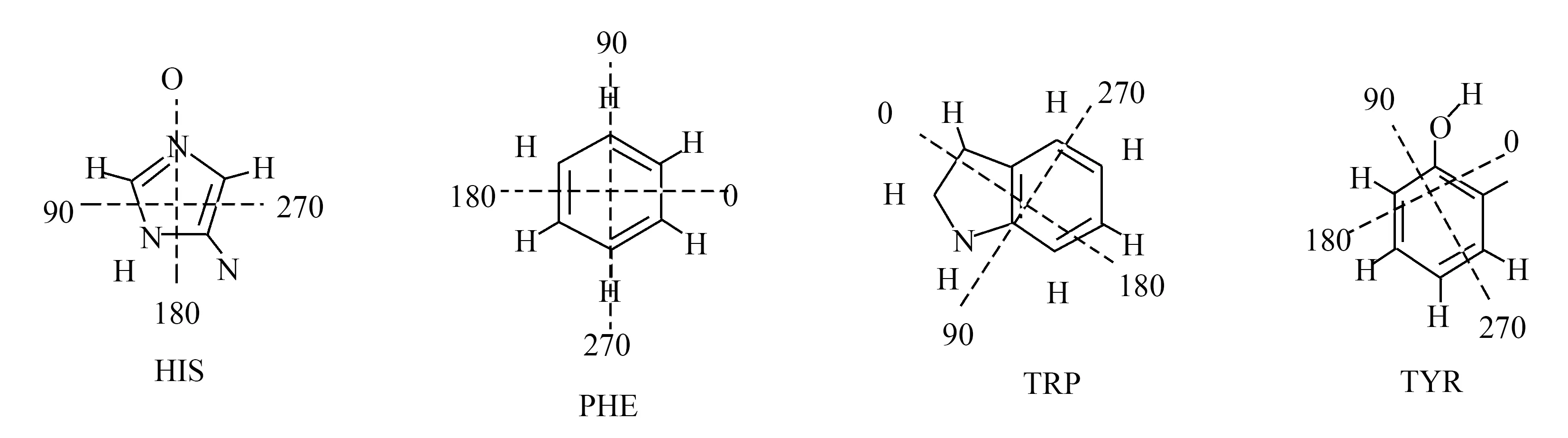
图3 水平位移R2示意图,虚线代表平移方向
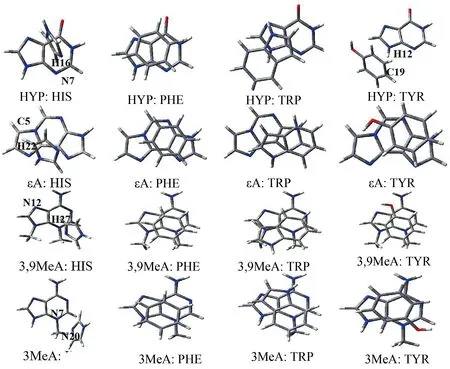
图4 二聚体结构示意图
2 计算细节与模型方法
2.1 计算细节
使用MP2/6-31G方法对受损腺嘌呤与氨基酸残基组成的二聚体进行结构优化,使用MP2/aug-cc-pVDZ方法进行能量计算,计算的公式为
ΔE=En+Ea-Edi.
(1)
其中,Edi表示氨基酸残基与受损腺嘌呤组成的二聚体的单点能,En表示受损腺嘌呤的单点能,Ea表示氨基酸残基的单点能,ΔE代表氨基酸残基与受损腺嘌呤之间的堆积能.
2.2 ABEEM极化力场模型
ABEEM极化力场除了设置原子位点,还定义了σ键、π键和孤对电子的位点,并且给出明确的电荷,这些位点的电荷随着结构和环境的变化而变化,反映了体系的电荷转移和由外部分子所引起的极化效应.
ABEEM极化力场与其他力场相比,最为突出的特色是静电相互作用,采取库仑相互作用形式,具体表达如式(2):
(2)
其中,q是原子位点、键位点及孤对电子位点的电荷,rij是任意两位点i和j间的距离,参数kij是校正因子,它充分考虑了电子与原子核之间的屏蔽效应及钻穿效应,具体表达为式(3).
(3)
其中,kHB是氢键拟合函数具体公式参见文献[14],kπH/ππ是堆叠拟合函数,具体公式为式(4),k′,C,D,rπH/ππ是参数,RπH/ππ是H原子和π位点的距离,或者两个分子中π位点之间的距离.
(4)
2.3 ABEEM极化力场参数调节
参数的调节是本工作的重点,也是最耗费时间的部分.模型分子包括4个损伤腺嘌呤、4个氨基酸残基,16个损伤腺嘌呤和氨基酸残基的堆叠二聚体.本工作主要调节相关π位点参数和堆叠拟合函数相关参数.π位点电荷参数通过线性回归和最小二乘法优化,拟合HF/STO-3G[14]水平的电荷.堆叠拟合函数通过拟合MP2/aug-cc-pVDZ方法的堆叠能确定,相关参数列于表1.
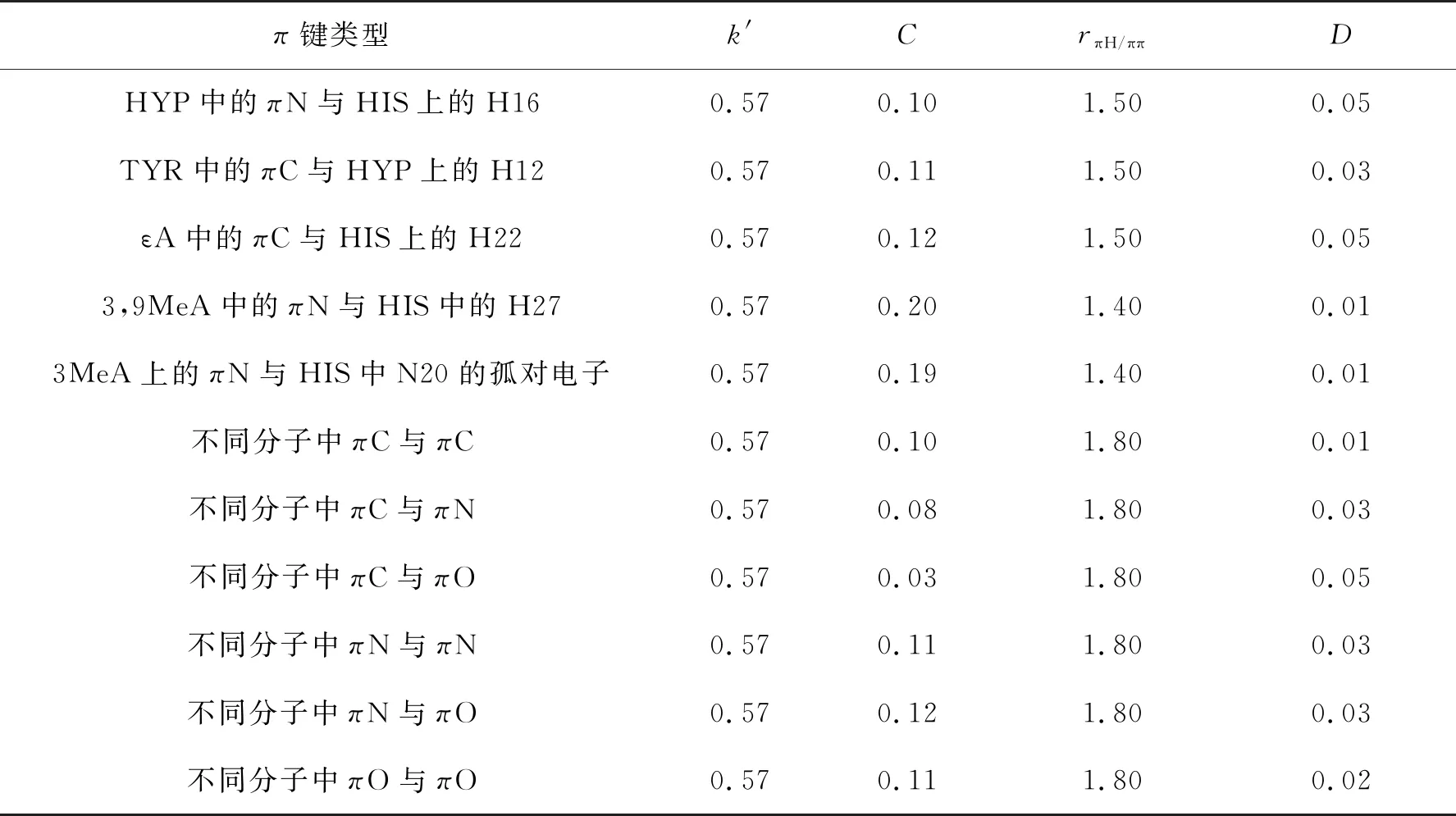
表1 堆叠拟合函数相关参数
3 结果与讨论
3.1 几何结构
用MP2/6-31G方法和ABEEM极化力场方法将几何结构进行优化,得到的稳定结构的参数平均绝对偏差如下:HYP与氨基酸体系的R1、R2的偏差分别为0 , 0.002 5 nm,α、θ的偏差分别为0.25,0°;εA与氨基酸体系的R1、R2的偏差分别为0,0.01 nm,α、θ的偏差分别为2.25,0.5°;3,9MeA与氨基酸体系的R1、R2的偏差分别为0,0.025 nm,α、θ的偏差分别为1.25 , 0.5°;3MeA与氨基酸体系的R1、R2的偏差分别为0,0.005 nm,α、θ的偏差分别为0.5 , 0.5°.上述结果表明ABEEM极化力场可以很好地模拟生物体系的堆积作用.
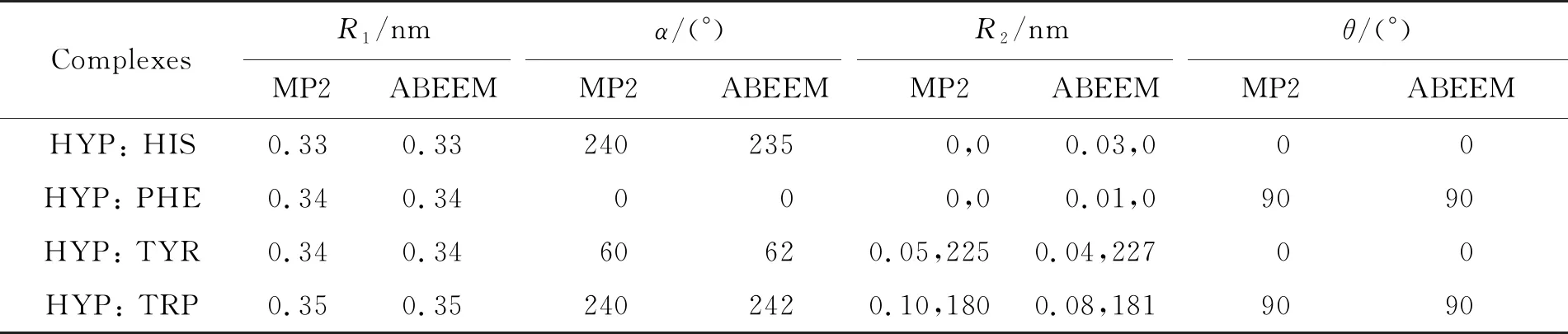
表2 HYP与氨基酸体系的4个最优参数
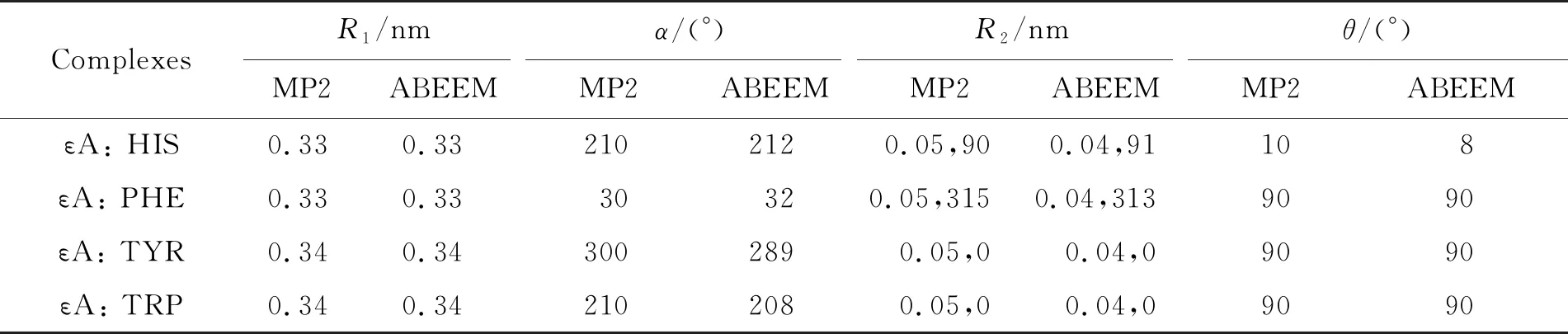
表3 εA与氨基酸体系的4个最优参数
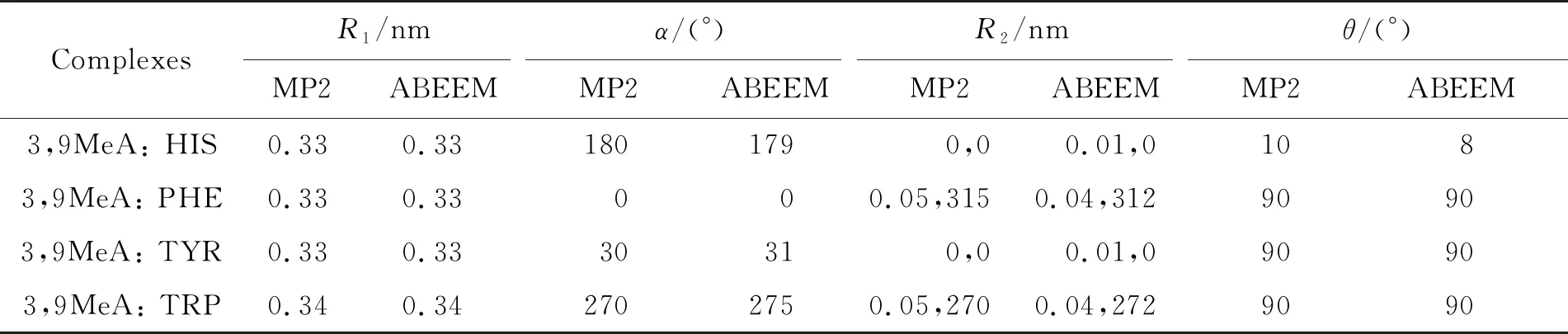
表4 3,9MeA与氨基酸体系的4个最优参数
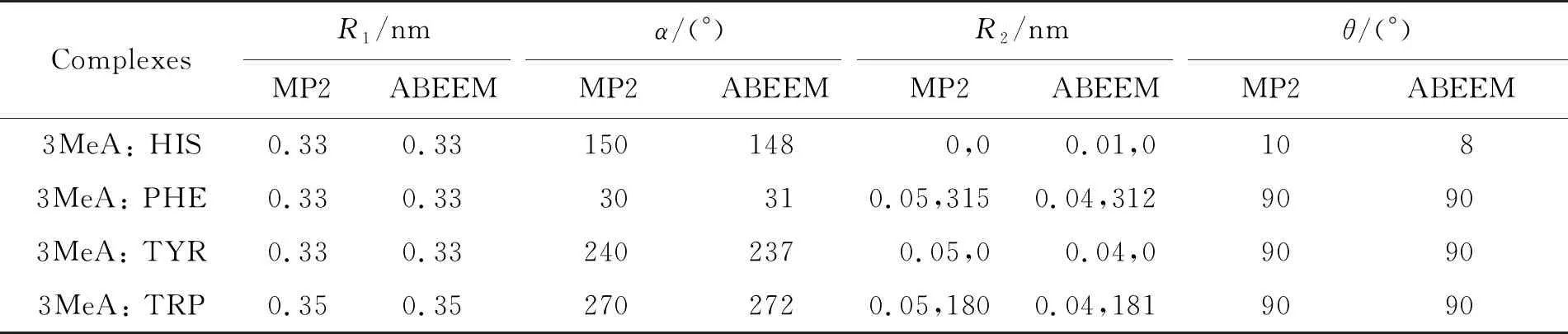
表5 3MeA与氨基酸体系的4个最优参数
3.2 电荷分布
比对HF/STO-3G方法下的电荷分布与ABEEM极化力场下的电荷分布发现,两者的线性相关系数都在0.97以上,说明ABEEM极化力场可以快速、准确地计算大分子体系的电荷分布.
偶极矩是判断电荷分布是否准确的物理量,将中性二聚体——HYP和εA分别与4种氨基酸形成的二聚体的偶极矩,在MP2/aug-cc-pVDZ和ABEEM极化力场下进行对比,二者的平均绝对偏差为0.39×10-30C·m,线性相关系数是0.98,说明ABEEM极化力场计算的电荷分布是合理的.只是斜率为0.747 3,说明ABEEM极化力场的偶极矩比MP2/aug-cc-pVDZ方法的偶极矩稍小.
3.3 堆叠能
用MP2/aug-cc-pVDZ和ABEEM极化力场计算出各损伤腺嘌呤、氨基酸单体与16种二聚体的单点能,可以发现:相同氨基酸下,各损伤碱基的堆叠能大小为3,9MeA>3MeA>εA>HYP,烷基化碱基的堆叠能要比中性受损碱基的堆叠能大,且随着烷基的数量增多而增大,3,9MeA的堆叠能甚至是HYP的2倍.对比MP2/aug-cc-pVDZ方法和ABEEM极化力场的堆积能发现,能量平均绝对偏差为1.06 kJ/mol,二者的线性相关系数是0.99,非常接近于1,且斜率为1.019 8.可以看出,无论是偏差的数值还是线性相关系数均很好地说明了ABEEM极化力场能够准确地模拟堆积相互作用.
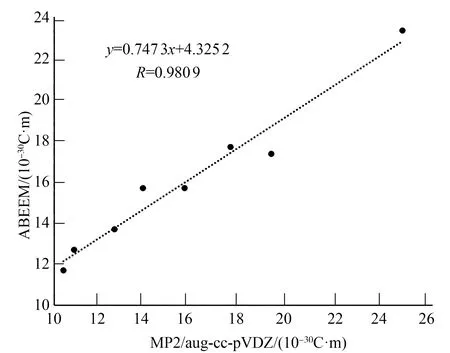
图5 偶极矩线性相关图
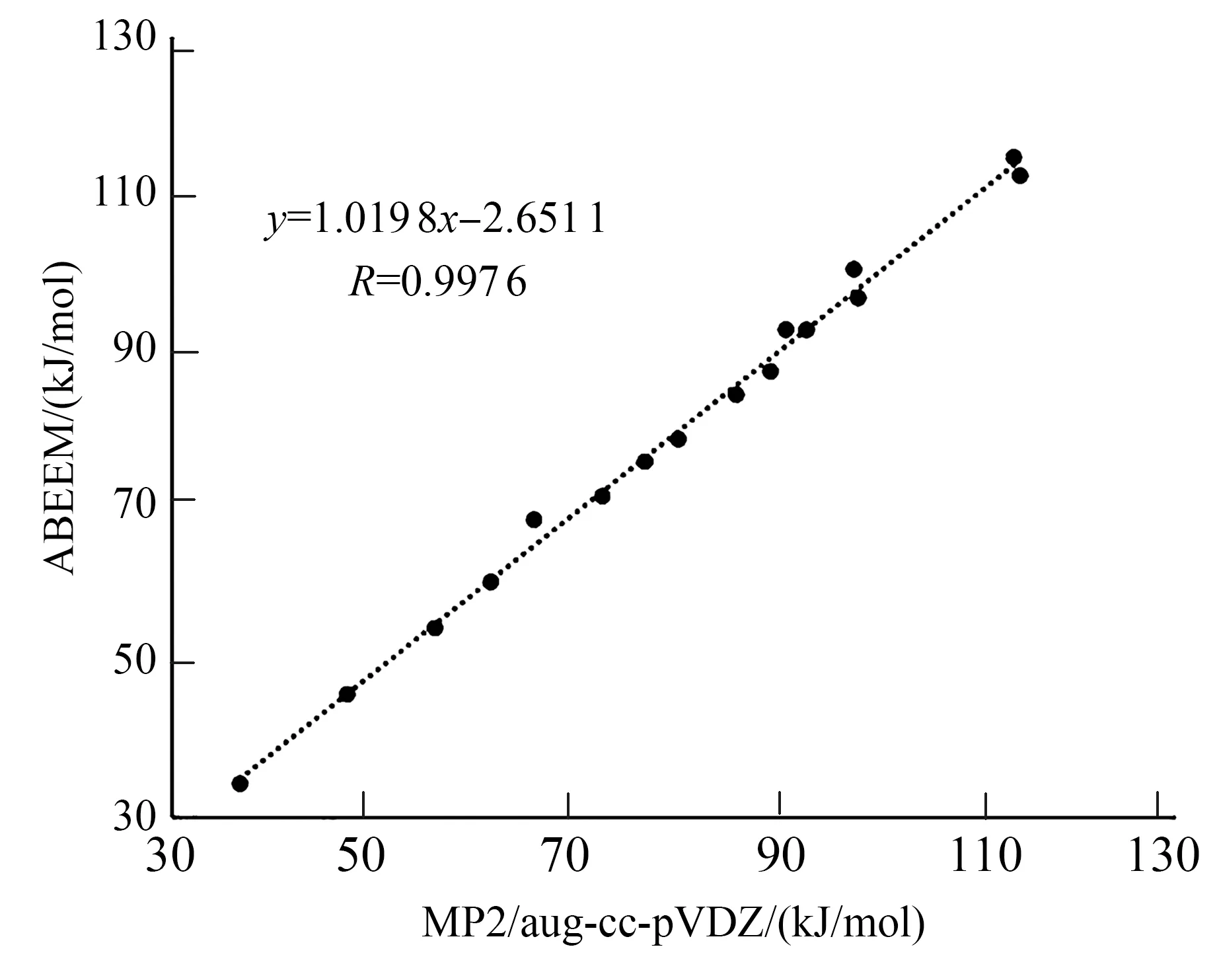
图6 堆叠能线性相关图
4 结 论
本文使用MP2/aug-cc-pVDZ//MP2/6-31G方法对HYP、εA、3,9MeA和3MeA分别与4种氨基酸残基所形成的二聚体体系进行结构优化和能量的计算,将结果作为调节ABEEM极化力场π键位点参数的依据,本文发展的ABEEM极化力场的最突出贡献就是添加了π键位点和堆叠作用函数,使模拟的堆积作用二聚体体系更加准确.将ABEEM极化力场计算得出的结果与量子力学结果进行对比,其电荷分布、偶极矩以及堆积能的线性相关系数分别是0.97、0.98和0.99,都非常趋近于1,有很好的拟合相关性,说明本工作发展的ABEEM极化力场可以精准地模拟氨基酸残基与受损腺嘌呤间的堆积相互作用.并且用于这16种堆积二聚体体系的参数具有可转移性,结合其考虑了分子间的静电相互作用的优势,ABEEM极化力场能够很好地应用于DNA与蛋白质生物大分子体系为修复机理的研究提供理论基础.

