基于R-shiny的临床数据分析应用
崔怡丹 魏 婷 孔伟名 俞章盛,△
【提 要】 目的 针对临床数据变量类型多、数据结构复杂的特征,基于R-shiny开发交互式分析临床数据并展示结果的应用。方法 基于R-shiny与R语言,搭建应用的UI(用户界面)和Server(后台功能),将相应代码封装并部署在网页上。以UK Biobank乳腺癌数据为例,对其临床及基因数据进行分析,查看R-shiny应用的分析效果。结果 构建出能够交互式分析临床数据的R-shiny应用,可供用户上传临床数据、生存型数据及基因数据进行分析。将UK Biobank数据作为示例进行分析,得到了具有临床与生物学意义的结果。结论 将R-shiny与临床数据分析结合,为使用者提供了分析临床数据的简便方式,并为UK Biobank数据提供新的交互式展示分析平台。
R语言是免费开源的编程语言[1],适用于各种计算机系统,具有强大的统计分析和图形绘制功能。R-shiny是RStudio公司基于R语言推出的Web开发工具[2],借助R语言强大的数据分析与图形绘制功能,帮助熟悉R语言的编程者开发以浏览器为依托、具有交互功能的应用。使用者无需学习R语言,仅通过鼠标点击的方式就可以轻松使用shiny应用。常见的shiny应用包含UI(用户界面)和Server(后台功能)两部分,其中UI的功能对应B/S架构中的B(Browser浏览器)端[3],Server对应S(Server应用)端。UI包括sidebarPanel(侧边栏,显示选项)与mainPanel(主页面,展示结果),负责展示用户界面布局并输出后台运算结果;Server负责抓取UI动态并在后台进行相应运算。通过UI与Server配合,能够实时对用户在UI页面的指令进行反馈。本应用使用shiny的navbarPage框架作为UI的架构基础,运用HTML与Javascript对其外观与功能进行完善。临床数据分析使用R语言实现后台运算,基因分析通过R调用plink[4]进行分析并将结果展示在shiny应用中。
UK Biobank是一项由英国国家卫生局支持的前瞻性开放获取队列研究[5]。该项目招募了英国各地超过500000位参与者,采集参与者的表型与遗传信息,包括生理指标、生活方式、生物学测量、血液尿液、身体及大脑图像、全基因组测序等数据[6]。基于UK Biobank数据的研究已较为广泛,包括对肾病[7]、甲状腺疾病[8]等的临床与基因数据研究。世界卫生组织发布的《2020世界癌症报告》指出[9],乳腺癌是全球女性患病率最高的癌症,同时也是全球女性癌症死亡的主要原因。寻找乳腺癌易感及影响乳腺癌患者生存时长的临床与基因因素是亟待解决的问题。因此,本研究选用UK Biobank乳腺癌数据作为shiny应用示例,展示本研究开发的临床数据分析应用,同时探究影响乳腺癌患病及预后的风险因素。
针对临床与基因数据特点,以R语言与shiny为基础开发临床数据分析应用,可交互式分析数据并展示结果,为不熟悉R语言与plink的使用者提供易上手的数据分析应用,为UK Biobank数据提供展示平台,为临床数据分析应用的开发提供新思路。
关键问题及解决方案
本应用包含四个功能:数据上传与描述性统计、回归分析、生存分析与基因分析。针对以下五个关键问题,我们探索出了相应解决方案。
1.如何架构UI能够兼顾功能多样与界面简洁?
临床与基因数据涉及多个分析方向,若显示在同一页面,难免会纷杂不清晰。比较多种shiny架构后,我们在navbarPage架构的基础上,用navbarMenu与tabPanel进行扩充。navbarPage是shiny中较为常用的架构之一,点击页面顶部横条的不同名称显示不同页面。navbarMenu是navbarPage架构中的可选功能,点击顶部横条的名称显示下拉式菜单,点击菜单中的名称显示不同页面。tabPanel是shiny中常用的基础架构,点击shiny应用主页面上方的不同标签名称显示不同页面。子功能较多且相互独立的模块通过navbarMenu展示,子功能具有一定关联性的模块通过tabPanel展示。同时,使用HTML与JavaScript语句对UI进行完善,例如通过HTML调整字体大小及添加使用说明,通过JavaScript控制输出图片的显示与隐藏等功能。
2.如何处理临床数据的变量类型?
R语言读取csv、Excel、txt格式文件时会将变量读取为连续型,或通过stringsAsFactors选项将所有变量读取为分类型,这显然不符合临床数据特征。针对这一问题,首先将数据读取为连续型,利用reactive函数与observe函数,在选择框中显示数据集变量名,使用者通过点击选择可将相应变量修改为分类型。
3.回归模型类型如何判断?
根据结局变量的不同类型,常见的回归模型包括线性回归模型(结局变量为连续型)和logistic回归模型(结局变量为分类型)。为简化操作,直接根据使用者选择的结局变量类型判断回归模型类型。若结局变量为连续型,glm函数的family参数设置为Gaussian;若结局变量为分类型,family参数设置为Binomial。
4.连续型变量如何进行log rank检验?
生存分析模块的log rank检验可以比较不同亚组生存分布。分类型变量可以直接根据变量水平区分亚组,但对于连续型变量,需要先规定其分组依据。为方便使用者,我们选用滑动条作为依托:当检验变量为连续型时,应用会显示一条阈值与该变量取值范围相等的滑动条,使用者拖动滑块,以滑块的取值对该连续型变量进行分组,并对两组的生存分布进行log rank检验。
5.数据量较大的基因数据如何分析?
一些临床研究包含基因数据,但基因数据量较大,如果在shiny中直接运算,上传数据会占用较长时间。因此,考虑使用shiny调用plink软件在本地运算,在shiny中展示本地储存的运算结果与日志。首先在后台通过bigsnpr::download_plink()语句在本地目标路径中安装plink软件,后台通过system(“plink-xxxx”)等语句调用plink进行文件格式转换、主成分分析、GWAS等运算操作。
shiny应用界面与功能介绍
本应用包含五个模块:应用介绍、数据上传及描述、回归分析、生存分析与基因分析,涵盖了临床研究数据分析的主要步骤。
“数据上传及描述”模块使用tabPanel显示可选功能,通过点击主页面上方的标签选择数据上传、图像描述及描述性统计。在数据上传页面可以选择内置的UK Biobank数据,也可上传格式为csv、Excel、txt的文件并设置变量类型。可通过三种方式对数据进行简要描述:前十行预览、数据结构、汇总描述(分别对应R语言head、str、summary函数)。还可以绘制变量的描述性图像:连续型变量绘制条形图,分类型变量绘制饼图。描述性统计页面展示tableone包输出的描述性表格,可以描述总体数据,也可分亚组进行对比描述。数据上传及描述页面见图1。
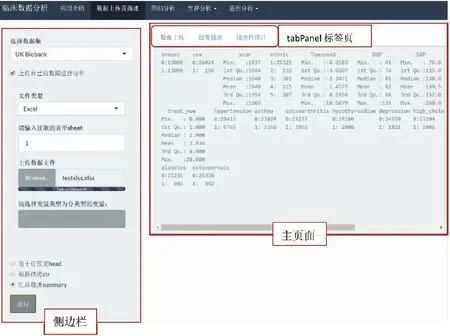
图1 shiny应用的数据上传及描述页面
“回归分析”模块根据选择的结局变量类型拟合线性回归或logistic回归模型,可以选择数据集中的任意变量作为模型自变量。若模型自变量较多,可通过添加惩罚项进行变量选择,可选的惩罚方式包括SCAD、MCP和Lasso三种。回归分析模块页面展示见图2。
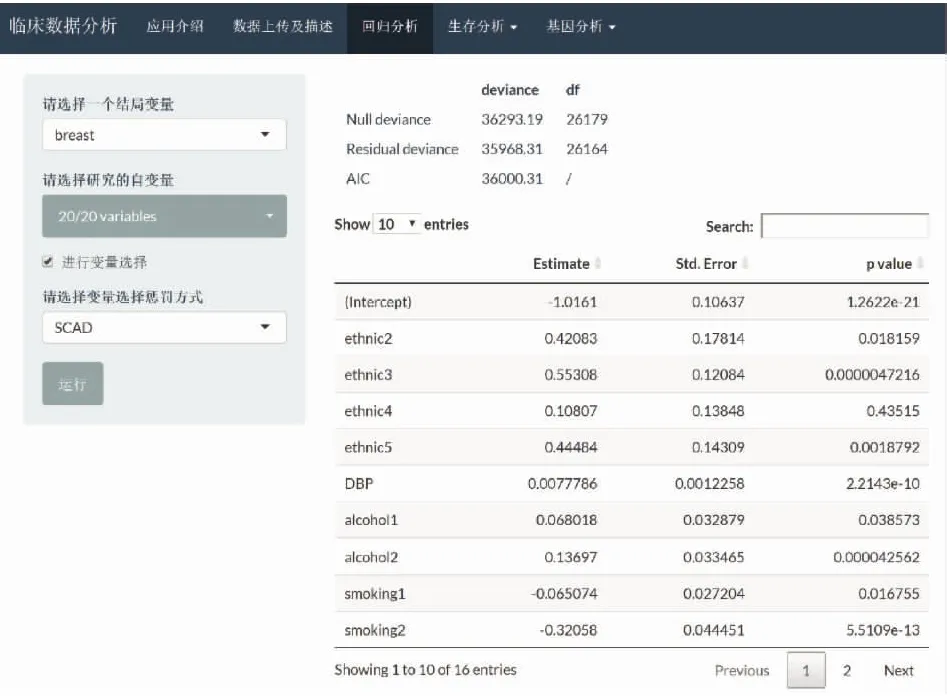
图2 shiny应用的回归分析页面
“生存分析”模块包括Kaplan-Meier图像、生存模型、log rank检验三部分。Kaplan-Meier图像通过ggplot2包绘制,需要依次选择生存时间与状态。生存模型包括Cox比例风险模型与可加风险模型两种,都可进行模型拟合与变量选择,并在主页面显示模型的参数估计与统计量。图3展示了navbarMenu与Cox比例风险模型页面。log rank检验可根据使用者选择的变量进行分组并比较亚组间的生存分布,连续型变量会根据滑动条的取值进行分组。图4展示了log rank检验变量为连续型时的页面。
“基因分析”模块相对独立于其他三个模块,主要功能包括数据类型转换、主成分分析、单因素及多因素GWAS分析三个功能。数据转化是将常见的ped/map或vcf格式文件转化为运算速度更快的二进制bed文件。主成分分析的功能是通过plink计算基因数据的十个主成分,读取计算结果绘制主成分图并展示在主页面。单因素GWAS分析直接计算表型与基因数据关联程度的P值;多因素GWAS分析计算有临床数据影响时表型与基因数据关联程度的P值。若基因数据较多,可勾选“计算矫正相关系数”以避免多重检验误差。图5展示基因GWAS分析页面。
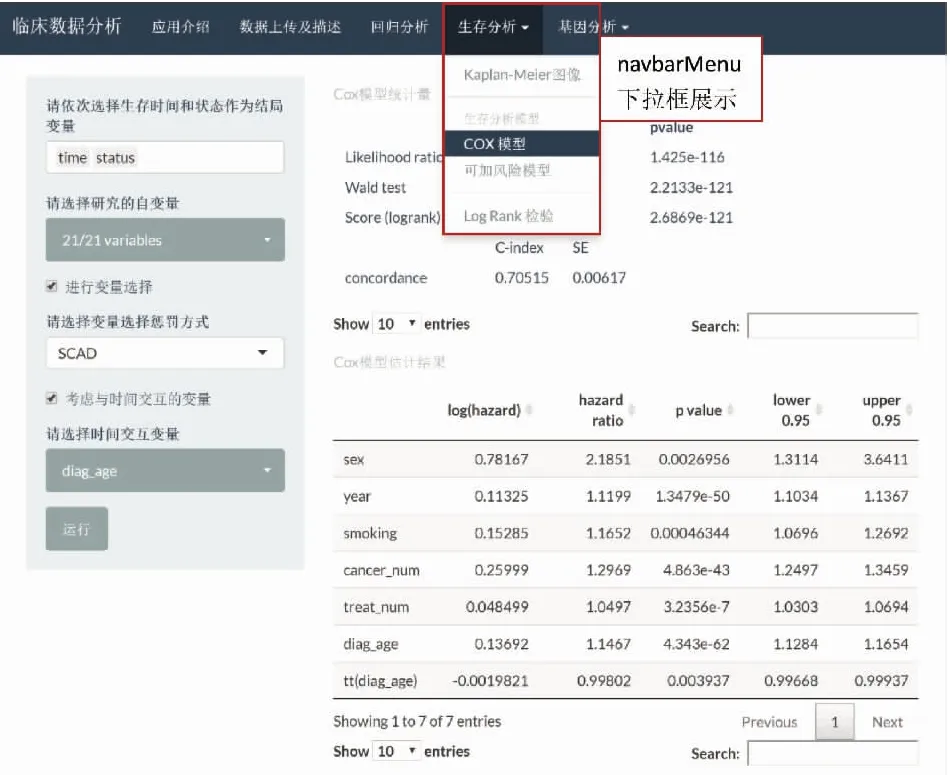
图3 shiny应用的生存分析页面

图4 shiny应用的log rank检验页面

图5 shiny应用的GWAS分析页面
部分应用示例结果展示
1.生存分析:Cox比例风险模型
点击页面上方“生存分析”按钮进入Cox比例风险模型页面。选择生存时间和状态后,将剩余变量选为模型自变量,勾选“变量选择”选项,选择SCAD惩罚,得到分析拟合结果见表1。性别、吸烟、确诊年龄等对乳腺癌患者生存的影响与临床研究一致[10-12]。

表1 生存分析Cox比例风险模型分析结果
2.基因分析
(1)主成分分析
点击基因分析按钮进入主成分分析页面,粘贴基因数据所在文件路径,计算主成分。之后根据计算结果绘制第一和第二主成分图,如图6,不同颜色代表不同人种。可以看出相同人种之间存在一定相关性,不同人种之间差异较显著。

图6 第一主成分与第二主成分示意图
(2)单因素GWAS分析
点击进入GWAS分析,选择需要分析的文件名并勾选“计算矫正相关系数”选项,选取Bonferroni矫正。得到39个显著位点,其中23个有研究支持,包括保护性和危害性SNP。例如rs3803662,一项关于乳腺癌患者的队列研究表明其突变与乳腺癌易感风险增高显著相关(OR=1.15)[13],这与我们的GWAS分析结果一致(OR=1.20)。具体结果见表2,其中A1为次等位基因,A2为参考基因。
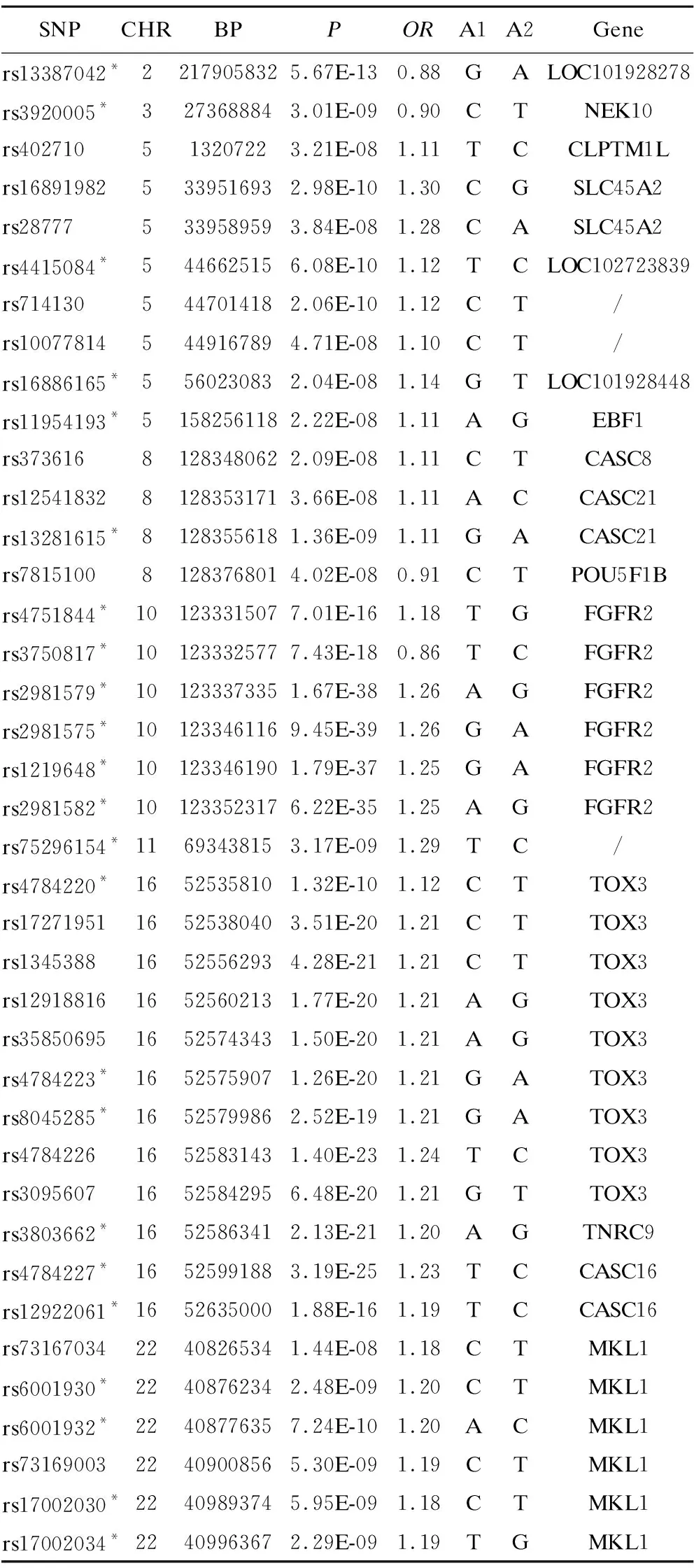
表2 GWAS分析影响罹患乳腺癌的SNP位点
讨 论
将临床数据分析与R-shiny结合,通过鼠标点击的方式选择感兴趣的分析方法及模型,并交互式地展示相应分析结果,方便不熟悉R语言的使用者轻松使用R语言的分析统计、图形绘制功能。本研究开发的应用能够完成常见的分析方法,如描述性统计、回归分析、生存分析、基因分析等;可以绘制常用的图形,如饼图、直方图、Kaplan-Meier图像等。在后续的研究中会对本应用继续改进,完善统计绘图功能,增强应用页面使用的友好性、便捷性、交互性。期望本应用能够协助不熟悉R语言的医疗工作人员进行数据分析,为临床数据分析相关应用的开发提供新思路,同时为UK Biobank数据提供新的可视化分析统计平台。

