纤枝短月藓BeLEA2基因的克隆及表达分析
李雪宝 王琦 鄢波



摘 要: 为探究纤枝短月藓LEA2基因的结构和表达特征,该研究以纤枝短月藓为材料,首次利用PCR克隆技术得到纤枝短月藓BeLEA2基因序列,并对该基因进行分析。结果表明:(1)该基因序列中含有2个外显子和1个内含子,其开放阅读框(ORF)为 456 bp,編码151个氨基酸,预测其相对分子质量为16 515.96 Da。(2)将纤枝短月藓与其他植物LEA2基因氨基酸序列进行比对,构建系统进化树,结果显示纤枝短月藓与小立碗藓的亲缘关系最近。(3)利用HiTail-PCR技术克隆获得1 072 bp的BeLEA2启动子序列,用PlantCARE在线工具对该启动子的顺式作用元件进行预测,结果表明该启动子除了含有核心启动子元件TATA-box和CAAT-box外,还含有ABRE、MYB、MYC、MYB结合位点(MBS)等其他顺式元件。(4)实时荧光定量PCR分析表明,BeLEA2基因在纤枝短月藓不同发育时期和不同组织中都有表达,且对脱水胁迫有响应。以上结果为进一步探究LEA2基因在苔藓植物中的功能及作用机制奠定了基础。
关键词: 纤枝短月藓, LEA2基因, 启动子, 脱水胁迫, 表达
中图分类号: Q943
文献标识码: A
文章编号: 1000-3142(2022)02-0277-09
Cloning and expression analysis of BeLEA2 gene from Brachymenium exile
LI Xuebao1,3, WANG Qi2, YAN Bo1,3*
( 1. College of Landscape Architecture and Horticulture Sciences, Southwest Forestry University, Kunming, 650224; 2. College of Life Sciences, Sichuan University, Chengdu 610065;
3. Southwest Research Center for Landscape Architecture
Engineering, State Forestry and
Grassland Administration, Kunming 650224 )
Abstract: The purpose of this study was to explore the structural and expression characteristics of LEA2 genes from Brachymenium exile. BeLEA2 gene was firstly isolated and analyzed by polymerase chain reaction (PCR). The results were as follows: (1) Gene structure analysis showed that BeLEA2 gene contained 2 exons and 1 intron and contained an open reading frame (ORF) of 456 bp encoding a protein of 151 amino acids, and its molecular mass was predicted to be 16 515.96 Da. (2) The phylogenetic analysis of LEA2 with other LEA2 in different plants revealed that BeLEA2 from B. exile and LEA2 from Physcomitrella patens belonged to the same branch of evolutionary distance. (3) The promoter sequence of the BeLEA2 gene of 1 072 bp was isolated from Brachymenium exile by high-efficiency hermal asymmetric interlaced polymerase chain reaction (HiTail-PCR) and analyzed by PlantCARE, the results showed that it had TATA-box, CAAT-box, ABRE, MYB, MYC, MYB binding site and other cis-acting elements. (4) Quantitative real-time PCR analysis indicated that BeLEA2 expressed in different stages and tissues of B. exile, and BeLEA2 responded to dehydration stress. These results lay a foundation for further study on the function of LEA2 gene in bryophytes.
Key words: Brachymenium exile, LEA2 gene, promoter, dehydration stress, expression
水分是植物体生长所必需的重要组成部分,缺水会干扰植物正常的生理活动,而干旱、高温、盐碱等逆境都会使植物体细胞大量失水(潘瑞炽, 2012)。同时我国也是世界上盐碱地面积最多的国家之一,干旱和荒漠化问题非常严重(张建锋等, 2002)。到2014年底,我国土地荒漠化面积约为26 115.93万hm2,约占我国国土面积的27.20%(屠志方等, 2016)。盐碱和干旱不仅减缓了我国林业的高速发展,同时也会给我国的生态环境造成很大的破坏。因此,进行耐盐/耐干旱的研究非常重要。
纤枝短月藓(Brachymenium exile)属于真藓科短月藓属(黎兴江, 2006),生长在裸露的岩壁和极端干旱的环境中,具有较强的耐干旱能力。苔藓植物中存在抗氧化防御系统,可以有效地应对外界的干旱胁迫,在面对一些极端的外界环境时可以表现出极强的抗逆性(张萍等, 2005)。通过研究植物的抗逆型基因,探究植物的耐盐/耐旱机理,培养耐盐/耐旱作物或林木对保持我国农业生态的可持续发展,改善我国生态环境具有极大的作用。
LEA蛋白(late embryogenisis abundant proteins),即胚胎发育后期丰富蛋白,是植物胚胎发育后期种子中大量积累的一类蛋白,最早发现于棉花胚胎发育后期的子叶中(Dure et al., 1981)。随后,在拟南芥(Arabidopsis thaliana)、小麦(Triticum aestivum)、番茄(Solanum lycopersicum)、水稻(Oryza sativa)和油菜(Brassica napus)等植物中也同样发现了LEA蛋白的存在(Wang et al., 2006;Hundertmark & Hincha, 2008;Cao & Li, 2015;Yu et al., 2016;Bhattacharya et al., 2019)。尽管LEA蛋白在植物界中具有较为广泛的分布,但它并不是植物界所特有的一类蛋白,在真菌、细菌和某些无脊椎动物中也同样存在LEA蛋白(Tunnacliffe et al., 2005; Shinozaki & Yamaguchi-Shinozaki, 2006; Hand et al., 2007)。
到目前为止,已从多种不同植物中分离出LEA基因的启动子序列,分析结果表明LEA基因的启动子中具有不止一种压力响应相关的顺式作用元件,表明LEA 基因与干旱等非生物胁迫的调控密切相关(Huang et al., 2016; I·brahime et al., 2019; Nagaraju et al., 2019)。通过分析LEA基因结构得出,其内含子数目的差异较小,同时内含子数目通常不多于3个,基因结构的保守性较高(Huang et al., 2016; Wu et al., 2018)。
本研究对纤枝短月藓BeLEA2启动子基因进行了分离,将纤枝短月藓与其他植物LEA2基因氨基酸序列加以比对,构建系统进化树;对纤枝短月藓进行脱水胁迫处理,并对LEA2基因进行实时荧光定量分析,以期为 LEA2基因的表达特性及其功能研究奠定理论基础。
1 材料与方法
1.1 植物材料
研究所需纤枝短月藓采于昆明市郊区,保存于西南林业大学实验室内。清洗后进行DNA的提取,用于BeLEA2基因和启动子的克隆。
分别选取两组长势相同且良好的纤枝短月藓,分别选取有性世代和无性世代的配子体部分和孢子体部分。一组采用新鲜植株不予以处理,另一组洗净后,给予脱水胁迫2 h处理,进行RNA的提取,逆转录为cDNA,用于表达分析。
1.2 纤枝短月藓BeLEA2基因的克隆
采用 TIANGEN公司的小量植物(叶)总DNA抽提试剂盒来提取纤枝短月藓的基因组DNA,采用OMEGA公司的总RNA提取试剂盒提取纤枝短月藓的总RNA,选用逆转录试剂盒(全式金),将上一步所得的RNA逆转录为cDNA。
根据本实验室的转录组测序结果(未公布),设计出该基因特异性引物LEA2-F:ATGGCGGGGTTGT
TGAACAAAG;LEA2-R:TTAGAAGATGTCGGACAGT
GTG。分别以cDNA和DNA为模板,进行PCR扩增。反应体系为2×Taq PCR Master Mix 20 μL:正向引物 1 μL、反向引物1 μL、ddH2O 16 μL、DNA模板2 μL。PCR扩增程序:预变性,95 ℃,3 min;变性,95 ℃,15 s,退火,60 ℃,30 s,延伸,72 ℃,90 s,35个循环;延伸,72 ℃, 10 min;4 ℃保存。PCR产物回收后,将其连接到克隆载体PMD18-T中,送生工生物工程股份有限公司进行测序。
1.3 纤枝短月藓BeLEA2基因启动子的克隆
将纤枝短月藓的DNA作为模板,依照BeLEA2基因DNA全长序列设计出三轮的特异性引物:LEA2-1R:CGAAGACGGTAGGTGATCTCG;LEA2-2R:ACGAT
GGACTCCAGTCCGGCCGTTGTGGATCATGACGTTACT
C;LEA2-3R:GGTGACGTTCCCGATGTCCAC。参照Liu & Chen(2007)的办法,使用HiTail-PCR方法进行启动子的扩增分离。
1.4 生物信息学分析
利用ExPASy-ProtParam分析氨基酸的理化性质;使用在线软件GSDS 2.0分析基因的结构信息;利用NetPhos 3.1 Server软件预测磷酸化位点;利用NetOGlyc 1.1 Server软件预测O-糖基化位点;利用NetNGlyc 1.0 Server 预测N-糖基化位点(徐志文等,2019);利用Prot Scale软件得到蛋白质亲疏水性图;利用SOPMA软件进行二级结构预测分析;利用Swiss-Model通过同源建模建立三级模型;利用DNAMAN 6.0软件获得多序列结构域比对图;利用MEGA X软件构建系统进化树;利用PlantCARE软件分析基因的启动子区域中所包括的转录调控元件。
1.5 实时荧光定量PCR分析
根据所获得的BeLEA2基因的序列,设计荧光定量专用引物,L2-F:GCGACAGGGAGATTACCTCC、L2-R:GTCGTAGTCGATATCCCAGTC。內参基因为Actin基因,设计引物A-F:CTGTACGGCAACATCGTGCTG,A-R:CCAGACACTGTACTTCCTCTC,以cDNA为模板,按照TB Green Premix Ex TaqTM Ⅱ说明书进行操作,反应体系为SYBR Premix Ex Taq 10 μL:ROX Reference Dye 0.4 μL、正向引物 0.8 μL、反向引物0.8 μL、ddH2O 6 μL、cDNA模板2 μL。反应程序:预变性,95 ℃,30 s;变性,95 ℃,5 s,退火,60 ℃,30 s,40个循环。每个样品设置3次重复,使用2-△△Ct 法计算BeLEA2基因的相对变量值。
2 结果与分析
2.1 BeLEA2基因的克隆与结构分析
以纤枝短月藓cDNA为模板,结合引物LEA2-F和LEA2-R进行PCR扩增,经测序得到456 bp的序列。以DNA为模板,结合引物LEA2-F和LEA2-R进行PCR扩增,经测序得到1 168 bp的序列,得到两条序列的长度不一致,表明序列中有内含子。
根据转录组序列分析表明,该基因包含82 bp 5’UTR和266 bp 3’UTR,其开放阅读框长度为456 bp,编码151个氨基酸(图1)。其基因组序列全长长度为1 168 bp,同时含有1个内含子和2个外显子(图2)。经Pfam分析得出,LEA2蛋白在45~140氨基酸位点含有LEA2结构域,表明该基因属于LEA2家族,将该基因命名为BeLEA2。
2.2 BeLEA2基因的生物信息学分析
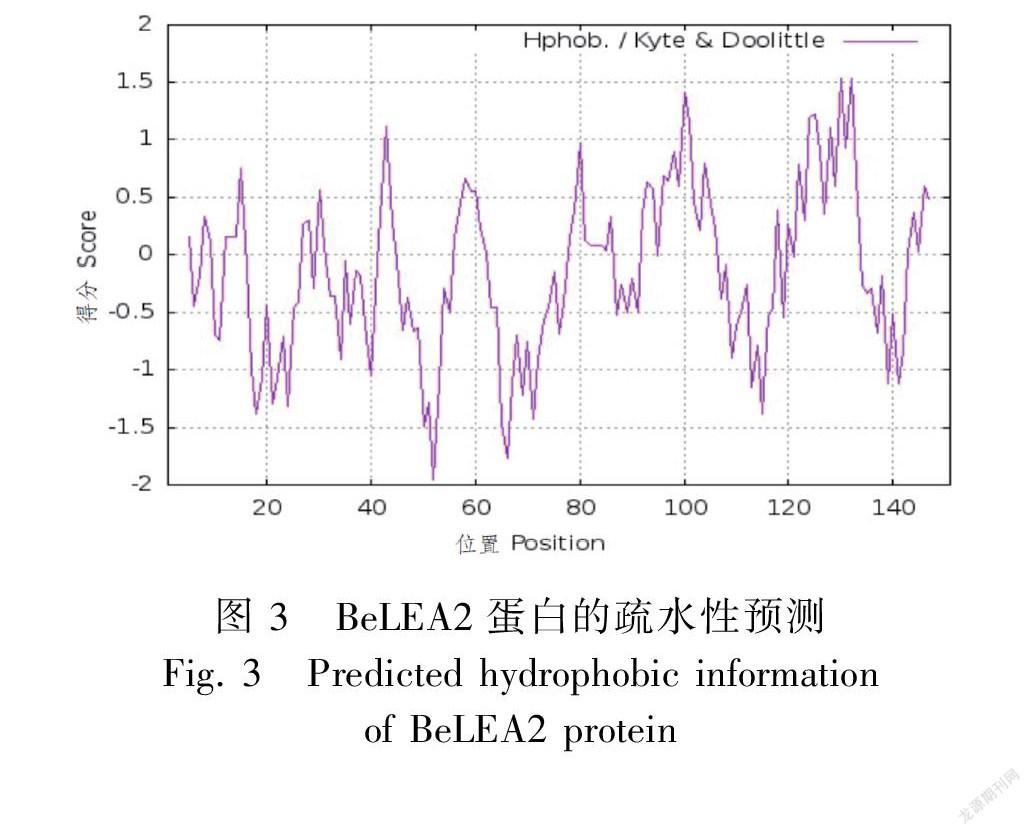
使用ProtParam软件预测BeLEA2编码蛋白质的相对分子质量为16 515.96 Da,理论等电点为5.74,分子式为C732H1179N197O224S6。BeLEA2所编码蛋白的氨基酸组成中,亮氨酸含量最高,占到了总氨基酸的9.9%;其次为缬氨酸和天冬氨酸,分别占总氨基酸的8.6%和9.3%。脂肪系数为101.32,平均亲水性为-0.071,是亲水蛋白(图3);不稳定指数为17.01,属于稳定性蛋白。
预测发现BeLEA2蛋白发生磷酸化修饰的位点共有10个,其中丝氨酸的磷酸化位点和苏氨酸的磷酸化位点各4个,而酪氨酸的磷酸化位点最少,为2个。BeLEA2蛋白中不含有发生O-糖基化和N-糖基化的位点。
使用SOPMA软件预测BeLEA2蛋白的二级结构。结果表明,该蛋白二级结构主要由无规则卷曲(Random coil)、β转角(Beta turn)、延伸链(Extended strand)和α螺旋(Alpha helix)构成。其中:无规则卷曲最多,占氨基酸序列的45.03%;其次为延伸链和α螺旋,分别占29.80%和21.19%;β转角最少(3.97%)。通过Swiss-Model对纤枝短月藓BeLEA2蛋白的三级结构进行预测分析,以拟南芥LEA14蛋白为模板进行同源建模,预测的BeLEA2蛋白三级结构中含有1个α螺旋和10个β折叠。
2.3 BeLEA2基因的启动子元件
以纤枝短月藓DNA为模板,通过HiTail-PCR扩增BeLEA2基因启动子序列,得到了1 072 bp的BeLEA2基因启动子序列。
通过对启动子元件进行分析,结果显示BeLEA2基因启动子具有典型的CAAT-box和TATA-box元件。在BeLEA2基因启动子区含大量非生物胁迫响应顺式作用元件, 主要包括茉莉酸甲酯响应元件(methyl jasmonate response element,CGTCA-motif);
脱落酸响应元件(abscisic acid responsive element,ABRE);还含有光响应作用元件Sp1和与分生组织有联系的作用元件CAT-box;与启动子响应调控或活性有关的元件CCAAT-box;MYB结合位点(MYB binding site,MBS);MYC和MYB元件,这两类元件都是转录因子作用元件,都和ABA或干旱诱导的表达调控有联系;除此之外,还含有G-box、F-box、A-box、I-box、TCT-motif、TCCC-motif、TGACG-motif、CCGTCC-motif、GATA-motif、GT1-motif等元件,其中ABRE元件含量较高。说明BeLEA2的转录将受到这些元件的影响和调控。
2.4 BeLEA2氨基酸序列比对分析
将BeLEA2编码的氨基酸序列在NCBI数据库中进行Blastp对比,结果表明其与小立碗藓(Physcomitrella patens,XP_024360796.1)、海枣(Phoenix dactylifera,XP_008795021.1)、花旗松(Pseudotsuga menziesii,CAA10047.1)、玉米(Zea mays,NP_001142311.1)、甘蔗(Saccharum officinarum,ACT53873.1)、北美云杉(Picea sitchensis,ADM74314.1)、菠萝(Ananas comosus,OAY80542.1)、高粱(Sorghum bicolor,XP_002441588.1)、黍(Panicum miliaceum,RLN27694.1)、木槿(Hibiscus syriacus,KAE8699781.1)、阿月浑子(Pistacia vera,XP_031249202.1)、月季(Rosa chinensis,XP_024175981.1)的LEA2蛋白同源,并且它们之间的相似性较高。利用DNAMAN 6.0软件将LEA2和其他物种的氨基酸序列进行多序列比对,结果显示,BeLEA2蛋白与其他物种LEA2蛋白具有高度的相似性,总体表现为C端较保守,N端保守性较差(图4)。
2.5 LEA2蛋白系统进化分析
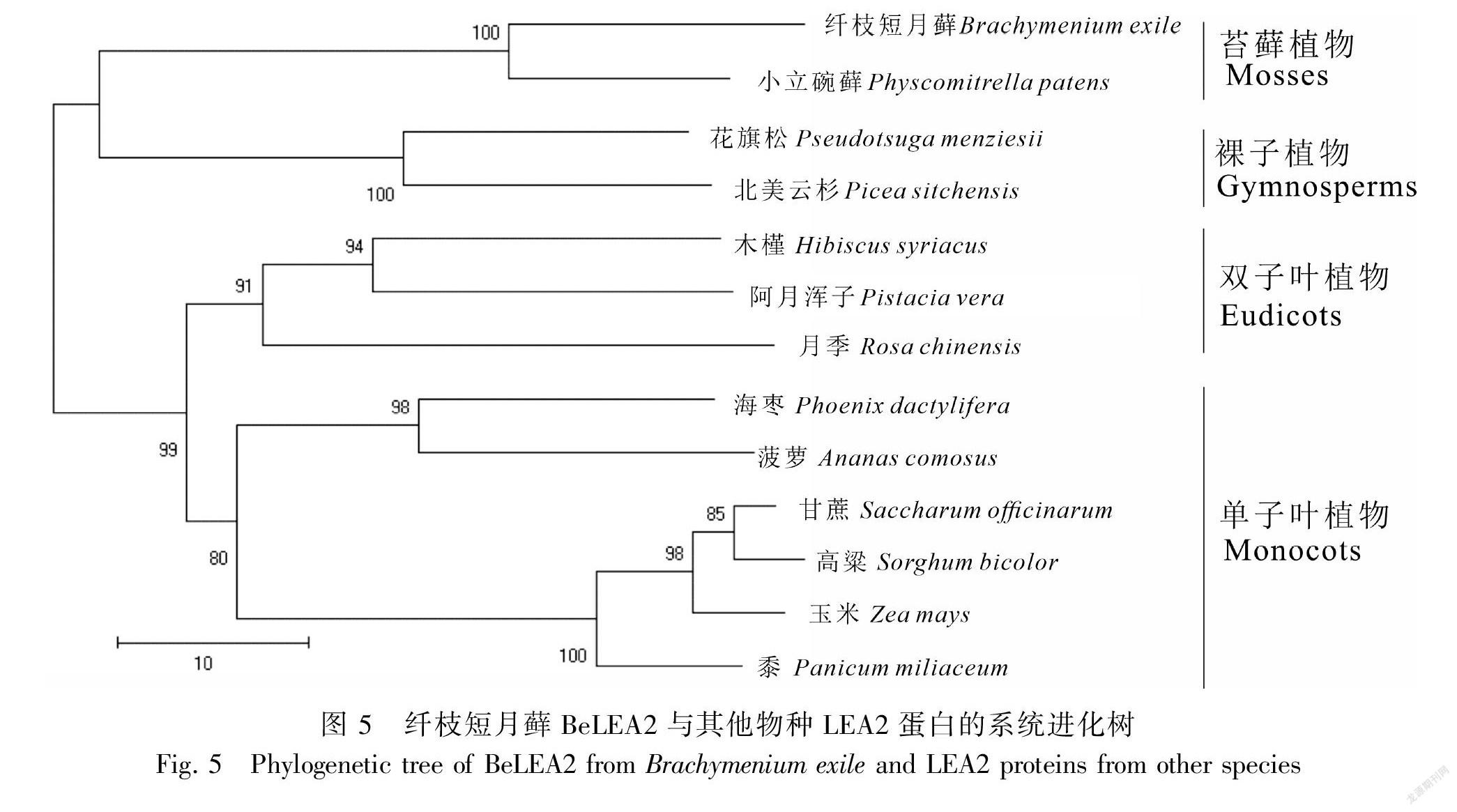
使用MEGA X软件的近邻相接法构建纤枝短月藓LEA2蛋白的系统进化树,进一步探究BeLEA2蛋白与其他物种的LEA2蛋白的进化关系。由图5可知,整个系统发育树分为2个大的分支,纤枝短月藓首先与小立碗藓聚为一支,其进化关系最为接近;裸子植物与苔藓植物聚在一支,其亲缘关系比较接近;而单子叶植物和双子叶植物的LEA2蛋白同属于两个不同的分支,同时与纤枝短月藓有较远的亲缘关系。
2.6 BeLEA2基因实时荧光定量分析
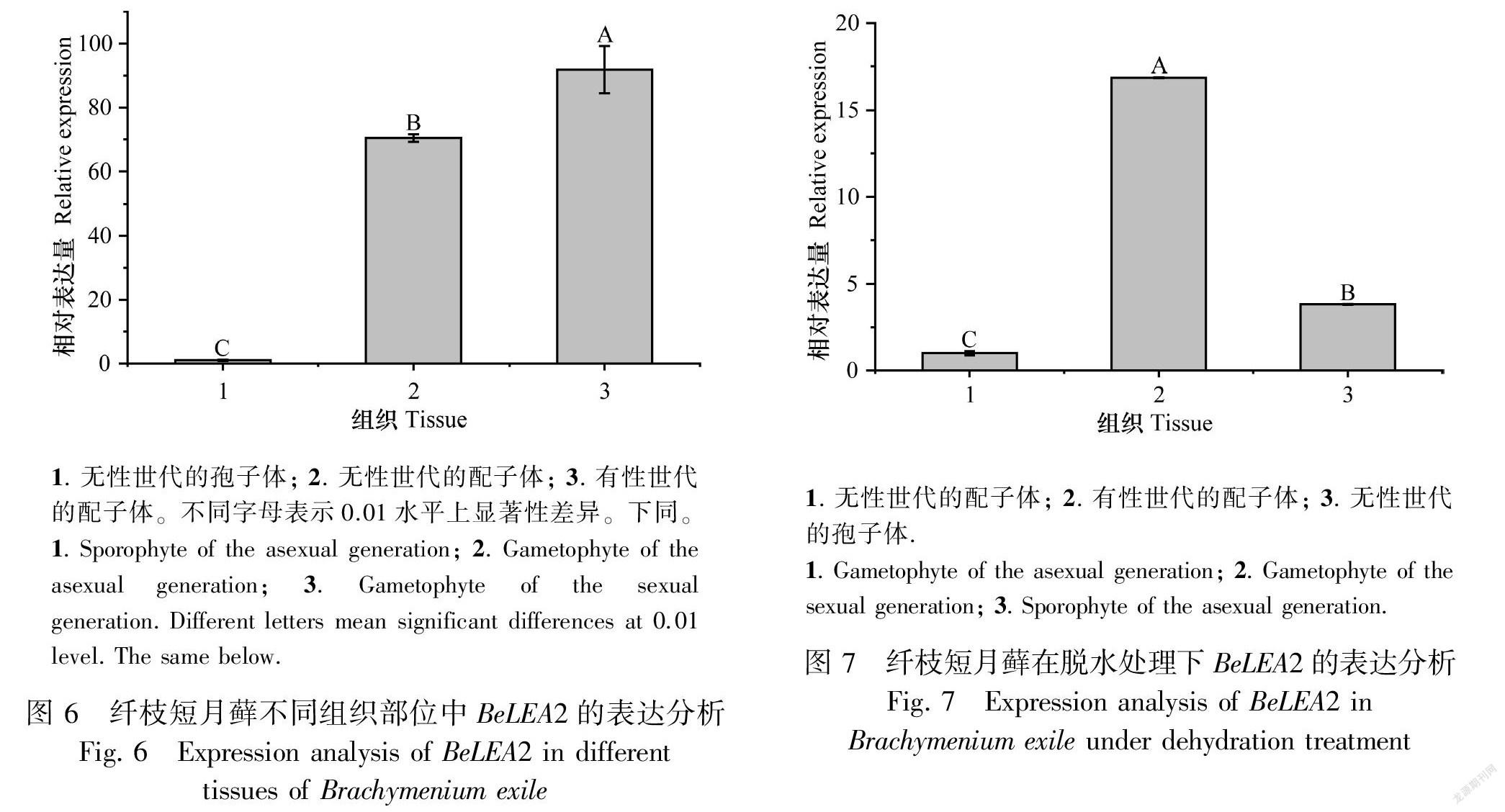
實时荧光定量分析的结果表明,在未作处理的条件下,BeLEA2基因在纤枝短月藓不同的发育时期和部位中都有表达,特别是在有性世代的配子体中表达量最多, 其次是无性世代的配子体,无性世代的孢子体最低,差异极显著(图6)。为了分析BeLEA2是否受到脱水胁迫的诱导,本研究将纤枝短月藓植株各部位都进行了2 h的自然脱水处理。在脱水胁迫条件下,BeLEA2基因在有性世代的配子体中表达量最高,其次是无性世代的孢子体,无性世代的配子体最低,差异极显著(图7)。分析结果证明,不管是正常条件还是脱水胁迫处理,BeLEA2蛋白都在有性世代时期表达量最高,表明BeLEA2基因可能在有性世代的生长发育过程中具有关键作用。
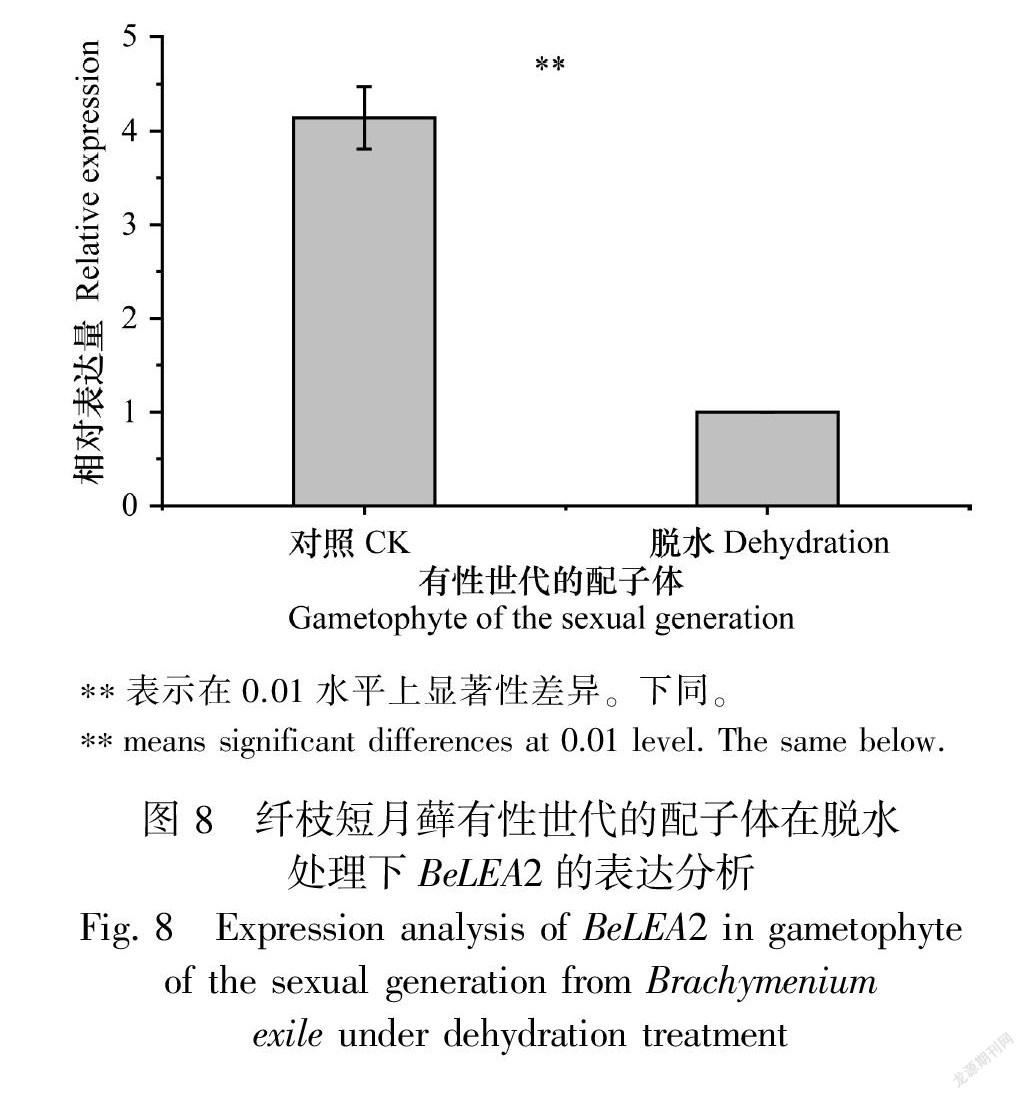
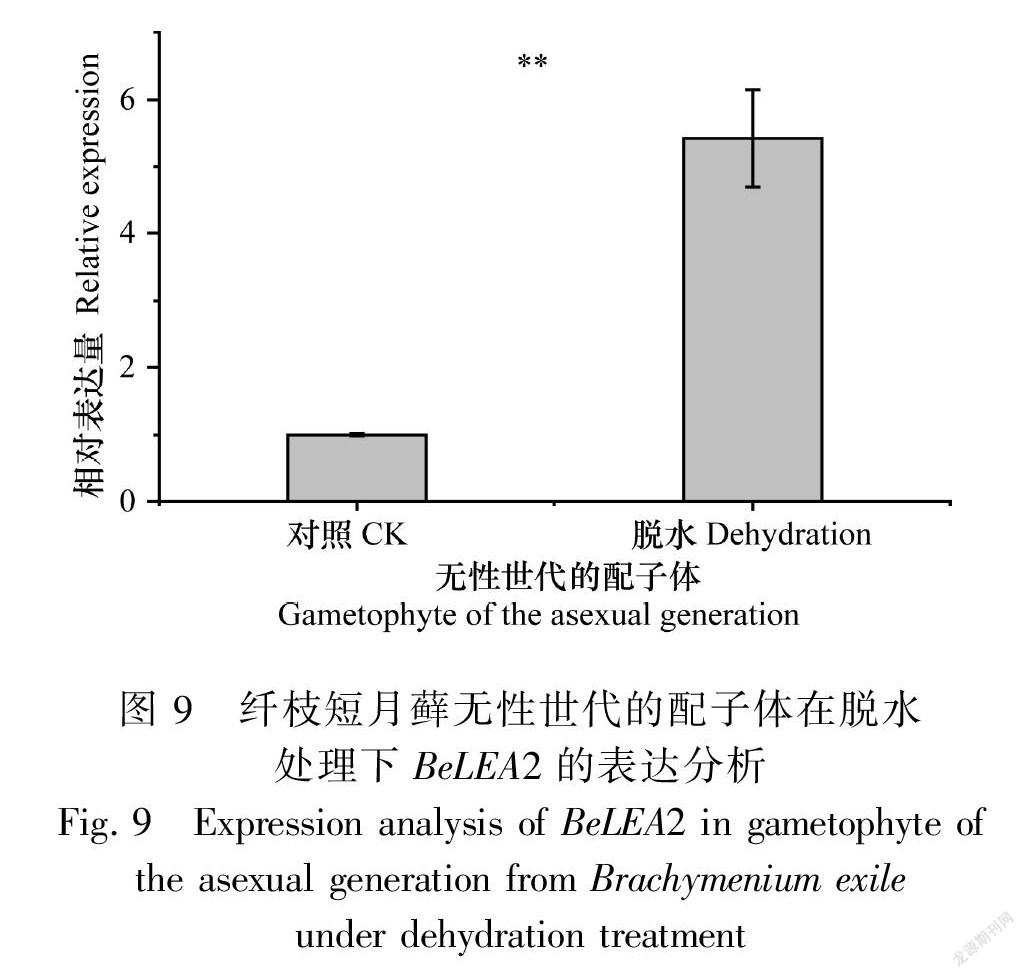
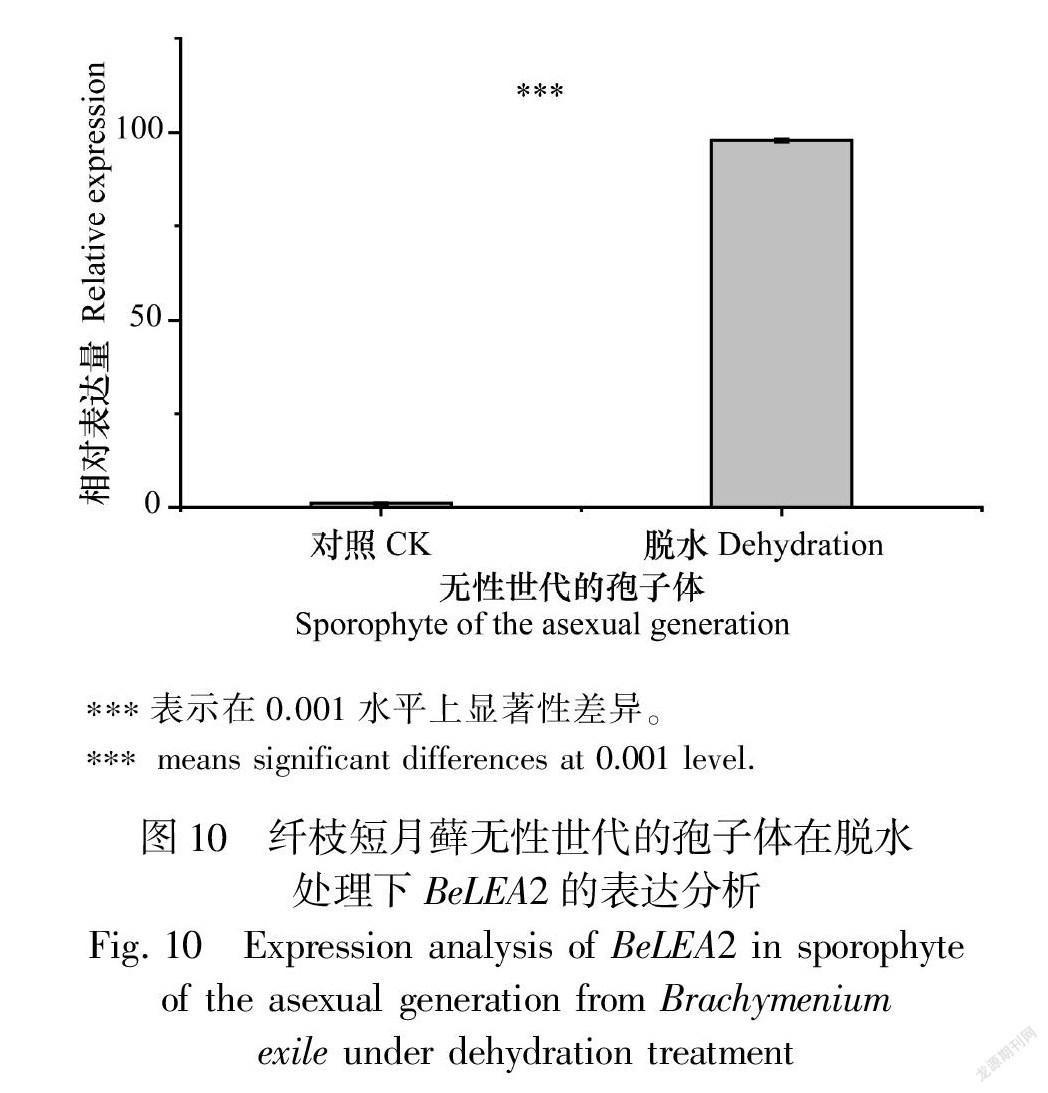
在脱水胁迫下,有性世代时期的配子体表达量降低(图8),差异显著;而无性世代时期的配子体表达量增加,约为正常植株的5.4倍(图9),差异显著;无性世代时期的孢子体表达量有所上升,约为正常植株的98倍(图10),差异极为显著。表明BeLEA2基因可能参与干旱胁迫的响应基因,这与启动子区顺式作用元件预测结果一致,揭示BeLEA2蛋白在脱水条件下可能对孢子体的发育具有重要作用。
3 讨论与结论
苔藓植物是一种结构简单的小型绿色植物,一直以来都以其特殊的生理结构在植物体的进化研究方面占据着重要地位。苔藓植物在世界各地均有着广泛的分布,在潮湿阴暗的地区尤为常见,在高温、干旱等恶劣环境中仍能够正常存活。目前,我国土地荒漠化现象日趋严重,由于地理特征、气候环境因素等影响,极易发生干旱、高温等极端天气,极大影响了植物的正常生长,甚至造成植物大面积枯萎、死亡。但在某些植物中存在着大量的抗逆机制,在植物面临胁迫时,可以通过一些功能性蛋白直接参与胁迫响应(Campo et al., 2014)。而LEA蛋白就属于此类功能性蛋白。根据相关研究和资料表明,LEA基因与干旱等非生物胁迫的调控密切相关(Hundertmark & Hincha, 2008; Magwanga et al., 2017; Muvunyi et al., 2018; I·brahime et al., 2019)。
水稻 (Oryza sativa) 幼苗中,OsLEA19a基因在受到干旱脅迫下会大量表达(胡廷章等,2011);小麦 (Triticum aestivum) 中的TaLEA5基因只有在干旱胁迫时才会表达(刘露露等,2014);将大豆 (Glycine max)LEA基因转化至大肠杆菌 (Escherichia coli)中,在高盐胁迫下的存活率大幅度提高(Lan et al., 2005)。综上所述,LEA基因参与了植物体干旱、高盐等胁迫的响应,这与本研究结果保持一致。
通过分析可知,BeLEA2基因ORF序列为456 bp,编码151个氨基酸,根据Pfam数据库对于LEA蛋白家族的分类,该基因属于LEA2家族。纤枝短月藓BeLEA2基因启动子中不仅含有CAAT-box和TATA-box等基本的元件,同时还有和压力响应相关的顺式作用元件,例如,逆境激素ABA的主要调控元件(ABRE、MYC和MYB)以及MYB的结合位点和茉莉酸甲酯响应元件等元件。推测BeLEA2基因可能会受这些非生物胁迫的调控(Yamaguchi-Shinozaki & Shinozaki, 2005; Du et al., 2013; Huang et al., 2016;李莎莎等,2018; Nagaraju et al., 2019)。这说明BeLEA2在纤枝短月藓的抗逆性中有非常重要的作用,具有深入研究的价值。
从纤枝短月藓LEA2建立的系统发育树分析可以得出:LEA2基因在不同的分类单元中都可以明显地区分开,传统分类的系统进化树和基于LEA2构建的结果一致。值得关注的是,苔藓植物LEA2基因与裸子植物LEA2基因的系统位置较为接近,这是否反映了它们的LEA2基因很可能具有相近的共同祖先,还需更多的研究资料查证。
实时荧光定量分析表明,不管是未经处理还是经脱水胁迫处理,BeLEA2基因在不同时期及不同部位中皆有表达,且在有性世代时期表达量最高,揭示BeLEA2基因可能在有性世代的生长发育过程中起重要作用。脱水胁迫后,BeLEA2基因表达量变化差异明显,在有性世代发育时期的表达下降,而在无性世代发育时期表达上升,揭示了该基因在纤枝短月藓不同生长发育时期对脱水胁迫的响应机制可能不同。
本研究首次从纤枝短月藓中克隆分析了BeLEA2基因,并进行了初步表达分析,结果显示纤枝短月藓中BeLEA2基因在脱水胁迫时有较高的表达,表明BeLEA2基因参与了纤枝短月藓脱水胁迫的响应。本研究对深入了解LEA2基因在苔藓植物生长发育过程中的功能,分析LEA2蛋白植物抗逆的分子机制以及深入探究苔藓植物的抗逆机制具有重要意义。
参考文献:
BHATTACHARYA S, DHAR S, BANERJEE A, et al., 2019. Structural, functional, and evolutionary analysis of late embryogenesis abundant proteins (LEA) in Triticum aestivum: A detailed molecular level biochemistry using in silica approach [J]. Comput Biol Chem, 82: 9-24.
CAO J, LI X, 2015. Identification and phylogenetic analysis of late embryogenesis abundant proteins family in tomato (Solanum lycopersicum) [J]. Planta, 241(3): 757-772.
CAMPO S, BALDRICH P, MESSEGUER J, et al., 2014 Overexpression of a calcium-dependent protein kinase confers salt and drought tolerance in rice by preventing membrane lipid peroxidation [J]. Plant Physiol, 165(2): 688.
DU DL, ZHANG QX, CHENG TR, et al., 2013. Genome-wide identification and analysis of late embryogenesis abundant (LEA) genes in Prunus mume [J]. Mol Biol Rep, 40(2): 1937-1946.
DURE L, GREENWAY SC, GALAU GA, 1981. Developmental biochemistry of cottonseed embryogenesis and germination: changing messenger ribonucleic acid populations as shown by in vitro and in vivo protein synthesis [J]. Biochemistry-USA, 20(14): 4162-4168.
HAND S, JONES D, MENZE M, et al., 2007. Life without water: expression of plant LEA genes by an anhydrobiotic arthropod [J]. J Exp Zool, 307(1): 62-66.
HU YZ, WU YM, CHEN ZG, et al., 2011. Molecular cloning, expression character and bioinformatics analysis of OsLEA19a from rice [J]. Acta Agric Boreal-Sin, 26(6): 73-78. [胡廷章, 吴应梅, 陈再刚, 等, 2011. 水稻OsLEA19a基因的克隆、表达及生物信息学分析 [J]. 华北农学报, 26(6): 73-78.]
HUANG Z, ZHONG XJ, HE J, et al., 2016. Genome-wide identification, characterization, and stress-responsive expression profiling of genes encoding LEA (late embryogenesis abundant) proteins in Moso Bamboo (Phyllostachys edulis) [J]. PLoS ONE, 11(11): e0165953.
HUNDERTMARK M, HINCHA DK, 2008. LEA (Late Embryogenesis Abundant) proteins and their encoding genes in Arabidopsis thaliana [J]. BMC Genet, 9(1): 118.
I·BRAHIME M, KIBAR U, KAZAN K, et al., 2019. Genome-wide identification of the LEA protein gene family in grapevine (Vitis vinifera L.) [J]. Tree Genetics & Genomes, 15(4): 55.
LAN Y, CAI D, ZHENG YZ, 2005. Expression in Escherichia coli of three different soybean late embryogenesis abundant (LEA) genes to investigate enhanced stress tolerance [J]. J Integr Plant Biol, 47(5): 613-621.
LIANG Y, XIONG ZY, ZHENG JX, et al., 2016. Genome-wide identification,structural analysis and new insights into late embryogenesis abundant (LEA) gene family formation pattern in Brassica napus [J]. Sci Rep, 6(1): 24265.
LI SS, DU TZ, AN KL, et al., 2018. Identification and expression analysis of SmLEA gene family in Salvia miltiorrhiza [J]. J Agric Biol Biotechnol, 26(9): 1514-1526. [李莎莎, 杜堂志, 安克莉, 等, 2018. 丹參SmLEA家族基因的鉴定和表达分析 [J]. 农业生物技术学报, 26(9): 1514-1526.]
LI XJ, 2006. Flora bryophtorum Sinicorum [M]. Beijing: Science Press. [黎兴江,2006. 中国苔藓志 [M]. 北京: 科学出版社.]
LIU LL, CHEN L, ZHANG CY, et al., 2014. Characterization of two LEA genes and their response to abiotic stresses in wheat [J]. Chin Agric Sci, 47(19): 3736-3745. [刘露露,陈雷,张春艳,等,2014. 两个小麦LEA基因的特征及其对非生物胁迫的响应 [J]. 中国农业科学, 47(19): 3736-3745.]
LIU YG, CHEN Y, 2007. High-efficiency thermal asymmetric interlaced PCR for amplification of unknown flanking sequences [J]. Biol Technique, 43(5): 649-656.
MAGWANGA R, LU P, KIRUNGU J, et al., 2017. Characterization of the late embryogenesis abundant (LEA) protein family and their role in drought stress tolerance in upland cotton [J]. BMC Genet, 19(1): 6.
MUVUNYI BP, YAN Q, WU F, et al., 2018. Mining late embryogenesis abundant (LEA) family genes in Cleistogenes songorica, a xerophyte perennial desert plant [J]. Int J Mol Sci, 19(11): 6.
NAGARAJU M, KUMAR SA, REDDY PS, et al., 2019.Genome-scale identification, classification, and tissue specific expression analysis of late embryogenesis abundant (LEA) genes under abiotic stress conditions in Sorghum bicolor L. [J]. PLoS ONE, 14(1): e0209980.
PAN RZ, 2012. Plant physiology [M]. Beijing: Higher Education Press. [潘瑞炽, 2012. 植物生理学 [M]. 北京: 高等教育出版社.]
SHINOZAKI K, YAMAGUCHI-SHINOZAKI K, 2006. Gene networks involved in drought stress response and tolerance [J]. J Exp Bot, 58(2): 221-227.
TU ZF, LI MX, SUN T, 2016. The status and trend analysis of desertification and sandification [J]. For Resourc Manag, (1): 1-5. [屠志方, 李梦先, 孙涛, 2016. 第五次全国荒漠化和沙化监测结果及分析 [J]. 林业资源管理, (1): 1-5.]
TUNNACLIFFE A, LAPINSKI J, MCGEE B, 2005. A putative LEA protein, but no trehalose, is present in anhydrobiotic bdelloid rotifers [J]. Hydrobiologia, 546(1): 315-321.
WANG XS, ZHU HB, JIN GL, et al., 2006. Genome-scale identification and analysis of LEA genes in rice (Oryza sativa L.) [J]. Plant Sci, 172(2): 414-420.
WU CL, HU W, YAN Y, et al., 2018. The late embryogenesis abundant protein family in cassava (Manihot esculenta Crantz): genome-wide characterization and expression during abiotic stress [J]. Molecules, 23(5): 1196.
XU ZW, REN XM, ZHAO M, et al., 2019. Cloning and expression analysis of storage protein hexamerin genes in Tenebrio molitor [J]. J SW For Univ (Nat Sci Ed), 39(4): 96-102. [徐志文, 任雪敏, 趙满, 等, 2019. 黄粉甲储存蛋白hexamerin基因的克隆及表达分析 [J]. 西南林业大学学报(自然科学版), 39(4): 96-102.]
YAMAGUCHI-SHINOZAKI K, SHINOZAKI K, 2005. Organization of cis-acting regulatory elements in osmotic- and cold-stress-responsive promoters [J].Trends Plant Sci, 10(2): 88-94.
ZHANG JF, SONG YM, XING SJ, et al., 2002. Saline soil amelioration and forestation techniques [J]. J NE For Univ, 30(6): 124-129. [张建锋, 宋玉民, 邢尚军, 等, 2002. 盐碱地改良利用与造林技术 [J]. 东北林业大学学报, 30(6): 124-129.]
ZHANG P, BAI XL, ZHONG XL, 2005. Advances in the desiccation tolerance of mosses [J]. Bull Bot, 22(1): 107-114. [张萍, 白学良, 钟秀丽, 2005. 苔藓植物耐旱机制研究进展 [J]. 植物学通报, 22(1): 107-114.]
(责任编辑 周翠鸣)
收稿日期: 2020-07-22
基金项目: 国家自然科学基金(31160177);云南省高校科技创新团队支持计划项目 [Supported by National Natural Science Foundation of China(31160177); Project for Innovative Research Team (in Science and Technology) in Colleges and Universities of Yunnan Province]。
第一作者: 李雪宝(1994-),硕士研究生,研究方向为植物分子生物学,(E-mail)421043791@qq.com。
*通信作者: 鄢波,博士,教授,主要从事植物分子生物方向研究工作,(E-mail)yanbodr@aliyun.com。
2404501186214

