肺腺癌LINC00982的TCGA表达谱分析及预后意义
彭瑞 张磊 王鹏 赵丽 王晔 牟晓峰






[摘要] 目的 通过TCGA数据库对肺腺癌(LUAD)病人相关测序资料进行分析,探讨与LUAD预后相关的长链非编码RNA(LncRNAs)。方法 从TCGA数据库下载LUAD的基因表达资料及临床信息,使用edge R包進行差异分析,Survival包行预后分析,加权基因共表达网络分析(WGCNA)包构建共表达网络,寻找与生存显著相关的基因模块,ClusterProfiler包对目标模块的mRNA进行基因本体论(GO)注释和KEGG富集分析。结果 共筛选得到15 473个差异表达基因(DEGs),进一步筛选得到187个差异表达的LncRNAs(DELncRNAs),其中80个DELncRNAs表达上调,107个DELncRNAs表达下调。将DELncRNAs纳入Cox危险比例回归模型进行分析,得到影响LUAD病人预后的15个LncRNAs。Kaplan Meier分析验证显示,Cox分析结果中低风险的LUAD病人较高风险者拥有更长的生存期。通过WGCNA生成40个模块,其中violet模块与生存相关性最高(r=0.680,P<0.05);violet模块中的LncRNAs与预后相关的DELncRNAs取交集,通过筛选得到LINC00982。对violet模块的mRNA进行富集分析后显示,LINC00982在血管生成、白细胞介素-6(IL-6)、淋巴免疫等有关的生物学过程(BP)中显著富集,在核染色体等有关的细胞成分(CC)中占比增加,与嘧菌酯域结合等有关的分子功能(MF)有关,主要参与IL-17、PD-L1等相关的信号通路。Pearson分析显示,PRDM16表达水平随LINC00982表达上调而升高(r=0.897,P<0.001),可能为LINC00982的靶基因。结论 LINC00982可能为预测LUAD病人预后的生物学标志物,通过靶控PRDM16参与LUAD的发生发展。
[关键词] 肺腺癌;长链非编码RNA;癌症基因组图谱;预后
[中图分类号] R730.261
[文献标志码] A
[文章编号] 2096-5532(2022)01-0079-06
doi:10.11712/jms.2096-5532.2022.58.016
肺癌为死亡率最高的一种恶性肿瘤,其发病率逐年上升[1]。其中非小细胞肺癌占所有肺癌病例的80%左右,错过早期诊治的病人普遍生存时间不超过18个月。寻找一种新型分子诊断靶点对于改善非小细胞肺癌病人的整体生存至关重要[2]。非小细胞肺癌常见亚型肺腺癌(LUAD)与肺鳞癌(LUSC)基因表达并不一致,探寻LUAD发病机制中的关键基因,对抑制癌细胞的增殖、迁移、侵袭具有重要意义[3]。长链非编码RNA(LncRNAs)是指超过200个核苷酸的非编码RNA,其可以调节许多生理和病理相关的细胞功能[4]。越来越多的研究结果显示,LncRNAs异常表达与癌症类型相关,这些异常表达的LncRNAs通过调控细胞增殖、迁移、存活来影响细胞内稳态[5]。许多研究都将LncRNAs与非小细胞肺癌的病理特征联系起来,认为其可作为预测非小细胞肺癌病人整体生存的生物学标志物[4]。癌症基因组图谱(TCGA)包含人类33种不同癌症的临床病理数据和多平台的分子图谱,为癌症病理机制的研究和分子靶点的开发提供了有力支持[6]。本文研究基于TCGA数据库中LUAD的LncRNAs表达谱构建Cox危险比例回归模型,并行生存分析,使用加权基因共表达网络分析(WGCNA)构建基因共表达网络,识别与病人预后相关的模块,富集分析注释模块基因的功能,探寻LUAD中的关键基因,为揭示LUAD的发病机制提供理论基础。
1 资料和方法
1.1 TCGA数据集和差异表达基因(DEGs)分析
从TCGA下载LUAD的基因表达资料,时间截止到2020年6月。该数据集包括585例测序资料,筛选出其中临床资料完整的59例正常样本和513例肿瘤样本。使用 edge R包对肿瘤样本与正常样本中的基因的表达量进行差异分析,筛选条件为log2 Fold Change绝对值>1,P<0.05。
1.2 生存分析
整合LUAD病人的临床信息,通过Survival包使用log-rank检验进行Kaplan Meier分析。另构建Cox风险比例回归模型,并行PH检验,报告风险比(HR)和95%置信区间(95%CI),比较差异表达的LncRNAs(DELncRNAs)与LUAD病人生存之间的联系。使用ggplot2包对风险回归模型和生存分析进行数据可视化。
1.3 WGCNA构建基因共表达网络
对TCGA中LUAD样本基因表达量的平均值进行大小排序,挑选513例肿瘤样本中基因表达量最高的15例样本,使用WGCNA包构建共表达模块,软阈值功率设置为30,对样本进行聚类分析,分析模块与性状之间的关系,寻找与生存显著相关的基因模块,分析基因显著性。提取该模块中的基因,将其与预后模型中得到的LncRNAs取交集,筛选出与预后相关的模块基因。
1.4 富集分析
使用ClusterProfiler包对目标模块的mRNA进行富集以了解模块对应的生物学功能,进行基因本体论(GO)注释和KEGG富集分析,使用ggplot2包绘制GO二级分类气泡图及KEGG气泡图,使用GDCRNAtools包计算mRNA与目标LncRNAs之间的Pearson相关系数(PCC),以PCC>0.8、P<0.05作为LncRNAs靶基因的筛选条件。
2 结 果
2.1 LUAD和正常样本中的DEGs
本文59例正常样本和513例LUAD样本共筛选得到15 473个DEGs,进一步筛选得到80个上调的DELncRNAs,107个表达下调的DELncRNAs(图1A),对相应DELncRNAs进行聚类分析,绘制热图(图1B)。其中最显著的前10个表达上调及下调DELncRNAs见表1、2。
2.2 与生存及预后相关的基因
基于DELncRNAs表达矩阵构建预后相关的预测模型,并进行多因素Cox风险比例回归模型分析,根据P<0.05筛选得到影响LUAD病人预后的15个LncRNAs(图2),多因素Cox风险比例回归模型PH检验结果见表3。对高表达及低表达的LncRNAs分别进行Kaplan Meier分析并绘制生存曲线,其中与预后最相关的6个LncRNAs的生存曲线见图3。由图3可见,高表达LINC00982、COLCA1、LINC01852、PWAR6、AL365361.1的LUAD病人较其低表达病人生存期长,TMPO-AS1高表达的LUAD病人预后较差。
2.3 共表达模块及与预后相关的模块基因
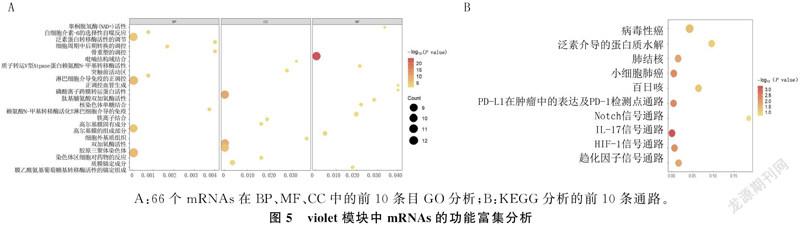
对15个表达量最高的肿瘤样本进行全基因分析,通过动态剪切树算法对样本的全基因组初步识别并分割,将具有相似表达谱的基因聚集于同一模块中,得到40个模块,每个颜色代表1个模块(图4A),对样本再次进行层次聚类分析以明确是否有离群异常值(图4B)。通过对样本临床信息和模块进行关联分析,发现2个模块与生存状态显著相关(图4C),其中violet模块相关程度最高(r=0.680,P<0.01),其生存相关基因与violet模块核心基因之间具有较高的相关性(图4D)。violet模块中仅有两个LncRNAs(LINC00982和AL161785.1)与图2中重叠,其中LINC00982生存分析显示差异有统计学意义,因此将LINC00982作为目标基因进行后续分析。
2.4 关键模块基因的功能分析
结果表明,violet模块中66个mRNA在血管生成、药物反应、细胞外基质、白细胞介素-6(IL-6)、淋巴免疫等有关的生物学过程(BP)中显著富集,在核染色体、高尔基体等有关的细胞成分(CC)中占比增加,与嘧菌酯域结合、二氧酶活性等有关的分子功能(MF)有关,见表4和图5。KEGG通路分析结果显示,这些基因主要参与IL-17、PD-L1、小细胞肺癌、HIF-1等相关的信号通路。Pearson相关分析结果显示,PRDM16表达与LINC00982表达之间相关性最高,两者表达呈显著正相关(r=0.897,P<0.001)。因此,将PRDM16作为LINC00982的靶基因。见图6。
3 讨 论
本文研究首先对TCGA数据库中LUAD病人572例的测序资料进行差异分析,共筛选出187个DELncRNAs,并根据病人的临床信息构建出15个LncRNAs的预测模型,进一步分析结果显示,这些模型DELncRNAs风险评分较高时往往伴随着较短的生存时间。LINC00982、COLCA1、LINC01852、PWAR6、AL365361.1低表达以及TMPO-AS1高表达是LUAD病人的独立预后因素,有望成为评估病人预后的生物学标志物。
在差异表达分析之外,WGCNA已经被确立为一种独立有效的数据挖掘方法,可以用来寻找高度相关的分子集群和模块,并识别模块中的枢纽基因,目前已经被应用于多项肺癌的研究中[7-8]。本文通过对TCGA数据库中LUAD病人中15个表达量最高的肿瘤样本进行WGCNA,共获得40个共表达模块,其中violet模块与生存状态相关程度最高;进一步将violet模块中的基因与影响病人预后的LncRNAs取交集,通过筛选得到了LINC009822和AL161785.1,其中LINC009822具有更好的预后预测价值,因此本文研究主要探讨了LINC00982在LUAD中的预后意义。
本文研究筛选出violet模块mRNA并进行富集分析,GO注释结果显示这些mRNA与血管生成的正向调节、细胞对药物的反应有关,也在IL-6以及淋巴系统免疫等炎症相关的BP中富集。OGAWA等[9]研究显示,IL-6可能成为肺癌新的治疗靶点,联合应用抗IL-6抗体能够增强顺铂靶向作用肺癌组织的敏感性,提高肺癌病人的生存率。此外,本文KEGG通路分析显示,violet模块mRNA主要参与IL-17、PD-L1、小细胞肺癌、HIF-1等相关的信号通路。另有研究结果显示,PD-L1在多种肿瘤细胞中均有表达,PD-L1介导T细胞对靶细胞的生物效应[10]。阻断PD-L1信号通路能增强细胞毒性T淋巴细胞的抗肿瘤活性,从而识别肿瘤特异性抗原,对多种肿瘤均有效;并且其毒副作用明显低于其他免疫疗法。目前,美国食品和药物管理局(FDA)已经批准将抗PD-L1药物用于非小细胞肺癌以及黑色素瘤的治疗中[11-12]。而IL-17作为肿瘤免疫中双向调节因子,一方面能够促进抗肿瘤细胞毒性T细胞反应,导致肿瘤消退;另一方面,IL-17可促进肿瘤细胞的血管生成和转移,导致肿瘤的扩散。有研究表明,肺癌病人肺泡灌洗液中的IL-17表达与调节T细胞中的细胞毒性T淋巴细胞抗原4表达呈负相关[13]。推测LINC00982在LUAD病人中可能通过调控IL-6、IL-17或PD-L1的表达,为肿瘤的抗免疫治疗提供新的分子靶点。
本文最后应用Pearson相关分析LINC009822與violet模块中mRNA表达的相关性,结果显示,PRDM16的表达与LINC00982的表达呈正相关,PRDM16可能为LINC00982的靶基因。L等[14]的研究发现,LUAD病人LINC00982和PRDM16表达和拷贝数的变异较低,而甲基化程度则较高,LINC00982和PRDM16基因低表达的LUAD病人预后不良,并且该研究在LUAD细胞株中进一步验证结果显示LINC00982和PRDM16表达下调。本文研究结果与其一致。另有研究指出,PRDM16在LUAD中表达下调,其表达水平与LUAD的主要临床病理特征和预后密切相关;体内外实验结果表明,PRDM16可以通过抑制LUAD上皮间充质转换(EMT)的调节因子MUC4的转录,从而抑制癌细胞的EMT。因此,PRDM16可能成为LUAD的一个转移抑制因子和潜在的治疗靶点[15]。
综上所述,LINC00982低表达是LUAD病人的预后相关危险因素,LINC00982可能通过调控PRDM16参与LUAD的发生发展。LINC00982可能成为LUAD预后评估的生物学标志物。
[参考文献]
[1]SIEGEL R L, MILLER K D, JEMAL A. Cancer statistics, 2019[J]. CA: A Cancer Journal for Clinicians, 2019,69(1):7-34.
[2]石少敏,赵建军,杨蕾. 抑制有氧糖酵解增加硼替佐米诱导的非小细胞肺腺癌细胞凋亡[J]. 中国实验诊断学, 2018,22(11):1999-2001.
[3]WANG T J, ZHANG L, TIAN P, et al. Identification of differentially-expressed genes between early-stage adenocarcinoma and squamous cell carcinoma lung cancer using meta-analysis methods[J]. Oncology Letters, 2017,13(5):3314-3322.
[4]LIU B, CHEN Y F, YANG J. LncRNAs are altered in lung squamous cell carcinoma and lung adenocarcinoma[J]. Oncotarget, 2017,8(15):24275-24291.
[5]HUARTE M. The emerging role of lncRNAs in cancer[J]. Nature Medicine, 2015,21(11):1253-1261.
[6]LIU J F, LICHTENBERG T, HOADLEY K A, et al. An integrated TCGA Pan-cancer clinical data resource to drive high-quality survival outcome analytics[J]. Cell, 2018,173(2):400-416.e11.
[7]WANG X H, LI G, LUO Q S, et al. Integrated TCGA analysis implicates lncRNA CTB-193M12.5 as a prognostic factor in lung adenocarcinoma[J]. Cancer Cell International, 2018,18:27.
[8]QI G Q, KONG W, MOU X Y, et al. A new method for excavating feature lncRNA in lung adenocarcinoma based on pathway crosstalk analysis[J]. Journal of Cellular Biochemistry, 2019,120(6):9034-9046.
[9]OGAWA H, KOYANAGI-AOI M, OTANI K, et al. Interleukin-6 blockade attenuates lung cancer tissue construction integrated by cancer stem cells[J]. Scientific Reports, 2017,7(1):12317.
[10]DAVIES L C, HELDRING N, KADRI N, et al. Mesenchymal stromal cell secretion of programmed death-1 ligands regulates T cell mediated immunosuppression[J]. Stem Cells (Dayton, Ohio), 2017,35(3):766-776.
[11]IWAI Y, HAMANISHI J, CHAMOTO K, et al. Cancer immunotherapies targeting the PD-1 signaling pathway[J]. Journal of Biomedical Science, 2017,24(1):1-11.
[12]SUL J, BLUMENTHAL G M, JIANG X P, et al. FDA approval summary: pembrolizumab for the treatment of patients with metastatic non-small cell lung cancer whose tumors express programmed death-ligand 1[J]. The Oncologist, 2016,21(5):643-650.
[13]KWIECIEN I, STELMASZCZYK-EMMEL A, POLUBIEC-KOWNACKA M, et al. Elevated regulatory T cells, surface and intracellular CTLA-4 expression and interleukin-17 in the lung cancer microenvironment in humans[J]. Cancer Immunology, Immunotherapy, 2017,66(2):161-170.
[14]L W, YU X, LI W X, et al. Low expression of LINC00982 and PRDM16 is associated with altered gene expression, da-maged pathways and poor survival in lung adenocarcinoma[J]. Oncology Reports, 2018,40(5):2698-2709.
[15]FEI L R, HUANG W J, WANG Y, et al. PRDM16 functions as a suppressor of lung adenocarcinoma metastasis[J]. Journal of Experimental & Clinical Cancer Research, 2019,38(1):1-16.
(本文編辑 黄建乡)
3681500338288

