作物根系表型鉴定评价方法的现状与展望
李龙,李超男,毛新国,王景一,景蕊莲
作物根系表型鉴定评价方法的现状与展望
李龙,李超男,毛新国,王景一,景蕊莲*
中国农业科学院作物科学研究所/农作物基因资源与基因改良国家重大科学工程,北京 100081
根系是作物固定植株并吸收土壤水分和养分的主要器官,其表型特征直接影响作物的生产力和适应性。优化根系表型被认为是实现第二次“绿色革命”的重要途径之一。然而,根系的隐匿性、复杂性和可塑性极大地制约着根系表型鉴定效率,导致根系优化进程远远滞后于地上部器官。随着光谱成像、机器学习和三维重建等新技术的快速发展,根系表型鉴定方法逐渐由传统取样观测向原位、无损、自动化检测转变,评价依据由二维形态指标向立体构型参数拓展,促进了根系表型鉴定效率大幅提升,根系表型数据快速增长。与此同时,海量数据也带来了信息冗余及利用率低等问题,对根系表型研究提出了规范化和共享化的时代新要求。本文概述了现行主要根系表型鉴定方法的原理和技术要点,从精准度、通量和成本等方面对不同方法进行系统比较,并从使用许可、运行平台和分析方式等方面对常用根系表型量化软件进行归纳总结;进一步提出今后重点研究方向,即开发高效的田间根系表型鉴定方法,建立根系可塑性鉴定评价技术体系,加强根系解剖结构的鉴定和利用,强化分子检测技术在根系表型鉴定中的应用,推进根系表型鉴定技术规范化和数据信息共享化,以期为合理选用和改进作物根系表型鉴定评价方法提供参考,促进作物根系改良。
作物;根系表型;室内鉴定;田间鉴定;评价方法
0 引言
根系是作物固定植株地上部并吸收土壤水分和养分的主要器官,也是多种激素和有机酸合成的重要场所,其表型特征与产量、品质和适应性均有密切的关系[1-2]。1978年,GreGORY等[3]发现小麦根系干物质随土层深度增加而递减,呈现“锥体”分布,“锥体”衰减缓慢(深层根系比重大)有利于小麦抵御干旱胁迫。随后,研究者针对不同作物根系表型与产量及耐逆性的关系开展了大量研究,相继提出适宜于不同生态环境或生产管理条件的作物“理想根型”[4]。例如,以耕层数量多、分布广为主要特征的耐低磷根型[5],以及抗旱耐低氮的“陡-廉-深”(steep-cheap-deep)根型[6]。依据“理想根型”定向改良作物根系有望进一步提高作物生产力和适应性,被认为是实现第二次“绿色革命”的重要途径[7]。然而,根系隐匿于土壤中,表型鉴定工作耗时耗力且准确性较低,极大地制约着“理想根型”在育种实践中的应用,致使根型优化进程远远滞后于地上部株型[8]。因此,提高根系表型鉴定效率是加速品种改良的迫切需求。
近年来,得益于成像技术及数据分析平台的快速发展,形式多样的新型根系表型鉴定方法不断涌现[9]。在鉴定技术上,从传统取样观测模式逐步向自动化图像提取模式转变[10];在数据分析深度上,从基本根系形态指标逐步向三维根系构型参数拓展[11]。然而,尽管近年来作物根系表型鉴定评价方法已经取得了长足的进步,但是仍难以兼具经济、高效和精准的特性[12]。因此,根据研究目标、观测对象及操作环境等因素合理选择根系表型鉴定方法依然是现阶段提高根系表型鉴定效率的主要方式[1]。本文系统评述了当前作物根系表型的鉴定评价方法,并提出未来的重点研究方向,以期为合理选用和改进作物根系表型鉴定方法提供参考。
1 室内根系表型鉴定方法
根据检测对象的生长环境,根系表型鉴定方法可分为室内鉴定与田间鉴定两大类[9]。室内鉴定对象种植于人工设施内,研究者可以根据试验需求控制光照、温度和湿度等生长环境。
1.1 二维根系表型鉴定方法
传统室内根系表型鉴定方法间的根本区别在于作物生长介质和容器的不同,因此,传统室内根系表型鉴定方法通常以作物生长所采用的介质或容器而命名。例如,萌发袋法、凝胶根室法、净盆法、根管法等。萌发袋法以种子萌发袋作为生长容器,萌发袋由袋子和纸芯2部分组成,将无菌水或营养液加入袋子中润湿纸芯,种子放置于袋子顶部纸芯凹槽中即可吸收水分而萌发,根系穿过纸芯顶部空隙向下方透明袋子中生长,透过袋子即可对根系进行观测或图像采集[13]。然而,萌发袋纸芯顶部空隙往往限制种子根的自然开张,从而影响种子根的角度。凝胶根室法以琼脂糖凝胶作为根系生长介质,将琼脂糖溶液倒入利用透明板材(玻璃或亚克力板)和塑料封条制作的凝胶槽,待其凝固后,在凝胶上方播放种子,然后用另一块透明板盖住凝胶槽,形成凝胶根室,将根室垂直放置,根系即可在凝胶根室中生长(图1),可用于观测种子根的自然开张角度[14]。萌发袋法和凝胶根室法均采用无土栽培模式,具有廉价、快速及分辨率高等优点,但在播种及根系生长过程中容易遭受细菌或真菌污染。为此,Richard等[15]研发出净盆(clear-pot)法,净盆为一种特制花盆(图1),盆壁四周均匀分布空隙,将种子播于空隙中,种子根发生后沿盆壁生长,通过拍摄盆壁可获取清晰的根系图像,该方法不仅解决了微生物污染的问题,而且环形的播种设计也大大提高了空间利用率。上述方法所用的根系生长容器体积较小,一般仅用于鉴定幼苗期初生根表型。初生根数量较少,例如,小麦初生根在第一片完全叶出现后便停止发生,而不定根、冠状根或侧根等次生根则是固定植株地上部并吸收土壤水分和养分的核心组织[16]。根管法是目前应用较为广泛的次生根表型鉴定方法,一般使用聚氯乙烯(polyvinyl chloride,PVC)硬管作为生长容器,首先沿管口直径将PVC硬管分为两半,而后用合拢套固定,垂直放置,管内装入按试验要求所配制的土壤,取样时将PVC硬管水平放置,去掉合拢套并打开PVC硬管,清洗土柱获取完整根系[17]。近年来,该方法有了进一步改进,即先将土壤装入软质塑料管中,再将软管置于提前掩埋好的PVC硬管中,测量时抽出软管冲洗土柱即可,掩埋好的PVC硬管可以重复使用,也免去了PVC硬管的掩埋和挖取,以及合拢套安装与拆解等复杂环节[8]。
1.2 三维根系表型鉴定方法
传统室内鉴定方法所获取的根系样本及其图像可用于提取根系平面几何构型,即长度、数目和直径等反映同一根系的不同单根在根轴二维平面上的分布特征,但是无法检测根系在生长介质中的三维空间配置和布局,即根系是如何通过分支相互联结的,其中涉及根系空间分布函数、各单根形态特征及拓扑结构等大量立体几何构型,这些构型特征很大程度上决定了根系在生长介质中的空间占有率和对水肥资源的吸收效率[9]。
根系三维构型的检测需借助以原位成像、动态拟合及三维建模等方法为核心的新一代室内数字化成像技术,主要包括X射线计算机断层扫描(X-ray CT)、磁共振成像(magnetic resonance imaging,MRI)、正电子发射断层显像(positron emission tomography,PET)和电阻抗断层成像(electrical impedance tomography,EIT)等技术[9, 18-20]。其中,X-ray CT是通过检测穿过物体的X射线衰减差异来反映物体内部不同位置的物质组成及其密度,该技术早在20世纪就应用于医学领域,而后逐渐拓展至植物三维根系构型研究[21],例如,水稻根系表型可塑性研究以及拟南芥、玉米和水稻侧根生长模式研究等[21-22]。不过,X-ray CT对根系的识别能力受土壤中非根物质影响较大,适用于检测生长在质地均匀且无黏土矿物和铁磁颗粒的非膨胀土中的根系[21]。MRI法是通过射频波和强磁场激励(stimulate)原子生成三维根系空间分布图(图1)。适度的磁共振激励能够强制原子核磁矩偏移到与作用磁场垂直的平面,停止激励后,原子核磁矩将恢复到静态磁场状态,原子核在重新排列的过程中释放能量并发出共振频率的射频信号,MRI对该信号进行检测并形成三维根系图像[20]。该方法对根系生长介质要求较高,适宜的介质有助于提取更精准的根系图像。Pflugfelder等[23]从8种介质中优选出5种适宜于MRI成像系统的生长介质,其中2种介质为人工配制的。Metzner等[24]对X-ray CT和MRI的成像能力进行评比,发现当根系生长容器体积较小时,X-ray CT对根系结构的辨析能力更强,而当容器体积较大时,MRI能够检测出侧根分布等更多的根系细节;此外,土壤湿度对两者的成像能力均有影响,而对X-ray CT的影响更大。因此,2种方法相结合能够获取不同环境下更加精确的三维根系图像[24]。PET法是通过可视化短半衰期放射性示踪元素(例如碳同位素)在根系中的分布来呈现三维根系图像,尽管该方法检测放射性示踪元素的灵敏度很高,但是分辨率较低(1.4 mm左右),通常与X-ray CT、MRI技术配合使用[25]。以上3种根系表型原位探测方法共有的局限性在于系统功能复杂且成本高,投入使用后需要配备高度专业化的操作人员。与之相比,EIT法成本低廉、检测速度快。EIT法是对土壤表面施加微弱电流,根据电压与电流之间的关系重构出土壤中电导率变化的分布情况,进而以电导率的分布变化间接反映根系布局。因此,该方法获取的根系图像仅属于功能图像,分辨率极低,无法用以观测具体的根系构型[18]。
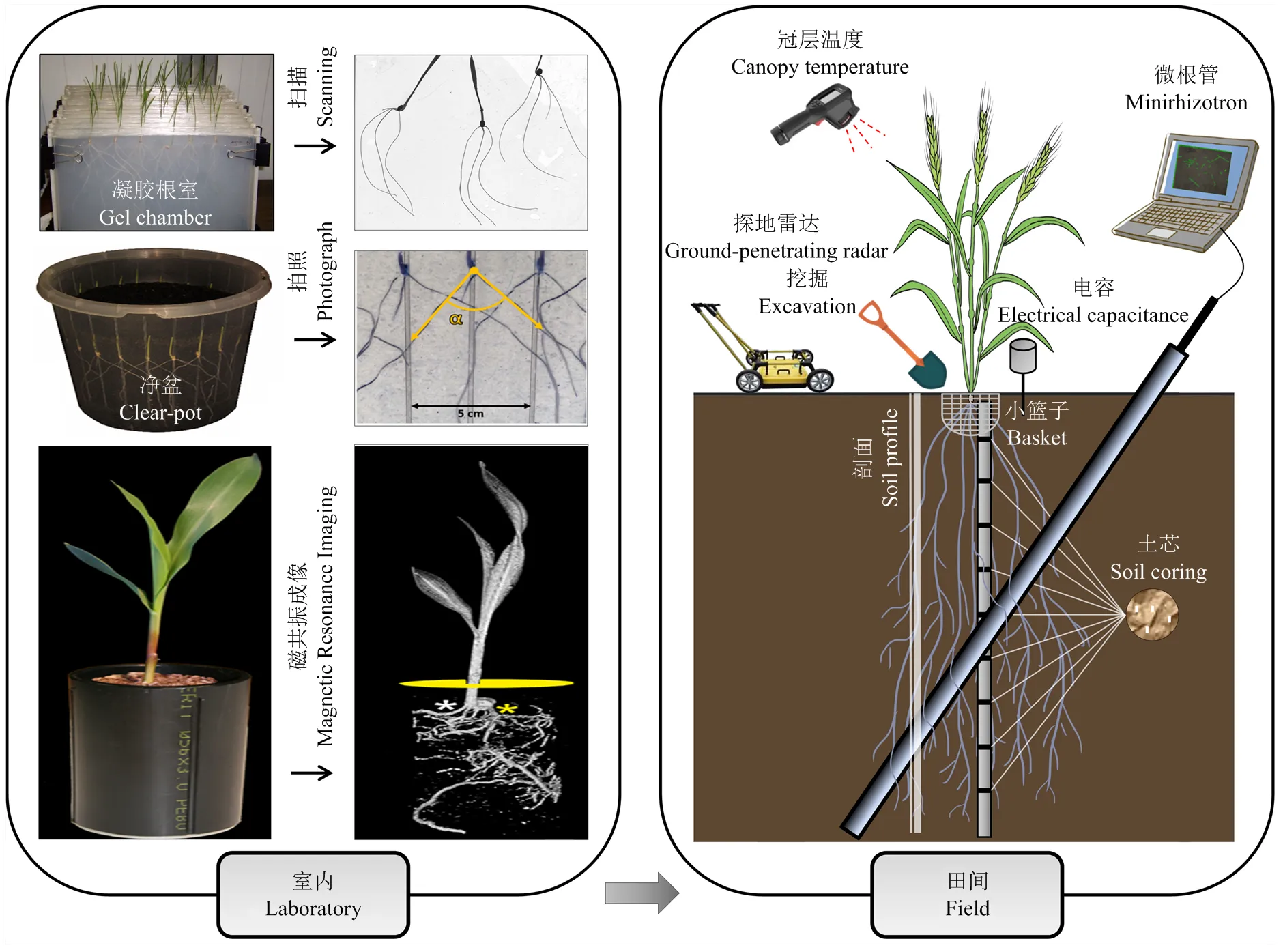
图中净盆和磁共振成像图分别引自Richard等[15]和van Dusschoten等[20]
2 田间根系表型鉴定方法
室内根系表型鉴定具有占地面积小,环境可控及重复性好等优点,但是人工环境下发育的根系始终无法准确表征田间生产环境下的根系构型[16]。因此,高通量田间根系表型鉴定是室内根系表型鉴定的必要补充,也是将根系表型研究应用于生产实践的迫切需求。田间根系表型鉴定方法可根据根系样本(图像)采集是否为原位采集分为破坏性鉴定法和原位鉴定法。
2.1 破坏性鉴定法
破坏性鉴定法主要包括挖掘法、土芯法、小篮子法、网袋法及剖面法等(图1),其中,前3种方法应用较为广泛。挖掘法是利用铁锹等工具将根系从土壤中挖出,清洗后进行观测,操作简单、易行且直观性强,但是对根系的损伤程度较大[26]。近年来,研究者针对不同作物制定挖掘技术规范,革新操作工具,使得该方法所获取的根系样本完整度不断提高。例如,Zheng等[27]针对玉米和高粱制定了“CREAMD”挖掘流程,利用高压气洗取代传统水洗方式,快速清除根系表面土壤,获取了30 cm土层以上较为完整的根系样本。然而,随着土层深度增加,根系挖掘难度及易损性增加,挖掘法的鉴定效率和准确性降低。因此,挖掘法一般用于受耕作、施肥及灌溉等因素影响最大的耕层(0—30 cm)根系的表型鉴定。深层根系表型可采用土芯法进行鉴定,其核心工具是土钻,用于钻取植株下方小于根生长体积的土芯。土芯处理方式主要有2种:(1)水平放置后等距截断,调查不同深度横截面上显现的根数;(2)冲洗土芯后回收根系样本。进而通过横截面根数及样本分析结果鉴定根深、根生物量及根长密度等指标[28]。然而,土芯法只能获取局部区域的根系样品,通过局部观测推断整体。因此,选用土芯法鉴定根系表型需配以合理的试验设计。例如,Wasson等[28]采用高密度点播法(hill plot)增加取样点根系密度,同时根据研究对象的生长特性来确定取样频率,增加样品重复数,从而提高了局部样本的代表性以及不同供试材料之间的对比度。小篮子法是近年来用于鉴定水稻、小麦和大麦等须根系作物根系延伸方向的重要方法[29-30]。该方法首先根据作物根系直径选取孔径适宜的小篮子(网格容器),将其埋入土壤中,在篮子中心位置播种,根系从篮子孔隙穿出,通过挖掘小篮子并调查不同位置孔隙的根数量可以判断根系的延伸方向,从而计算根系在不同土层的分布比例[31]。目前,基于该方法所获取的根系表型信息,已挖掘到水稻根深相关重要基因和。高表达可以促使根系向下生长,因而吸收更多的深层土壤水分增强水稻抗旱性;而高表达能够促使水稻根系在表土层中伸展,从而减轻盐渍土壤缺氧而导致的减产[29, 32]。
2.2 原位鉴定法
破坏性鉴定法技术难度低、易操作且直观性强,但是取样观测耗时耗力,比较粗放。随着现代高新技术的不断进步,人们已经探索出更加智能、高效的田间根系表型原位鉴定方法,主要包括微根管法、探地雷达法及电容法等,可以对根系进行实时监测。微根管法最早由Bates于1937年提出[33],在作物播种前将透明管束埋入植株下方的土壤中,待根系长出后,将柱形扫描探头插入微根管扫描管壁上的根系图像,通过解析图像提取根深、根长密度、根表面积及根数目等指标(图1)[8]。由于扫描探头只能获取靠近管壁上的根系信息,因此,该方法属于局部观测法,播种时应注重等距密植,以增加根系附着在微根管上的几率,并减少人为误差。微根管表面的根长受微根管影响,根沿管壁的生长速度与自然生长速度不同,而根数目受管壁影响较小,是该方法的重点指标[34]。传统的微根管系统中扫描探头长度一般为20 cm,需要分段扫描才能获取完整的根系图像,扫描图像中的根系也需通过肉眼识别、手工绘制,操作过程十分耗时[34]。针对这一问题,Svane等[35]研制出自动化微根管监测平台,利用Videometer MR多光谱成像系统获取光谱图像,经过Videometer软件提取根系图像,大大提高了微根管法的检测效率。此外,传统的微根管系统所采用的RGB可见光成像技术是根据颜色差异识别根系,当根系和土壤之间色差较小时,容易发生误判。Videometer MR多光谱成像系统则基于光谱特征识别根系,准确性高。不仅如此,Wang等[36]基于机器学习算法开发了SegRoot软件,初步实现了微根管扫描图像中根系与土壤背景的自动化分离。这些技术革新推动着微根管检测系统的不断完善,有望使其成为田间根系表型高通量原位检测的优选方法。探地雷达法和电容法是2种低分辨率的地球物理学方法,基于土壤物理学参数间接评价根系发育状况。探地雷达法是利用一个天线发射高频宽带(1MHz-1GHz)电磁波,利用另一个天线接收来自地下介质界面的反射波,进而探测地下介质结构的一种电磁法,其测定速度快,但是对土质要求高,目前仅适用于沙质土壤,另外,该方法难以分辨细根(直径小于5 mm),适宜于检测较为粗壮的根系[37]。电容法是依据根系质量与根际电容值之间的线性关系而提出的,最初应用于林木根系表型研究,近年来逐渐应用于玉米、小麦及大麦等作物根系生物量的检测[38]。电容值的读取受根系所在土层、根系类型及发育时期影响较大,目前,对于这些因素的影响机制仍知之甚少。除探地雷达法和电容法之外,根系表型间接评价方法还包括替代性状法。替代性状是指与根系性状显著相关且便于检测的性状,通过检测此类性状能够间接评价根系表型[39]。例如,多项研究表明冠层温度与根系深度显著负相关,与浅根材料相比,深根材料能够从含水量丰富的深层土壤中汲取更多水分,通过蒸腾作用散发热量,使植物体维持较低的代谢温度。因此,冠层温度可作为替代性状用于根系深度的判定[8, 40]。但值得注意的是,冠层温度易受光照、温度、风速及大气湿度等农田小气候影响。因此,应选择晴朗、无风且气温稳定的时间段测量,并尽量缩短测量时间,保证测量不同供试材料时的外界环境基本一致。近年来,无人机观测技术的应用大大提高了冠层温度的测定速度和精度,为田间检测作物根系深度提供了间接、高通量的技术支撑[41]。然而,目前已发现的根系表型可替代性状还十分有限,冠层温度与根系性状的相关系数偏低。因此,现阶段的替代性状法仅适用于对根系表型进行辅助判断,也就是大概率判断,而非绝对判断[28, 40]。
综上所述,田间根系表型鉴定方法种类繁多,不同方法各有优点,但也有其自身的局限性(表1)。根系表型鉴定方法的选择决定试验效果,选择时应考虑以下6个方面:(1)“精确度”,对目标性状鉴定结果的精细程度和准确性;(2)“工作量”,操作过程所投入的劳动力;(3)“鉴定范畴”,所获取的表型参数多样性;(4)“动态性”,能否对根系进行实时监测或反复测定;(5)“成本”,仪器设备投资总量;(6)“通量”,单位时间内检测的最大样本量,用以评价实际操作的便捷性。一般来讲,研究对象较少时,应重点考虑精确度、鉴定范畴及动态变化,尽可能地获取全面信息,开展深入分析;研究对象较多时,应着重考虑工作量、成本及高通量,尽量缩短检测时间,从而减少因环境变化而产生的表型误差。另外,不同鉴定方法的原理差别较大,对于不同类型根系的检测能力和效果不同。例如,探地雷达法在检测不同直径的根系时表现出不同的辨识能力[37]。因此,在选择田间根系表型鉴定方法时除了考虑其特点外,还要注意被检测植物的根系特点。
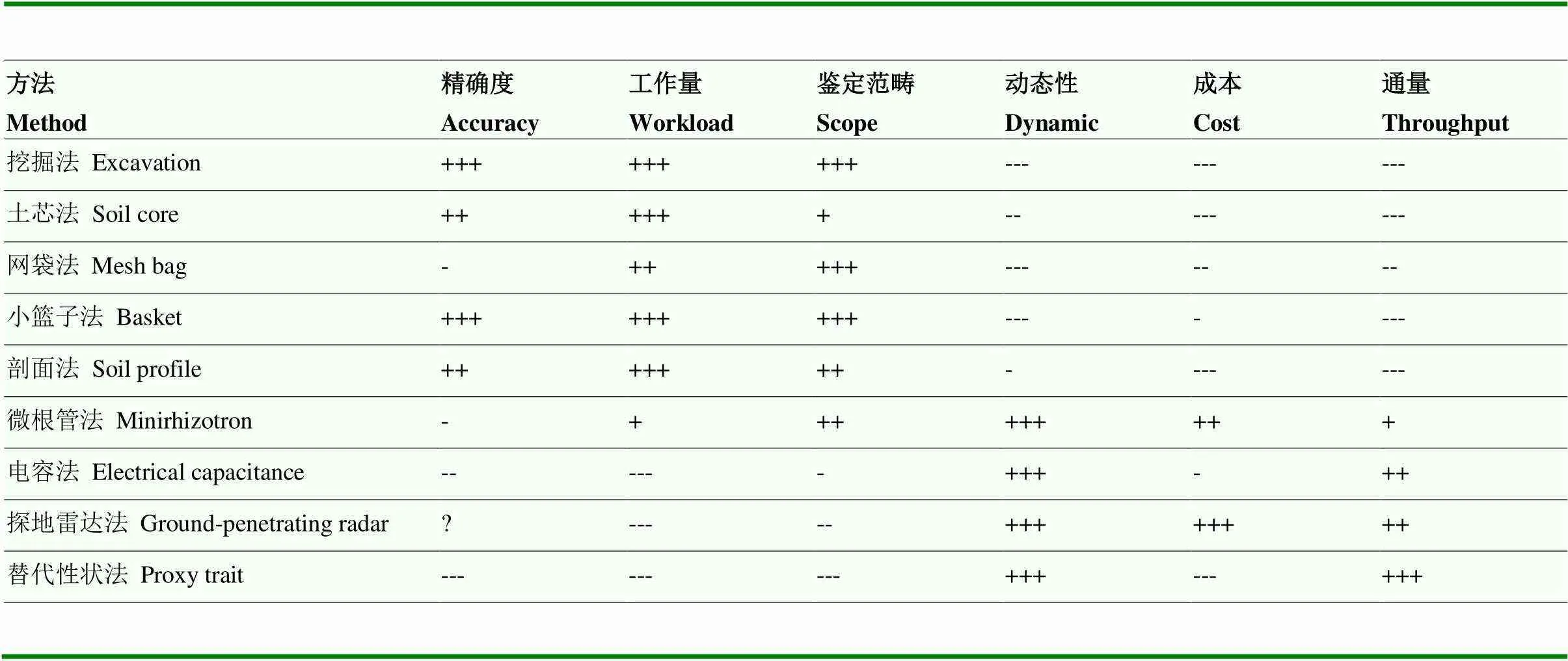
表1 田间根系表型鉴定方法评价
+++:极高;---:极低;?:精确度与待测植物根直径有关
+++: very high; ---: very low; ?: The accuracy depends on the root diameter of the plant to be measured
3 根系表型参数提取方法
随着根系表型鉴定技术推陈出新,根系样本的采集形式也逐渐由实体向图像转变,同时推动着根系表型参数提取方法由“一把尺子一杆秤”的肉眼观测转变为智能化软件解析[9]。通过肉眼观测根系组织所获取的表型量化指标十分有限,主要包括重量、数量和长度等二维形态指标,且观测对象主要针对种子根、冠状根及节根等宏观组织,而无法考察各级侧根、根毛及根尖等细微组织。根系表型分析软件能够将立体和细微结构特征均纳入考察范围,极大地丰富了根系样本的量化参数。因此,开发和利用根系图像分析软件对于提高根系表型量化水平至关重要。目前已报道的根系图像分析软件多达近百种,研究者可根据软件使用许可、运行平台、分析模式、批量处理能力及三维成像能力等多个方面进行选择(表2)。
WinRHIZO是应用较早的一套根系图像分析软件,通常与不同型号的EPSON根系扫描仪匹配使用。该软件依据根系图像的像素及投影面积获取根长、根直径及根体积等基本形态指标,根据颜色分级确定根系存活以及生长状况,根据分支角度、交叉点和连通性等特征确定各级根的数量及拓扑结构。目前已广泛应用于作物根系构型的鉴定评价、时空演变及遗传解析等众多研究中[59-61]。该软件采用自动化分析模式,即自动识别图片背景中的根系样本并提取表型数据,但在分析前需手动载入每张照片并指定分析区域,不能对图像进行批量处理;此外,闭源收费的管理模式也限制了该软件的进一步改造和普及。RootNav是一款应用较为广泛的免费开源根系图像分析软件,该软件采用半自动化分析模式,即在软件识别根系的基础上,用户还可通过手动辅助识别。例如,用户可以指定图像中根基和根尖的位置,RootNav将根据此信息和图像中的像素强度生成主根和侧根模型,通过进一步的人工校对,可准确显示各级根系网络;另外,用户还可以改变图像中不同区域的阈值参数,以提取不同亮度背景下的根系表型,从而减少因图像亮度不均而产生的表型误差[52]。不仅如此,开源模式推动RootNav不断升级,已发布的RootNav 2.0版本将机器学习技术纳入到根系识别中,大大强化了RootNav自动识别根系构型的能力[62]。随着计算机语言和网络技术的普及,开源已成为软件发展的必然趋势,一些根系图像分析软件甚至被用作已有图像分析软件的插件。例如,基于Image J图像分析软件的根系分析插件SmartRoot、GT-RootS及Root Hair Sizer,对于熟悉Image J软件的用户来讲,安装过程和操作界面十分友好。其中,SmartRoot采用半自动分析模式,需要手动描出每条根,虽然分析效率较低,但可对每一条根单独命名,从而获取更为精细的根系构型信息;此外,该软件对图片分辨率要求较低,支持多种图像格式,具有强大的硬件兼容性[57]。GT-RootS采用自动化分析模式,通过指定图像存放路径和输出路径,可实现批量处理(图2),并将量化分析结果自动保存在同一个文档中,极大地提高了根系图像的量化效率[48]。Root Hair Sizer则是一款专门用于检测根毛表型的软件,基于西格摩德(Sigmoidal)模型,可提取根毛长度,根分化区位置及根毛生长速率等参数[63]。
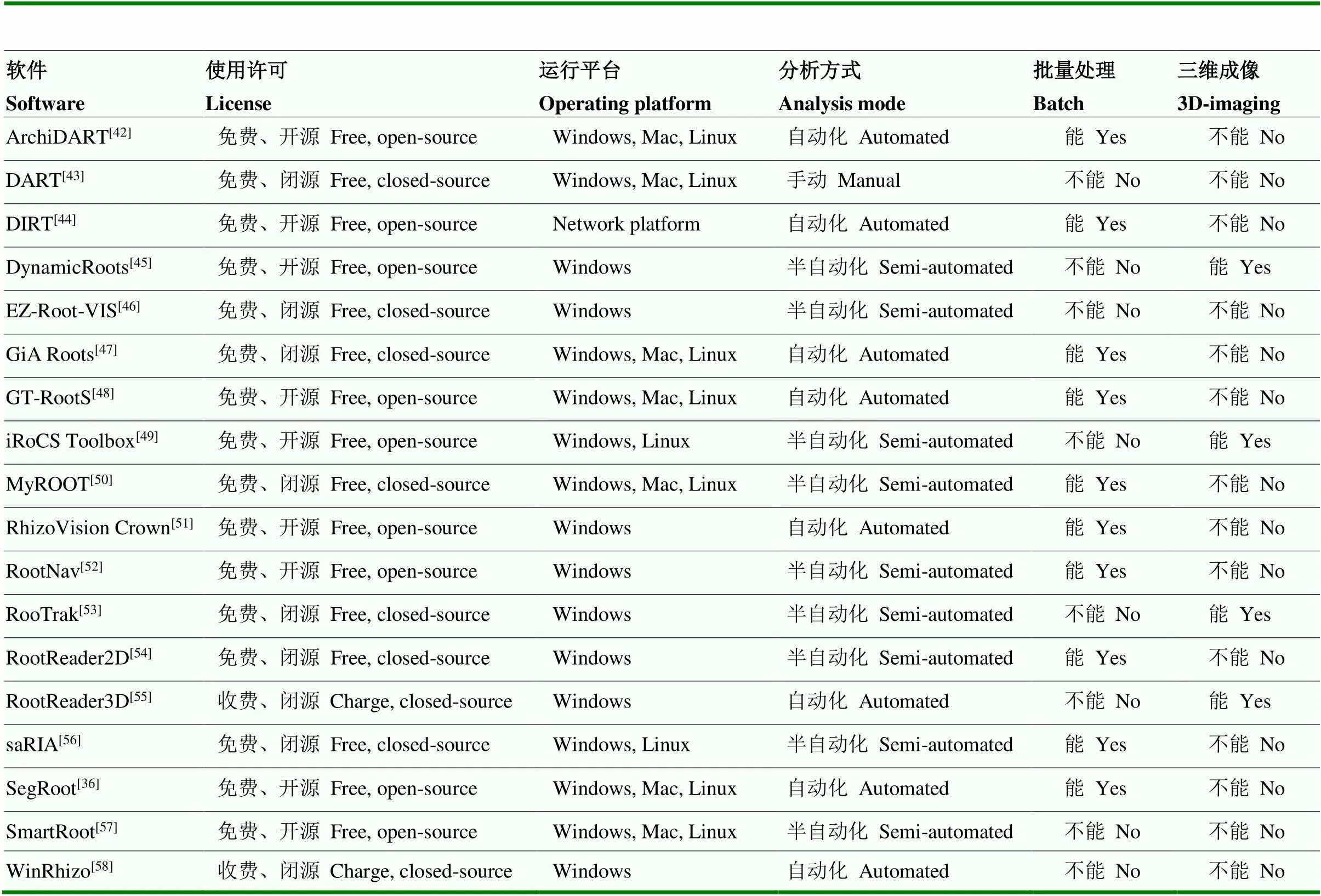
表2 常用根系表型分析软件
上述软件主要用于二维根系平面构型分析,而目前针对三维根系构型定量分析软件的研发相对薄弱。其主要原因在于提取三维根系构型参数不仅需要解读图像像素多少、颜色等级和尺寸大小,还需要构建空间分布函数,大大增加了软件设计难度;另外,用于分析三维根系构型的原始图像大多是由多视角相机系统、X射线计算机断层扫描仪及磁共振成像仪等昂贵硬件设备生成,软件用户群体较小[64]。RootReader3D、RooTrak及NMRooting是目前比较常用的三维根系表型分析软件。其中,Clark等[55]开发的RootReader3D软件可以利用多视角相机系统采集的根系图像创建三维根系模型,提取根角度、根表面积和根体积等27个量化参数。该软件仅适宜于分析背景单一的根系图像,不能消除图像中非根物质的影响,例如,生长于透明培养液中的水稻幼苗根系图像[65]。RooTrak是早期由Mairhofer等[53]开发的一款用于复杂介质背景下根系表型可视化的软件,主要用于分析X-ray CT法生成的图像,即将图像视为沿Z轴方向的一系列X-Y平面堆积图,通过构建多个局部模型并跟踪特定区段来完善根系网络结构,利用视觉跟踪框架的模型引导功能辨识由X射线衰减产生的模糊根系,从而呈现较为完整的三维根系构型图。然而,该软件分析用时较长且无法提取量化参数。Teramoto等[19]进一步针对X-ray CT开发了RSAvis3D根系图像量化技术,通过在X-ray CT扫描中使用较高的电压和电流来增加土壤背景和根系的对比度,并利用三维中值滤波和边缘检测算法提高对根系的辨识能力。在高性能计算机支持下,RSAvis3D用时10 min即可对单个样品完成高质量根系图像扫描和重建(33 s即可获取粗略根系图像),仅需2 min便可完成根系图像的量化分析。该技术初步实现了土壤背景下根系表型参数的高通量提取。此外,van Dusschoten等[20]基于Python语言开发的NMRooting软件能够借助功能强大的Mayavi可视化库及Igraph工具包对MRI技术所获取的根系图像进行量化分析。即便如此,现有的三维根系表型分析软件仍无法完全排除根系自身重叠性以及土壤中非根物质的影响,难以根据原始图像提取完整根系表型参数。为此,研究者开发了三维根系重建技术,即结合计算机模拟算法,推演与实际根系形态相似的根系三维几何模型。例如,POSTMA等[66]开发的OPENSIMROOT软件能够对X-ray CT和MRI技术所获取的根系图像建立模型,根据根系生长轨迹模拟出未显示的部分根系,从而提高三维根系构型的完整度。三维重建技术是根系表型鉴定的一个扩展领域,目前仍处于探索阶段,随着参数提取算法和数据融合方法的逐渐成熟,该技术必将助力实现立体根系构型的精准鉴定[11]。
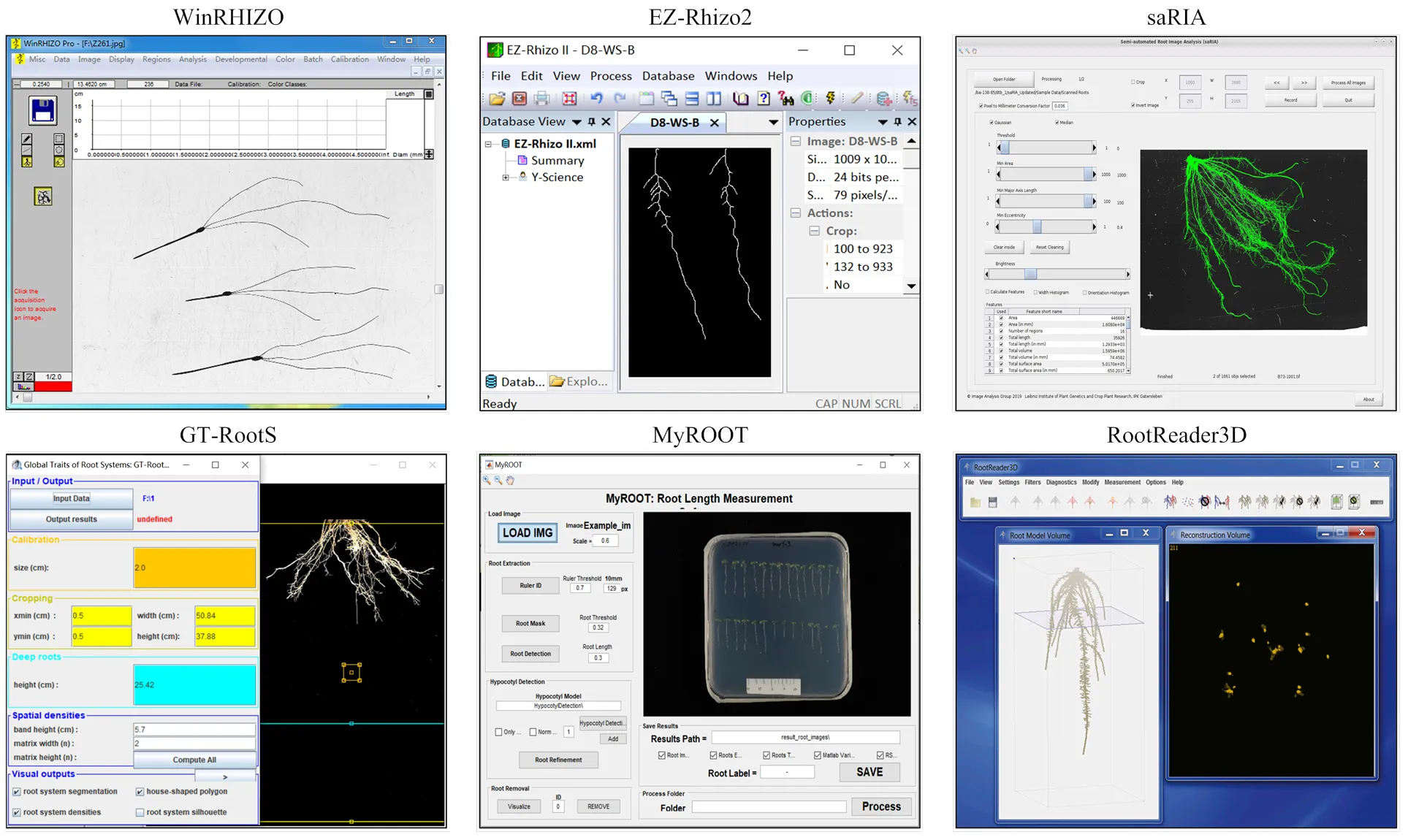
图2 常用根系表型分析软件用户界面
4 根系表型数据共享平台
利用多样化的根系分析软件,研究者们已获取海量根系图像及表型数据,其中不乏重复性的冗余信息,造成资源浪费;同时也隐藏着极具研究价值的信息,例如,不同生态环境下相同种质的根系表型数据可用于研究根系表型可塑性,进而分析作物对环境变化的响应。因此,亟需建立数据共享平台提高根系表型信息利用率。标准化数据存储格式是构建根系表型数据共享平台的基本前提。为此,Lobet等[67]设计开发了RSML(root system markup language)标准化根系表型数据格式,该格式基于XML(extentsible markup language)标准,可存储二维和三维根系表型的元数据、几何尺寸以及根系生长路径函数等内容,目前已成功应用于7个根系表型分析软件(EZ-Rhizo、GLO- RIA、RootNav、RhizoScan、Root System Analyser、RooTrak、SmartRoot),同时还分别针对Excel、R、Python、Image J和ArchiDART开发了软件扩展包用于数据解析,使其能够适应不同计算机系统或软件环境,实现了研究人员或机构之间的无缝协作。此外,Das等[44]在高性能计算集群的支持下开发了DIRT根系表型分析与共享平台(http://dirt. iplantcollaborative.org/),用户可将根系图像批量上传至该平台,快速获取多达70多种根系表型量化参数,同时该平台还允许数据的所有者共享、编辑、下载和删除已上传的根系图像或元数据。Lobet等[68]建立了Quantitative Plant植物表型量化平台(https://www. quantitative-plant.org/),该平台目前已汇集多达179种植物表型(包括根系及地上部表型)分析软件、31个表型数据库以及98种植物数字化模型,平台提供软件下载链接、用户反馈、数据共享及新软件发布等服务,帮助研究者们快速锁定最佳研究工具及方案,并及时分享研究心得。由此可见,根系表型数据共享平台建设已取得诸多进展,但在软件兼容性和数据积累方面仍有很大提升空间,今后应继续加快推进根系表型数据资源规范存储、共享开放和开发应用。
5 展望
作物表型鉴定是认识作物和培育新品种的基础。过去几十年,中国种质资源工作者已对大量作物种质进行了表型精准鉴定[69]。这些工作主要针对地上部性状,极少涉及根系,根系表型成为作物种质信息库中的重要缺口。根系表型鉴定的难点在于根系的隐匿性(生长介质阻碍直接观测)、复杂性(组织结构错综复杂)及可塑性(易受环境影响)[70]。因此,未来作物根系表型鉴定方法的创新仍将围绕这三个问题,重点开展以下工作:
5.1 开发高效的田间根系表型鉴定方法
目前,通过结合人工介质培养、光谱成像及自动化根系表型量化技术,已基本实现室内二维根系表型高通量精准鉴定[9]。以X-ray CT、MRI为代表的室内三维根系表型检测技术也取得了突破性进展,该技术今后所面临的挑战是实现自动化检测,使其通量媲美二维根系表型鉴定平台,以便进行大规模的作物根系表型研究[71]。相比而言,土壤的粘结性极大地影响着田间根系表型鉴定,利用自动化微根管监测平台和机器识别技术也仅能获取部分(附着管壁的)根系表型信息,尚未有田间环境下的高通量三维根系形态检测方案。因此,今后需加强根系原位探测技术和遥感观测技术研发,提高田间根系表型鉴定效率和动态观测能力,同时结合三维重建技术突破局部观测的限制,提取根系全局三维信息,实现田间根系表型的高效精准鉴定。
5.2 建立根系可塑性鉴定评价技术体系
作物在遭受生物和非生物胁迫后,其根系表现出生理、发育及形态变化,统称为根系可塑性[72]。例如,在低磷条件下,玉米冠状根数量、分布范围及侧根密度均显著增加(大),而在低氮条件下则显著减少(小)[72-73]。根系可塑性是作物对环境变化的最直接响应,如何提高根系应对环境变化的可塑性潜力已成为作物增产增效研究的热点问题[74]。目前,根系表型鉴定方法主要用以表征单一环境下的根系形态,缺乏根系可塑性的评价方法及判定依据。因此,今后应充分结合遗传和环境因素,建立根系可塑性鉴定评价技术体系。
5.3 加强根系解剖结构的鉴定和利用
根系解剖结构是根系发育状况的直接体现,如木质化程度、导管数量及表皮附属结构特征等均会影响根系的功能,进而影响作物生产力及适应环境变化的能力[75]。然而,由于根系解剖结构的观测需要借助专门仪器设备,技术门槛高且样品制备过程繁琐,现有观测技术难以快速提取根系解剖结构信息。因此,应探讨根系解剖结构高效鉴定策略,包括高通量的根系切片设备、自动化显微成像平台及根横截面的量化方法等[76],加强作物根系解剖结构的鉴定和利用。
5.4 强化分子检测技术在根系表型鉴定中的应用
等位基因(位于同源染色体的相同位置上具有不同DNA序列形式的基因)序列差异是决定作物品种表型多样性的本底差异[77]。近年来,随着测序技术和生物信息学的快速发展,等位基因发掘效率不断提升。目前在玉米、水稻和小麦等作物中已经相继分离鉴定到大量根系性状相关等位基因[78]。在此基础上,应开发实用性分子标记或育种芯片,为根系表型建立“分子指纹”,应用分子标记或全基因组选择辅助鉴定根系表型。
5.5 推进作物根系表型鉴定技术规范化和数据信息共享化
规范化和共享化是作物种质资源研究的发展趋势,也是开展规模化作物种质资源精准鉴定的重要前提[69]。目前,中国已针对不同作物制定了一系列的表型鉴定规范及评价标准,涉及农艺性状、生物胁迫抗性性状、非生物胁迫抗性性状及土壤养分利用性状等,但其中均缺乏根系表型相关内容[79]。因此,应统筹兼顾成本、通量及精准度,充分结合传统方法及新型技术,确定适用于不同作物的根系样本采集方法及鉴定流程,归纳根系表型性状量化参数,建立根系表型鉴定评价技术规范,构建参数化根系表型分类标准及共享数据库。
总之,未来通过显微成像、遥感观测、人工智能及大数据分析等技术的深度结合,将实现精准化、规模化、自动化和共享化根系表型鉴定,突破作物根系选择和遗传改良的技术瓶颈,深化对“理想根型”的认知和实践,通过根系改良提高作物产量潜力和适应性。
[1] TRACY S R, NAGEL K A, POSTMA J A, FASSBENDER H, WASSON A, WATT M. Crop improvement from phenotyping roots: Highlights reveal expanding opportunities. Trends in Plant Science, 2020, 25: 105-118.
[2] THORUP-KRISTENSEN K, HALBERG N, NICOLAISEN M, OLESEN J E, CREWS T E, HINSINGER P, KIRKEGAARD J, PIERRET A, DRESBOLL D B. Digging deeper for agricultural resources, the value of deep rooting. Trends in Plant Science, 2020, 25: 406-417.
[3] GREGORY P J, MCGOWAN M, BISCOE P V. Water relations of winter wheat. 2. Soil water relations. Journal of Agricultural Science, 1978, 91: 103-116.
[4] VAN DER BOM F J T, WILLIAMS A, BELL M J. Root architecture for improved resource capture: Trade-offs in complex environments. Journal of Experimental Botany, 2020, 71: 5752-5763.
[5] SUN B, GAO Y, LYNCH J P. Large crown root number improves topsoil foraging and phosphorus acquisition. Plant Physiology, 2018, 177: 90-104.
[6] LYNCH J P. Root phenotypes for improved nutrient capture: An underexploited opportunity for global agriculture. New Phytologist, 2019, 223: 548-564.
[7] BAILEY-SERRES J, PARKER J E, AINSWORTH E A, OLDROYD G E D, SCHROEDER J I. Genetic strategies for improving crop yields. Nature, 2019, 575: 109-118.
[8] LI L, PENG Z, MAO X G, WANG J Y, CHANG X P, REYNOLDS M P, JING R L. Genome-wide association study reveals genomic regions controlling root and shoot traits at late growth stages in wheat. Annals of Botany, 2019, 124: 993-1006.
[9] ATKINSON J A, POUND M P, BENNETT M J, WELLS D M. Uncovering the hidden half of plants using new advances in root phenotyping. Current Opinion in Biotechnology, 2019, 55: 1-8.
[10] BEKKERING C S, HUANG J, TIAN L. Image-based, organ-level plant phenotyping for wheat improvement. Agronomy-Basel, 2020, 10: 1287.
[11] TAKAHASHI H, PRADAL C. Root phenotyping: Important and minimum information required for root modeling in crop plants. Breeding Science, 2021, 71: 109-116.
[12] WASSON A P, NAGEL K A, TRACY S, WATT M. Beyond digging: Noninvasive root and rhizosphere phenotyping. Trends in Plant Science, 2020, 25: 119-120.
[13] WALLER S, WILDER S L, SCHUELLER M J, HOUSH A B, FERRIERI R A. Quantifying plant-borne carbon assimilation by root-associating bacteria. Microorganisms, 2020, 8: 700.
[14] LIU X L, LI R Z, CHANG X P, JING R L. Mapping QTLs for seedling root traits in a doubled haploid wheat population under different water regimes. Euphytica, 2013, 189: 51-66.
[15] RICHARD C A I, HICKEY L T, FLETCHER S, JENNINGS R, CHENU K, CHRISTOPHER J T. High-throughput phenotyping of seminal root traits in wheat. Plant Methods, 2015, 11: 13.
[16] RICH S M, CHRISTOPHER J, RICHARDS R, WATT M. Root phenotypes of young wheat plants grown in controlled environments show inconsistent correlation with mature root traits in the field. Journal of Experimental Botany, 2020, 71: 4751-4762.
[17] PAEZ-GARCIA A, MOTES C M, SCHEIBLE W R, CHEN R J, BLANCAFLOR E B, MONTEROS M J. Root traits and phenotyping strategies for plant improvement. Plants, 2015, 4: 334-355.
[18] CORONA-LOPEZ D D J, SOMMER S, ROLFE S A, PODD F, GRIEVE B D. Electrical impedance tomography as a tool for phenotyping plant roots. Plant Methods, 2019, 15: 49.
[19] TERAMOTO S, TAKAYASU S, KITOMI Y, ARAI-SANOH Y, TANABATA T, UGA Y. High-throughput three-dimensional visualization of root system architecture of rice using X-ray computed tomography. Plant Methods, 2020, 16: 66.
[20] VAN DUSSCHOTEN D, METZNER R, KOCHS J, POSTMA J A, PFLUGFELDER D, BUEHLER J, SCHURR U, JAHNKE S. Quantitative 3D analysis of plant roots growing in soil using magnetic resonance imaging. Plant Physiology, 2016, 170: 1176-1188.
[21] ROGERS E D, MONAENKOVA D, MIJAR M, NORI A, GOLDMAN D I, BENFEY P N. X-ray computed tomography reveals the response of root system architecture to soil texture. Plant Physiology, 2016, 171: 2028-2040.
[22] BAO Y, AGGARWAL P, ROBBINS N E, STURROCK C J, THOMPSON M C, TAN H Q, THAM C, DUAN L, RODRIGUEZ P L, VERNOUX T, MOONEY S J, BENNETT M J, DINNENY J R. Plant roots use a patterning mechanism to position lateral root branches toward available water. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111: 9319-9324.
[23] PFLUGFELDER D, METZNER R, VAN DUSSCHOTEN D, REICHEL R, JAHNKE S, KOLLER R. Non-invasive imaging of plant roots in different soils using magnetic resonance imaging (MRI). Plant Methods, 2017, 13: 102.
[24] METZNER R, EGGERT A, VAN DUSSCHOTEN D, PFLUGFELDER D, GERTH S, SCHURR U, UHLMANN N, JAHNKE S. Direct comparison of MRI and X-ray CT technologies for 3D imaging of root systems in soil: Potential and challenges for root trait quantification. Plant Methods, 2015, 11: 17.
[25] JAHNKE S, MENZEL M I, VAN DUSSCHOTEN D, ROEB G W, BUEHLER J, MINWUYELET S, BLUEMLER P, TEMPERTON V M, HOMBACH T, STREUN M, BEER S, KHODAVERDI M, ZIEMONS K, COENEN H H, SCHURR U. Combined MRI-PET dissects dynamic changes in plant structures and functions. The Plant Journal, 2009, 59: 634-644.
[26] OYIGA B C, PALCZAK J, WOJCIECHOWSKI T, LYNCH J P, NAZ A A, LEON J, BALLVORA A. Genetic components of root architecture and anatomy adjustments to water-deficit stress in spring barley. Plant, Cell & Environment, 2020, 43: 692-711.
[27] ZHENG Z, HEY S, JUBERY T, LIU H, YANG Y, COFFEY L, MIAO C, SIGMON B, SCHNABLE J C, HOCHHOLDINGER F, GANAPATHYSUBRAMANIAN B, SCHNABLE P S. Shared genetic control of root system architecture betweenand. Plant Physiology, 2020, 182: 977-991.
[28] WASSON A P, REBETZKE G J, KIRKEGAARD J A, CHRISTOPHER J, RICHARDS R A, WATT M. Soil coring at multiple field environments can directly quantify variation in deep root traits to select wheat genotypes for breeding. Journal of Experimental Botany, 2014, 65: 6231-6249.
[29] KITOMI Y, HANZAWA E, KUYA N, INOUE H, HARA N, KAWAI S, KANNO N, ENDO M, SUGIMOTO K, YAMAZAKI T, SAKAMOTO S, SENTOKU N, WU J, KANNO H, MITSUDA N, TORIYAMA K, SATO T, UGA Y. Root angle modifications by thehomolog improve rice yields in saline paddy fields. Proceedings of the National Academy of Sciences of the United States of America, 2020, 117: 21242-21250.
[30] VOSS-FELS K P, ROBINSON H, MUDGE S R, RICHARD C, NEWMAN S, WITTKOP B, STAHL A, FRIEDT W, FRISCH M, GABUR I, MILLER-COOPER A, CAMPBELL B C, KELLY A, FOX G, CHRISTOPHER J, CHRISTOPHER M, CHENU K, FRANCKOWIAK J, MACE E S, BORRELL A K, EAGLES H, JORDAN D R, BOTELLA J R, HAMMER G, GODWIN I D, TREVASKIS B, SNOWDON R J, HICKEY L T.modulates root system architecture in wheat and barley. Molecular Plant, 2018, 11: 226-229.
[31] LOU Q, CHEN L, MEI H, WEI H, FENG F, WANG P, XIA H, LI T, LUO L. Quantitative trait locus mapping of deep rooting by linkage and association analysis in rice. Journal of Experimental Botany, 2015, 66: 4749-4757.
[32] UGA Y, SUGIMOTO K, OGAWA S, RANE J, ISHITANI M, HARA N, KITOMI Y, INUKAI Y, ONO K, KANNO N, INOUE H, TAKEHISA H, MOTOYAMA R, NAGAMURA Y, WU J, MATSUMOTO T, TAKAI T, OKUNO K, YANO M. Control of root system architecture byincreases rice yield under drought conditions. Nature Genetics, 2013, 45: 1097-1102.
[33] BATES G H. A device for the observation of root growth in the soil. Nature, 1937, 139: 966-967.
[34] MAJDI H. Root sampling methods - applications and limitations of the minirhizotron technique. Plant and Soil, 1996, 185: 255-258.
[35] SVANE S F, DAM E B, CARSTENSEN J M, THORUP- KRISTENSEN K. A multispectral camera system for automated minirhizotron image analysis. Plant and Soil, 2019, 441: 657-672.
[36] WANG T, ROSTAMZA M, SONG Z, WANG L, MCNICKLE G, IYER-PASCUZZI A S, QIU Z, JIN J. Segroot: A high throughput segmentation method for root image analysis. Computers and Electronics in Agriculture, 2019, 162: 845-854.
[37] ALANI A M, LANTINI L. Recent advances in tree root mapping and assessment using non-destructive testing methods: A focus on ground penetrating radar. Surveys in Geophysics, 2020, 41: 605-646.
[38] STREDA T, HABERLE J, KLIMESOVA J, KLIMEK-KOPYRA A, STREDOVA H, BODNER G, CHLOUPEK O. Field phenotyping of plant roots by electrical capacitance-a standardized methodological protocol for application in plant breeding: A review. International Agrophysics, 2020, 34: 173-184.
[39] WASSON A P, RICHARDS R A, CHATRATH R, MISRA S C, PRASAD S V S, REBETZKE G J, KIRKEGAARD J A, CHRISTOPHER J, WATT M. Traits and selection strategies to improve root systems and water uptake in water-limited wheat crops. Journal of Experimental Botany, 2012, 63: 3485-3498.
[40] LI X, INGVORDSEN C H, WEISS M, REBETZKE G J, CONDON A G, JAMES R A, RICHARDS R A. Deeper roots associated with cooler canopies, higher normalized difference vegetation index, and greater yield in three wheat populations grown on stored soil water. Journal of Experimental Botany, 2019, 70: 4963-4974.
[41] JIN X, ZARCO-TEJADA P J, SCHMIDHALTER U, REYNOLDS M P, HAWKESFORD M J, VARSHNEY R K, YANG T, NIE C, LI Z, MING B, XIAO Y, XIE Y, LI S. High-throughput estimation of crop traits: A review of ground and aerial phenotyping platforms. IEEE Geoscience and Remote Sensing Magazine, 2021, 9: 200-231.
[42] DELORY B M, LI M, TOPP C N, LOBET G. Archidart v3.0: A new data analysis pipeline allowing the topological analysis of plant root systems version 1; referees: 2 approved, 1 approved with reservations. F1000Research, 2018, 7: 22.
[43] LE BOT J, SERRA V, FABRE J, DRAYE X, ADAMOWICZ S, PAGES L. Dart: A software to analyse root system architecture and development from captured images. Plant and Soil, 2010, 326: 261-273.
[44] DAS A, SCHNEIDER H, BURRIDGE J, ASCANIO A K M, WOJCIECHOWSKI T, TOPP C N, LYNCH J P, WEITZ J S, BUCKSCH A. Digital imaging of root traits (DIRT): A high- throughput computing and collaboration platform for field-based root phenomics. Plant Methods, 2015, 11: 51.
[45] SYMONOVA O, TOPP C N, EDELSBRUNNER H. Dynamicroots: A software platform for the reconstruction and analysis of growing plant roots. PLoS One, 2015, 10: e0127657.
[46] SHAHZAD Z, KELLERMEIER F, ARMSTRONG E M, ROGERS S, LOBET G, AMTMANN A, HILLS A. EZ-root-VIS: A software pipeline for the rapid analysis and visual reconstruction of root system architecture. Plant Physiology, 2018, 177: 1368-1381.
[47] GALKOVSKYI T, MILEYKO Y, BUCKSCH A, MOORE B, SYMONOVA O, PRICE C A, TOPP C N, IYER-PASCUZZI A S, ZUREK P R, FANG S, HARER J, BENFEY P N, WEITZ J S. GiA roots: Software for the high throughput analysis of plant root system architecture. BMC Plant Biology, 2012, 12: 116.
[48] BORIANNE P, SUBSOL G, FALLAVIER F, DARDOU A, AUDEBERT A. GT-RootS: An integrated software for automated root system measurement from high-throughput phenotyping platform images. Computers and Electronics in Agriculture, 2018, 150: 328-342.
[49] SCHMIDT T, PASTERNAK T, LIU K, BLEIN T, AUBRY-HIVET D, DOVZHENKO A, DUERR J, TEALE W, DITENGOU F A, BURKHARDT H, RONNEBERGER O, PALME K. The iRoCS Toolbox-3D analysis of the plant root apical meristem at cellular resolution. The Plant Journal, 2014, 77: 806-814.
[50] GONZALEZ A, SEVILLANO X, BETEGON-PUTZE I, BLASCO- ESCAMEZ D, FERRER M, CANO-DELGADO A I. MYROOT 2.0: An automatic tool for high throughput and accurate primary root length measurement. Computers and Electronics in Agriculture, 2020, 168: 105125.
[51] SEETHEPALLI A, GUO H, LIU X, GRIFFITHS M, ALMTARFI H, LI Z, LIU S, ZARE A, FRITSCHI F B, BLANCAFLOR E B, MA X, YORK L M. RhizoVision Crown: An integrated hardware and software platform for root crown phenotyping. Plant Phenomics, 2020, 2020: 3074916.
[52] POUND M P, FRENCH A P, ATKINSON J A, WELLS D M, BENNETT M J, PRIDMORE T. RootNav: Navigating images of complex root architectures. Plant Physiology, 2013, 162: 1802-1814.
[53] MAIRHOFER S, ZAPPALA S, TRACY S R, STURROCK C, BENNETT M, MOONEY S J, PRIDMORE T. RooTrak: Automated recovery of three-dimensional plant root architecture in soil from X-ray microcomputed tomography images using visual tracking. Plant Physiology, 2012, 158: 561-569.
[54] CLARK R T, FAMOSO A N, ZHAO K, SHAFF J E, CRAFT E J, BUSTAMANTE C D, MCCOUCH S R, ANESHANSLEY D J, KOCHIAN L V. High-throughput two-dimensional root system phenotyping platform facilitates genetic analysis of root growth and development. Plant, Cell & Environment, 2013, 36: 454-466.
[55] CLARK R T, MACCURDY R B, JUNG J K, SHAFF J E, MCCOUCH S R, ANESHANSLEY D J, KOCHIAN L V. Three-dimensional root phenotyping with a novel imaging and software platform. Plant Physiology, 2011, 156: 455-465.
[56] NARISETTI N, HENKE M, SEILER C, SHI R, JUNKER A, ALTMANN T, GLADILIN E. Semi-automated root image analysis (saRIA). Scientific Reports, 2019, 9: 19674.
[57] LOBET G, PAGES L, DRAYE X. A novel image-analysis toolbox enabling quantitative analysis of root system architecture. Plant Physiology, 2011, 157: 29-39.
[58] PANG W, CROW W T, LUC J E, MCSORLEY R, GIBLIN-DAVIS R M, KENWORTHY K E, KRUSE J K. Comparison of water displacement and WINRHIZO software for plant root parameter assessment. Plant Disease, 2011, 95: 1308-1310.
[59] BEYER S, DABA S, TYAGI P, BOCKELMAN H, BROWN- GUEDIRA G, MOHAMMADI M, IWGSC. Loci and candidate genes controlling root traits in wheat seedlings - a wheat root GWAS. Functional & Integrative Genomics, 2019, 19: 91-107.
[60] FERNANDO K M C, EHOCHE O G, ATKINSON J A, SPARKES D L. Root system architecture and nitrogen uptake efficiency of wheat species. Journal of Agricultural Sciences, 2021, 16: 37-53.
[61] VESCIO R, ABENAVOLI M R, SORGONA A. Single and combined abiotic stress in maize root morphology. Plants-Basel, 2021, 10: 5.
[62] YASRAB R, ATKINSON J A, WELLS D M, FRENCH A P, PRIDMORE T P, POUND M P. RootNav 2.0: Deep learning for automatic navigation of complex plant root architectures. Gigascience, 2019, 8: giz123.
[63] GUICHARD M, ALLAIN J M, BIANCHI M W, FRACHISSE J M. Root hair sizer: An algorithm for high throughput recovery of different root hair and root developmental parameters. Plant Methods, 2019, 15: 104.
[64] PAULUS S. Measuring crops in 3D: Using geometry for plant phenotyping. Plant Methods, 2019, 15: 103.
[65] UGA Y, ASSARANURAK I, KITOMI Y, LARSON B G, CRAFT E J, SHAFF J E, MCCOUCH S R, KOCHIAN L V. Genomic regions responsible for seminal and crown root lengths identified by 2D & 3D root system image analysis. BMC Genomics, 2018, 19: 273.
[66] POSTMA J A, KUPPE C, OWEN M R, MELLOR N, GRIFFITHS M, BENNETT M J, LYNCH J P, WATT M. OPENSIMROOT: Widening the scope and application of root architectural models. New Phytologist, 2017, 215: 1274-1286.
[67] LOBET G, POUND M P, DIENER J, PRADAL C, DRAYE X, GODIN C, JAVAUX M, LEITNER D, MEUNIER F, NACRY P, PRIDMORE T P, SCHNEPF A. Root system markup language: Toward a unified root architecture description language. Plant Physiology, 2015, 167: 617-627.
[68] LOBET G, DRAYE X, PÉRILLEUX C. An online database for plant image analysis software tools. Plant Methods, 2013, 9: 38.
[69] 刘旭, 李立会, 黎裕, 方沩. 作物种质资源研究回顾与发展趋势. 农学学报, 2018, 8: 1-6.
LIU X, LI L H, LI Y, FANG W. Crop germplasm resources: Advances and trends. Journal of Agriculture, 2018, 8: 1-6. (in Chinese)
[70] LI C N, LI L, REYNOLDS M P, WANG J Y, CHANG X P, MAO X G, JING R L. Recognizing the hidden half in wheat: Root system attributes associated with drought tolerance. Journal of Experimental Botany, 2021, 72: 5117-5133.
[71] PATURKAR A, SEN GUPTA G, BAILEY D. Making use of 3D models for plant physiognomic analysis: A review. Remote Sensing, 2021, 13: 2232.
[72] GAO K, CHEN F, YUAN L, ZHANG F, MI G. A comprehensive analysis of root morphological changes and nitrogen allocation in maize in response to low nitrogen stress. Plant, Cell & Environment, 2015, 38: 740-750.
[73] VYSOTSKAYA L, AKHIYAROVA G, FEOKTISTOVA A, AKHTYAMOVA Z, KOROBOVA A, IVANOV I, DODD I, KULUEV B, KUDOYAROVA G. Effects of phosphate shortage on root growth and hormone content of barley depend on capacity of the roots to accumulate ABA. Plants-Basel, 2020, 9: 1722.
[74] SCHNEIDER H M, LYNCH J P. Should root plasticity be a crop breeding target? Frontiers in Plant Science, 2020, 11: 546.
[75] SIDDIQUI M N, LEON J, NAZ A A, BALLVORA A. Genetics and genomics of root system variation in adaptation to drought stress in cereal crops. Journal of Experimental Botany, 2021, 72: 1007-1019.
[76] WANG C, LI X, CARAGEA D, BHEEMANAHALLIA R, JAGADISH S V K. Root anatomy based on root cross-section image analysis with deep learning. Computers and Electronics in Agriculture, 2020, 175: 105549.
[77] 武晶, 黎裕. 基于作物种质资源的优异等位基因挖掘: 进展与展望. 植物遗传资源学报, 2019, 20: 1380-1390.
WU J, LI Y. Mining superior alleles in crop germplasm resources: Advances and perspectives. Journal of Plant Genetic Resources, 2019, 20: 1380-1390. (in Chinese)
[78] DEJA-MUYLLE A, PARIZOT B, MOTTE H, BEECKMAN T. Exploiting natural variation in root system architecture via genome- wide association studies. Journal of Experimental Botany, 2020, 71: 2379-2389.
[79] 刘旭. 四十年改革开放几代人梦想成真——记中国作物种质资源40年发展巨变. 中国种业, 2019, 1: 1-6.
LIU X. Forty years of reform and opening-up and several generations' dreams come true-Recording the great changes in the development of crop germplasm resources in China in the past 40 years. China Seed Industry, 2019, 1: 1-6. (in Chinese)
Advances and Perspectives of Approaches to Phenotyping Crop Root System
LI Long, LI ChaoNan, MAO XinGuo, WANG JingYi, JING RuiLian*
Institute of Crop Sciences, Chinese Academy of Agricultural Sciences/National Key Facility for Crop Gene Resources and Genetic Improvement, Beijing 100081
Roots are the vital organs for fixing the plant shoots and absorbing soil water and nutrients. The phenotypic characteristics of roots directly affect crop productivity and adaptability. Optimizing root phenotypes is considered to be one of the important ways to achieve the second “Green Revolution”. However, the invisibility, complexity and plasticity of root system greatly restrict the efficiency of root phenotyping, which makes the root optimization process lag far behind that of aboveground organs. With the rapid development of new technologies, i.e. spectral imaging, machine learning and three-dimensional reconstruction, the approaches to phenotyping roots gradually changed from traditional sampling observation to in-situ, nondestructive and automatic detection, and the evaluation basis expanded from two-dimensional morphological indices to three-dimensional parameters, which promoted the efficiency of root phenotyping and dramatically enriched the data of root phenotype. Meanwhile, the massive data exhibited problems, such as data redundancy and low use efficiency of information resources, which put forward new requirements, i.e. standardization and shareability, for root phenotype studies. This paper summarized the principles and technical keys of main approaches to phenotyping roots, and compared systematically in terms of precision, cost and throughput. The commonly used software for quantification of root phenotype were listed out from the aspects of license, operating platform, analysis mode and so on. The important research direction in the future was put forward, that is, to develop effective approaches to phenotyping roots in the field, to establish the evaluation system for root plasticity, to strengthen the identification and utilization of root anatomical characters, to strengthen the application of molecular detection techniques in root phenotyping, and to promote standardization of root phenotyping techniques and data sharing. The aim is to provide reference for the reasonable selection and improvement of approaches to phenotyping crop root system, so as to promote crop root improvement.
crop; root phenotype; phenotyping in the laboratory; phenotyping in the field; evaluation method
2021-07-21;
2021-08-09
国家重点研发计划项目(2017YFD0300202)、中国农业科学院科技创新工程重大科研任务(CAAS-ZDRW202002)
李龙,E-mail:lilong01@caas.cn。通信作者景蕊莲,E-mail:jingruilian@caas.cn
(责任编辑 李莉)

