微生物群落对氨胁迫响应的宏基因组学研究
彭 韵,李 蕾,伍 迪,杨屏锦,彭绪亚,王小铭
微生物群落对氨胁迫响应的宏基因组学研究
彭 韵,李 蕾*,伍 迪,杨屏锦,彭绪亚,王小铭
(重庆大学三峡库区生态环境教育部重点实验室,重庆 400045)
为探析厌氧消化微生物群落对氨胁迫的响应,在串联批次实验中引入氨氮(TAN)胁迫,结合宏基因组学分析,研究了不同TAN浓度下,厌氧消化系统的过程参数、群落结构/组成及其功能基因/代谢途径的响应.结果显示,TAN为3000mg/L时,系统性能逐步优化;TAN为6000mg/L时,甲烷产率逐步下降,并伴随着丙酸、丁酸积累.从群落组成上看,两个氨氮梯度下,抗性和冗余性保障了水解酸化菌代谢底物的能力.产氢产乙酸和产甲烷方面,低氨氮组两类功能菌群丰度持续增加,但高氨氮组增加幅度低甚至出现丰度下降,指示该环节可能受到了氨胁迫.进一步分析两阶段的功能基因变化,发现产甲烷和互营乙酸氧化途径在氨胁迫下表现出抗性和冗余性,但高浓度TAN(6000mg/L)抑制了丁酸和丙酸代谢途径中关键基因和的表达.可见,氨抑制失稳的关键环节是产氢产乙酸阶段.
餐厨垃圾;厌氧消化;氨抑制;宏基因组学
厌氧消化技术能将有机废物转化为沼气,但是厌氧消化易遭遇过程失稳,其中氨抑制是全规模厌氧消化系统中主要的失稳诱因[1-2].不少研究者探索了氨胁迫下微生物群落结构的响应,早期研究普遍认为水解酸化细菌受氨氮(TAN)的影响较少[3-6];但氨胁迫会强烈抑制产甲烷菌,其中乙酸型产甲烷菌,如,对氨氮尤其敏感,相比之下,混合营养型的耐受性强得多[6-9].氨胁迫会使反应器内的主导甲烷菌由向演替[10-11].也有研究者着重关注氨胁迫对产甲烷代谢途径的影响,通过同位素检测发现,与低氨氮反应器以乙酸营养型产甲烷途径为主不同,高氨胁迫反应器的产甲烷途径多以互营乙酸氧化(SAO)联合氢营养型产甲烷途径(HM)为主[9].但矛盾的是,有些反应器已经是主导,依然在3500mg/L左右的氨氮下就出现失稳[12].更有研究者直接用互营乙酸氧化细菌(SAOB)进行生物强化,却发现根本无法改善氨抑制现象[13].
由于氨抑制失稳的反应器往往伴随着丙酸、丁酸等脂肪酸的大量积累[12],研究者逐步意识到产氢产乙酸阶段受氨抑制的程度可能甚于产甲烷阶段[14-17].如Li等[14]在反应器中塑造氨胁迫环境(TAN 为2500mg/L),并以丙酸为唯一碳源,发现丙酸降解菌(e和)相对丰度减少,导致丙酸积累,但少量丙酸降解产生的乙酸却可被立即转化为沼气.Zhang等[17]直接对比了氨胁迫下,产氢产乙酸菌和产甲烷菌相对丰度的变化,发现产氢产乙酸菌(和)对高浓度氨氮(6000mg/L)非常敏感,连续高氨氮暴露下,其丰度不能恢复并且没有被具有相似功能的丙酸降解菌代替,相比之下,暴露于高浓度氨氮,乙酸型产甲烷菌出现了主导菌的演替,氢型产甲烷菌表现出抗性,维持了群落产甲烷功能.Peng等[12]开展了长期的餐厨垃圾干式厌氧消化实验,发现随着内源氨积累,乙酸在积累一段时间后能够稳定到一个特定水平,相比之下,丙酸和戊酸却在乙酸停止积累后呈现更为迅猛的积累,且最终造成了反应器不可逆的抑制.同时,微生物交互分析证实产氢产乙酸菌()与TAN负相关.
目前对氨抑制失稳机理研究中微生物的测定一般采用的是高通量测序技术,这种方法虽然能较为准确地识别出系统中的微生物类别,却无法分析微生物的功能.因此,对群落功能的描述只能根据其他文献中报道的亲缘关系较为接近的已鉴定物种的功能进行推断[18],而实际上不同微生物的功能存在极大差异[10],因此该方法准确度有限.宏基因组学技术对环境中的全基因组进行测序,不仅可以全面地分析微生物群落具有的功能及代谢途径,使得反应器性能、微生物群落结构和功能/代谢特征之间的相关性研究成为可能,还有助于验证或者解释通过16S rRNA方法获得的关于氨胁迫对群落组成的影响机理,以进一步明确氨抑制失稳根源.采用宏基因组学研究氨抑制失稳机理的研究鲜有报道.基于此,本研究开展了串联批次实验,基于宏基因组学技术观察氨胁迫下,微生物群落结构/分类学组成及其功能基因/代谢途径的响应.本研究的目的是找出氨抑制失稳的关键环节,并从功能基因层面解析该环节受到扰动的原因,为解析氨抑制失稳提供参考.
1 材料与方法
1.1 接种物和底物
接种污泥来自实验室稳定运行的中温餐厨垃圾厌氧消化器,使用前进行预孵化去气.接种污泥的pH值、总固体含量(TS)、挥发性固体含量(VS)和TAN分别为7.89±0.2、(2.73±0.26)%、(2.04±0.07)%和(1723.43±166.78)mg/L.为保证底物的均匀性和实验结果的可靠性,采用根据实际餐厨垃圾性质配制的模化垃圾(湿基质量比,蔬菜:肉:米饭:植物油=35%:8%:50%:7%)为底物,模化垃圾分装到自封袋内于-18℃冰冻保存.临用前1d置于4℃冰箱中解冻.模化垃圾的TS、VS分别为(27.70±0.19)%、(27.54±0.14)%;蛋白质及粗脂肪含量分别为(17.21±0.63)%和(23.32±1.88)%;理论甲烷产率为(507.43±7.31)mLCH4/gVS.
1.2 反应器及运行
根据本团队前期研究,TAN为3000,6000mg/L时反应器性能都会受到扰动,但TAN为3000mg/L的实验组在连续批次实验中系统性能可完全恢复,TAN为6000mg/L的实验组性能则不断恶化[17],因此,本研究针对这2个浓度进行研究.
共开展3批次实验.第1批次实验在总容积1L,有效容积800mL的反应器(R1_3000)内进行.运行至日产气量小于累计产气量的1%时停止,继续孵化至完全不产气后开展第2批次实验.第2批次以R1_3000的消化渣作为接种物,R1_3000的消化渣被均分为2份,其中1份消化渣被暴露在原来的TAN浓度下(R2_3000),而另1份则被暴露至6000mg/L的TAN浓度下消化(R2_6000).第2批次实验中各反应器产气完成后,以反应器内全部消化渣作为接种物继续开展第3批次实验(R3_3000和R3_6000).为了保证各批次反应器中污泥浓度及有效容积的一致性,2、3批次实验在总容积500mL,有效容积400mL的反应器内开展.
所有反应器均调节接种污泥浓度为5gVSS/L,底物与接种物浓度比设置为1.氨氮以NH4Cl溶液的形式添加,随后向每个反应器内添加20%(体积比)的营养液[17],并添加蒸馏水达到有效容积.反应器内的初始pH值采用1mol/L的HCl溶液或3mol/L的NaOH溶液调至7.5.向反应器内充5min氮气,以排空瓶内的氧气,保证厌氧环境.反应器置于(36±1)℃的恒温水浴锅内发酵,每次记录气体体积前手动摇晃1min.
在每个批次反应末期,取液体样品,每次采样后立即在4℃下以10000r/min的转速冷冻离心15min,获得的上清液用于分析理化指标,污泥样品则保存至-80℃冰箱中,以备进行宏基因组学分析.
1.3 性能参数分析
产生的甲烷通过排NaOH(3mol/L)法进行收集,并换算成标态下(273.15K,101.325kPa)的体积[19]. pH值、TS、VS和TAN采用国家标准方法测定[20].挥发性脂肪酸(VFAs)(乙酸、丙酸、丁酸)用气相色谱(7890A,Agilent,美国)测定.底物的蛋白质和脂肪含量分别采用凯氏定氮法(KDN-1,上海)和索氏提取法测定[21-22].碳水化合物用差减法计算[12].根据式(1)将获得的底物组分用于理论甲烷产率计算[23]:
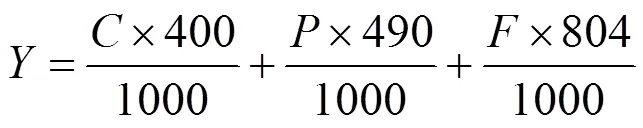
式中:是理论甲烷产率, mLCH4/gVS;是底物中碳水化合物含量, mg/gTS;是底物中蛋白质含量, mg/gTS;是底物中脂肪含量, mg/gTS.
1.4 动力学模型
考虑到餐厨垃圾极易降解,反应器启动初期底物被快速水解酸化产生大量VFAs,继而转化为甲烷,可使反应器出现第1个产气高峰.而本研究向各反应器中引入了氨氮扰动,氨氮逐步扩散至微生物细胞内,会对其活性产生抑制,由此迅速引发产气下降甚至停滞;直至微生物适应后,系统才会恢复产气,慢慢呈现第2个产气高峰.鉴于此,采用origin 9.3用含有前后两个产气高峰和中间产气平台的修正的两级Gompertz模型[24]对各反应器的产气情况进行模拟.根据模拟结果,各实验组实际甲烷产率和模拟甲烷产率的RMSE极小,2极高(0.99~1.00),表明该模型模拟效果很好.模型方程见式(2).

式中:Y是最终甲烷产率, mLCH4/gVS;Y1和Y2分别是第1阶段和第2阶段的甲烷产率, mLCH4/gVS;R1和R2分别是第1阶段和第2阶段的比产甲烷活性, mLCH4/(gVS·d);1和2分别是第1阶段和第2阶段的停滞期, d;e为自然对数,等于2.718.
1.5 宏基因组学分析方法
用FastDNA®Spin Kit for Soil试剂盒进行样品DNA抽提.分别利用TBS-380、NanoDrop200、1%琼脂糖凝胶电泳检测DNA浓度、纯度和完整性.通过Covaris M220将DNA片段化,筛选约400bp的片段,使用NEXTFLEX Rapid DNA-Seq构建PE文库.通过上海美吉生物技术有限公司Illumina NovaSeq测序平台进行宏基因组测序,原始数据已提交至NCBI数据库(序列号:SRP231897).
每个样品获得94440174~123356644条原始序列.利用fastp(v0.20.0)对原始序列进行质控,去除长度小于50bp、平均碱基质量值低于20以及含N碱基的序列,获得优化序列.使用基于succinct de Bruijn graphs原理的拼接软件Megahit(v1.1.2)对优化序列进行拼接组装,在拼接结果中筛选³300bp的重叠群作为最终的组装结果.使用MetaGene对拼接结果中的重叠群进行基因预测.选择核酸长度³100bp的基因,并将其翻译为氨基酸序列.使用CD-HIT(v4.6.1)对所有样品预测出来的基因序列进行聚类(相似度³90%、覆盖率³90%),每类取最长的基因作为代表序列,构建非冗余基因集.使用SOAPaligner(v2.21),分别将每个样品的高质量序列与非冗余基因集进行比对(相似度³95%),统计基因在对应样品中的丰度信息.基因丰度计算方法为Reads Per Kilobase Million,即每1百万条序列中,每个基因以1000个碱基为单位,比对上的序列条数.
采用基于bray_curtis距离算法的样本层级聚类分析计算了微生物样本之间的相似度.使用Diamond (v0.8.35)将非冗余基因集的氨基酸序列与NR数据库和KEGG数据库进行比对(BLAST比对参数设置期望值e-value为1e-5),以获得物种和功能注释.为识别在两样本组中具有显著差异的微生物,基于费舍尔精确检验和fdr多重检验校正对两样本进行了组间差异检验,设置值为0.05.
2 结果与讨论
2.1 氨氮对反应器性能的影响
从表1可知,第1批次实验中,氨氮浓度为3000mg/L时,甲烷回收率(实际甲烷产率/理论甲烷产率)仅88.25%,这显著低于批次实验条件下稳定运行反应器的甲烷回收率(通常在94%以上[25-26]),也显著低于本团队前期研究[17]中报道的本底氨氮浓度(1800mg/L)下反应器的甲烷回收率(96.53%±2.66%).可见R1_3000产气性能受到了氨氮的影响.当将R1_3000中污泥继续暴露在3000mg/L的TAN浓度下时,R2_3000的甲烷产率和R2都显著增加(< 0.05),同时乙酸、丙酸浓度显著下降(<0.05),丙酸甚至降至0mg/L,表明其性能在连续的暴露中逐渐恢复了.R2_3000较长的2可能代表此阶段微生物正在重组适应新环境[27-28].继续暴露于相同氨氮浓度,R3_3000甲烷产率保持稳定,R2也维持在较高水平,2及乙酸含量进一步下降.说明此氨氮胁迫下,微生物代谢功能已恢复并维持到稳定状态,系统运行良好.Tian等[29]也观察到TAN为3300~3800mg/L时, 中温厌氧反应器运行稳定的现象.
注:*各类酸浓度是指实验末期酸的浓度.
当将来自R1_3000的种泥暴露于6000mg/L时,R2_6000组反应器的2显著增加(<0.05),指示微生物也发生了重组;R2显著提高,甲烷产率也呈现轻微上浮,暗示产气性能似乎在优化.但同时,反应器末期出现了更大量的VFAs积累,特别是丙酸,高达(428.36±56.79)mg/L.继续暴露于6000mg/L时, R3_6000组反应器的R2下降,甲烷产率不仅显著低于R2_6000组反应器(<0.05),也显著低于R1_3000组反应器(<0.05).VFAs方面,乙酸含量继续下降,但丙酸积累却没有丝毫改善(>0.05),丁酸尽管积累程度不如丙酸,但也呈现持续积累趋势.这是可以理解的,因为从热力学角度来看,在标况下(即101325Pa, 298.15K以及反应浓度为1mol/L时),丁酸和丙酸氧化反应的标准吉布斯自由能分别为48.4, 76.2kJ[30],两者的氧化均无法自发进行,但与丁酸相比,丙酸氧化反应需要吸收的能量更多,因此更难降解.前期在其他扰动因素造成的过程失稳中,丙酸和丁酸也曾被鉴定为主要的过程失稳原因[15,31-32].综上,TAN为6000mg/L时,乙酸没有明显积累,指示其代谢未受阻遏,但丙酸、丁酸在连续暴露实验中积累,因此它们的代谢可能是造成系统失稳的关键.
2.2 氨氮对微生物群落组成的影响
由表2可见,来自同一接种源的微生物样品暴露于不同TAN后,群落出现了越来越明显的差异;但同一TAN下,不同批次样品的群落相似性指数逐步增大,这与前期通过16S rRNA分析方法得到的研究结果一致[17,33].来自同一接种源(R1_3000组反应器)的R2_3000和R2_6000组微生物样品的群落相似性为88.53%,继续暴露在对应氨氮浓度下, R3_3000和R3_6000组样品相似性指数下降至83.77%.表明不同TAN浓度使群落朝着不同的方向演替.相比之下,R1_3000与R2_3000的相似性仅78.23%,这与R2_3000反应器较长的2是对应的.但R2_3000与R3_3000的群落相似性达到86.94%,暗示群落在朝着相同的方向演替.TAN为6000mg/L的实验组也有群落相似性逐渐增大的规律,R2_6000与R3_6000的群落相似性达到90.31%,表明长期暴露会使微生物的动态演替趋于平衡.通过解析此反应器的群落演替来揭示氨抑制失稳机理是可行的.
采用组间差异检验找出了不同氨氮浓度下,各反应器末期具有显著差异的微生物,并将其按功能分为水解酸化菌、产氢产乙酸菌和产甲烷菌.如图1a可见,不同氨氮浓度下,相对丰度具有显著差异的水解酸化菌共检测到12种.其中,7种微生物在R3_6000中显著富集(<0.05),5种微生物相对丰度在R3_6000中显著少于R3_3000(<0.05).总体上, TAN为3000mg/L时,主导水解酸化菌有10种,总相对丰度为28.94%;TAN为6000mg/L时,主导水解酸化菌有11种,总相对丰度为33.11%.多样性和高丰度保障了水解酸化菌在氨胁迫下代谢底物的能力,因此水解产酸阶段并非导致氨抑制反应器失稳的关键,这与以前的研究结论一致[17,34].
如图1b可见,在2个氨氮浓度下相对丰度具有显著差异的产氢产乙酸菌包括、、、、.据报道,主要降解C4~C18的脂肪酸[17],与R1_3000相比,该菌相对丰度在R2_3000增加了1.02倍,在R3_3000继续增加,相对丰度达到R1_3000的3.11倍,但在高氨氮浓度下,尽管R2_ 6000和R3_6000都出现明显的丁酸积累(表1),但相对丰度远低于低氨氮组(R2_3000和R3_3000),暗示系统丁酸代谢功能可能受到了高氨氮的影响.Du等[35]也指出由于丁酸降解过程发生在热力学平衡附近,微小的环境变化(如中间化合物)都会导致生物代谢途径受到影响.除以外的菌都是丙酸降解菌[36-37],低氨氮反应器中丙酸降解菌总相对丰度在R2_3000有所下降,在R3_3000出现回弹,反应末期无丙酸积累;但当TAN为6000mg/L时,尽管两个实验组的丙酸降解菌总相对丰度与R1_3000相比仅略有下降,但系统内丙酸积累到131.70~472.90mg/L,暗示该阶段代谢功能可能受到扰动.这些结果表明,微生物群落能够抵抗或成功适应低浓度TAN(3000mg/L),但可能无法在高浓度TAN(6000mg/L)下维持丁酸、丙酸互营氧化功能,丁酸、丙酸的互营降解可能是氨胁迫下反应器性能恶化的关键环节.

表2 不同样本之间的相似性指数

不同氨氮浓度下相对丰度具有显著差异的产甲烷菌仅有氢营养型和混合营养型.但鉴于检测到的主导甲烷菌仅3种,乙酸营养型也列于图1c中.从演替趋势来看,2个氨氮浓度下,除外,和相对丰度都较R1_3000显著上升(<0.05),导致R3_3000和R3_ 6000总产甲烷菌相对丰度分别增加了89.18%和33.64%.说明两个氨氮浓度下,产甲烷菌都得到了富集.而高氨氮反应器中产甲烷菌相对丰度的增加幅度不如低氨氮反应器,除氨氮胁迫外,也可能是产甲烷菌底物受限造成的.虽然2个氨氮浓度下,相对丰度均显著下降(<0.05),但研究报道,随着氨水平增加,当受到抑制后,可代替进行乙酸型产甲烷[17,38],因此的富集也许可以代行乙酸型产甲烷功能.而耐受性强的则维持了群落氢型产甲烷功能[39].因此,产甲烷阶段不一定是氨抑制失稳的关键环节.
2.3 氨氮对微生物代谢途径的影响
分析产氢产乙酸和产甲烷阶段功能基因的响应,而由于本研究及前期研究普遍认为水解酸化阶段不是导致氨抑制失稳的关键[3],本研究未关注此阶段.
2.3.1 产氢产乙酸阶段 水解酸化产物,如丙酸、丁酸等,不能直接被产甲烷菌利用,而要先被产氢产乙酸菌转化为乙酸和H2,再分别被乙酸型和氢型产甲烷菌降解.产氢产乙酸阶段包括丁酸及更长链脂肪酸的氧化、丙酸互营氧化和SAO.其中,丁酸及更长链脂肪酸主要通过β氧化途径降解,根据KEGG数据库和本研究检测到的基因,简化脂肪酸β氧化途径如图2a,脂肪酸β氧化产乙酸的过程就是脂肪酸的羧基在细胞质基质中与辅酶A结合,生成酰基辅酶A,并循环往复地被催化脱去乙基,直至生成乙酰辅酶A,乙酰辅酶A通过三羧酸循环生成乙酸.
图2b展示了β氧化途径相关基因的丰度变化,通过对样本进行差异检验,发现3个基因的相对丰度在不同氨氮浓度下具有显著差异,分别是烯酰辅酶A水合酶编码基因()、乙酰辅酶A酰基转移酶编码基因()和丁酰辅酶A脱氢酶编码基因().其中,和均在TAN为3000mg/L时得到显著富集,在TAN为6000mg/L时其相对丰度则维持在R1_3000水平,说明这2个基因氨耐受性强,但低氨氮环境更有助于它们的高度表达,这可能是TAN为3000mg/L的反应器中微生物群落丁酸氧化功能更优,系统性能得以恢复的原因之一.丁酸的互营氧化过程中,关键步骤是丁酰辅酶A的氧化,涉及到酰基辅酶A氧化酶编码基因()//酰基辅酶A脱氢酶编码基因(),在低氨氮环境中显著上调(< 0.05),而在高氨氮环境中受到严重抑制(<0.05),导致负责丁酰辅酶A氧化的基因总相对丰度下降了46.78%.由此可见,丁酸互营氧化途径确实受到了高浓度氨氮的影响,证实了2.1节的推论.
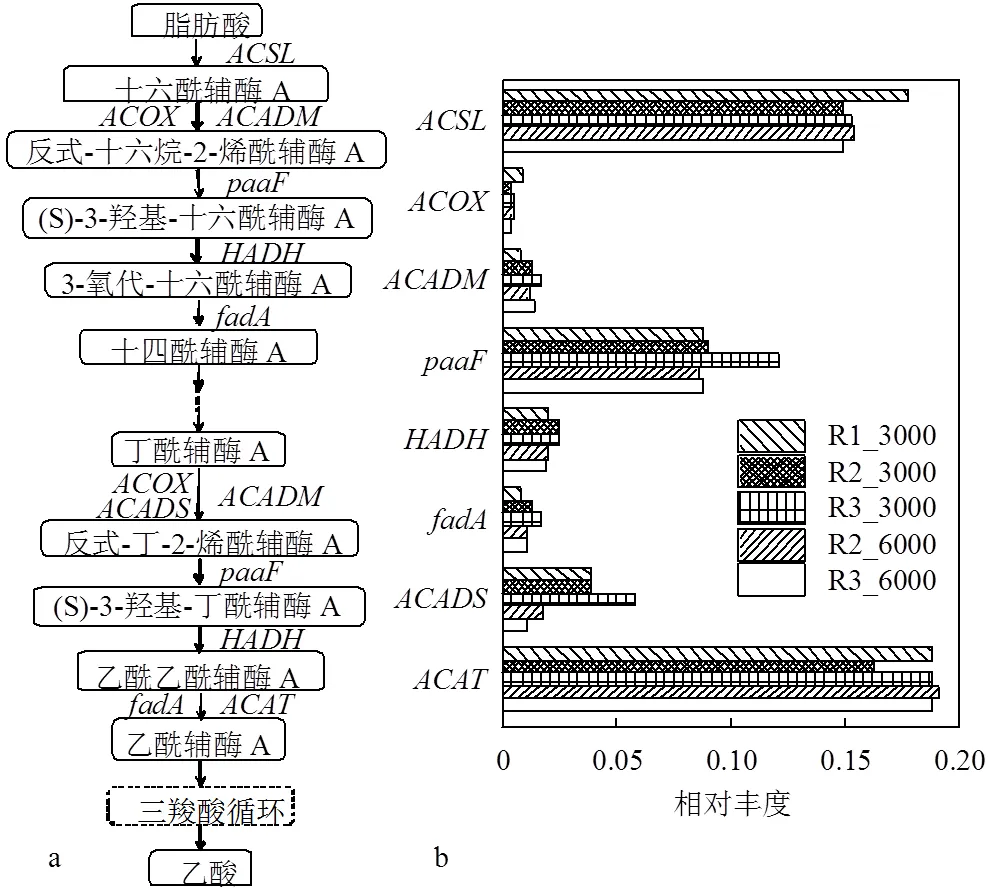
图2 脂肪酸β氧化途径及相关基因丰度变化
丙酸互营氧化途径主要为甲基丙二酰辅酶A途径(MMC途径).如图3a所示,丙酸首先被活化为丙酰辅酶A,然后通过MMC途径进行互营降解,转化为琥珀酰辅酶A后进入三羧酸循环,被进一步氧化为乙酸.对于丙酸活化过程,由图3b可知,丙酸辅酶A转移酶编码基因()和乙酸辅酶A连接酶编码基因()在2个氨氮浓度下都保持稳定.磷酸乙酰转移酶编码基因()在TAN为3000mg/L时受到抑制作用,与Yu等[27]的研究结果类似.催化辅酶A和CH3-CO-Pi生成Acetyl-CoA,CH3-CO-Pi可以可溶性CH3-CO-PO4K2、CH3-CO-PO4Na2及其他金属离子的形式存在.在产甲烷菌的胞内结构中,大多数金属离子会不受控的富集,同时氨分子向细胞壁扩散的可控性较差,也会因质子泵而在胞内结构中积累,2者反应生成CH3-CO-PO4[NH4]2,预期转化率超过90%,且反应产物为不溶性沉淀[27].而根据研究报道,氨氮对的抑制率可达95.57%[27].因此,CH3- CO-Pi更多的被用于与氨分子反应转化为CH3- CO-PO4[NH4]2沉积物,由于底物缺乏,丙酸通过丙酰磷酸生成丙酰辅酶A路径受到抑制.但是乙酰辅酶A合成酶编码基因()相对丰度在R3_3000开始上升,增长率为8.91%,丙酸活化过程代谢途径的转变,有助于维持丙酸活化功能.Yu等[27]运行了5组TAN浓度不等(171.17~3043.83mg/L)的实验室规模的反应器,也发现了丙酸活化途径从生成丙酰磷酸转变为生成丙酰腺苷酸.相比之下,TAN为6000mg/L时,相对丰度并无显著变化(>0.05),但在两批次氨氮暴露过程中,相对丰度持续下降,较R1_3000下降约24.28%,在一定程度上影响了丙酸活化功能.
对于MMC途径,丙酰辅酶A羧化酶α链编码基因()与类似,在R2_3000和R2_6000中丰度均显著下降(<0.05),但持续暴露在低氨氮环境中,该基因相对丰度开始上调,增长率为17.45%,而在高氨氮环境中,该基因的受抑制情况毫无改善, 其在R3_6000组样品中的相对丰度较R1_3000组少54.73%.相比之下,连续暴露下甲基丙二酰辅酶A编码基因()和甲基丙二酰辅酶A变位酶编码基因()的相对丰度也有所下降,但其在两个氨氮浓度下(R2_3000和R2_6000、R3_3000和R3_6000)并没有显著差异(>0.05),因此它们可能不是氨抑制失稳的关键基因.

图3 MMC途径及相关基因丰度变化
由此可见,MMC途径的关键基因受到不可逆抑制是高氨氮环境丙酸大量积累的主要原因,进一步证实了丙酸互营氧化过程是反应器失稳的根源.
SAO是产氢产乙酸阶段中的特殊环节,该环节在乙酸代谢受阻时,发挥重要作用[39],将乙酸转化为H2和CO2进而被氢型产甲烷菌代谢.大多数已鉴定的SAOB都是通过反向Wood-Ljungdahl途径(WL途径)进行乙酸氧化[40].反向WL途径由2个分支组成,甲基分支和羧基分支.在甲基分支中,乙酰辅酶A被氧化为甲基进而氧化为CO2,而在羧基分支中,乙酰辅酶A被氧化为CO进而氧化为CO2(图4a).

图4 WL途径及相关基因丰度变化
如图4b所示,TAN为3000mg/L时,甲基分支中亚甲基四氢叶酸还原酶编码基因()、四氢叶酸连接酶编码基因()的表达受到抑制,但羧基分支相关基因均得到富集,总相对丰度上调约11.76%,维持了群落互营乙酸氧化功能;TAN为6000mg/L时,羧基分支部分一氧化碳脱氢酶编码基因()的表达受到抑制,但甲基分支中、亚甲基四氢叶酸脱氢酶编码基因()得到富集,有助于维持群落互营乙酸氧化功能,从而避免了系统乙酸的积累,与Ruiz-Sánchez等[41]的研究结果一致.由此可见,此环节并不是氨抑制失稳的关键环节.很多研究都指出反应器受到氨扰动后,除了乙酸型产甲烷途径,乙酸还会通过SAO-HM产甲烷[30,39],氢和还原力通过SAO途径得到释放,是厌氧微生物应对氨氮抑制的重要适应性机制[32].此外,虽然SAO代谢途径没有受损,但本研究却没有检测到常见的SAOB,推测系统中可能存在未知的SAOB,后续可结合宏转录组学和宏蛋白质组学技术进一步验证.
2.3.2 产甲烷阶段 厌氧消化的最后1个阶段是产甲烷,根据底物类型的不同,产甲烷途径包括:乙酸型产甲烷(M00357)、还原CO2型产甲烷(M00567)、甲醇型产甲烷(M00356)以及甲胺型产甲烷(M00563)4种[42].

图5 产甲烷途径及相关基因丰度变化
根据KEGG数据库,对于M00357,乙酸通过两种机制转变为乙酰辅酶A,其一是乙酸经乙酸激酶和磷酸乙酰转移酶的催化利用1分子ATP转变为乙酰辅酶A,主要采用这种机制;另一种是乙酸通过乙酰辅酶A合成酶和2分子ATP被转化为乙酰辅酶A,代表微生物是[42].乙酰辅酶A再分别由乙酰辅酶A羧化酶、四氢甲蝶呤S-甲基转移酶催化生成甲基辅酶M.对于M00567,甲酰甲烷呋喃脱氢酶催化还原CO2为甲酰基共价连接于甲烷呋喃的氨基基团形成甲酰甲烷呋喃,接下来甲酰甲烷呋喃在四氢甲烷蝶呤甲酰转移酶、甲酰四氢甲烷蝶呤环化水解酶等酶的催化下形成甲基辅酶M[42].M00356和M00563都属于甲基营养途径,甲基化合物在特殊的辅酶M甲基转移酶系统作用下将甲基基团转移到HS-CoM的巯基上生成甲基辅酶M[42].4个途径都包括的共有部分为甲基辅酶M转化为甲烷及辅酶M-S-S-辅酶B转化为辅酶B两步.
4条完整途径都在本研究中被检测到,由图5可见,R3_3000中4条途径相关基因相对丰度之和均大于R3_6000,尽管R3_3000产甲烷代谢功能整体优于R3_6000,但是从基因相对丰度的变化来看,4条产甲烷代谢途径都表现出对氨的极强抗性,尤其是甲基型产甲烷途径和共有代谢途径在两个氨氮浓度下均被显著富集,说明氨氮并没有抑制群落产甲烷功能.具体来看,甲基型产甲烷途径所独有的基因和4条途径共有的基因在氨氮的刺激下均显著上调(<0.05),其中,四氢甲烷蝶呤S-甲基转移酶编码基因()的富集有助于增强鲁棒性[43],解释了物种分析中的显著富集现象.氢型产甲烷途径所独有的基因在不同批次的驯化下均保持稳定,SAO途径可为氢型产甲烷途径提供电子和碳源[27],结合SAO途径的稳定再次暗示了系统中可能存在未知SAOB.对于乙酸型产甲烷途径所独有的基因,高氨氮环境中,被强烈抑制(>0.05),但乙酸激酶编码基因()和均保持稳定,如前所述,主要采用前者进行产甲烷,主要采用后者进行产甲烷,结合物种分析中和相对丰度的变化,有效证明了本研究中代替维持了乙酸型产甲烷的推论.
另一方面,丙酸和丁酸代谢需要丙酸互营氧化功能、丁酸互营氧化功能和耗氢的CO2还原型产甲烷功能共同作用,CO2还原途径是厌氧消化过程中电子流动的关键,氢型产甲烷菌通过该途径降解低浓度H2/甲酸盐,有助于推动产氢产乙酸阶段的顺利进行[44],后者基因功能的稳定也说明失稳状态下,丙酸、丁酸代谢受阻仅归因于前端丙酸、丁酸互营氧化过程被氨氮抑制.总之,群落产甲烷功能并不是氨抑制失稳的根本原因.
3 结论
3.1 氨氮影响厌氧消化系统性能和产气动力学. TAN为3000mg/L时,长期暴露可使厌氧消化系统性能优化;TAN为6000mg/L时,长期暴露无法恢复系统的性能.
3.2 多样性和高丰度保障了水解酸化菌在氨胁迫下代谢底物的能力,水解酸化阶段不是系统运行失败的原因.
3.3 高氨氮环境中,甲基型产甲烷途径相关基因被富集,氢型产甲烷途径相关基因保持稳定,乙酸型产甲烷途径由经乙酰辅酶A合成酶催化直接生成乙酰辅酶A转变为由乙酸激酶和磷酸乙酰转移酶联合催化生成乙酰辅酶A,维持了群落乙酸型产甲烷功能.因此产甲烷代谢途径对氨的极强抗性及其本身的代谢冗余性,也保障了群落产甲烷功能.
3.4 产氢产乙酸阶段中,高氨氮抑制了关键基因和的表达,影响了群落互营丁酸和丙酸氧化功能,由此引起高氨氮反应器中丁酸和丙酸的积累,反应器产气性能下降.鉴于此,互营丁酸和丙酸氧化是高氨氮反应器过程失稳的关键环节.
[1] Wu D, Li L, Zhao X F, et al. Anaerobic digestion: A review on process monitoring [J]. Renewable and Sustainable Energy Reviews, 2019,103:1-12.
[2] Li L, Peng X Y, Wang X M, et al. Anaerobic digestion of food waste: A review focusing on process stability [J]. Bioresource Technology, 2018,248:20-28.
[3] Chen H, Wang W, Xue L N, et al. Effects of ammonia on anaerobic digestion of food waste: Process performance and microbial community [J]. Energy & Fuels, 2016,30(7):5749-5757.
[4] Bi S J, Mw D, Westerholm M, et al. The metabolic performance and microbial communities of anaerobic digestion of chicken manure under stressed ammonia condition: A case study of a 10-year successful biogas plant [J]. Renewable Energy, 2021,167:644-651.
[5] Wijesinghe D T N, Suter H C, Scales P J, et al. Lignite addition during anaerobic digestion of ammonium rich swine manure enhances biogas production [J]. Journal of Environmental Chemical Engineering, 2021, 9(1):104669.
[6] Meng X S, Sui Q W, Liu J B, et al. Relieving ammonia inhibition by zero-valent iron (ZVI) dosing to enhance methanogenesis in the high solid anaerobic digestion of swine manure [J]. Waste Management, 2020,118:452-462.
[7] Niu Q G, Qiao W, Qiang H, et al. Microbial community shifts and biogas conversion computation during steady, inhibited and recovered stages of thermophilic methane fermentation on chicken manure with a wide variation of ammonia [J]. Bioresource Technology, 2013,146:223-233.
[8] Yang Z Y, Wang W, He Y F, et al. Effect of ammonia on methane production, methanogenesis pathway, microbial community and reactor performance under mesophilic and thermophilic conditions [J]. Renewable Energy, 2018,125:915-925.
[9] Bi S J, Qiao W, Xiong L P, et al. Improved high solid anaerobic digestion of chicken manure by moderate in situ ammonia stripping and its relation to metabolic pathway [J]. Renewable Energy, 2020, 146:2380-2389.
[10] Buhlmann C H, Mickan B S, Jenkins S N, et al. Ammonia stress on a resilient mesophilic anaerobic inoculum: Methane production, microbial community, and putative metabolic pathways [J]. Bioresource Technology, 2019,275:70-77.
[11] Zhang N, Peng H J, Li Y, et al. Ammonia determines transcriptional profile of microorganisms in anaerobic digestion [J]. Brazilian Journal of Microbiology, 2018,49(4):770-776.
[12] Peng X Y, Zhang S Y, Li L, et al. Long-term high-solids anaerobic digestion of food waste: Effects of ammonia on process performance and microbial community [J]. Bioresource Technology, 2018,262: 148-158.
[13] Fotidis I A, Karakashev D, Angelidaki I. Bioaugmentation with an acetate-oxidising consortium as a tool to tackle ammonia inhibition of anaerobic digestion [J]. Bioresource Technology, 2013,146:57-62.
[14] Li Y, Zhang Y, Kong X Y, et al. Effects of ammonia on propionate degradation and microbial community in digesters using propionate as a sole carbon source [J]. Journal of Chemical Technology and Biotechnology, 2017,92(10):2538-2545.
[15] Bonk F, Popp D, Weinrich S, et al. Ammonia inhibition of anaerobic volatile fatty acid degrading microbial communities [J]. Frontiers in Microbiology, 2018,9:2921.
[16] Wang H Z, Yan Y C, Gou M, et al. Response of propionate-degrading methanogenic microbial communities to inhibitory conditions [J]. Applied Biochemistry and Biotechnology, 2019,189(1):233-248.
[17] Zhang H, Peng Y, Yang P J, et al. Response of process performance and microbial community to ammonia stress in series batch experiments [J]. Bioresource Technology, 2020,314:123768.
[18] Ruiz-Sánchez J, Campanaro S, Guivernau M, et al. Effect of ammonia on the active microbiome and metagenome from stable full-scale digesters [J]. Bioresource Technology, 2018,250:513-522.
[19] 何 琴,李 蕾,瞿 莉,等.餐厨垃圾干式厌氧消化污泥膨胀微生态特征 [J]. 中国环境科学. 2018,38(3):1010-1017.
He Q, Li L, Qu L, et al. Microbial characteristics of bulking sludge in high-solids anaerobic digestion of kitchen waste [J]. Chinese Environmental Science, 2018,38(3):1010-1017.
[20] 国家环境保护总局水与废水监测分析方法编委会.水与废水监测分析方法[M]. 北京:中国环境科学出版社, 2002.
State Environmental Protection Administration. Determination methods for examination of water and wastewater [M]. Beijing:China Environmental Science Press, 2002.
[21] Chen S S, He J, Wang H Y, et al. Microbial responses and metabolic pathways reveal the recovery mechanism of an anaerobic digestion system subjected to progressive inhibition by ammonia [J]. Chemical Engineering Journal, 2018,350:312-323.
[22] Ma X X, Yu M, Song N, et al. Effect of ethanol pre-fermentation on organic load rate and stability of semi-continuous anaerobic digestion of food waste [J]. Bioresource Technology, 2020,299:122587.
[23] Li D, Liu S C, Mi L, et al. Effects of feedstock ratio and organic loading rate on the anaerobic mesophilic co-digestion of rice straw and cow manure [J]. Bioresource Technology, 2015,189:319-326.
[24] Gomes C S, Strangfeld M, Meyer M. Diauxie studies in biogas production from gelatin and adaptation of the modified Gompertz model: Two-phase Gompertz model [J]. Applied Sciences-basel, 2021, 11(3):1067.
[25] Gu J, Liu R, Cheng Y, et al. Anaerobic co-digestion of food waste and sewage sludge under mesophilic and thermophilic conditions: Focusing on synergistic effects on methane production [J]. Bioresource Technology, 2020,301:122765.
[26] Li L, He Q, Zhao X F, et al. Anaerobic digestion of food waste: Correlation of kinetic parameters with operational conditions and process performance [J]. Biochemical Engineering Journal, 2018,130:1-9.
[27] Yu D W, Zhang J Y, Chulu B, et al. Ammonia stress decreased biomarker genes of acetoclastic methanogenesis and second peak of production rates during anaerobic digestion of swine manure [J]. Bioresource Technology, 2020,317:124012.
[28] Kurade M B, Saha S, Kim J R, et al. Microbial community acclimatization for enhancement in the methane productivity of anaerobic co-digestion of fats, oil, and grease [J]. Bioresource Technology, 2020,296:122294.
[29] Tian H L, Fotidis I A, Mancini E, et al. Acclimation to extremely high ammonia levels in continuous biomethanation process and the associated microbial community dynamics [J]. Bioresource Technology, 2018,247:616-623.
[30] Pan X F, Zhao L, Li C X, et al. Deep insights into the network of acetate metabolism in anaerobic digestion: focusing on syntrophic acetate oxidation and homoacetogenesis [J]. Water Research, 2021, 190:116774.
[31] Li Y, Sun Y M, Yang G X, et al. Vertical distribution of microbial community and metabolic pathway in a methanogenic propionate degradation bioreactor [J]. Bioresource Technology, 2017,245:1022-1029.
[32] 邓玉营.共培养菌群强化秸秆厌氧消化及微生物学机制研究 [D]. 无锡:江南大学, 2017.
Deng Y Y. Enhanced anaerobic digestion of straw via co-cultivated consortia and its underlying microbial mechanisms [D]. Wuxi: Jiangnan University, 2017.
[33] 张 虹,李 蕾,彭 韵,等.氨氮对餐厨垃圾厌氧消化性能及微生物群落的影响 [J]. 中国环境科学, 2020,40(8):3465-3474.
Zhang H, Li L, Peng Y, et al. Effects of ammonia on anaerobic digestion of food waste: Process performance and microbial community [J]. China Environmental Science, 2020,40(8):3465-3474.
[34] Yin D M, Westerholm M, Qiao W, et al. An explanation of the methanogenic pathway for methane production in anaerobic digestion of nitrogen-rich materials under mesophilic and thermophilic conditions [J]. Bioresource Technology, 2018,264:42-50.
[35] Du J, Yin Q D, Gu M Q, et al. New insights into the effect of ethanol and volatile fatty acids proportions on methanogenic activities and pathways [J]. Environmental Research, 2021,194:110644.
[36] Saha S, Jeon B H, Kurade M B, et al. Interspecies microbial nexus facilitated methanation of polysaccharidic wastes [J]. Bioresource Technology, 2019,289:121638.
[37] Xia X X, Zhang J C, Song T Z, et al. Stimulation of Smithella -dominating propionate oxidation in a sediment enrichment by magnetite and carbon nanotubes: Propionate syntrophy in the presence of conductive materials [J]. Environmental Microbiology Reports, 2019,11(2):236-248.
[38] 孙航宇,杨紫怡,李潇男,等. ADM1模型对生物强化厌氧产甲烷体系的模拟 [J]. 中国环境科学, 2020,40(3):1049-1058.
Sun H Y, Yang Z Y, Li X N, et al. Simulation of anaerobic digestion based on bioaugmentation by ADM1 [J]. Chinese Environmental Science, 2020,40(3):1049-1058.
[39] Westerholm M, Dolfing J, Schnurer A. Growth characteristics and thermodynamics of syntrophic acetate oxidizers [J]. Environmental Science & Technology, 2019,53(9):5512-5520.
[40] Westerholm M, Moestedt J, Schnurer A. Biogas production through syntrophic acetate oxidation and deliberate operating strategies for improved digester performance [J]. Applied Energy, 2016,179:124-135.
[41] Ruiz-Sánchez J, Guivernau M, Fernandez B, et al. Functional biodiversity and plasticity of methanogenic biomass from a full-scale mesophilic anaerobic digester treating nitrogen-rich agricultural wastes [J]. Science of the Total Environment, 2019,649:760-769.
[42] 方晓瑜,李家宝,芮俊鹏,等.产甲烷生化代谢途径研究进展 [J]. 应用与环境生物学报, 2015,21(1):1-9.
Fang X Y, Li J B, Rui J P, et al.Research progress in biochemical pathways of methanogenesis [J]. Chinese Journal of Applied and Environmental Biology, 2015,21(1):1-9.
[43] Yan M, Treu L, Campanaro S, et al. Effect of ammonia on anaerobic digestion of municipal solid waste: inhibitory performance, bioaugmentation and microbiome functional reconstruction [J]. Chemical Engineering Journal, 2020,401:126159.
[44] Leng L, Yang P X, Singh S, et al. A review on the bioenergetics of anaerobic microbial metabolism close to the thermodynamic limits and its implications for digestion applications [J]. Bioresource Technology, 2018,247:1095-1106.
Metagenomic analysis on the responses of microbial community to ammonia stress.
PENG Yun, LI Lei*, WU Di, YANG Ping-jin, PENG Xu-ya, WANG Xiao-ming
(Key Laboratory of Three Gorges Reservoir Region’s Eco-Environment, Ministry of Education, Chongqing University, Chongqing 400045, China)., 2022,42(2):777~786
A series of batch experiments were conducted to investigate the responses of anaerobic digestion (AD) microbial community exposed to ammonia (TAN) stress. In combination with Metagenomic sequencing analysis, this paper investigated the responses of process parameters, community structure/composition and functional gene/metabolic pathways of AD to the process disturbance induced by different TAN concentrations. The overall performance of AD system was gradually optimized at the concentration of 3000mg/L TAN while when TAN reached 6000mg/L, an accumulation of propionate and butyrate was observed with a concomitant decrease of methane yield. At both concentration levels, the resistance and redundancy of hydrolytic and acidogenic bacteria can ensure their metabolic performance for substrate degradation. However, the total relative abundance of acetogens and methanogens at the low concentration level continued to increase, whereas a low increase rate and even a decrease in the total relative abundance was observed at the high level, suggesting the potential process inhibition. By further analyzing the functional gene changes in the two process stages, The methanogenesis and syntrophic acetate oxidation pathways showed resistance and redundancy under the ammonia stress, but the high concentration of TAN (6000mg/L) inhibited the expression of key genesandin the syntrophic degradation of butyrate and propionate. Therefore, the key contributor to the ammonia inhibition turned out to be acetogenesis.
food waste;anaerobic digestion;ammonia stress;metagenomics
X705
A
1000-6923(2022)02-0777-10
彭 韵,(1993-),女,重庆人,重庆大学博士研究生,研究方向为固体废物污染控制与资源化.发表论文5篇.
2021-06-22
国家自然科学基金资助项目(51708057);重庆市自然科学基金资助项目(cstc2018jcyjAX0743)
* 责任作者, 副教授, lileich17@cqu.edu.cn

