特早熟甘蓝型春油菜恢复系核心种质构建
林晓阳 杜德志 柳海东 李 钧
(青海大学农林科学院/春油菜遗传改良重点实验室/国家油菜改良中心青海分中心/农业农村部春油菜科学观测实验站/青海省春油菜研究开发中心/青海省春油菜工程技术研究中心,810016,青海西宁)
油菜作为我国重要的油料作物,每年种植面积可达667万hm2(1亿亩)[1-2]。春油菜属于一年生草本植物,种植区域主要分布在青海、甘肃、新疆、内蒙古及西藏等气温较低、无霜期较短的地区,约占我国油菜种植面积的10%~15%[3-5]。在我国高海拔、无霜期较短的春油菜区,油菜种植主要以白菜型油菜为主,存在产量低、品质差的问题;甘蓝型油菜(Brassica napus L.)较白菜型油菜产量高、品质优,但生育期较长制约了其在高海拔地区的推广种植。选育能在高海拔地区正常成熟的高产优质甘蓝型油菜品种对提高高海拔地区春油菜产量具有重要意义。
甘蓝型油菜的F1代具有较强的杂种优势。自傅廷栋等[6]发现波里马雄性不育系以来,恢复系的合理选择和应用成为三系配套杂交种选育的一个重要环节。种质资源作为新品种选育的遗传基础,通过构建核心种质,可以用部分样本代表遗传资源群体,为亲本的杂交组配提供理论指导[7]。目前,我国已构建了水稻[8]、大豆[9]和小麦[10]等主要农作物核心种质。核心亲本作为核心种质的子集,主要是为组配强优势杂交种做准备。同时其筛选并不是一成不变的,不同育种目标有不同侧重点。随着具有更优良性状品种的选育,可以不断扩充和更新具有目标性状的核心种质库,增强优势杂交亲本的组配几率,从而提高育种效率[11]。核心种质的建立主要借助于 SSR(simple sequence repeat)和 SNP(single nucleotide polymorphism)等分子标记手段。曾晓珊等[12]利用48对SSR引物构建了27个水稻核心种质的指纹图谱,从中筛选到的优势核心种质均为籼稻;杨勇[13]利用173对SSR引物构建了甘蓝型油菜核心种质的指纹图谱。上述研究为籼稻和甘蓝型油菜的杂交组合及杂种优势的预测提供了科学指导。
构建核心种质,重要的是如何从群体中选出核心子集。对于给定的群体,已经提出了许多用于核心集的算法。如Brown提出的仅取决于群体大小而不取决于每个群体多样性的 P方法和 L方法;Schoen和 Brown提出的 M 方法等[14]。而 Core Hunter算法直接从整个群体中采样,依托 REMC(replica exchange monte carlo)本地搜索技术,可以防止搜索陷入局部最优状态;同时,可自主选择优化多个度量,并进行权重的分配,进而找到具有较高亲本间平均遗传距离和较高总体多样性的核心集;与其他算法相比,该方法灵活、高效,且所保留的核心子集明显较小[15]。用能在高海拔地区正常成熟的优质特早熟甘蓝型油菜资源作为亲本,杜德志等[16]选育出“青杂3号”、“青杂4号”、“青杂7号”、“青杂8号”和“青杂10号”5个特早熟甘蓝型油菜杂交种,提高了高海拔地区油菜的产量及品质。选择优良的特早熟甘蓝型油菜恢复系对特早熟甘蓝型油菜品种的培育具有重要意义。本研究在简化基因组测序技术的基础上,利用 Core Hunter算法对97份特早熟甘蓝型油菜恢复系进行核心种质的构建,可以更好地对特早熟甘蓝型春油菜恢复系种质资源进行评价和利用,减少育种工作中恢复系与不育系选配杂交组合的工作量,同时为特早熟甘蓝型春油菜优良组合的亲本选配提供恢复系资源。
1 材料与方法
1.1 试验材料
材料来自青海省农林科学院春油菜研究所。供试的97份材料为早熟甘蓝型油菜品系与青藏高原白菜型油菜“门源小油菜”进行种间杂交(母本为甘蓝型D9946、父本为白菜型浩油11号),其中F1代在帐子内进行自由授粉,在下一代中选取偏甘蓝型植株进行多代自交,即后代系谱选育而成的特早熟甘蓝型春油菜恢复系品系。供试材料初花期在42~50d。
1.2 田间试验
2019年4月25日,将97份恢复系播于青海省农林科学院春油菜研究所试验田(101°45′ E,36°43′ N),采用随机区组设计,每个恢复系3个重复,每个重复2行区,行长2m,行间距30cm,株距15cm。
1.3 测序分析
1.3.1 酶切方案设计 对97份材料幼嫩叶片进行取样 1~2cm2,送至北京百迈客生物科技有限公司进行测序分析。选取甘蓝型油菜宁油 7号基因组(http://ibi.zju.edu.cn/bnpedigome/download.php?con=ny7)为参考基因组进行酶切预测,组装基因组大小为993.78Mb,GC含量36.32%。
1.3.2 Illumina平台测序及产出数据的质量分析
酶切预测后,选取合适的酶切组合对97份基因样品进行酶切。将酶切后片段(specific-locus amplified fragment标签)的3′端加ploy A、连接 Dual-index[17]测序接头,经过特定的PCR引物扩增后,进行纯化处理再混合,然后将扩增产物进行琼脂糖凝胶电泳进行分离,选取目的片段进行切胶,经文库质检合格后选用水稻日本晴(Oryza sativa ssp. japonica)作为对照,将测序得到的数据在Illumina测序平台进行测序分析。
1.3.3 SLAF标签和SNP标记开发 对测序得到的数据进行酶切效率评估。利用 SOAP[18]软件将Control测序中产生的读长(reads)与甘蓝型油菜参考基因组作比对。根据生物信息学分析结果,利用BWA[19]将SLAF-seq测序得到的reads以油菜基因组作为参考基因组进行比对,利用 GATK[20]和SAMtools[21]2种方法开发全基因组范围的SNP标记,选取基因型频率第2高的基因型所在频率(一般MAF>0.05)[22]作为具有代表性的SNP,将二者交集作为最终可靠的SNP标记库。
1.3.4 核心种质构建及评价指标 利用 Core Hunter软件构建该群体的核心种质并进行评价[23],从改良的罗杰斯距离(MR)、爱德华和卡瓦利距离(CE)、香农指数(SH)、期望杂合度(HE)、多态性信息含量(PIC)、有效等位基因(NE)和等位基因覆盖度(CV)等方面进行考量。MR将每个等位基因视为一个单独的维度,是标准欧几里德距离的改进,CE则采用选择性漂移模型,样本的突变率低且选择性压力快速变化;SH是1种合适的度量,试图尽可能多地保留子集中的稀有等位基因;HE将每个基因座内的多样性考虑在内;CV表示核心子集中的等位基因与原始集合相比所占的百分比;PN与CV相反,是未覆盖的等位基因[19]。
2 结果与分析
2.1 酶切建库与测序评估
确定以RsaⅠ+HaeⅢ为酶切组合,在Illumina测序平台以水稻日本晴作为对照进行测序分析,得到大小为364~414bp的SLAF标签119 741个,各染色体上SLAF标签的数量分布见图1,SLAF标签在基因组中分布大致均匀。结果显示,1条序列两端在参考基因组上的比对跨度介于50~1000bp的读长占总读长双端比对的比对效率为95.16%;1条序列两端在参考基因组上的比对跨度小于50bp,或大于1kb的读长占总读长的比例为1.21%;未比对到基因组上的读长占总读长的比例为3.63%;比对效率基本正常。与水稻日本晴数据双端比对的比对效率为 95.16%,酶切效率为 87.54%,因此 SLAF建库正常。
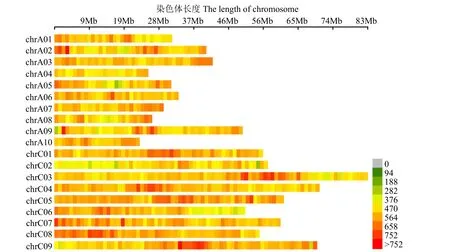
图1 SLAF标签在染色体上的分布Fig.1 SLAF tags distribution on chromosome
为保证项目数据分析可靠,采用去掉接头126bp×2的读长进行数据分析和评估。共得到367.5Mb读长数据,测序平均Q30(碱基测序出错概率为0.001)为94.70%;平均GC含量43.27%。用于评估试验建库准确性的水稻日本晴测序获得0.43Mb的读长数据量。测序质量值越高对应的碱基测序错误率越低,故此次建库及测序合理[23]。
2.2 SLAF标签与SNP标记的鉴定
选择油菜基因组为参考基因组进行电子酶切预测,共得到249.91Mb读长,获得527 872个SLAF标签,标签的平均测序深度为 14.91X,其中多态性的SLAF标签共有171 414个,以及842 248个群体SNP。SLAF标签在A、C基因组上分别有174 963和306 011个(表1);C基因组上标签数量明显多于A基因组,约为A基因组的2倍。利用A、C基因组分别开发多态性SLAF标签70 441个和86 086个。
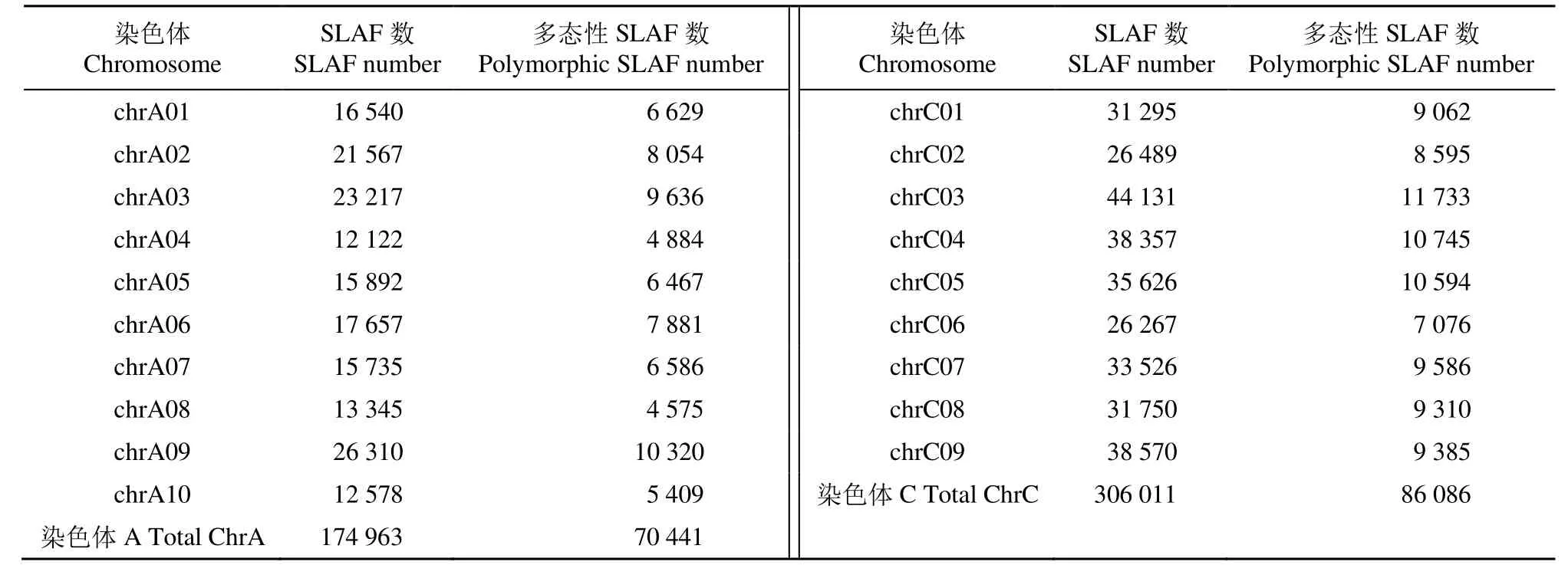
表1 SLAF标签和多态性SLAF标签染色体分布Table 1 Chromosome distribution of SLAF tags and polymorphic SLAF tags
共获得842 248个SNP标记,根据SNP在染色体上的分布,绘制SNP在染色体上的分布图(图2)。A基因组上SNP分布明显较C基因组更为富集。此外,SNP完整度介于23.77%~38.73%;杂合率介于5.20%~15.33%,纯合度较高。
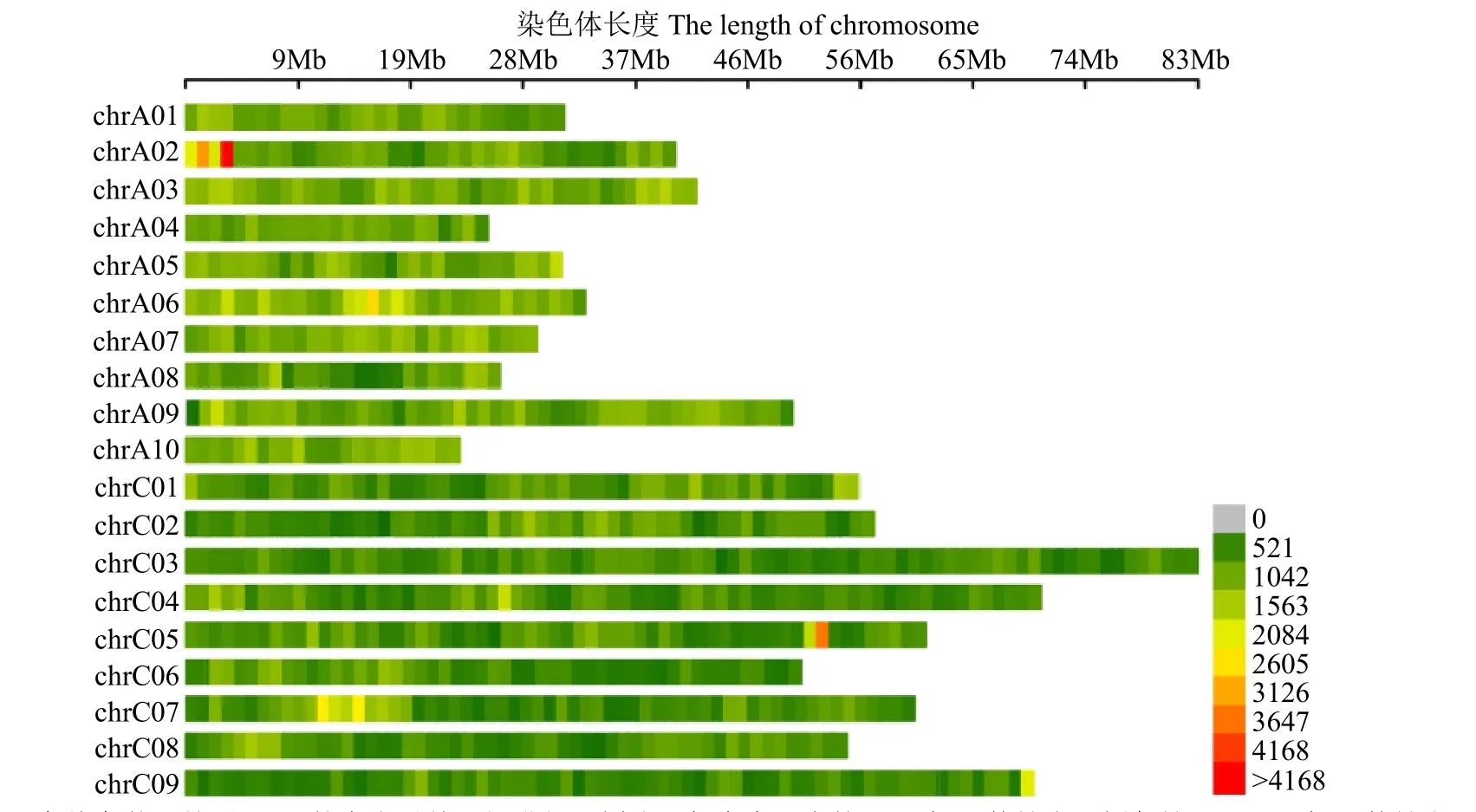
图2 SNP在染色体上的分布Fig.2 SNP distribution on chromosome
2.3 系统发育和遗传结构分析
使用MEGA X[24]软件对得到的SNP位点进行遗传分析,基于neighbor-joining的算法,在bootstrap重复1000次、Kimura 2-parameter模型下,构建97份特早熟甘蓝型油菜种质资源的系统发育树,分析不同种质之间的遗传进化关系(图3)和群体结构。97份材料可分为5类:第1类只包含1份种质;第2类包含26份种质;第3类包含17份种质;第4类包含17份种质;第5类包含36份种质。
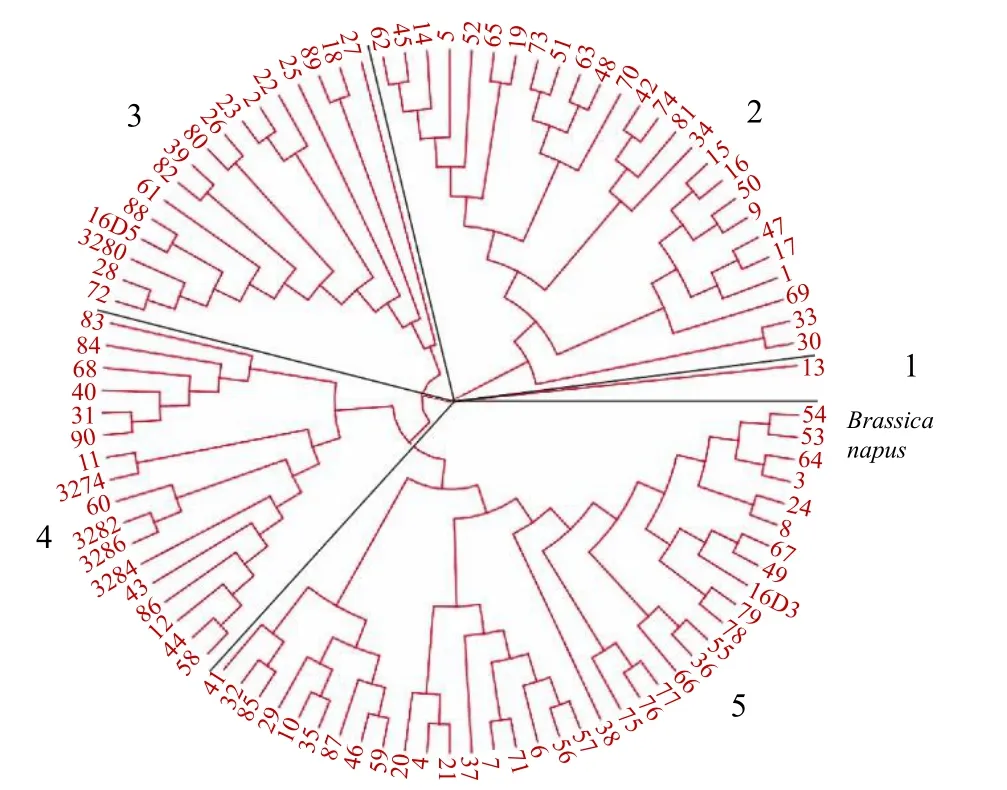
图3 基于SLAF-seq技术的甘蓝型春油菜恢复系NJ进化树Fig.3 NJ phylogenetic tree of Brassica napus L. restorer based on SLAF-seq
2.4 恢复系间遗传距离分析
基于neighbor-joining算法计算97份甘蓝型油菜恢复系之间的遗传距离。遗传距离介于 0.0026~0.6628,平均遗传距离为0.3510。由表2可知,97份恢复系间排列前10位的遗传距离值均≥0.60。可以看出,遗传距离前10位的组合主要集中于编号86、12、44、58 和编号 10、78、16D3、49、67 材料之间的组合。
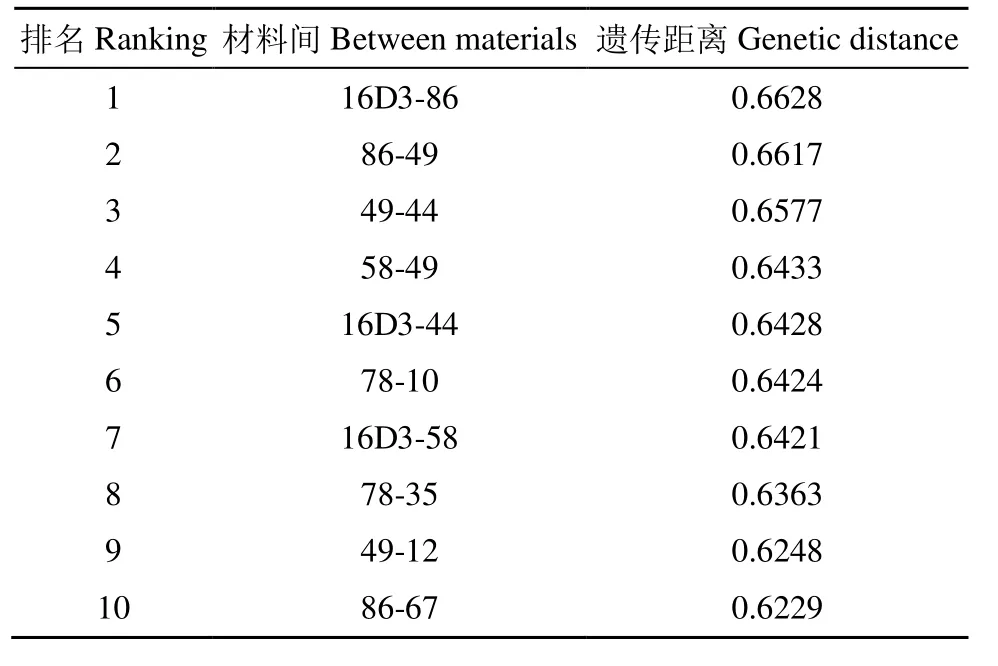
表2 甘蓝型油菜各恢复系之间排位前10位的遗传距离Table 2 Genetic distances of the top 10 restorer lines in Brassica napus L.
2.5 核心种质构建与评价
根据有效SNP,利用Core Hunter软件构建97份甘蓝型油菜恢复系核心种质,得到2个较优核心子集,分别命名为C30和C40。其中,C30包括29份恢复系资源(图4),C40包括38份恢复系资源(图5)。
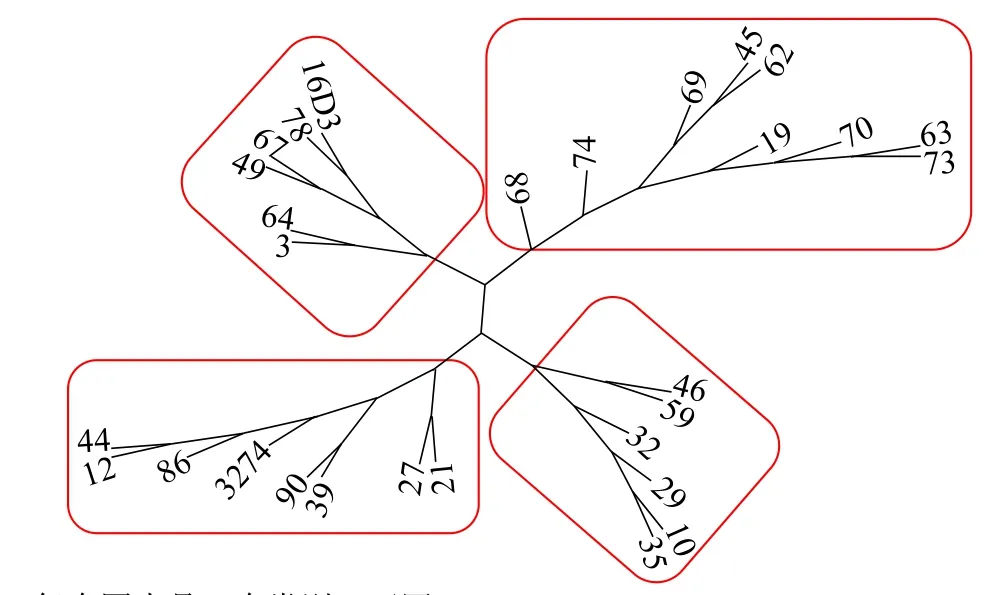
图4 C30核心种质聚类图Fig.4 Clustering map of C30 core collection
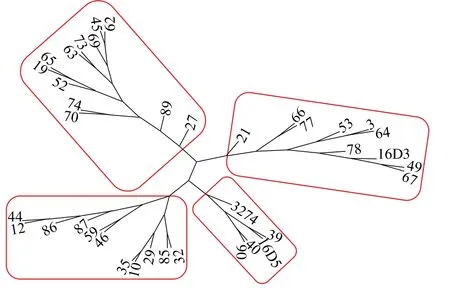
图5 C40核心种质聚类图Fig.5 Clustering map of C40 core collection
由表3可知,C30和C40核心子集的PIC分别为0.20和0.29。其中,C30核心种质的MR大小在0.15~0.47范围内,CE范围为0.16~0.48,2个子集相差不大。2个核心子集的HE相同,均为0.33;SH均为11.35。CV分别为99.89%(C30)和99.96%(C40),均达到 99%以上。进一步对C30和 C40进行聚类分析,2个核心子集均可分为4类(图4-图5)。原始样品聚类类型中编号13的样品单列一类,未被选入C30和C40核心种质群中;其他4类所包含材料类型和原始材料间聚类基本一致。因此,C30和C40这2个核心子集分别所包含的样品可以代表97份优良甘蓝型油菜恢复系的核心种质(表3);由于2个核心子集的各项数据差异不大,且C30子集的材料均被包含在C40中,最终将C30作为核心子集。这些结论为后期最优杂交组合配制中种质的选配提供依据。
3 讨论
核心种质的筛选有助于更好地利用现有种质资源,核心种质群离不开核心种质的构建。构建核心种质即从整体中选取一定数量较少的材料代表一个作物种及其近缘野生种的最大遗传多样性[25]。对于油菜核心种质的研究,国内外主要通过形态分析和分子标记的技术手段来获得大量的遗传背景信息。新疆农业大学在2001年以RAPD资料建立起新疆部分栽培油菜的核心资源[26];何余堂等[27]从199个白菜型油菜资源中初步建立了含有 28个品种的核心资源;赵绪涛等[23]对118份甘蓝型油菜进行核心种质的构建。本研究通过SLAF-seq技术获得了249.91Mb的读长数据,开发得到527 872个SLAF标签和842 248个群体SNP,为提高育种遗传效率和开发特早熟甘蓝型油菜特异性 SNP提供参考,可进一步加快甘蓝型油菜的遗传育种改良。
虽然97份材料来源于同一组合,但5类材料之间仍具有一定的遗传差异,可指导杂交育种,遗传差异大的材料杂交后代可能具有超亲优势。使用Core Hunter软件,得到了C30和C40这2个核心子集,从各项指数看,这2个子集都能够代表原始材料,最终选取C30子集作为核心子集。但是甘蓝型油菜相比于其他2种类型的油菜,具有明显较小的遗传差异。因此局限于材料的来源,为更好地增加遗传多样性,还需要继续扩宽亲本特早熟甘蓝型油菜的种质资源,增加遗传丰度。
青海特早熟甘蓝型油菜较其他地区油菜品种能够早熟1个月,具有在2900m海拔以上且年平均温度在0.5℃以下的区域种植的特点,比该地区主栽品种浩油11号增产约10%[28]。特早熟甘蓝型油菜的育种目标是要使植株对光、温的需求接近白菜型油菜的水平,其中生育期是主要的育种目标[29],在特早熟甘蓝型油菜育种中选取开花期作为熟性选择的指标[30]。目前在我国高海拔地区已经选育出了特早熟甘蓝型油菜品种“青杂4号”和“青杂7号”,“青杂4号”适合种植在海拔2900~3100m的地区,“青杂7号”适合种植在海拔2750~2950m的地区,但在3100m以上的高海拔地区仍然主栽白菜型油菜,因此,选育更早熟的甘蓝型油菜品种对于这些地区油菜品种改良具有重要意义。
4 结论
通过 SLAF-seq对 97份特早熟甘蓝型油菜恢复系构建了核心亲本子集,有利于特早熟恢复系种质资源的保存、评价和利用,从而减少育种工作中恢复系与不育系选配杂交组合的工作量,为生产中代替高海拔地区白菜型油菜的优良特早熟甘蓝型油菜杂交种提供更多可能和选择,扩大春油菜区甘蓝型油菜的种植区域,以及提高高海拔地区油菜的产量和品质。
利用SNP构建核心亲本子集,得到包含29份材料的C30子集和包含38份材料的C40子集,通过各项数据表明,这2个核心子集能代表整个资源的群体遗传多样性水平,并最终将C30子集作为核心子集。构建了97份特早熟甘蓝型油菜恢复系的核心种质,为种质资源的开发、利用和保存提供了参考。

