胃癌转移相关长链非编码RNA差异表达谱的研究
王亚芳,刘向强,章思偲,刘理礼(中国人民解放军南部战区总医院放射治疗科,广州 5000;中国人民解放军南部战区总医院消化内科;中国人民解放军空军军医大学第二附属医院肿瘤科;通讯作者,E-mail:lilyfmmu@6.com)
胃癌是全球最常见的恶性肿瘤之一,为世界范围内第三高发肿瘤。根据2018年中国恶性肿瘤报告显示,在我国胃癌发病率位居第二位,仅次于肺癌;其所致死亡率已高居第三位[1]。侵袭和转移是恶性肿瘤的重要特征,是导致胃癌患者死亡的最主要原因[2]。然而胃癌的侵袭转移是一个涉及多分子、多步骤、多机制的过程[3];因此寻找能够多层面调控转移相关基因的枢纽分子是解决问题的根本所在。
长链非编码RNA(lncRNA)是一类转录本长度超过200 nt的非编码RNA,占人类总RNA的4%~9%,远高于仅占人类总RNA 1%~2%的蛋白编码基因[4,5]。研究表明其具有“一对多”和“多对一”的枢纽功能,可在多层面、多阶段调控基因表达[3-6]。近年来越来越多的研究证实lncRNAs的异常表达在胃癌发生发展过程中发挥着重要的调控作用,并参与了胃癌增殖、侵袭转移等多条相关信号通路,与胃癌患者的预后密切相关[3,7-9]。因此,获得胃癌转移相关的差异lncRNA,靶向lncRNA分子的干预能够更有效地预测和识别高转移风险的胃癌,并促进新治疗策略的研发。
本研究应用高通量lncRNA芯片技术,比较未发生远处转移胃癌组织与发生远处转移胃癌组织中lncRNA的差异表达情况,以筛选与胃癌侵袭转移相关的lncRNAs,为阐明胃癌侵袭转移的分子机制和开发胃癌诊断治疗的靶标提供新的思路。
1 材料与方法
1.1 材料
胎牛血清(美国Hyclone公司);RPMI-1640培养液,胰酶(美国,Gibco公司);提取RNA的Trizol(美国,Invitrogen公司);反转录试剂盒和SYBR Green real-time PCR Masker Mix试剂盒(日本,TaKaRa公司);real-time PCR引物由TaRaKa公司设计并合成;Human lncRNA expression arrays的芯片选择、探针设计、图像采集、数据分析等由上海康成生物工程有限公司完成。
1.2 方法
为明确发生远处转移的胃癌与未发生远处转移胃癌组织中是否具有差异性表达的lncRNAs,我们利用高通量lncRNA芯片技术分析发生远处转移的胃癌原发灶组织与未发生远处转移的胃癌原发灶组织中的lncRNAs表达差异,利用箱式图、火山图、散点图等分析芯片数据的可靠性,并进一步挑选具有统计学意义的lncRNA分子进行细胞验证及功能研究,予以寻找可抑制胃癌转移的关键lncRNA分子。
1.2.1 细胞及新鲜组织收集 2对发生远处转移/未发生远处转移胃癌细胞系:SGC7901-M(metastasis)/SGC7901-NM(non-metastasis)及MKN28-M(metastasis)/MKN28-NM(non-metastasis),以上细胞株均冻存于西京消化病医院肿瘤生物学国家重点实验室,于液氮罐中长期保存。相关细胞系在含10%胎牛血清的RPMI-1640培养液(Gibco)中培养。收集经手术或穿刺病理证实为腺癌的胃癌组织6例,经全身影像学检查后,证实其中3例胃癌组织未发生远处转移(M0),另3例发生远处转移(M1)。全部未接受放疗或化疗。以未发生远处转移胃癌组织作为无转移胃癌组(阴性组),以远处转移的肿瘤组织作为高转移胃癌组(阳性组)。全部标本经-80 ℃冻存。取材前向患者告知试验用途,签署知情同意书,并上报医院伦理委员会批准。
1.2.2 组织总RNA的提取与检测 组织样本的预处理是先将组织样品在液氮中磨成粉末,待液氮挥发迅速加入TRIzol研磨。上述样本均按照Trizol(Invitrogen)说明书提取总RNA,并用无RNase的DNase Ⅰ进行处理以去除基因组DNA的污染。紫外分光光度计测定吸光度值OD260和OD280,对总RNA进行纯度分析;并计算OD260/OD280值,结合甲醛变性凝胶电泳,分析RNA质量。
1.2.3 芯片的选择 利用Arraystar公司12×135k的lncRNA芯片,覆盖目前NCBI Refseq、UCSC Known Gene、NRED、RNAdb、UCRs等多个数据库的lncRNAs。
1.2.4 探针设计 Arraystar lncRNA芯片设计探针为约60 met的长寡核苷酸,这些长寡核苷酸探针在高严格杂交条件下可得到高灵敏度及高特异性的理想实验结果。针对每条序列都设计了多条探针,增加了信号的可靠度。
1.2.5 cDNA样品合成、标记和杂交 利用Invitrogen公司提供的ds-cDNA合成试剂盒进行逆转录,合成双链互补DNA;利用Nimblegen公司的单色DNA标记试剂盒进行双链互补DNA的标记,使用分光光度计检测荧光标记效率,以保证后续芯片实验结果的可靠性;利用Nimblegen提供的杂化系统进行排列杂交,然后利用Nimblegen清洗液试剂盒进行清洗。
1.2.6 图像采集和数据分析 利用Axon基因芯片4000B芯片扫描仪(分子设备公司)扫描芯片的荧光强度,然后将扫描图像(TIFF格式)输入Nimble Scan软件(版本号2.5)读取原始数据,以进行网格对齐与表达数据分析。通过分位数标准化以及NimbleScan软件将表达数据进行规范,然后将规范化处理后的数据上传至Agilent GeneSpring Software软件(版本11.0)进一步分析,通过设置数据滤过的比值获取差异表达的lncRNAs。差异表达lncRNAs或mRNAs通过Pvalue/FDR(false discovery rate,错误发现率)筛选,P值利用t检验计算,并根据BenjaminiHochberg FDR方法进行修正(即FDR值),筛选标准为|Fold change|≥2.0且FDR<0.05。对差异表达的基因进行GO和KEGG分析,使用编写脚本进行层次聚类和关联分析。
1.2.7 逆转录实时定量PCR验证 挑选差异倍数较大的特异性差异分子lncRNA-uc341进行real-time PCR验证。分别以来自胃癌发生远处转移细胞系SGC7901-M和MKN28-M以及对应的未发生远处转移的胃癌细胞系SGC7901-NM及MKN28-NM的cDNA分子为模板,扩增目的基因,以GAPDH基因作为内参照。采用Bio-Rad荧光定量PCR仪,SYBR Green荧光染料(TaKaRa公司)掺入法相对定量。每个样本同时做3个复孔,根据获得的Ct值进行基因相对定量分析,以2-ΔΔCt计算高转移细胞系与低转移细胞系中目的基因的相关表达水平。lncRNA-uc341上游引物序列:5′-AGAGCGGAAGGAAGAAGAGGTG-3′,下游引物序列:5′-GTCTTGCTAATCTCCAGGCGG-3′;GAPDH上游引物序列:5′-TGACTTCAACAGCGACACCCA-3′,下游引物序列:5′-CACCCTGTTGCTGTAGCCAAA-3′。
1.2.8 统计学方法 以SPSS 17.0软件对数据进行统计学分析,计量资料采用独立样本t检验分析,以P<0.05表示差异有统计学意义。
2 结果
2.1 lncRNA芯片杂交图像
lncRNA芯片信号清晰,无边缘化效应(见图1)。

图1 未发生远处转移胃癌组织与发生远处转移胃癌组织的lncRNA芯片Figure 1 lncRNA array images of gastric cancer without metastasis and gastric cancer with high-metastasis
2.2 箱式图分析
箱式图体现了芯片扫描后各个样本荧光密度值标准化之后的分布,其中红色代表标准化后溢出的数据,即偏倚出去的异常值;蓝色箱子中间的一条线代表了数据的中位数,即样本数据的平均水平;横线的上下分别代表了数据的上四分位数和下四分位数。从图2我们可以观察到其平均值基本保持在同一水平,验证了芯片扫描读数的稳定性及基因的真实性,排除了因实验技术误差而导致结果误差的可能性。
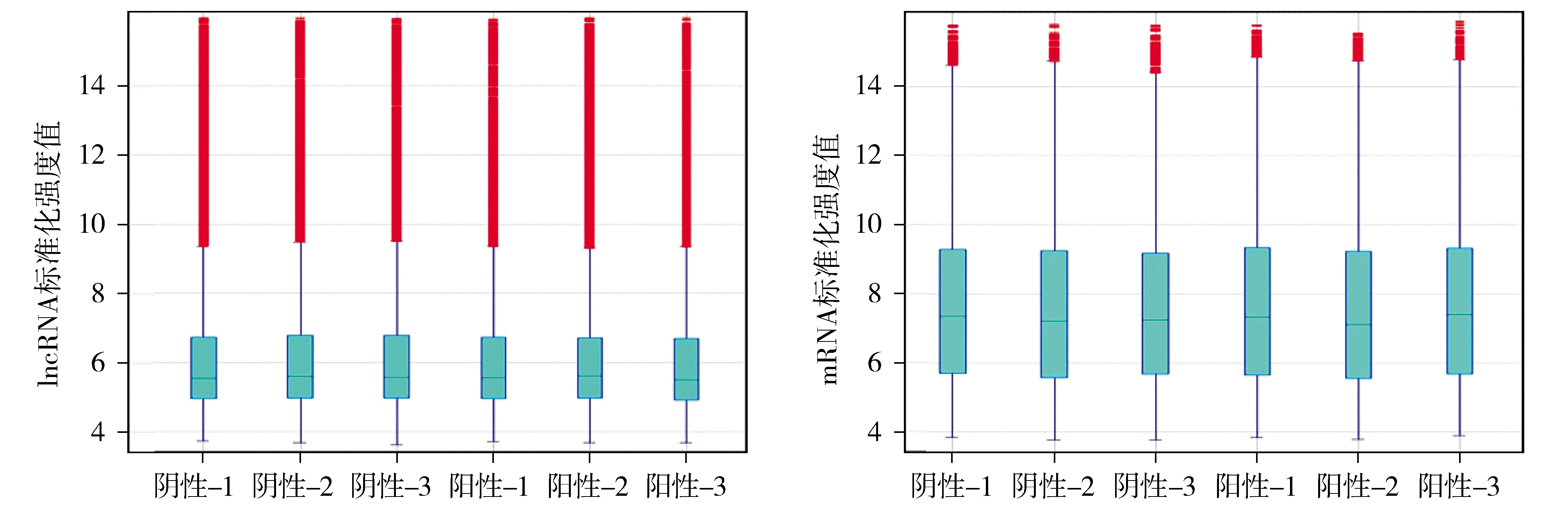
红色表示偏倚出去的异常值;蓝色线表示中位数和上下四分位数图2 lncRNA芯片中lncRNA及mRNA数据的箱体图Figure 2 Box plot of lncRNA and mRNA data in lncRNA microarray
2.3 散点图分析
芯片数据的散点图由芯片分析的原始数据经标准化处理后在一个二维执教坐标系屏幕中绘制而成,结果可以观察到两组之间数据总体分布趋势集中(见图3),体现了芯片数据处理的可靠性。散点图中,自蓝色至红色代表该区域的基因数目,可在图中看到大多数基因处于差异较小的区域。我们亦观察到绿线之外的散点在信号强弱上具有明显差异(见图3),提示未发生远处转移与发生远处转移的胃癌组织中有相关基因表达的差异性。
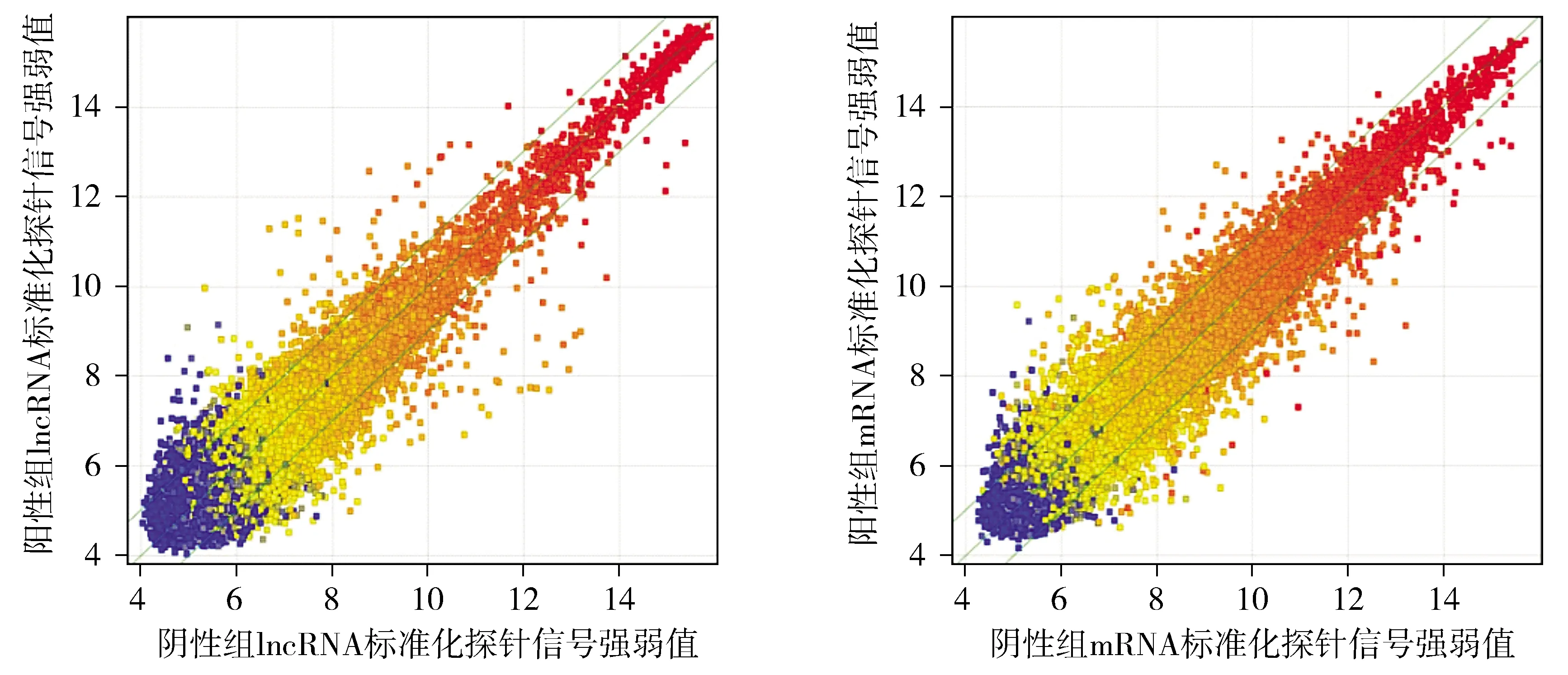
图3 lncRNA芯片中lncRNA及mRNA数据的散点图Figure 3 Scatter plot of lncRNA and mRNA data in lncRNA microarray
2.4 火山图
芯片火山图可直观地显示芯片结果中重要的两个指标:变化倍数和P值。我们可以观察到阴性与阳性两组之间具有差异表达的基因(红色)(见图4)。
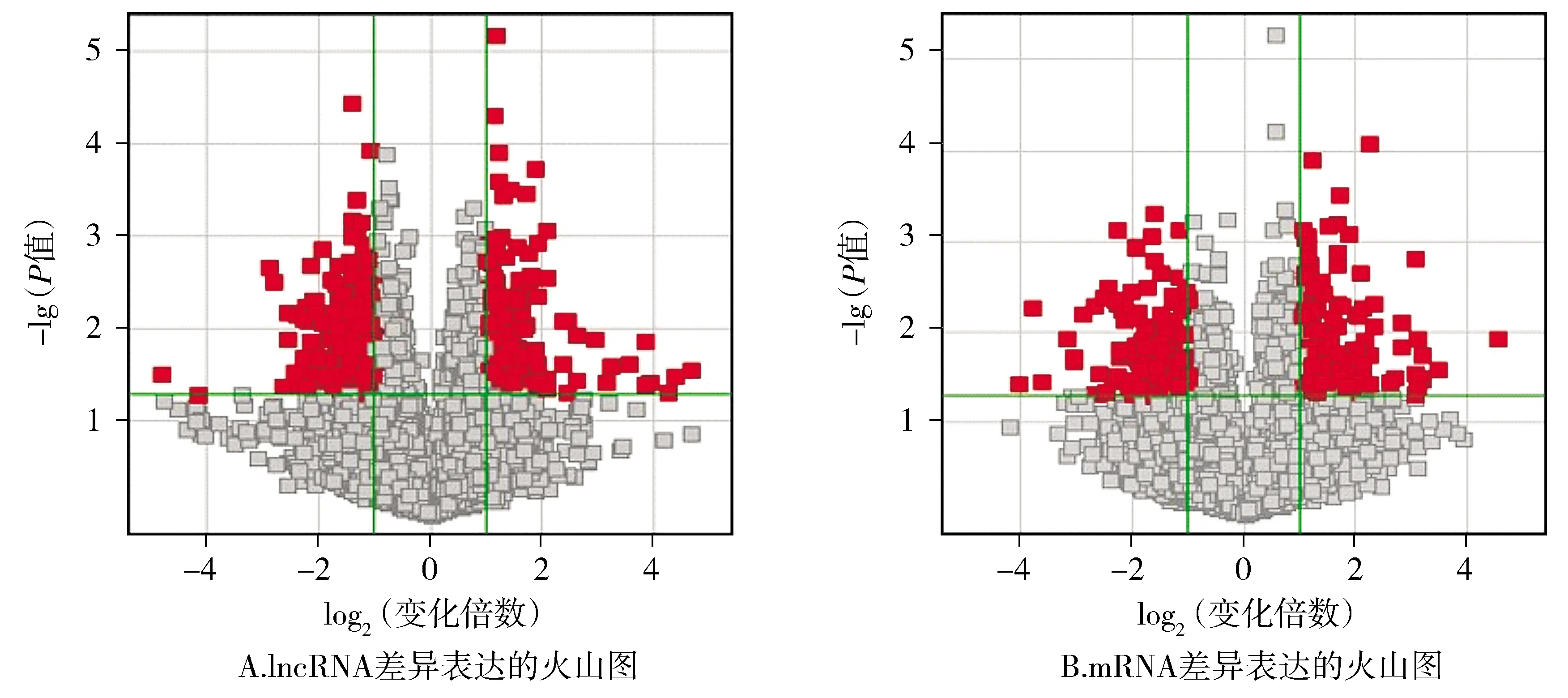
图4 差异性表达的lncRNA及mRNA的火山图Figure 4 Volcano plots of differentially expressed lncRNA and mRNA
2.5 差异表达的lncRNAs和mRNAs
根据芯片分析结果,阳性组(发生远处转移胃癌组织)与阴性组(未发生远处转移胃癌组织)相比,有549个lncRNAs差异性表达具有统计学意义,其中阳性组中表达升高的为230个,表达下降的为319个;同时检测到两组之间有386个mRNAs差异性表达具有统计学意义,其中阳性组中上调的为208个,下调的为178个。在差异性表达的lncRNAs中,阳性组中表达升高5倍以上的共9个,10倍以上的共4个;表达下降5倍以上的共11个,10倍以上的共2个。在阳性组中表达升高倍数在5倍以上的lncRNAs信息见表1;在阳性组中表达降低倍数在5倍以下的lncRNAs信息见表2。
2.6 DEGs的GO分析
使用DAVID数据库,对差异性表达的lncRNAs靶基因进行Gene Ontology的生物学分类,可获得lnc-RNAs靶基因对应的显著性功能,从而了解lncRNAs可能参与的生物学功能。差异性表达的mRNAs共富集到198条biological process(BP)术语、21条cellular component(CC)术语、36条molecular function(MF)术语。差异性表达的mRNAs富集到的GO术语前10位(按P值排序的差异显著性排名)见图5-7。差异性表达的mRNAs在生物过程中,主要富集到的功能有对生长激素刺激的反应、淋巴结浸润、血管平滑肌收缩、TCR信号通路的调控等相关过程(见图5);在细胞组分上,显著富集到的组分有细胞质膜、多泡体、肌球蛋白丝、SWI/SNF复合体等(见图6);在分子功能上,显著富集到的功能有肿瘤坏死因子受体活化、结合肿瘤坏死因子、结合白介素-1等(见图7)。
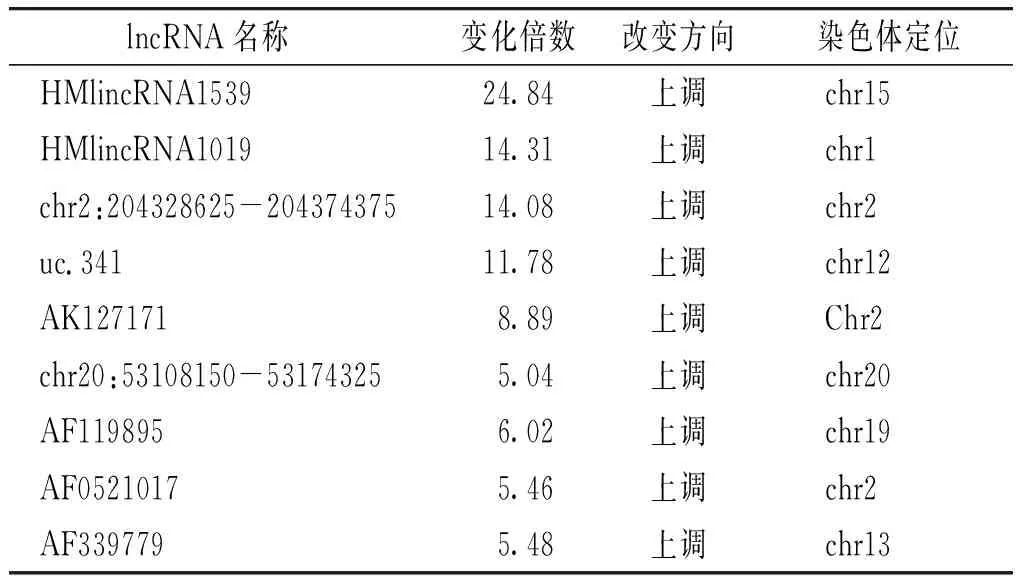
表1 发生远处转移胃癌组织与未发生远处转移胃癌组织中的差异性表达的lncRNAs(上调≥5倍)
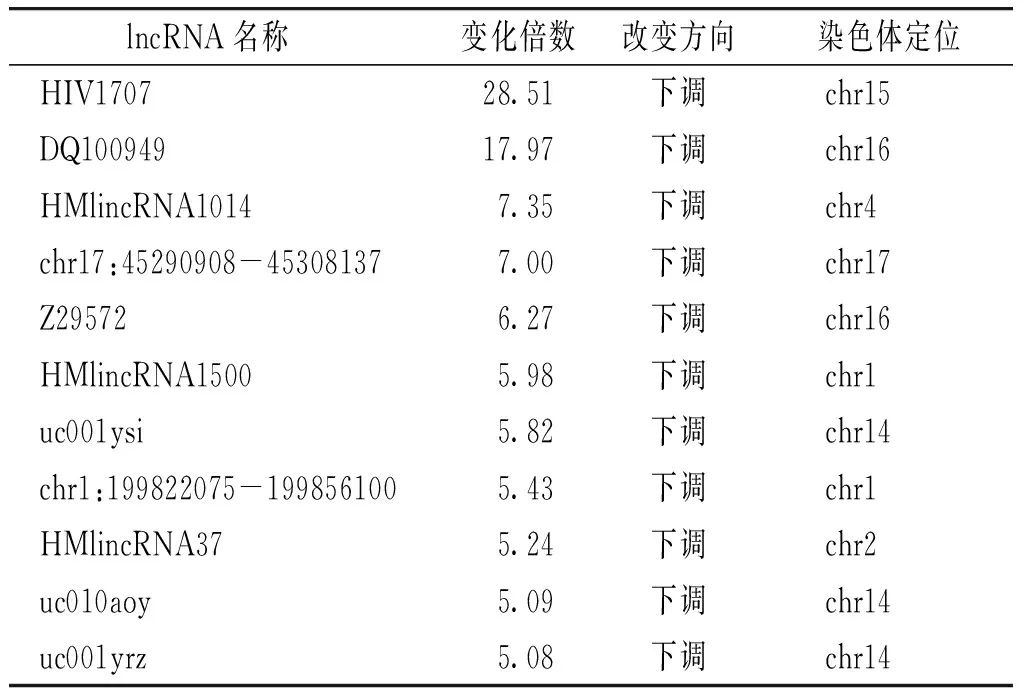
表2 发生远处转移胃癌组织与未发生远处转移胃癌组织中的差异性表达的lncRNAs(下调≥5倍)
2.7 DEGs的KEGG生物学通路分析
对差异性表达的mRNAs经KEGG生物学通路分析(pathway analysis),发现其主要在7条信号通路中富集程度比较高,其中最重要的是细胞因子及其受体相互作用信号通路(见图8)。
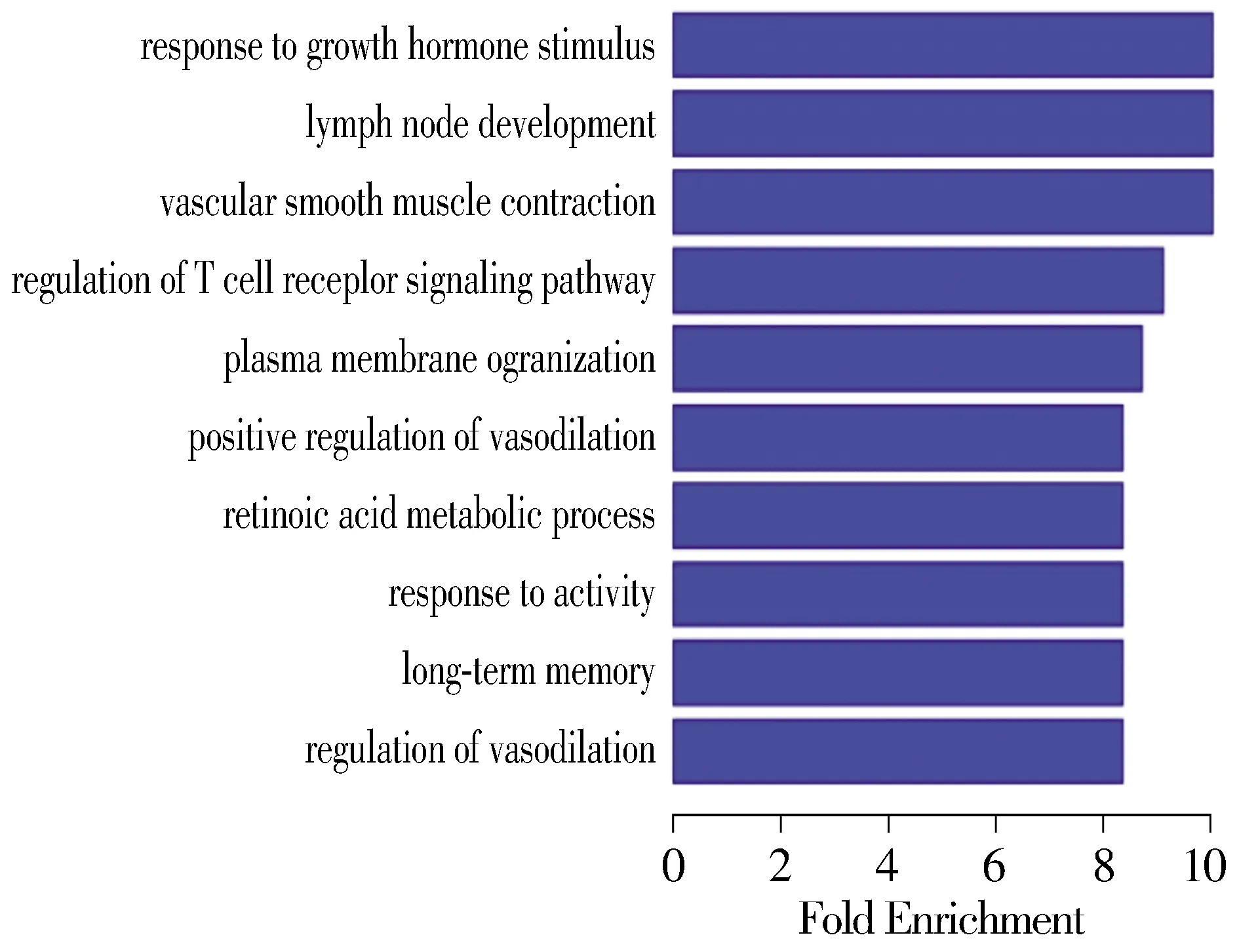
图5 GO聚类分析差异性表达mRNAs参与的生物过程Figure 5 GO cluster analysis of differentially expressed mRNAs in biological process
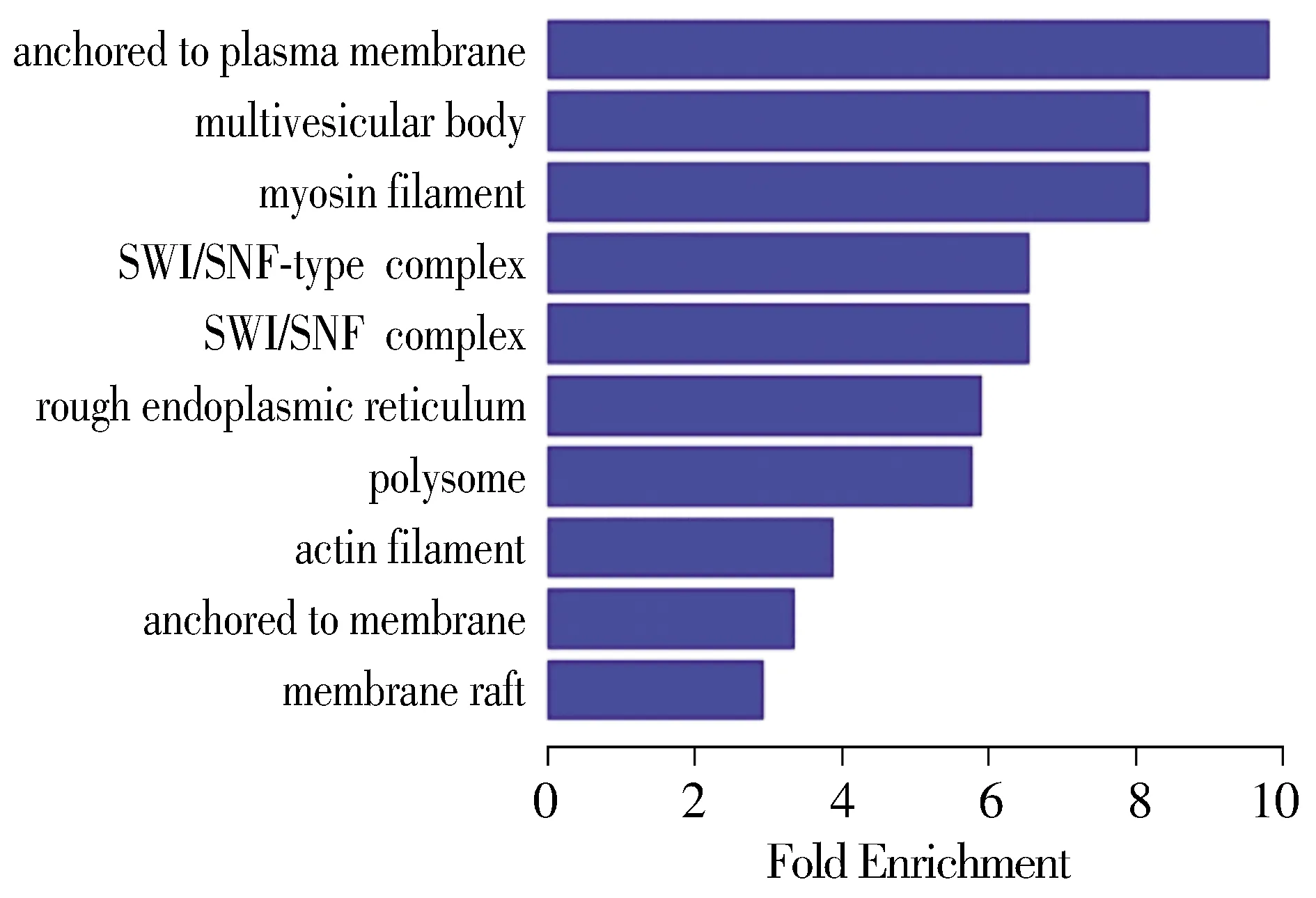
图6 GO聚类分析差异性表达mRNAs参与的细胞成分Figure 6 GO cluster analysis of differentially expressed mRNAs in cellular component
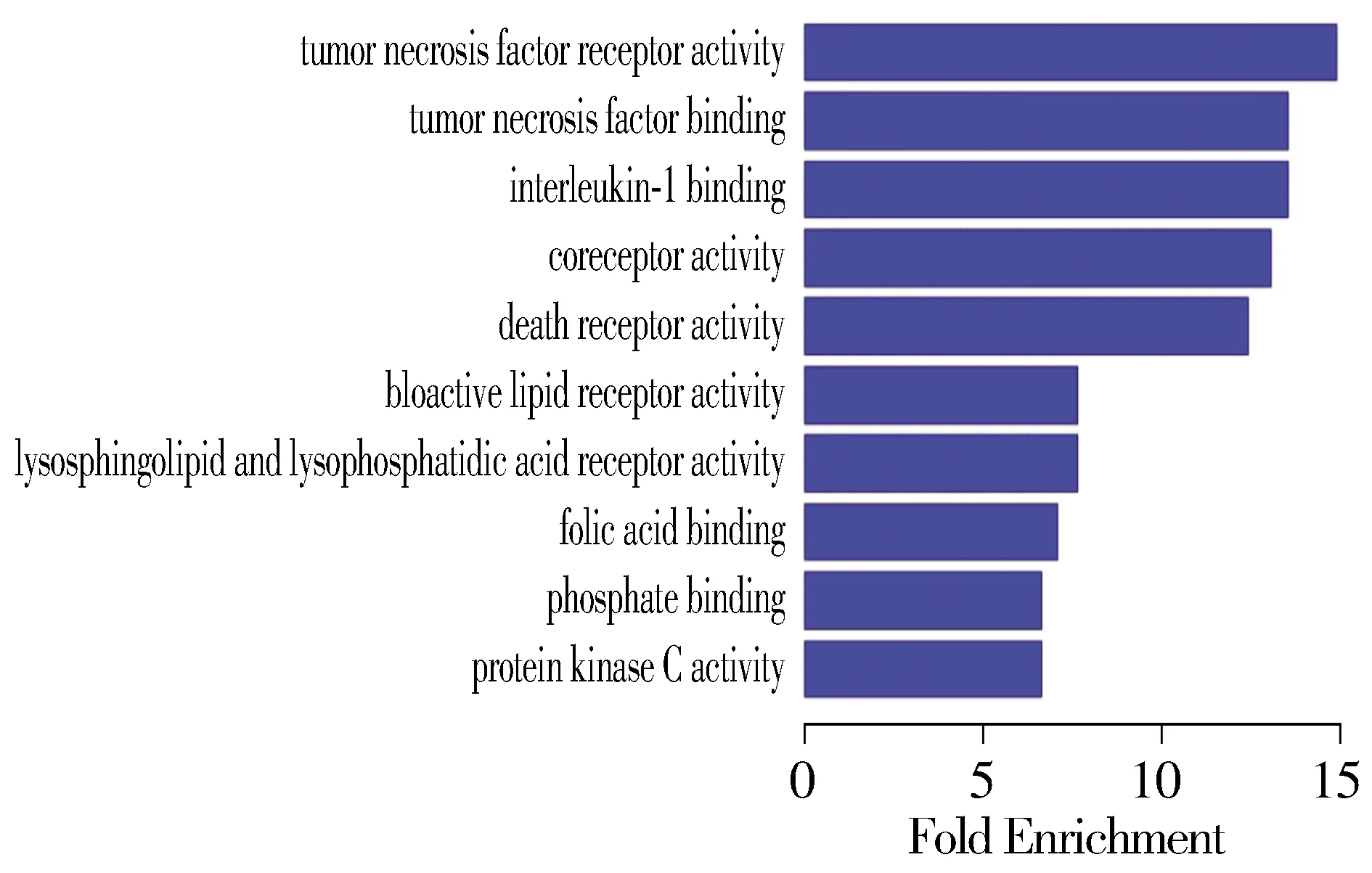
图7 GO聚类分析差异性表达mRNAs参与的分子功能Figure 7 GO cluster analysis of differentially expressed mRNAs in molecular function
2.8 逆转录实时定量PCR验证结果
与发生远处转移的胃癌细胞系(MKN28-M及SGC7901-M)相比,lncRNA-uc341在未发生转移的胃癌细胞系(MKN28-NM及SGC7901-NM)中的表达降低,并具有统计学意义(见图9)。
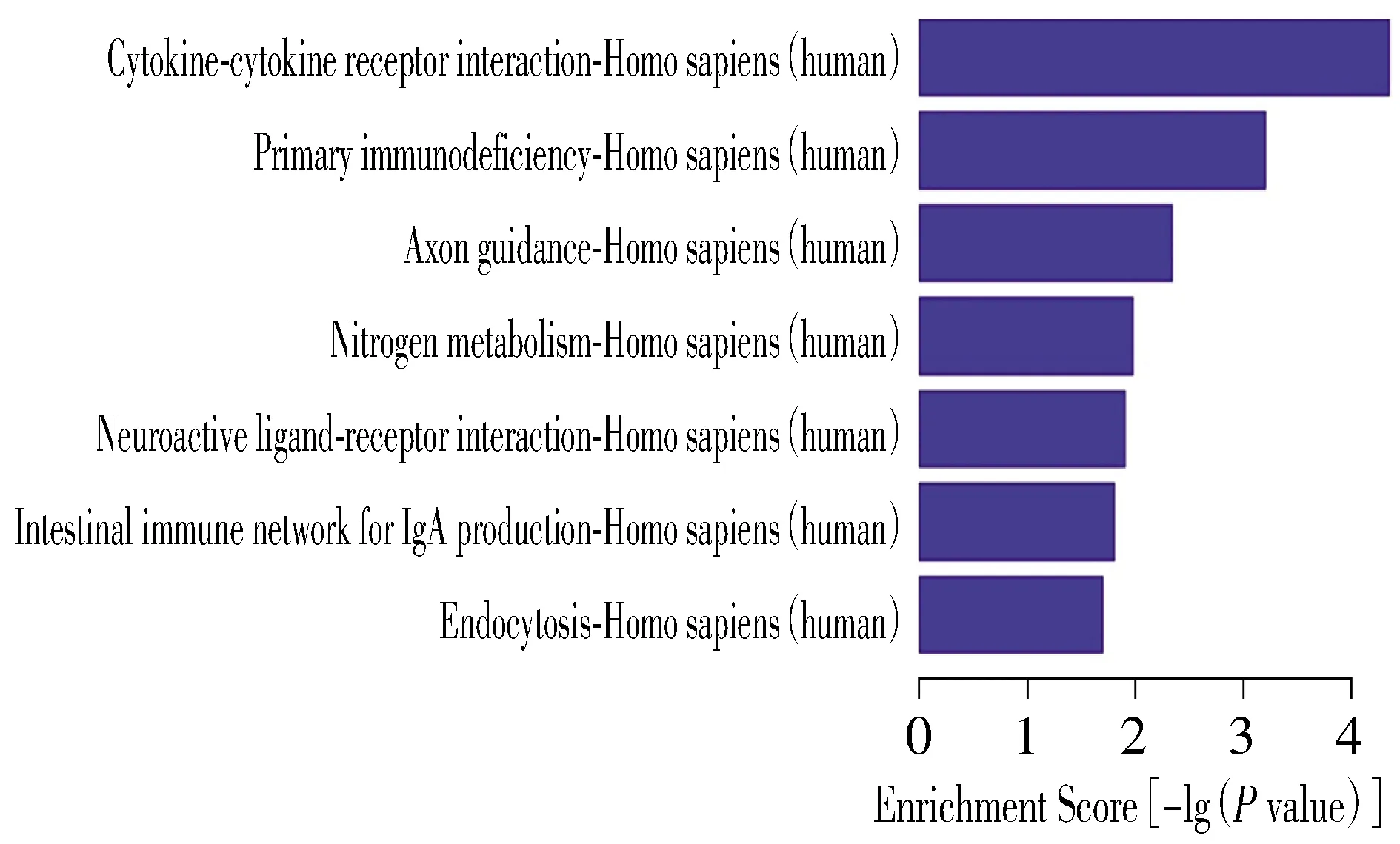
图8 差异性表达基因可能参与的调控机制通路Figure 8 KEGG pathways of differentially expressed genes
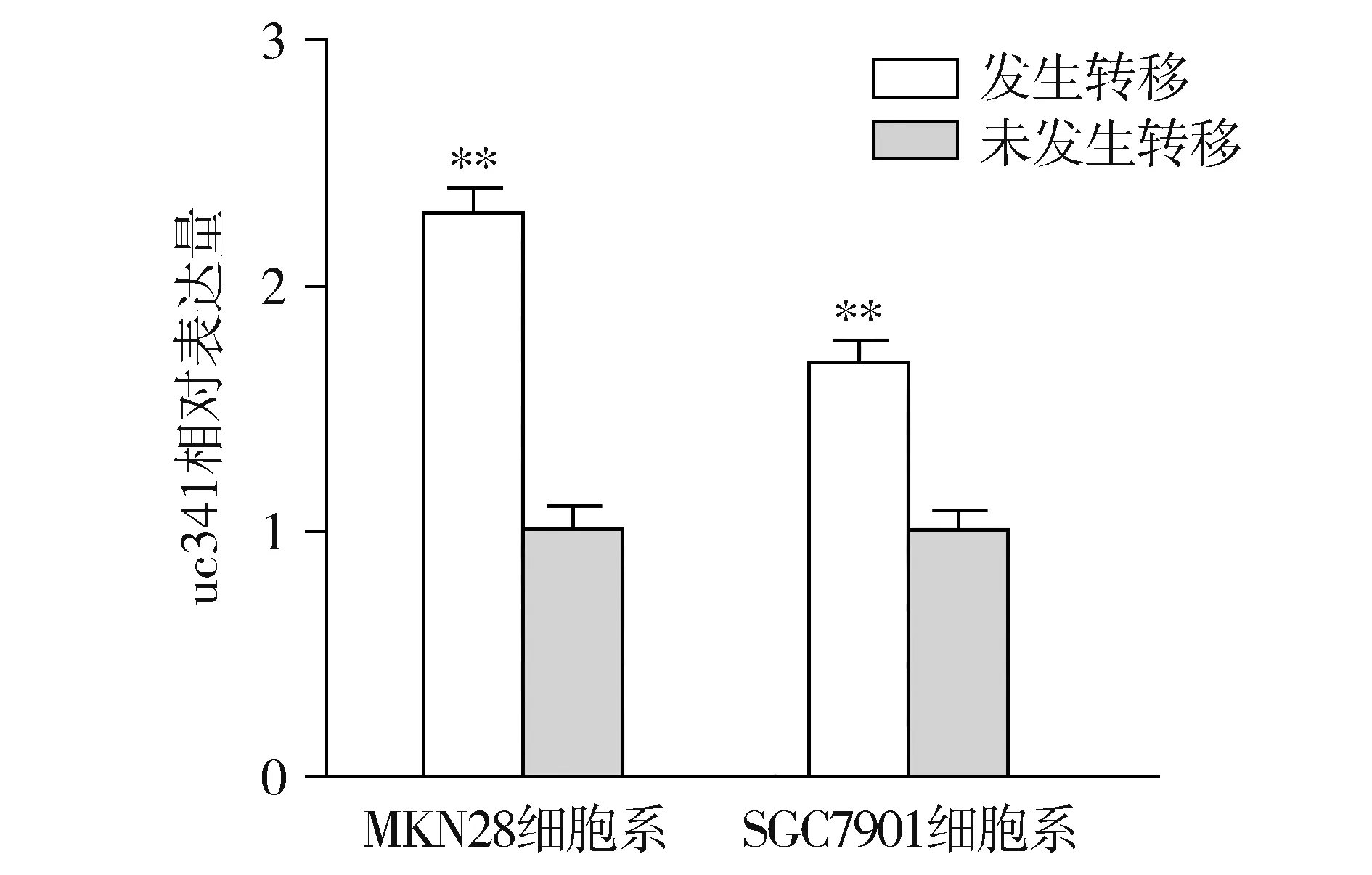
与未发生转移相比,**P<0.01图9 定量PCR检测差异表达分子lncRNA-uc341的表达Figure 9 Differentially expressed lnRNA-uc341 in MKN28 cells and SGC7901 cells by qRT-PC
3 讨论
胃癌是我国最常见的恶性肿瘤之一,发病率及死亡率分别高居第二、三位。因早期症状隐匿且进展迅速,约40%的胃癌患者发现时已伴有转移。尽管在胃癌的诊断及治疗方面取得了部分进展,但转移性胃癌患者的5年生存率仍不足30%[1,2,10-12]。胃癌的侵袭转移是一个多分子、多表型变化的多步骤过程[3]。因此,寻找可在多层面、多阶段调控胃癌转移相关的特异性因子,阐明胃癌转移的分子机制是解决问题的关键所在。
近年来,具有器官及时空特异性的lncRNA研究揭示了其调控作用的多样性,尤其是其在整个转移级联反应中的关键作用[3,13-15]。lncRNA是一类长度超过200个核苷酸的RNA转录本。已有大量研究证实其通过多种机制参与了肿瘤侵袭转移的各个步骤过程[3,6]。lncRNA-H19作为支架分子,促进EZH2和H3K27me3与E-cad的启动子结合,抑制E-cad的表达,促进了膀胱癌的侵袭转移[16];lncRNA-ATB通过参与TGF-β/ZEB1/ZEB2通路诱导肝癌发生EMT及侵袭转移[17];lncRNA-MALAT1通过定位到核斑点和改变共转录的选择性剪接来调节肺癌侵袭转移[18]。另有研究证实lncRNA-SChLAP1的过表达对前列腺切除术后10年内发生转移具有独立预测作用[19];在胃癌相关研究中,已证实lncRNA-PANDAR可以竞争性结合p53蛋白调控CDKN1A基因转录,从而促进胃癌细胞的发展及淋巴结转移,提示其可作为胃癌诊断的标记物和预后检测的靶标[20];lncRNA-HOXC-AS3通过调节转录因子YBX1与其靶基因启动子区域之间关联,从而介导胃癌增殖、侵袭转移[21]。但目前大多数胃癌相关lncRNA的筛选多集中于癌旁与癌组织的差异性表达谱筛选,且只有一小部分进行了相关功能机制研究。因此,获得胃癌侵袭转移相关的lncRNAs,靶向转移相关lncRNA分子的干预能够更有效地预测和控制多因素、多基因变化的胃癌侵袭转移。
为筛选与胃癌侵袭转移相关的特异性lncRNA,我们借助Arraystar公司的lncRNA芯片,以3例未发生远处转移胃癌组织对比3例发生远处转移胃癌组织,精确筛选与胃癌转移相关lncRNA;并进一步对其进行相关的生物功能及信号通路的深入分析预测。最终筛选出与胃癌侵袭转移相关的具有统计学意义的差异性表达lncRNAs。其中在胃癌侵袭转移组中有230个上调的lncRNAs,319个下调的lncRNAs;差异倍数≥5倍的上调lncRNA为9个,下调lncRNA为12个。以上数据提示我们这些差异性表达的lncRNAs可能参与了GC的侵袭转移过程。随着lncRNA在肿瘤进展中研究的深入,部分lncRNA的生物学功能及调控机制也逐渐清晰,但对于阐明占据总RNA 4%~9%的lncRNA的功能机制仍只是冰山一角。本研究通过进一步GO分析发现,这些差异性表达lncRNA功能涉及到肿瘤细胞的生长、代谢、浸润、免疫调节等多种生物过程,提示我们lncRNA可能通过参与生物过程的不同环节促进胃癌的侵袭转移。进一步进行KEGG pathway分析后,获得了相关差异分子可能参与的肿瘤通路。差异性的mRNA参与了7条信号通路,这些通路大部分都包含有细胞因子、免疫、代谢等相关分子和路径。富集程度最高的是细胞因子受体相互作用通路,其中包括了TGF-β信号通路、EGF、VEGF等信号通路,与目前的关于肿瘤侵袭转移信号通路研究基本吻合;已有多项研究证实lncRNA通过调控TGF-β信号通路参与肿瘤侵袭转移[17,22,23]。为进一步验证芯片结果的准确性,我们依据差异倍数及分子特点,挑选总长为314 bp的uc341进行进一步验证,利用胃癌高低转移细胞系进行RT-PCR检测后,初步证实uc341在高转移胃癌细胞系中表达高于低转移细胞系,与芯片中uc341的表达趋势一致;但因胃癌细胞对验证分子转移特性的相对有限性,下一步我们拟利用生物信息网络分析,深入探讨芯片中具有统计学差异的相关分子,在大样本的胃癌组织中进行临床验证分析,并进一步分析其发挥功能的机制,以期寻找到与胃癌侵袭转移密切相关的靶点。
综上所述,本研究初步证实了发生远处转移的的胃癌组织与未发生远处转移的胃癌组织相比,lncRNA表达谱的变化具有统计学意义,这些变化的lncRNAs可能与胃癌的侵袭转移有关,但肿瘤组织具有高度的异质性,因此这些lncRNA是否可以成为胃癌侵袭转移预测的标记物或者治疗的靶点,还需要进一步的临床标本验证分析及深入的功能机制研究。

