花生高亲和硝酸盐转运蛋白基因AhNRT2.7a响应低氮胁迫的功能研究
王娟,陈皓宁,2,石大川,于天一,闫彩霞,孙全喜,苑翠玲,赵小波,牟艺菲,王奇,李春娟,单世华
花生高亲和硝酸盐转运蛋白基因响应低氮胁迫的功能研究

1山东省花生研究所,山东青岛 266100;2中国海洋大学食品科学与工程学院,山东青岛 266000;3青岛市农业科学研究院,山东青岛 266100
【目的】氮素在作物生产中对生物量和产量起关键作用。高亲和硝酸盐转运蛋白基因NRT2在植物响应低氮胁迫时被激活并具有维持氮吸收和转运的作用。通过筛选花生低氮耐受相关的NRT2基因并解析其生物学功能,为培育高氮素利用率的花生新品种提供理论参考,最终有助于实现节氮、高效的绿色生产目标。【方法】检测正常氮浓度及1/20正常氮浓度(15 mmol·L-1)条件下5个具有典型跨膜结构的花生NRT2基因(、、、和)的时空表达情况。以花育6309的cDNA为模板,对进行克隆和生物信息学分析,并通过亚细胞定位确定的表达部位。进一步构建异源过表达的拟南芥株系,分别在正常以及低氮胁迫条件下测定其叶绿素含量、氮积累量以及谷氨酰胺合成酶(GS)、谷氨酸合成酶(GOGAT)、硝酸还原酶(NR)、亚硝酸还原酶(NiR)、谷氨酸脱氢酶(GDH)5个氮代谢关键酶的活性。【结果】5个NRT2基因中有4个NRT2基因在花生响应低氮胁迫条件下大量表达。其中,能够响应低氮胁迫,并在花生茎和叶中高表达。获得的cDNA序列,全长为1 380 bp,编码459个氨基酸。蛋白结构分析显示其为具有12个典型跨膜结构域的膜蛋白。该氨基酸序列与栽培种花生(L.)相似性高达99.56%,其次是野生亲本AA和BB基因组花生。亚细胞定位显示其主要定位于细胞质膜上。构建异源过表达的转基因拟南芥植株,在不同供氮条件下,成熟叶和幼叶叶片的叶绿素相对含量均显著高于野生型拟南芥。同时,对上述5个氮代谢相关酶活性以及氮、磷、钾积累量测定显示,转基因植株中2个氮代谢相关酶(GS和NR)的酶活性以及氮积累量均比野生型拟南芥有显著升高。【结论】花生中4个NRT2基因响应低氮胁迫,其中能够提高植物氮代谢过程的氮素利用率。的表达在促进氮代谢过程的同时,促使碳代谢作用的进一步加强。因此,适合作为花生氮素高效利用为目的的候选基因。
花生;;;氮素效率;氮代谢相关酶
0 引言
【研究意义】花生(L.)是重要的油料作物和经济作物,在保障中国食用油脂安全方面具有重要作用[1]。花生除了根瘤菌固氮外,根系吸收土壤中的硝态氮也是氮素利用的一种重要方式,占花生需氮量的50%—60%[2-4]。氮素参与了氨基酸、蛋白质和磷脂等必需物质的合成过程,同时也作为信号分子调控植物的形态建成、生理响应以及相关基因的表达。因此,氮素是影响花生生长发育和产量形成的重要因素[5]。合理施用氮肥不仅可以提高花生的产量和品质,而且能够更有效地发挥根瘤的固氮作用。然而,目前投入的氮肥仅约30%可以被吸收并转化为农业生产力,过量施用氮肥不仅导致花生根瘤数目减少,自身固氮能力下降[6],同时还造成一系列的环境污染问题,如土壤酸化板结、氮磷失衡、水体污染、病虫害易发等[7-10]。因此,选育高氮素利用率(nitrogen use efficiency,NUE)的低氮耐受型花生新品种,对花生生产的绿色协调发展具有重要意义。【前人研究进展】陆地植物中氮素的吸收、转运和储存主要由硝酸盐转运蛋白(nitrate transporter,NRT)来完成[5]。当NO3-浓度低于1 mmol·L-1时,其吸收主要依靠由高亲和硝酸盐转运蛋白(NRT2)转运蛋白组成的高亲和转运系统(high-affinity transport system,HATS)完成。NRT2转运蛋白属于膜蛋白,一般具有典型的10—12个跨膜蛋白结构,且只把硝酸盐作为特异性底物[11-12]。NRT2能够在植物响应低氮胁迫时被激活并发挥主导作用[5, 13],具有维持低氮条件下氮转运的作用[14-15]。因此,NRT2基因适合作为筛选高氮素利用率的低氮耐受型花生新品种的目标基因。目前,植物高亲和硝酸盐转运蛋白基因(NRT2)功能机制研究取得一定的进展。如,拟南芥()7个AtNRT2家族基因成员中,和负责拟南芥中硝酸盐的吸收,主要在主根的较老部分表达;在主根的幼嫩部分和侧根的远端区域表达;在主根和侧根的根毛区表达,二者在受到低氮胁迫时才会诱导表达[16-17]。玉米(s)ZmNRT2.1与NAR2蛋白相互作用,在其初生根的诱导型高亲和转运系统(iHATS)的硝酸盐吸收中起主要作用[18-19]。水稻()的5个OsNRT2基因中,在根周围的薄壁细胞中表达,负责木质部NO3-从根到地上部的长距离运输[20]。在叶片韧皮部表达,在NO3-再活化和韧皮部pH平衡中起关键作用[21]。豆科植物苜蓿()在根中高表达,并与苜蓿氮素吸收有关[22]。以上研究表明,不同作物中,各NRT2基因的表达部位具有组织特异性,多数作物的NRT2基因均具有在低氮条件下促进植物对硝酸盐的吸收和转运的功能,从而参与了响应低氮胁迫的生理过程[23-24]。除此之外,NRT2家族基因还可能与作物的氮素利用效率相关。Chen等[25]通过过量表达获得转基因植株,与野生型相比,其生物量、籽粒产量和氮素利用率均出现显著提升。Fan等[21]发现在水稻中超表达能够极大提高水稻的氮素利用率和产量。然而,目前尚未见有关花生基因与氮素利用效率的报道。【本研究切入点】栽培种花生属于异源四倍体(AABB)植物,亚基因组间的高度相似性以及花生遗传转化技术不稳定性对花生基因功能验证造成了困难。近年来,研究表明花生基因组信息的释放[26-28]以及拟南芥、烟草等模式植物转基因异源研究体系为花生功能基因研究提供了有效途径[29]。花生氮素利用相关基因功能研究是进行花生遗传改良的重要理论基础。目前已发掘和鉴定的花生氮素利用相关途径关键基因还十分有限,相关基因的功能解析和表达调控的研究也尚未见报道。前期王娟等[30]利用栽培种花生全基因组关联分析和不同组织的转录组信息共鉴定10个NRT2基因家族成员。其中,、、、和是具备典型跨膜结构的膜蛋白[31-32]。参与了硝态氮吸收和转运,但是,该基因在花生中是否能够响应低氮胁迫,相关生物学功能尚不清楚。【拟解决的关键问题】本研究以花育6309为试验材料,对以上5个NRT2家族基因展开响应低氮胁迫的时空表达分析,并对进行克隆、序列分析和亚细胞定位,通过构建异源表达植株来解析其功能,为通过遗传改良选育具有高氮素利用率的低氮耐受型花生新品种提供理论参考。
1 材料与方法
1.1 正常氮和低氮条件下花生植株的培养
以花育6309为试验材料,共选取100粒健康种子进行催芽处理,即在水中进行24 h浸种处理后,挑选60粒发芽一致的种子置于滤纸浸润的培养皿上,于28℃恒温培养箱中培养48 h。待种子出芽后,将花生插入带孔的水培方盒中长至四叶期时,摘除花生子叶,缓苗后在不同供氮条件下培养。正常氮处理为改良Hoagland营养液培养(Coolaber,中国北京),低氮处理则为1/20正常氮浓度(15 mmol·L-1)的低氮Hoagland营养液,其中,缺少的Ca2+、K+分别用CaCl2·H2O和K2SO4补足。置于16 h光照/8 h黑暗培养室中培养,每2—3 d更换1次Hoagland营养液,培养过程中营养液保持通气状态。
1.2 不同供氮条件下花生表型测定
观察不同供氮条件下花生植株的表型,测定内容如下:
根系形态:每个时期取3株花生植株的根系,用吸水纸吸干根系表面水分,用Epson7500双面光源扫描仪(爱普生有限公司,中国北京)扫描根系并保存图像,通过WinRHIZO根系分析系统(瑞金特仪器有限公司,加拿大魁北克)测定并计算不同直径(0—1 mm、1—2 mm及> 2 mm)根长、根表面积及根体积。
干物质重:每处理取3株花生植株,并将茎叶和根分开,105℃杀青30 min,80℃烘至恒重后,分别称重并记录。
叶绿素含量测定:使用叶绿素含量测定仪(SPAD-502)对不同供氮条件下的花生成熟叶和幼叶中的叶绿素相对含量进行测定。每次重复测定4—5次,取平均值。使用Origin 9.8分析软件进行数据分析,采用检验进行差异显著性分析。
1.3 不同供氮条件下取样策略及时空表达分析
为检测NRT2家族基因在花生生长发育过程中的表达是否响应低氮胁迫,在正常氮/低氮条件下,以花育6309为材料,在不同发育时间的根、茎、叶等组织进行取样,采用TaKaRa Mini BEST Plant RNA Extraction Kit试剂盒(TaKaRa生物技术有限公司,中国北京)进行RNA提取,并使用PrimeScrit™ RT Kit with gDNA eraser反转录试剂盒(perfect real-time, TaKaRa生物技术有限公司,中国北京)合成cDNA。利用Primer Premier 6.0设计实时荧光PCR特异引物,以为内参基因(表1)。使用TB Green Premix Ex Taq™ Ⅱ试剂盒(TaKaRa生物技术有限公司,中国北京)进行实时荧光定量PCR(quantitative real-time PCR,qRT-PCR)检测,反应体系为20 μL,包含TB Green Premix Ex Taq II 10 μL、引物(F/R)各0.8 μL、cDNA模板2 μL和ddH2O 6 μL。反应程序为95℃ 3 min;95℃ 5 s,60℃ 10 s,测荧光值,38个循环;95℃ 15 s,65—95℃每增加0.5℃读取荧光1次,绘制熔解曲线。每个反应3个重复,采用2-△△CT法进行计算。利用Origin 9.8软件和Microsoft Excel 2019软件处理和分析试验数据。

表1 5个NRT2基因及其引物序列
1.4 AhNRT2.7a的克隆
根据栽培种花生基因组测序注释信息,结合NCBI数据库相关序列,检索获得的CDS序列,使用Primer Premier 6.0设计特异引物,以花育6309的cDNA为模板进行基因片段扩增。扩增体系为2× EasyTaq PCR SuperMix(+dye)12.5 μL、DNA模板0.5 μL、正向引物(10 μmol·L-1)1 μL、反向引物(10 μmol·L-1)1 μL,补ddH2O至25 μL。PCR反应程序为95℃ 5 min;94℃ 50 s;58℃ 1 min,72℃50 s,34个循环;72℃ 7 min。用1%琼脂糖(US EVERBRIGHT,中国苏州)凝胶电泳检测、回收,与pMD-18T载体连接,并转化大肠杆菌DH5α,经过质粒提取和酶切鉴定后,将所获得的阳性克隆送至华大科技公司测序。
1.5 花生AhNRT2.7a的生物信息学分析
利用NCBI Blast(http://blast.ncbi.nlm.nih.gov)对测序结果进行检索,DNAMAN软件分析基因的全长序列和氨基酸序列。使用在线软件Exon-Intron Graphic Make(http://www.wormweb.org/exonintron)绘制基因结构图,采用在线软件Compute pI/Mw(http://web.espasy.org/protparam)计算蛋白质分子量和等电点,SOPMA(http://npsa-pbil.ibcp.fr/cgi-bin/ npsa_automat.pl?page=npsa_sopm.html)和PHYRE2(http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index)预测蛋白质二级结构和三级结构,PSORT(http://psort.hgc.jp/form.html)进行亚细胞定位预测,TMHMM(http://www.cbs.dtu.dk/services/TMHMM/)预测蛋白质的氨基酸疏水性和跨膜结构,ProScale(http://web.expasy.org/protscale/)计算疏水性。对花生与其他物种中硝酸盐转运蛋白基因进行系统进化分析。利用Clustal X软件的聚类比较和MEGA7.0软件的邻位相近法(Neighbor-Joining)建树,研究与其他物种中同源基因的系统进化关系。
1.6 亚细胞定位
使用高保真酶Pfu扩增获得去掉终止密码子并在两端连有酶切接头的的CDS序列,通过酶切、连接进入pCAMBIA1301-eGFP载体,获得AhNRT2.7a-GFP融合表达载体。对拟南芥植株进行原生质体提取,取330 μL原生质体,加入5—10 μg(20 μL)AhNRT2.7a-GFP融合表达载体,加入350 μL PEG4000溶液介导转化,28℃,40 r/min暗培养20—24 h,将转化好的细胞进行FM4-64染色后,用激光共聚焦显微镜观察并记录。
1.7 转基因拟南芥的获得
将克隆获得的花生基因与改造过的过表达载体SN1301进行重组,并转化农杆菌。采用蘸花法转化拟南芥,用含潮霉素的1/2 MS固体培养基筛选转化子。根据抗病或感病植株对潮霉素的分离比例,筛选出含有单一插入(全长编码区)的株系,从而获得纯合株系,并进一步获得转基因拟南芥T3株系。
1.8 低氮处理下野生型和转基因植株的氮积累量及氮代谢相关酶活性生理指标的测定
以无菌蛭石为基质,定期浇灌正常和低氮Hoagland营养液,分别培养野生型和转基因拟南芥植株。待处理至8周左右,取正常和低氮条件下的转基因和野生型拟南芥叶片,采用碱水解法、碳酸氢钠浸提法和乙酸铵浸提法分别测定不同供氮条件下的氮、磷、钾含量。同时,由于植物氮代谢过程与氮代谢相关酶的活性紧密相关,取不同供氮浓度下的转基因株系和野生型拟南芥叶片(>50 mg),在液氮中研磨至粉末状,置于PBS匀浆液(pH=7.2—7.4,浓度为0.01 mol·L-1),匀浆比为10%。匀浆后,冰浴5 min,5 000 r/min离心15 min,取上清液,按照酶联免疫分析试剂盒(MDBio. Inc,,中国台湾)操作步骤分别测定谷氨酰胺合成酶(glutamine synthetase,GS)、谷氨酸合成酶(glutamate synthase,GOGAT)、谷氨酸脱氢酶(glutamate dehydrogenase,GDH)、硝酸还原酶(nitrate reductase,NR)和亚硝酸还原酶(nitrite reductase,NiR)这5种氮代谢相关酶的活性。使用BCA法蛋白含量测定试剂盒(ADS-W- SP002)检测样本中总蛋白的含量,并计算组织样本每毫克蛋白中的酶活性表达量。
2 结果
2.1 低氮胁迫下花生植株的表型
通过对正常和低氮条件下花生地上和地下部的干重分别进行测定(图1-a—b),花生地上和地下部分的干重均随生长而持续积累,但是,与低氮条件相比,正常供氮条件下的花生地上和地下两部分的干重显著增加。此外,正常供氮条件下,花生叶的叶绿素含量基本保持稳定,而低氮条件下,花生叶中叶绿素的含量呈显著下降趋势(图1-c)。
正常供氮和低氮条件下,花生的根长、根面积和根体积均会随着处理时间的增加总体呈上升趋势(图1-d—f)。正常供氮条件下,根长增加9.30倍,根体积增加3.61倍,根面积增加6.38倍;低氮条件下,根长增加4.31倍,根体积增加2.10倍,根面积增加2.99倍。通过检验,2种培养条件下,根长、根面积和根体积的生长差异显著。
2.2 花生5个NRT2基因的表达模式
利用qRT-PCR对花生不同发育时期组织中NRT2基因的表达模式进行分析。与低氮条件相比,正常供氮条件下的表达水平显著升高,说明花生可能受氮诱导表达(图2)。虽然和为同源基因,但二者表达模式存在明显差异。低氮胁迫条件下,在花生的根和茎中被诱导表达,而主要在茎和叶中被诱导表达,说明2个同源基因的表达部位存在差异。和2.7主要在花生的茎和叶中表达,且表达量均受低氮胁迫诱导,其在叶中大量表达的时期为第四周(w4),明显晚于在茎中大量表达的时期(第三周,w3,图2)。
2.3 AhNRT2.7a的cDNA全长克隆
鉴于能够响应低氮胁迫,并在花生茎和叶中高表达。根据栽培种花生基因组基因注释信息,设计引物并扩增、测序,获得长度为1 380 bp的CDS全长序列(图3),并且5′和3′端非编码区长度分别为104和289 bp。
2.4 AhNRT2.7a氨基酸同源性及进化树分析
根据NCBI网站BlastX的同源比对结果,构建花生高亲和硝酸盐转运蛋白AhNRT2.7a与野生花生2个亲本(,)、牧豆树()、开心果树()等NRT2.7蛋白的进化树(图4)。AhNRT2.7a蛋白的氨基酸序列与栽培种花生(,XP_025658933.1)相似性最高,达99.56%;其次与野生亲本BB基因组花生(,XP_016207043.1)、野生亲本AA花生基因组(,XP_020980850.1)的相似性分别为99.47%和97.36%。
运用ClustalX2对包含(At5g14570)在内的拟南芥7个NRT2家族基因与进行氨基酸序列比对,发现与拟南芥的氨基酸序列同源性较高(图5),说明二者可能存在相似的理化性质。
2.5 AhNRT2.7a蛋白结构分析
DNAMAN软件分析结果表明,该基因cDNA序列编码459个氨基酸,预测具有12个典型跨膜结构域(图6)。Compute pI/Mw在线软件分析显示,该蛋白分子量为49.35 kD,理论等电点为8.52。
经SPOMA在线软件预测,分别对花生AhNRT2.7a和拟南芥AtNRT2.7的蛋白质二级结构进行预测,发现AhNRT2.7a蛋白质二级结构中α-螺旋约占44.23%,片层结构约占18.74%,β转角约占6.54%,无规则卷曲约占30.50%。AtNRT2.7蛋白质二级结构中α-螺旋约占45.44%,片层结构约占17.85%,β-转角约占5.68%,无规则卷曲约占31.03%(图7-a和图7-b)。使用PHYRE2在线软件对三级结构建模,结果显示,2种蛋白的三级结构比较相似,推测二者功能存在一定差异(图7-c和图7-d)。
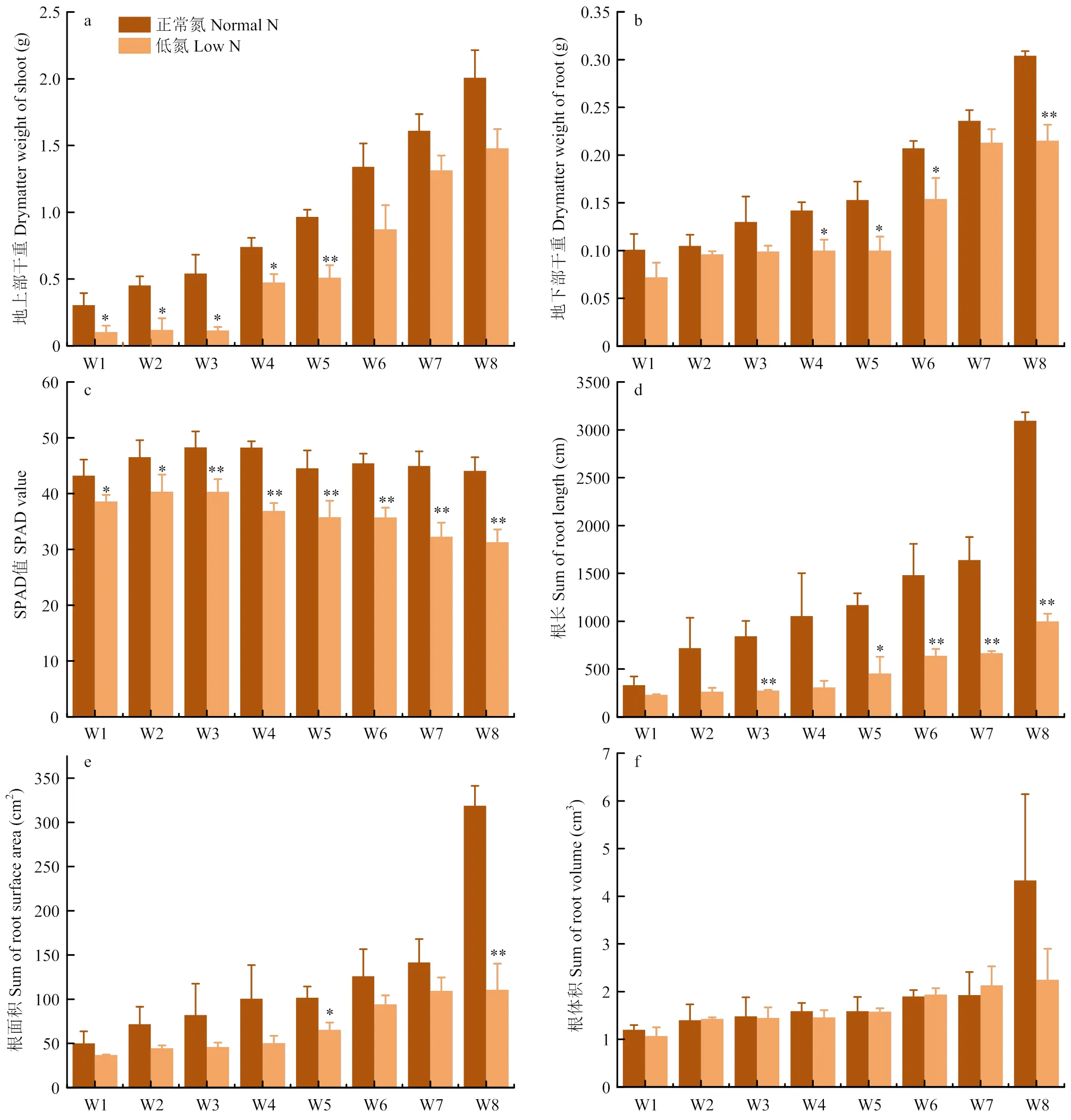
a:地上部干重;b:地下部干重;c:叶绿素含量;d:根长;e:根面积;f:根体积。W1—W8:低氮处理后第一周到第八周。*:在0.05水平差异显著;**:在0.01水平差异显著。下同
2.6 AhNRT2.7a蛋白的亚细胞定位
通过构建AhNRT2.7a-GFP融合表达载体,进行原生质体瞬时表达,发现对照组GFP蛋白在整个原生质体内均有分布,而AhNRT2.7a-GFP融合蛋白主要位于质膜,因此,推测AhNRT2.7a蛋白主要定位于细胞的质膜上(图8)。
2.7 AhNRT2.7a异源表达拟南芥植株的构建与生理参数的测定
将花生与改造的过表达载体SN1301连接。使用农杆菌介导转化野生型拟南芥,获得T1代阳性植株10株,T2代阳性植株6株。经过连续3代鉴定和潮霉素培养基筛选,成功获得3株异源表达拟南芥株系1、株系2和株系6(图9)。运用实时荧光定量PCR检测,显示在3个株系中均出现大量表达,其中,株系2和株系6的表达量分别是株系1的6倍和4倍(图10)。
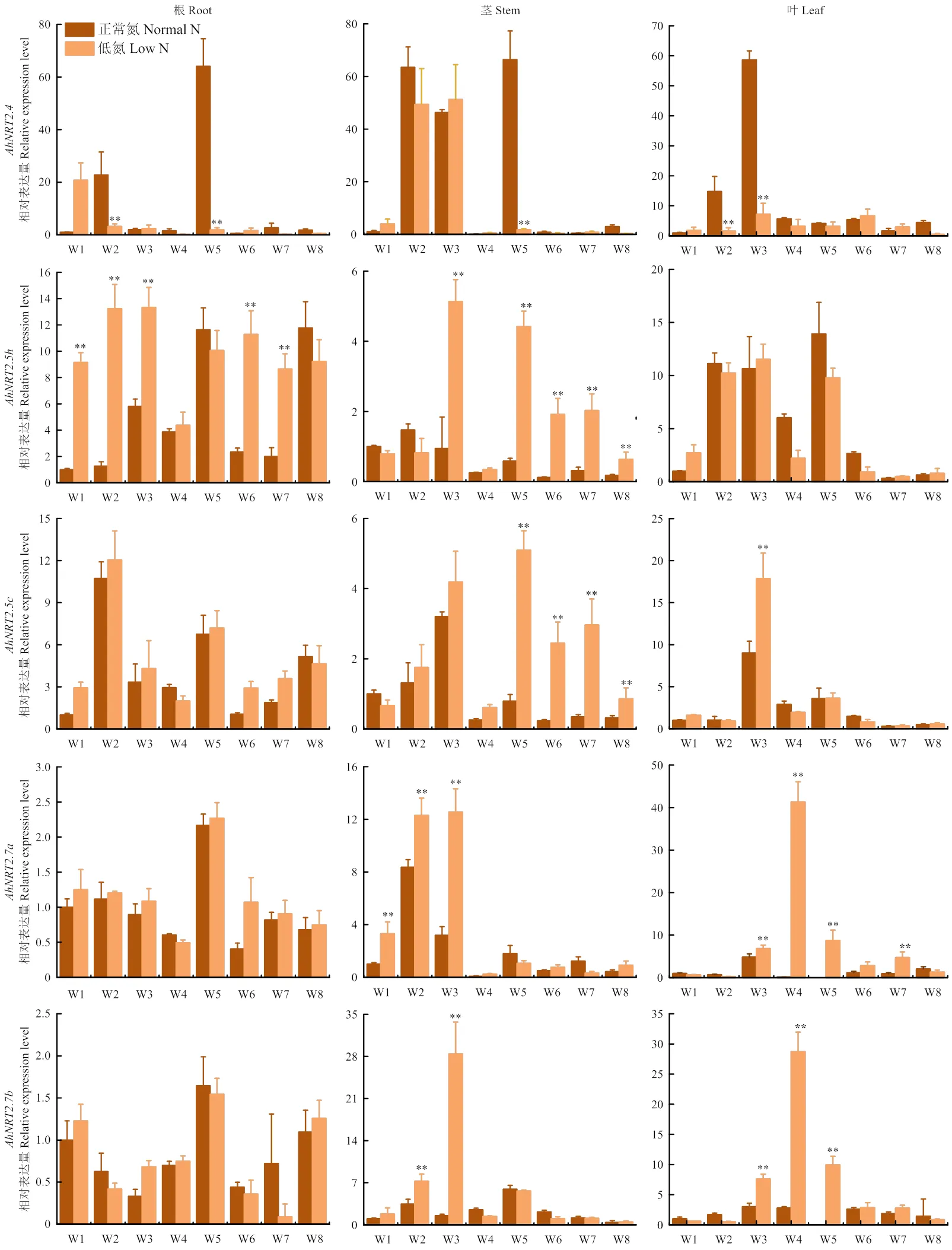
图2 正常供氮和低氮条件下5个NRT2基因的时空表达分析

图3 AhNRT2.7a的基因结构
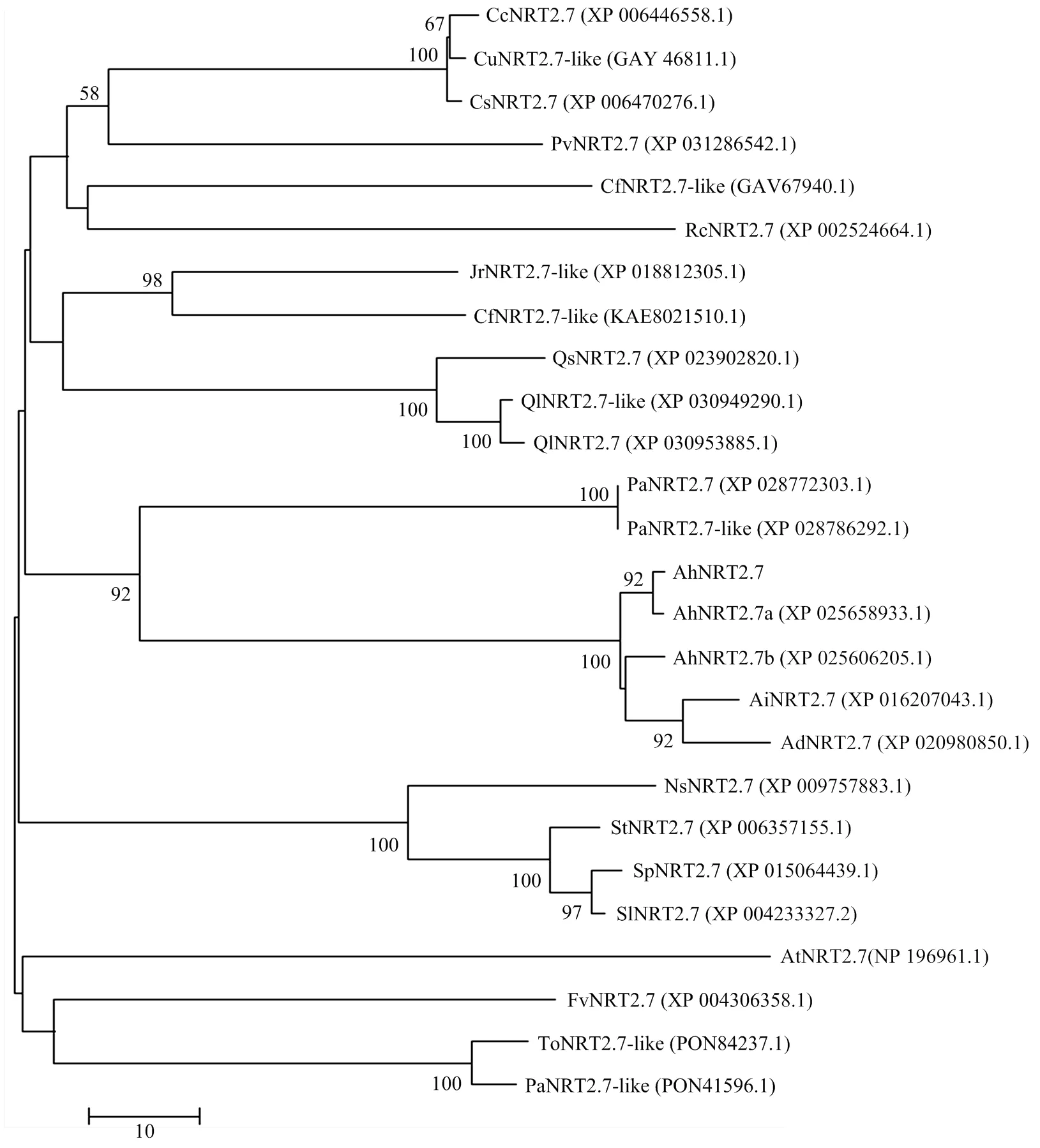
CcNRT2.7:中华辣椒,XP_006446558.1;CsNRT2.7:黄瓜,XP_006470276.1;PvNRT2.7:开心果,XP_031286542.1;RcNRT2.7:月季,XP_002524664.1;JrNRT2.7:核桃,XP_002524664.1;QsNRT2.7:栓皮栎,XP_023902820.1;QlNRT2.7:白栎,XP_030953885.1;PaNRT2.7:白牧豆树,XP_028772303.1;AhNRT2.7a:栽培种花生,XP_025658933.1,XP_025606205.1;AiNRT2.7a:野生花生,XP_016207043.1;AdNRT2.7a:野生花生,XP_020980850.1;AtNRT2.7:拟南芥,NP_196961.1;NsNRT2.7:美花烟草,XP_009757883.1;StNRT2.7:马铃薯,XP_006357155.1;SpNRT2.7:潘那利番茄,XP_015064439.1;SlNRT2.7:番茄,XP_004233327.2;FvNRT2.7:野草莓,XP_004306358.1;PaNRT2.7-like:白牧豆,PON41596.1
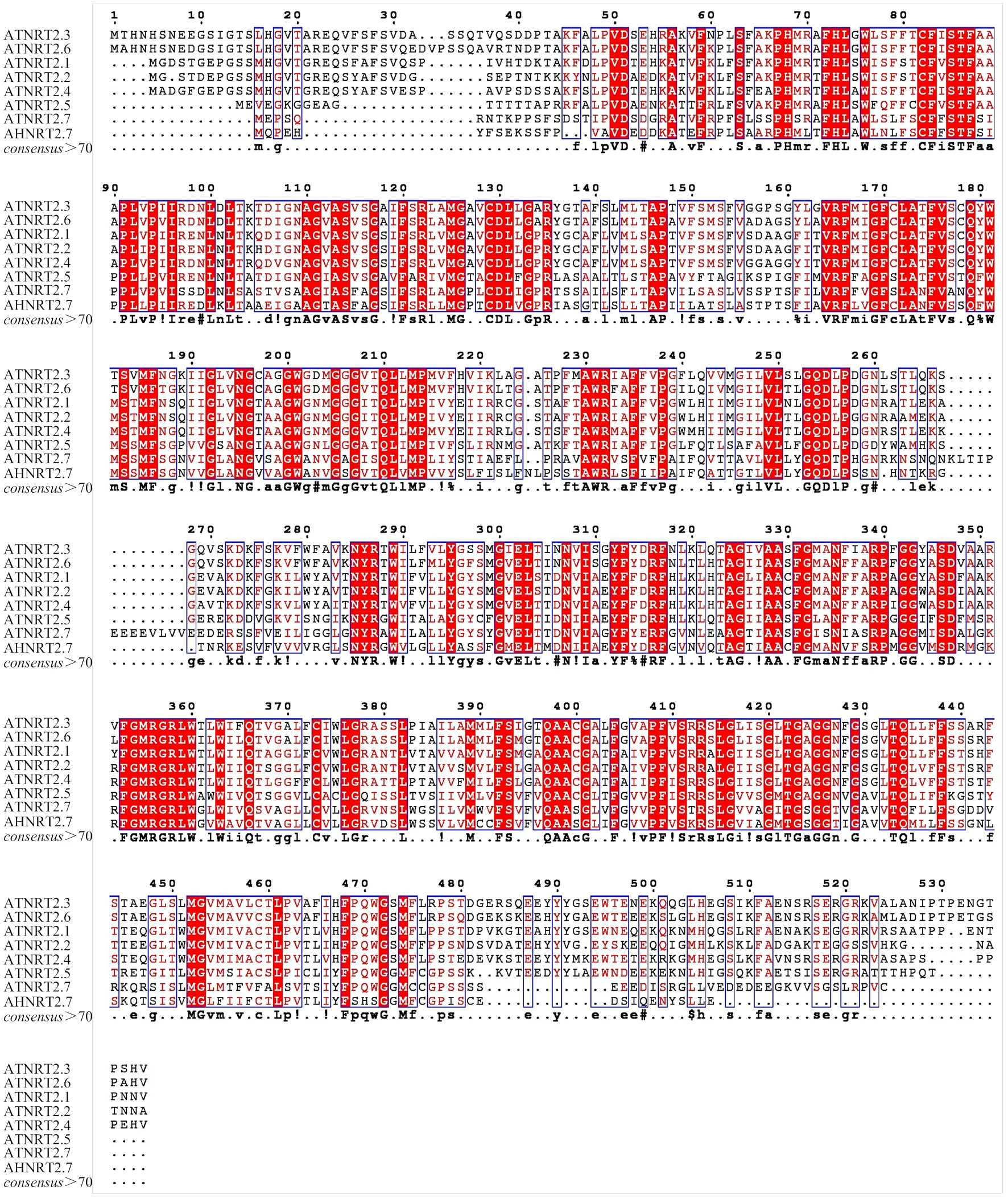
图5 AhNRT2.7a与拟南芥NRT2家族基因氨基酸的序列比对
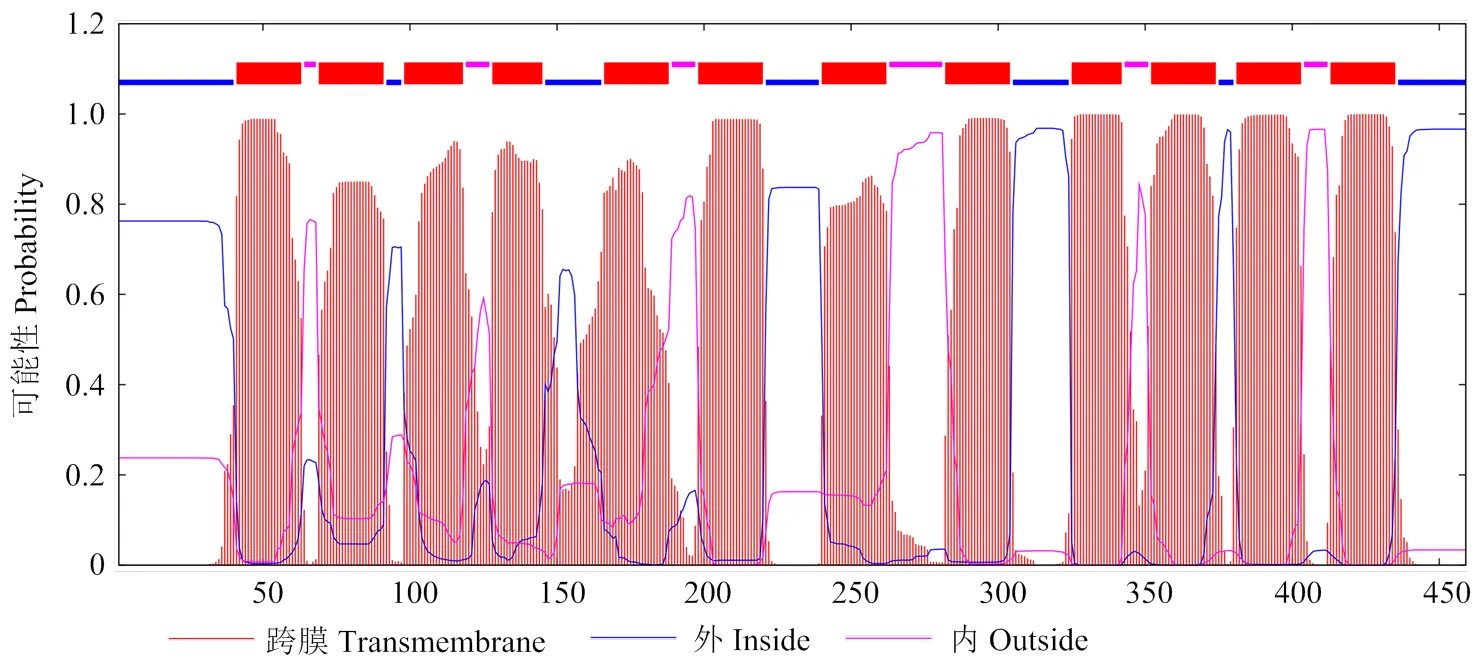
图6 AhNRT2.7a蛋白跨膜区预测
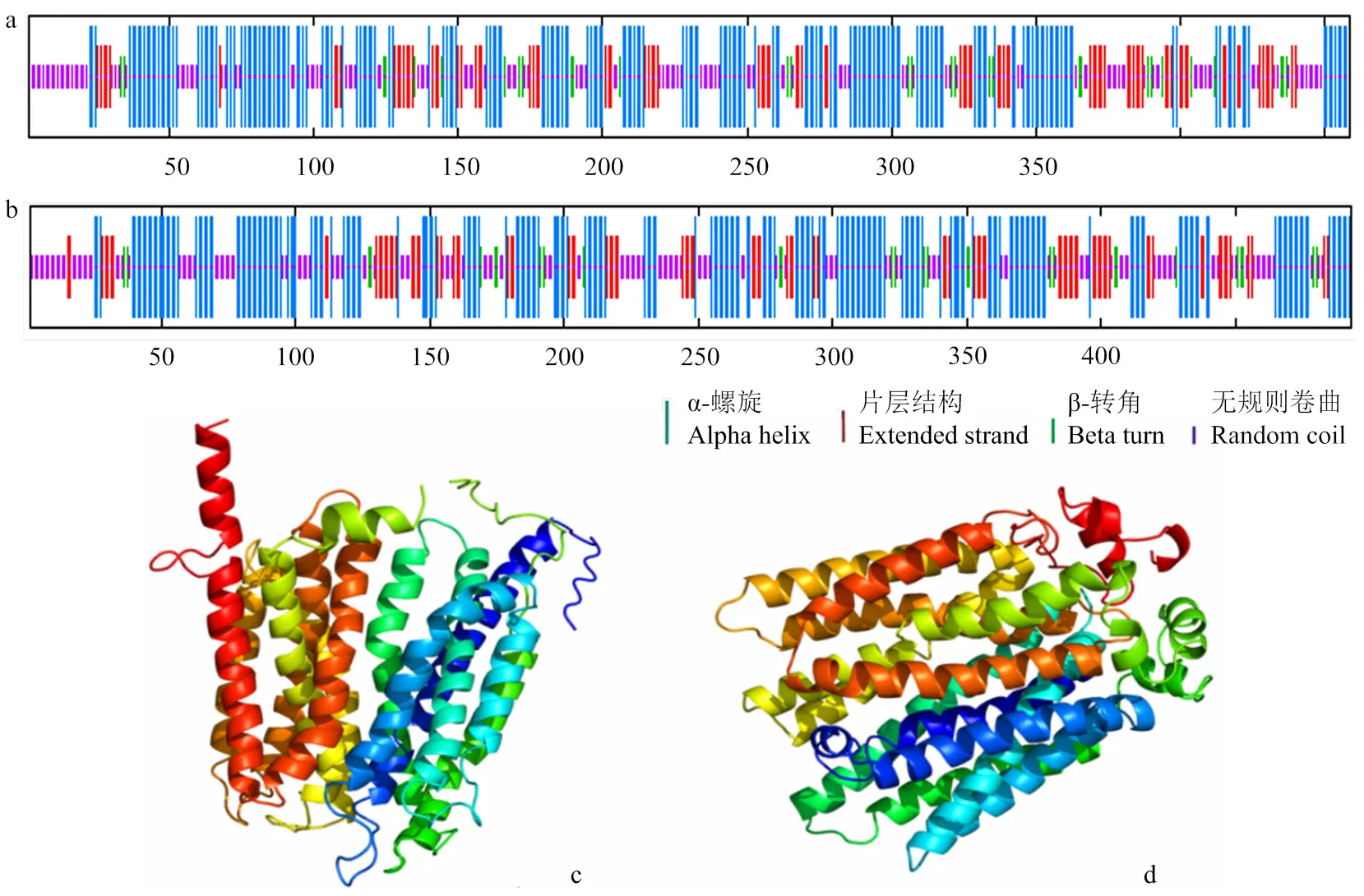
a、b:AhNRT2.7a和AtNRT2.7的二级结构预测;c、d:AhNRT2.7a和AtNRT2.7的蛋白3D建模
以灭菌蛭石为培养基种植异源表达拟南芥以及野生型拟南芥,分别在正常氮含量和1/20氮含量的Hoagland营养液中培养8周左右,发现异源过表达株系的生长情况(植株大小、叶片数目和形态等)明显优于野生型(图11-a)。2种供氮条件下分别取拟南芥野生型和转基因株系的成熟叶和幼叶,异源表达株系中2组叶片的叶绿素相对含量均显著高于野生型拟南芥(图11-b),表明拟南芥中异源表达能够显著提高植株的叶绿素含量,而有研究已指出碳代谢与根瘤固氮呈密切正相关。
与低氮胁迫相比,在正常供氮条件下,野生型和转基因拟南芥的地上部干重均增加(图12)。低氮和正常供氮条件下,野生型的地上部干重均显著小于3个转基因拟南芥株系。低氮条件下,转基因株系地上部干重约增长42%,而正常供氮条件下,转基因株系地上部干重约增长35%。
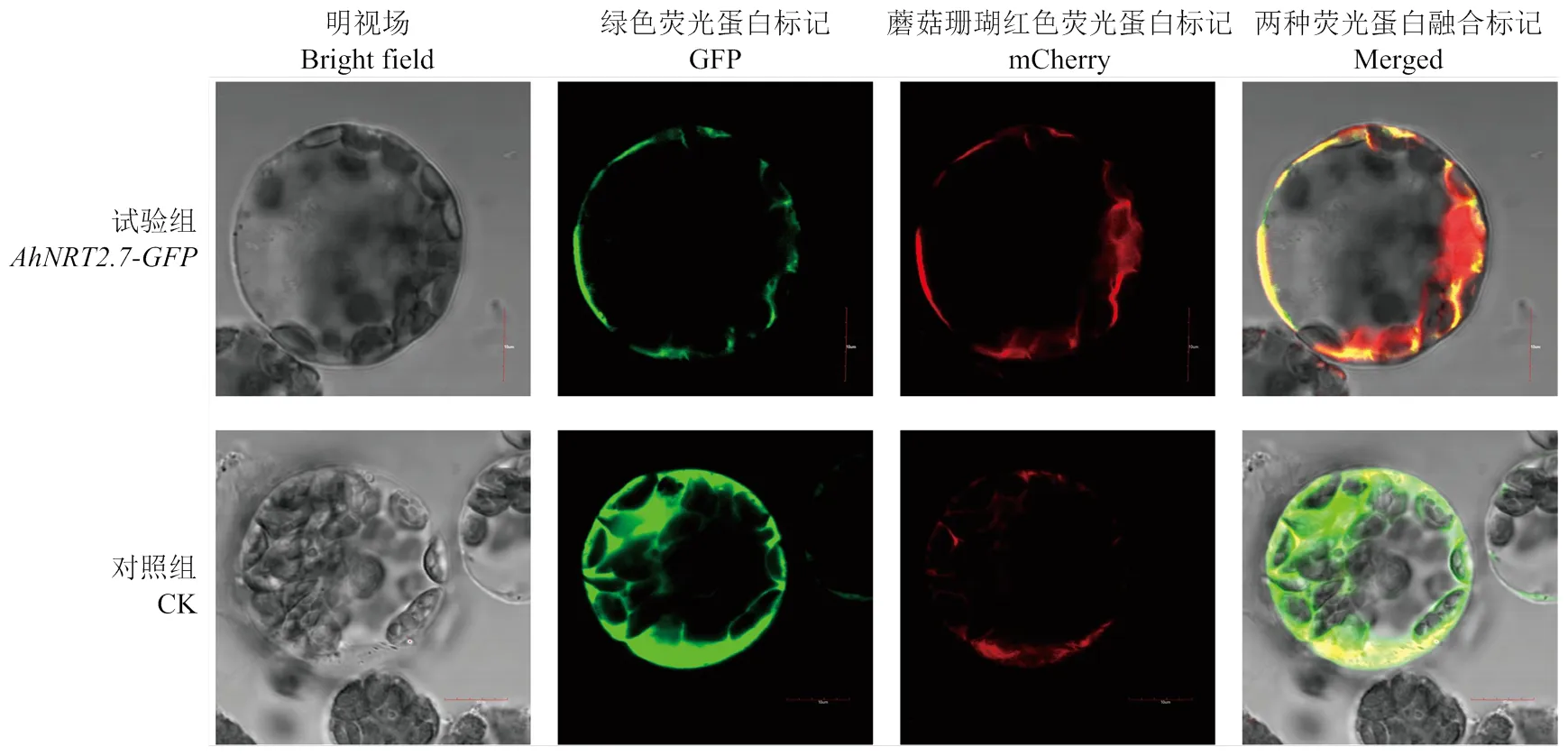
图8 AhNRT2.7a的亚细胞定位
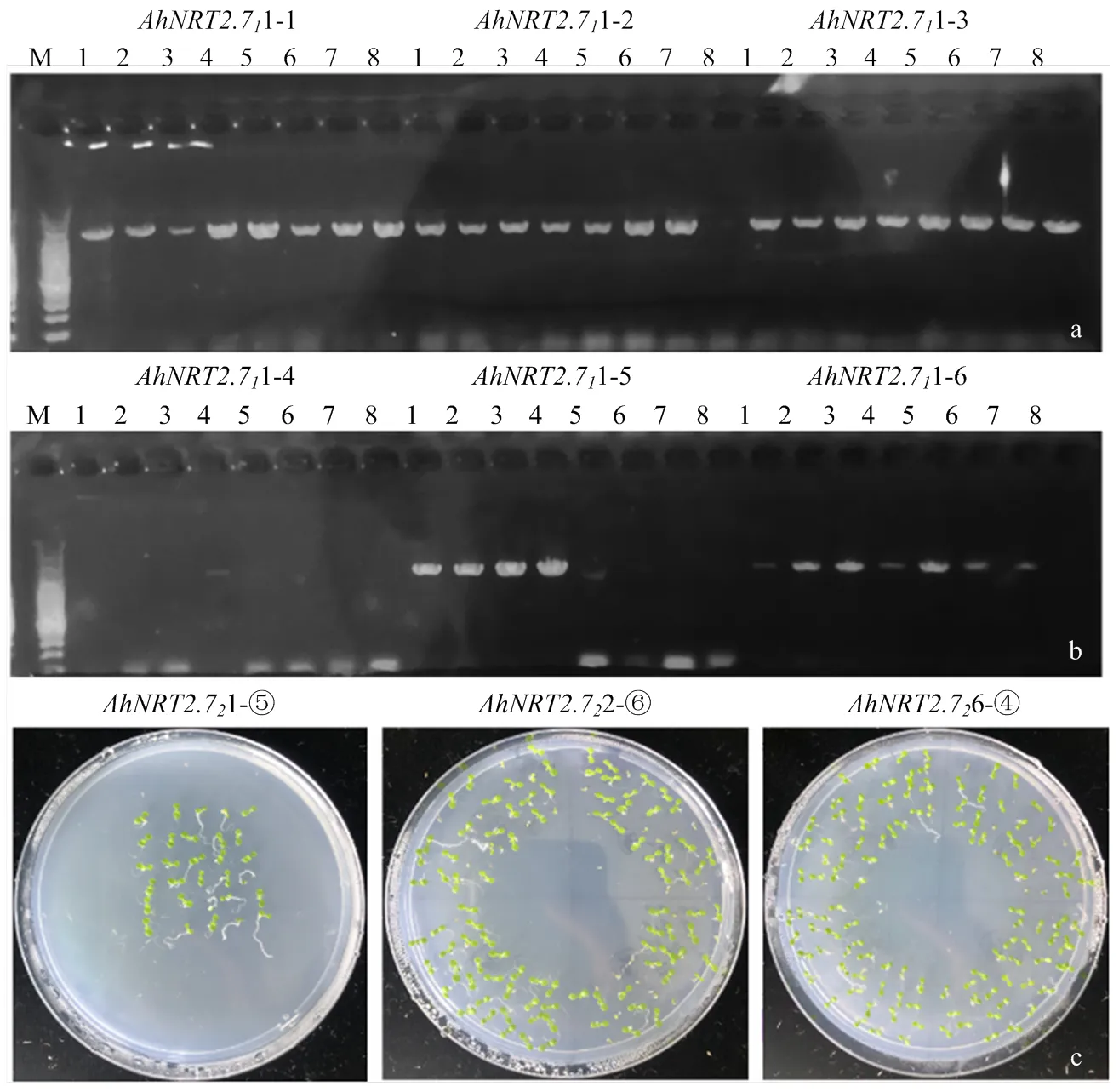
a:T0代阳性苗鉴定;b:T1阳性苗鉴定;c:T2阳性苗潮霉素筛选
2.8 转AhNRT2.7a拟南芥中氮代谢关键酶活性的变化
测定3个转基因株系与野生型拟南芥的氮、磷、钾积累量以及氮代谢相关酶(如谷氨酰胺合成酶(GS)、谷氨酸合成酶(GOGAT)、硝酸还原酶(NR)、亚硝酸还原酶(NiR)、谷氨酸脱氢酶(GDH))的活性,与野生型拟南芥相比,转基因拟南芥中硝酸还原酶(NR)活性、谷氨酰胺合成酶(GS)活性、氮积累量均显著升高(图13)。结果表明,能够影响拟南芥的氮吸收效率,即可能参与花生的氮代谢过程,又能够通过影响氮的吸收,进一步影响其他元素的吸收和同化效率。
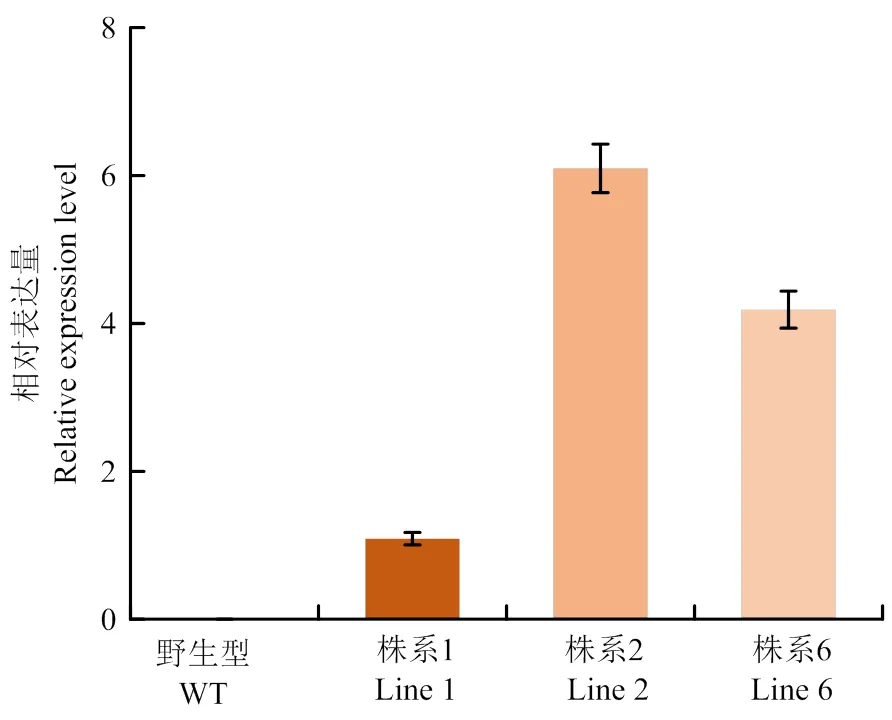
图10 转基因拟南芥株系AhNRT2.7a的表达分析
同时,在低氮胁迫条件下,与野生型拟南芥相比,转基因株系氮的积累量显著增高(图14)。与正常供氮条件相比,野生型拟南芥在低氮胁迫下的氮积累量显著下降,而3株转基因株系的氮积累量几乎没有下降。此外,低氮胁迫下转基因株系钾的积累量也显著升高,而磷的积累量几乎没有变化。
3 讨论
氮素吸收与利用是作物的生长发育和产量形成的重要保证[2]。提高作物氮素利用率是作物育种重要的研究方向[8]。氮素利用率是一个涉及遗传因素和环境因素的复杂性状[5, 31-32]。随着相关研究的深入,发现影响氮素利用率的主要瓶颈是作物本身,即作物不同品种之间的总吸氮量、氮转化和同化有明显的差异[33-35]。因此,选育低氮耐受型作物新品种,减少氮肥施用量,充分发挥其利用土壤中氮素的能力,能够从根本上提高氮素利用率。由NRT2家族组成的高亲和转运系统负责低氮条件下硝态氮的吸收和转运,是通过遗传改良获得低氮耐受型花生新品种的重要目标基因。但是,花生中该家族基因的功能研究尚未见报道。本研究对该家族能够响应低氮胁迫的家族成员进行了筛选,并且对不同供氮条件下差异表达最显著的进行功能分析,表明能够提高植物氮代谢过程的氮素利用率,可为遗传改良获得高氮素利用率的花生新品种提供重要依据。
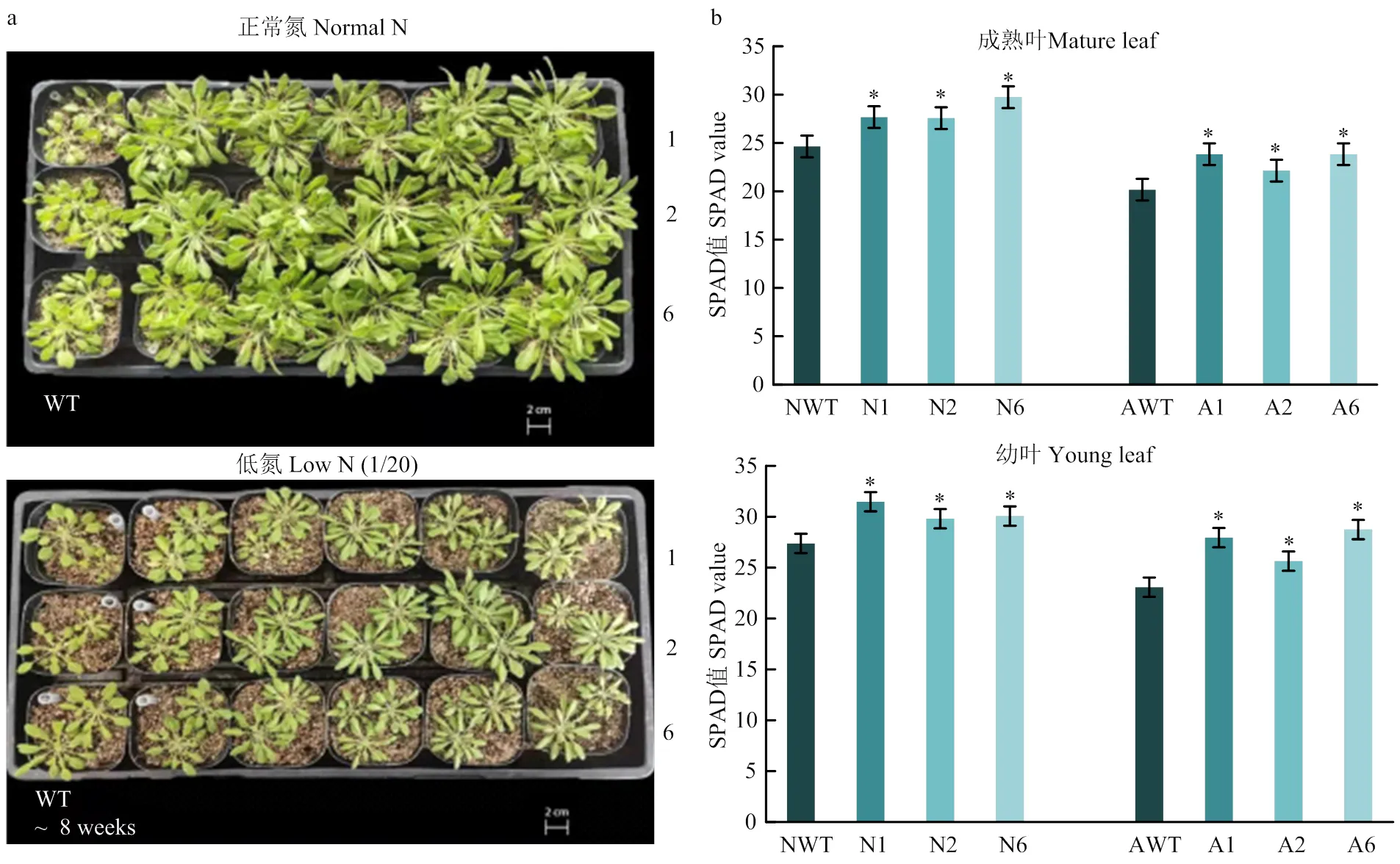
a:不同氮含量条件下拟南芥转基因株系的生长情况;b:拟南芥转基因株系成熟叶与幼叶叶绿素含量相关SPAD值的测定。N:正常氮;A:低氮。1、2、6:转基因拟南芥株系;WT:野生型。下同
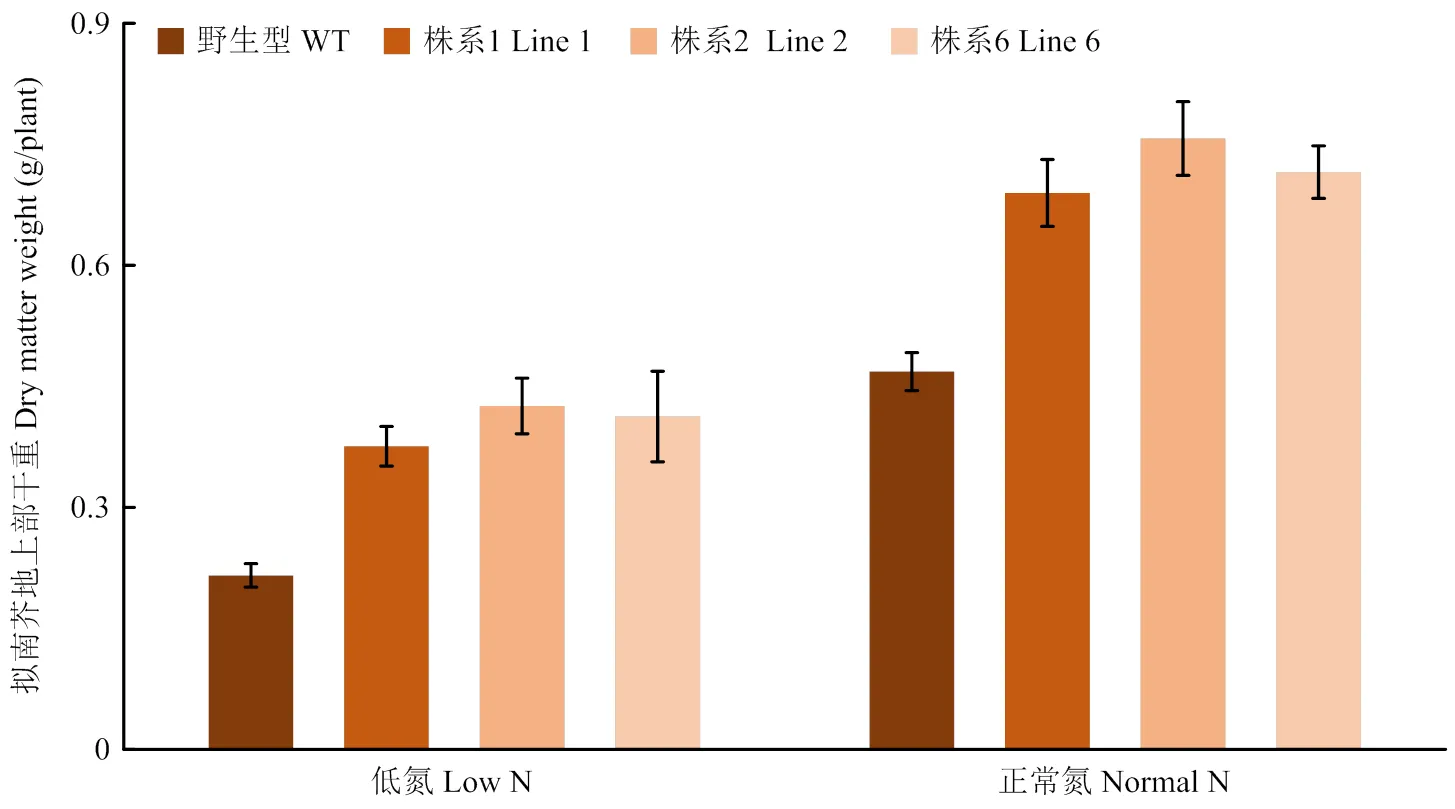
图12 不同供氮条件下转基因拟南芥株系地上部干重的变化
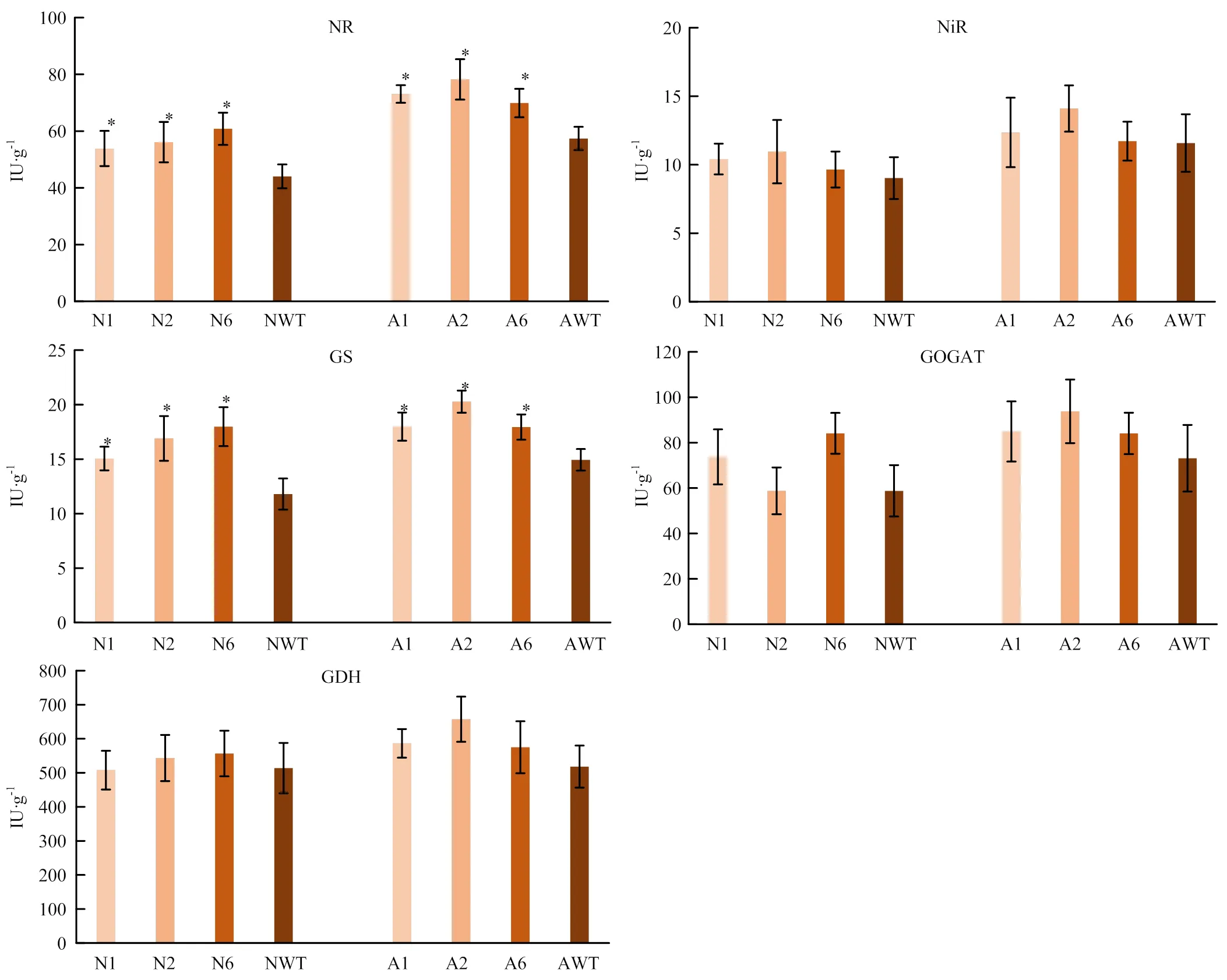
NR:硝酸还原酶;NiR:亚硝酸还原酶;GS:谷氨酰胺合成酶;GOGAT:谷氨酸合成酶;GDH:谷氨酸脱氢酶
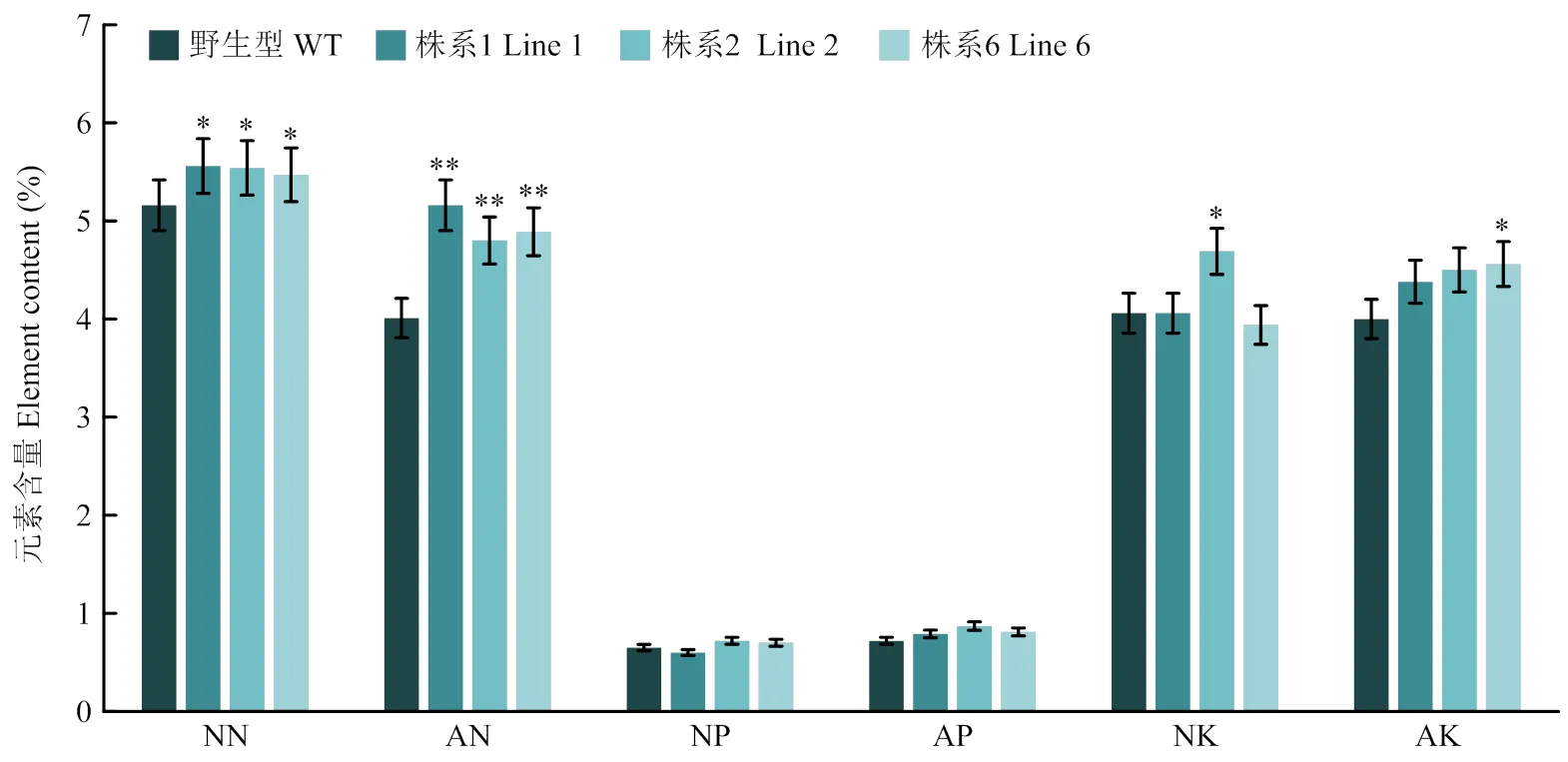
图14 不同供氮条件下转基因株系中氮、磷、钾积累的测定
3.1 低氮胁迫下NRT2家族基因的表达模式
栽培种花生全基因组基因注释和转录组数据的释放有利于花生中重要功能基因的挖掘[26-28]。前期根据预测的氨基酸序列分析发现,其中5个NRT2具有家族典型的跨膜结构域[29]。本研究首先根据不同供氮条件下花生各个发育时期不同组织中基因的表达模式分析,发现主要受到氮的诱导表达;和可能参与花生的不同组织响应低氮胁迫;和在低氮条件下的花生茎叶组织中被诱导表达。总之,响应低氮胁迫且相对表达量升高最为显著,更适合作为研究花生响应低氮胁迫的目标基因。
经过同源序列比对,花生和拟南芥属于同源基因,该同源基因在拟南芥种子中特异性表达,并且是位于液泡膜中的唯一NRT2转运蛋白[36-37]。突变体的种子硝酸盐含量降低了35%—70%,说明负责将硝酸盐转运到液泡中[21]。有研究表明,拟南芥家族成员和在低氮胁迫下参与了高亲和硝酸盐吸收过程[17, 38]此外,小麦()和在低氮胁迫下的表达量在不同的时间点均显著上升,说明上述2个基因响应低氮胁迫并维持正常的生命活动[39]。水稻参与调控不同浓度的NO3-诱导的侧根发生和NO3-在体内的转运以及低氮条件下的氮素再分配[40]。综上可知,NRT2家族基因在功能分化上是具有物种特异性的。并且,即使是同源的NRT2基因,在表达模式和生物学功能上存在物种水平的差异。
3.2 低氮胁迫下AhNRT2.7a功能解析
由于目前花生中尚未建立成熟的遗传转化体系,因此,将花生基因在拟南芥、烟草等模式植物中异源表达并研究相应的功能是花生功能基因研究的有效途径。如Liu等[41]挖掘出与种子大小相关的关键基因并经拟南芥转化研究表明是由增强脂肪酸和甘油三酯合成来增加种子重量。本研究通过构建拟南芥异源表达植株,发现与野生型拟南芥相比,各项生理参数包括根长、干物质重以及固氮量有显著提高,表明在拟南芥中异源表达能够显著提高植株的氮素利用率。无论是正常供氮还是低氮胁迫的条件下,植株的实际生长状况也出现了明显的改善。
植物的碳代谢和氮代谢的关系非常密切[42]。光合碳代谢与NO3-同化都发生在叶绿体内,碳氮代谢都需要消耗来自CO2同化和光合以及其他电子传递链的有机碳和能量。总之,碳代谢为根瘤固氮提供能量,而根瘤固氮为花生叶片提供氮素营养,二者共同调节花生碳氮代谢及营养平衡。通过在拟南芥中异源表达,植株中叶绿素含量显著提高,增幅在5%—10%,这意味着的表达在促进氮代谢过程的同时,也促使碳代谢作用的进一步加强。
NO3-的同化过程中,先后被硝酸还原酶(NR)和亚硝酸还原酶(NiR)还原成铵,再在谷氨酰胺合成酶(GS)和谷氨酸合成酶(GOGAT)的催化下,形成其他氨基酸或含氮化合物[17, 33, 43]。因此,氮代谢相关酶活性是与植物的氮素利用率直接相关的。低氮条件下的酶活性要显著强于正常氮条件下的酶活性,说明拟南芥本身就存在通过提高氮代谢相关酶活性来应对低氮胁迫的响应机制。而在拟南芥中异源表达,无论在何种供氮条件下,均能显著提高氮代谢相关酶活性。这说明可能是通过促进氮代谢相关酶活性的方式,产生了异源表达拟南芥高效响应低氮胁迫的影响。同时,3个转基因株系中酶活性情况并不完全相同:株系1和株系2在不同供氮条件下的酶活性仍然表现出一定的差异,即低氮条件下的酶活性要显著强于正常氮条件下的酶活性;而株系6在不同供氮条件下的酶活性基本一致。关于这种差异的机理仍需要后续研究来解析。
4 结论
花生5个高亲和硝酸盐转运蛋白基因(NRT2)中,响应低氮胁迫且相对表达量升高最为显著,较适合作为研究花生响应低氮胁迫的目标基因。拟南芥同源基因在种子中特异性表达并且是液泡膜中的唯一NRT2转运蛋白,而花生低氮条件下在茎、叶组织中显著表达,说明即使是同源的NRT2基因,在表达模式和生物学功能上存在物种水平的差异。拟南芥异源表达能够显著提高植株的氮素利用率。并且,无论是正常供氮还是低氮胁迫条件下,转基因植株的实际生长状况均出现了明显的改善。的表达在促进氮代谢过程的同时,还促使了碳代谢作用的进一步加强。表明适合作为花生氮素高效利用为目的的候选基因。
[1] 廖伯寿. 我国花生生产发展现状与潜力分析. 中国油料作物学报, 2020, 42(2): 161-166.
LIAO B S.Analysis on development status and potential of peanut production in China. Chinese Journal of Oil Crops Sciences, 2020, 42(2): 161-166. (in Chinese)
[2] 李向东, 万勇善, 张高英, 吴爱荣, 马晓东. 夏花生覆膜对根瘤中固氮酶和叶片硝酸还原酶活性影响的研究. 作物学报, 1996, 22(1): 96-100.
LI X D, WAN Y S, ZHANG G Y, WU A R, MA X D. Effects of film mulching on nitrogenase and nitrate reductase activities in root nodules of peanut in summer. Acta Agronomica Sinica, 1996, 22(1): 96-100. (in Chinese)
[3] 李向东, 王晓云, 张高英, 万勇善, 李军. 花生衰老的氮素调控. 中国农业科学, 2000, 33(5): 1-7.
LI X D, WANG X Y, ZHANG G Y, WAN Y S, LI J. Regulation of nitrogen in peanut senescence. Scientia Agricultura Sinica, 2000, 33(5): 1-7. (in Chinese)
[4] ROY S, LIU W, NANDETY R S, CROOK A, MYSORE K S, LISLARIU C I, FRUGOLI J, DICKSTEIN R, UDVARDI M K. Celebrating 20 years of genetic discoveries inand symbiotic nitrogen fixation. The Plant Cell, 2019, 32(1): 15-41.
[5] WANG Y Y, CHENG Y H, CHEN K E, TSAY Y F. Nitrate transport, signaling, and use efficiency. Annual Review of Plant Biology, 2018, 69: 85-122.
[6] 刘颖, 张佳蕾, 李新国, 张正, 万书波. 豆科作物氮素高效利用机制研究进展.中国油料作物学报, 2022, 44(3): 476-482.
LIU Y, ZHANG J L, LI X G, ZHANG Z, WAN S B. Research progress on nitrogen efficient utilization mechanism of leguminous crops. Chinese Journal of Oil Crop Sciences, 2022, 44(3): 476-482. (in Chinese)
[7] CHEN C Z, LU X F, LI J Y, YI H Y, GONG J M.is another essential component in the regulation of nitrate reallocation and stress tolerance. Plant Physiology, 2012, 159: 1582-1590.
[8] 于飞, 施卫明. 近10年中国大陆主要粮食作物氮肥利用率分析. 土壤学报, 2015, 52(6): 1311-1324.
YU F, SHI W M. Analysis of nitrogen use efficiency of main grain crops in mainland China in recent 10 years. Acta Pedologica Sinica, 2015, 52(6): 1311-1324. (in Chinese)
[9] WEST P C, GERBER J S, Engstrom P M, Engstrom P M, Mueller N D, Brauman K A, Carlson K M, Cassidy E S, Johnston M, Macdonald G K, Ray D K. Leverage points for improving global food security and the environment. Science, 2014, 345(6194): 325-328.
[10] 张卫峰, 马林, 黄高强, 武良, 陈新平, 张福锁. 中国氮肥发展、贡献和挑战. 中国农业科学, 2013, 46(15): 3161-3171.
ZHANG W F, MA L, HUANG G Q, WU L, CHEN X P, ZHANG F S. Nitrogen fertilizer development, contribution and challenge in China. Scientia Agricultura Sinica, 2013, 46(15): 3161-3171. (in Chinese)
[11] LERAN S, VARALA K, BOYER J C, CHIURAZZI M, CRAWFORD N, DANIEL-VEDELE F, DAVID L, DICKSTEIN R, FERNANDEZ E. A unified nomenclature of NITRATE TRANSPORTER1/PEPTIDE TRANSPORTER family members in plants. Trends in Plant Science, 2014, 19: 5-9.
[12] GALVAN A, FERNANDEZ E. Eukaryotic nitrate and nitrite transporter. Cellular and Molecular Life Science, 2001, 58: 225-233.
[13] 张合琼, 张汉马, 梁永书, 南文斌.植物硝酸盐转运蛋白研究进展. 植物生理学报, 2016, 52(336): 4-12.
ZHANG H Q, ZHANG H M, LIANG Y S,NAN W B. Research progress of nitrate transporter in plants. Plant Physiology Journal, 2016, 52(336): 4-12. (in Chinese)
[14] OKAMOT M, KUMAR A, LI W B, WANG Y, SIDDIQI M Y, CRAWFORD N M, GLASS A D M. High-affinity nitrate transport in roots ofdepends on expression of the-like gene AtNRT3.1. Plant Physiology, 2006, 140: 1036-1046.
[15] FORDE B G. Nitrate transporters in plants: structure, function and regulation. Biochimica et Biophysica Acta-Biomembranes, 2000, 1465(1/2): 219-235.
[16] ORSEL M, CHOPIN F, LELEU O, SMITH S H, KRAPP A, DANIEL-VEDELE F, MILLER A J. Characterization of a two- component high-affinity nitrate uptake system inPhysiology and protein-protein interaction. Plant Physiology, 2006, 142: 1304-1317.
[17] LEZHNEVA L, KIBA T, FERIA-BOURRELLIER A B, LAFOUGE F, BUOTET-MERCEY S, ZOUFAN P, SAKAKIBARA H, DANIEL-VEDELE F, KRAPP A. Thenitrate transporter NRT2.5 plays a role in nitrate acquisition and remobilization in nitrogen-starved plants. Plant Journal for Cell and Molecular Biology, 2014, 80(2): 230-241.
[18] PENUELAS J, SARDANS J. The global nitrogen-phosphorus imbalance. Science, 2022, 375: 6578.
[19] LIU J, CHEN F, Olokhnuud C, Glass A D M, Tong Y, Zhang F, Mi G. Root size and nitrogen uptake activity in two maize () inbred lines differing in nitrogen use efficiency. Journal of Plant Nutrition and Soil Science, 2009,172(2): 230-236.
[20] TANG Z, FAN X, LI Q, FENG H, MILLER A J, SHEN Q, XU G. Knockdown of a rice stelar nitrate transporter alters long-distance translocation but not root influx. Plant Physiology, 2012, 160(4): 2052-2063.
[21] FAN X, TANG Z, TAN Y, ZHANG Y, LUO B, YANG M, XU, G. Overexpression of a pH-sensitive nitrate transporter in rice increases crop yields. Proceedings of the National Academy of Sciences of the USA, 2016, 113(26): 7118-7123.
[22] PELLIZZARO A, CLOCHARD T, PLANCHT E, LIMAMI A M, MORERLEORER-LE PAVEN M C. Identification and molecular characterization ofandfamilies. Physiologia Plantarum, 2015, 154(2): 256-269.
[23] VON WITTGENSTEIN N J, LE C H, HAWKINS B J, EHLTING J. Evolutionary classification of ammonium, nitrate, and peptide transporters in land plants. BMC Evolutionary Biology, 2014, 14: 11.
[24] 宋田丽, 周建建, 徐晨曦, 蔡晓峰, 戴绍军, 王全华, 王小丽. 植物硝酸盐转运蛋白功能及表达调控研究进展. 上海师范大学学报(自然科学版), 2017, 46(5): 740-750.
SONG T L, ZHOU J J, XU C X, CAI X F, DAI S J, WANG Q H, WANG X L. Research progress on function and expression regulation of Nitrate transporter in plants. Journal of Shanghai Normal University (Nature Sciences), 2017, 46(5): 740-750. (in Chinese)
[25] CHEN J, ZHANG Y, TAN Y, ZHANG M, ZHU L, XU G, FAN X. Agronomic nitrogen-use efficiency of rice can be increased by drivingexpression with thepromoter. Plant Biotechnology Journal, 2016, 14(8): 1705-1715.
[26] ZHUANG W J, CHEN H, YANG M, WANG J P, PANDEY M K, ZHANG C, CHANG W C, ZHANG L S, ZHANG X T. The genome of cultivated peanut provides insight into legume karyotypes, polyploid evolution and crop domestication. Nature Genetics, 2019, 51(5): 865-876.
[27] BERTIOLI D J, JENKINS J, CLEVENGER J, Dudchenko O, Gao D y, Seijo G, Leal-Bertioli S C M, Ren L h, Farmer A D, Pandey M K, Samoluk S S, Abernathy B, Agarwa G, Ballén-Taborda C, Cameron C, CampbelL J, Chavarro C, Chitikineni A, Chu Y, Dash S, Baidouri M E, Guo B z, Huang w, Kim K D, Korani W, Lanciano S, Lui C G, Mirouze M, Moretzsohn M C, Pham M , Shin J H, Shirasawa K, Sinharoy S, Sreedasyam A, Weeks N T, Zhang X Y, Zheng Z, Sun Z q, Froenicke L, Aiden E L, Michelmore R i, Varshney R K, Holbrook C C, Cannon E K S, Scheffler B E, Grimwood J, Ozias-Akins P, Cannon S B, Jackson S A, Schmutz J. The genome sequence of segmental allotetraploid peanutL. Nature Genetics, 2019, 51(5): 877-884.
[28] CLEVENGERl J, CHU Y, SCHEFFLER B, OZIAS-AKINS P. A developmental transcriptome map for allotetraploid. Frontiers in Plant Science, 2016, 7: 1446.
[29] ZHAO Y H, MA J J, Li M, Deng L, Li G H, XIA H, ZHAO S Z, Hou L, LI P C, MA C L, YUAN M, REN L, GU J Z, GUO B Z, ZHAO C Z, WANG X J. Whole-genome resequencing-based QTL-seq identifiedgene encoding a R2R3-MYB transcription factor controlling peanut purple testa colour. Plant Biotechnology Journal, 2020, 18: 96-105.
[30] 王娟, 石大川, 陈皓宁, 吴丽青, 闫彩霞, 陈静, 赵小波, 孙全喜, 苑翠玲, 牟艺菲, 单世华, 李春娟. 花生高亲和硝酸盐转运蛋白基因家族生物信息学分析. 中国油料作物学报, 2022, 44(2): 316-323.
WANG J, SHI D C, CHEN H N, WU L Q, YAN C X, CHEN J, ZHAO X B, SUN Q X, YUAN C L, MU Y F, SHAN S H, LI C J. Bioinformatics analysis of high affinity nitrate transporter gene family in Peanut. Chinese Journal of Oil Crop Sciences, 2022, 44(2): 316-323. (in Chinese)
[31] LERAN S, VARALA K, BOYER J C, CHIURAZZI M, LACOMBE B. A unified nomenclature of NITRATE TRANSPORTER1/PEPTIDE TRANSPORTER family members in plants. Trends in Plant Science, 2014, 19: 5-9.
[32] WANG J, YAN C X, LI Y, LI C J, ZHAO X B, YUAN C L, SUN Q X, SHAN S H. GWAS discovery of candidate genes for yield-related traits in peanut and support from earlier QTL mapping studies. Genes, 2019, 10(10): 803.
[33] TANG W J, YE J, YAO X M, ZHAO P Z, XUAN W, TIAN T L, ZHANG Y Y, XU S, AN H Z, CHEN G M. Genome-wide associated study identifies NAC42-activated nitrate transporter conferring high nitrogen use efficiency in rice. Nature Communications, 2019, 10(1): 5279.
[34] 武姣娜, 魏晓东, 李霞, 张金飞, 谢寅峰. 植物氮素利用率的研究进展. 植物生理学报, 2018, 54(9): 1401-1408.
WU J N, WEI X D, LI X, ZHANG J F, XIE Y F. Research progress of nitrogen use efficiency in plants. Plant Physiology Journal, 2018, 54(9): 1401-1408. (in Chinese)
[35] TENG W, HE X, TONG Y. Transgenic approaches for improving use efficiency of nitrogen, phosphorus and potassium in crops. Journal of Integrative Agriculture, 2017, 16(12): 2657-2673.
[36] LI H, HU B, CHU C. Nitrogen use efficiency in crops: lessons fromand rice. Journal of Experimental Botany, 2017, 68(10): 2477-2488.
[37] GABRIEL K, NIGEL M C, GLORIA M C, TSAY Y F. Nitrate signaling: adaptation to fluctuating environments. Current Opinion in Plant Biology, 2015, 13(3): 265-272.
[38] KECHID M, DESBROSSES G, ROKHSI W, VAROQUAN F, DJEKOUN A, TOURAINE B. Theandgenes are involved in growth promotion ofby the plant growth- promoting rhizobacterium (PGPR) strainSTM 196. New Phytologist, 2013, 198(2): 514-524.
[39] 轩红梅, 王永华, 魏利婷, 杨莹莹, 王利娜, 康国章, 郭天财. 小麦幼苗叶片中硝酸盐转运蛋白和家族基因对氮饥饿响应的表达分析. 麦类作物学报, 2014, 34(8): 1019-1028.
XUAN H M, WANG Y H, WEI L T, YANG Y Y, WANG L N, KANG G Z, GUO T C. Expression analysis of nitrate transporterandfamily genes in response to nitrogen starvation in wheat seedling leaves. Journal of Triticeae Crops, 2014, 34(8): 1019-1028. (in Chinese)
[40] WEI J, ZHANG Y, FENG H, QU H, FAN X, YAMAJI N, XU G.encodes a dual-affinity nitrate transporter and functions in nitrate-regulated root growth and nitrate distribution in rice. Journal of Experimental Botany, 2018, 69(5): 1095-1107.
[41] LIU Y Y, SHAO L B, ZHOU J, LI R C, PANDEY M K, HAN Y, CUI F, ZHANG J L, GUO F, CHEN J, SHAN S H, FAN G Y, ZHANG H, SEIM I, LIU X, LI X G, VARSHNEY R K, LI G W, WAN S B. Genomic insights into the genetic signatures of selection and seed trait loci in cultivated peanut. Journal of Advanced Research, 2022, https://doi.org/10.1016/j.jare.2022.01.016
[42] 郑永美, 周丽梅, 郑亚萍, 吴正锋, 孙学武, 于天一, 沈浦, 王才斌. 花生主要碳代谢指标与根瘤固氮能力的关系. 植物营养与肥料学报, 2021, 27(1): 75-86.
ZHENG Y M, ZHOU L M, ZHENG Y P, WU Z F, SUN X W, YU T Y, SHEN P, WANG C B. Relationship between carbon metabolism and nitrogen fixation ability of peanut nodule. Journal of Plant Nutrition and Fertilizer, 2021, 27(1): 75-86. (in Chinese)
[43] CAMPBELL W H. Nitrate reductase structure, function and regulation: bridging the gap between biochemistry and physiology. Annual Review of Plant Biology, 1999, 50(1): 277-303.
Functional Analysis ofin Response to Low-Nitrogen in Peanut

1Shandong Peanut Research Institute, Qingdao 266100, Shandong;2College of Food Science and Engineering, Ocean University of China, Qingdao 266000, Shandong;3Qingdao Academy of Agricultural Sciences, Qingdao 266100, Shandong
【Objective】Nitrogen (N) plays a key role in determining biomass and yield in crop production., the high affinity nitrate transporter genes, are mainly activated under low nitrogen stress condition and have been implicated in nitrate absorption and remobilization. This study will screengene family responding to low-nitrogen condition (1/20 of the normal level) and conduct a preliminary functional analysis ofin order to provide target genes for breeding new peanut varieties with higher nitrogen utilization efficiency (NUE),which will help to achieve the goal of to improve crop production with less N fertilizer demand and environmental degradation. 【Method】The spatio-temporal expression patterns under normal and low-nitrogen conditions of five peanut NRT2 genes,,,,and, were investigated. Using the cDNA of Huayu6309 as template, full length ofCDS was cloned and bioinformatic analyzed. Subcellular localization of AhNRT2.7a was conducted by construction of transient expression vector and transformation ofprotoplasts. In order to explore the gene function of, heterologous overexpression of thegene inwere performed. Transgenic plants were used to determine chlorophyll content, nitrogen accumulation and the enzymatic activities of glutamine synthetase (GS), glutamate synthetase (GOGAT), nitrate reductase (NR), nitrite reductase (NiR) and glutamate dehydrogenase (GDH) under normal and low-nitrogen conditions. 【Result】Four NRT2 genes of peanut were highly expressed in response to low nitrogen stress, andwas highly expressed in the stems and leaves. The total length of 1 380 bp was obtained, encoding a 459-amino acid protein with a molecular weight of 49.35 kD. The total of 12 typical transmembrane protein domains with hydrophobic regions was predicted. Bioinformatics analysis showed that the amino acid sequence had 99.56% sequence similarity with the cultivated peanut (L.), followed by the wild-parents AA () and BB (). Subcellular localization analysis revealed that AhNRT2.7a was located in the cell membrane. Transgenicplants for over-expressingwere conducted. Relative content of chlorophyll in mature and young leaves was significantly higher than that in wild-typeunder different nitrogen supply. Meanwhile, the activity of five enzymes involved in nitrogen metabolism were examined. Furthermore, uptake, assimilation and re-mobilization of N, concentration of phosphorus and potassium were determined. The results have revealed that the activity of the two nitrogen metabolizing enzymes (NR and GS) and nitrogen accumulation in transgenic plants were significantly higher than in wild-type. 【Conclusion】These results indicated thatcould enhance the nitrogen use efficiency (NUE) in plants, and also improve carbon metabolism.seems promising as a candidate gene in breeding new peanut varieties with higher NUE.
peanut; nrt2;; nitrogen efficiency; enzymes related to nitrogen metabolism

10.3864/j.issn.0578-1752.2022.22.003
2021-07-12;
2022-09-08
山东省自然科学基金面上项目(ZR2021MC124)、山东省泰山学者(ts201712080)、山东省良种工程(2020LZGC001)、山东省农业产业体系(SDAIT-04-02)、山东省农科院创新工程(CXGC2022E20)
王娟,E-mail:wangjuan_1984@163.com。通信作者李春娟,E-mail:peanutlab@163.com。通信作者单世华,E-mail:shansh1971@163.com
(责任编辑 李莉)

