结合ZY-1 02D光谱与纹理特征的干旱区植被类型遥感分类
帅 爽,张 志,张 天,肖成志,陈 思,马梓程,谢翠容
结合ZY-1 02D光谱与纹理特征的干旱区植被类型遥感分类
帅 爽1,3,张 志2※,张 天2,肖成志2,陈 思3,马梓程3,谢翠容3
(1. 中国地质大学(武汉)地质调查研究院,武汉 430074; 2. 中国地质大学(武汉)地球物理与空间信息学院,武汉 430074;3. 湖北省国土测绘院,武汉 430010)
高光谱遥感技术已广泛应用于植被类型制图。然而,稀疏植被冠层覆盖和土壤背景影响仍然是干旱区植被类型遥感分类的主要挑战,单独利用遥感数据光谱或纹理特征难以获得可靠的分类精度和稳定性。广义正态分布优化算法(Generalized Normal Distribution Optimization,GNDO)的特征优选结果在质量和稳定性方面相较传统优化算法具有优势,但目前还未应用于高光谱波段选取研究。为探索结合ZY-1 02D光谱与纹理特征进行干旱区植被类型遥感分类的可行性,验证GNDO方法应用于高光谱波段选取的有效性,同时探讨不同数量训练像元条件下,各特征选取方法的选择结果差异和对植被类型分类精度的影响,该研究以青海省都兰县宗加镇为例,在随机选取各分类类别不同数量训练像元(30、50、100、150、200)基础上,分别利用遗传算法(Genetic Algorithm,GA)、粒子群优化算法(Particle Swarm Optimization,PSO)、灰狼优化算法(Grey Wolf Optimization,GWO)以及GNDO算法进行高光谱波段选取并对比结果,同时利用灰度共生矩阵(Gray-Level Co-occurrence Matrix,GLCM)方法提取纹理特征,将提取的光谱特征和纹理特征组合成30组分类数据集,利用随机森林(Random Forest,RF)方法完成植被类型自动分类,对比不同分类数据集的分类精度。结果显示:蓝波段(400~450 nm)、红边波段(700~750 nm)和红波段(600~650 nm)对区分植被类型最敏感;基于光谱特征的分类数据集中,使用200个训练像元和GNDO方法进行特征优选获取的分类数据集(GNDO200)获得了最高的总体分类精度(80.44%);随着训练像元的增加,各分类数据集总体分类精度整体均呈上升趋势,不同的特征选择方法的分类精度对训练像元数量表现出不同的依赖程度;图像纹理特征的加入,明显提升了植被分类精度,将使用200个训练像元和GWO方法进行波段优选的结果与纹理特征结合的分类数据集(GWO200+TEX)获得了最高的总体分类精度(82.86%)。该研究验证了ZY1-02D国产高光谱卫星数据光谱纹理特征结合进行干旱区植被类型划分的潜力,证实了GNDO方法对高光谱波段选取的有效性,为高光谱植被类型制图中光谱、纹理特征选取提供了一种思路。
遥感;算法;ZY1-02D;植被类型分类;特征选取;GNDO
0 引 言
干旱-半干旱生态系统在调节全球碳平衡方面起着重要作用。然而,在该区域区分植被物种及其在区域尺度碳动态中的作用仍具有挑战性[1]。高光谱卫星数据包含连续的反射率光谱信息,可提供丰富的区分植被类型所需的植被生理和化学信息[2–5]。不同类型星载(Hyperion[6]、CHRIS[3])与机载(APEX[7]、AISA[8])高光谱数据已被用于全球不同区域植被类型分类研究。2019年9月中国成功发射的资源一号O2D(ZY-1 02D)星包含高光谱传感器(Advanced HyperSpectral Imager,AHSI)和多光谱传感器(Visible and Near Infrared Camera,VNIC),是首颗民用高光谱业务卫星,大大提升了国产卫星对于植被类型精细识别与监测的能力[9-10]。
尽管星载高光谱数据为植被类型识别提供了更强的光谱探测能力,但干旱区植被类型制图仍面临诸多挑战。一是“异物同谱”现象。尽管高光谱图像相比传统的多光谱图像能更好地区分相似地物,但仅基于光谱特征区分不同植被类型的能力有限[11]。另外,混合光谱问题也是制约星载高光谱植被分类精度的重要因素[12],特别是在地表覆被为灌木、土壤和草地混合的干旱-半干旱地区,某一像元光谱多是灌木、土壤和草地等端元的混合光谱。有研究表明正确组合多遥感特征可有效提升分类精度[13]。二是高维数据产生的休斯效应(Hughes phenomenon)[14]影响图像分类精度。对高光谱数据进行光谱特征(波段)选取降低数据维度是缓解休斯效应的有效途径[15]。在保持或提高分类精度的前提下,利用特征优化算法从高光谱数据集中挑选一些最有效的光谱特征(波段)以降低特征空间维数,提高泛化能力[15]。特征选择方法依据是否独立于后续的学习算法, 分为过滤式(Filter)和封装式(Wrapper)[16]。与Filter方法相比,Wrapper方法总体准确率更高[17]。Vaiphasa等[18]证实基于遗传算法(Genetic Algorithm,GA)的高光谱波段选择方法能够在植被物种水平上解决光谱相似性问题。Monteiro等[19]将粒子群优化算法(Particle Swarm Optimization,PSO)应用于大豆作物甜度高光谱反演中的特征波段选取,获得了相较传统主成分分析方法更高的反演精度。Zhang等[20]提出了一种广义正态分布优化算法(Generalized Normal Distribution Optimization,GNDO)并应用于光伏模型的参数提取,验证了该方法的参数提取结果在质量和稳定性方面优于PSO和灰狼优化算法(Grey Wolf Optimization,GWO)等方法,但目前还没有研究尝试利用GNDO方法进行高光谱波段选取。三是图像分类精度受训练样本规模制约。Sabat-Tomala等[21]利用机载高光谱数据进行植被类型分类时,发现总体分类精度随训练像元数量的减少而降低,像元数量每减少10个,精度约降低2%。另外,不同分类器的植被分类精度对训练像元数量的依赖程度也不同。当训练像元从30减少到10,最大似然法(Maximum Likelihood,ML)总体分类精度显著下降约28%,而随机森林(Random Forest,RF)和支持向量机(Support Vector Machines,SVM)方法的总体分类精度仅下降2%~3%。然而,还少有研究关注训练像元数量对高光谱波段选取及其选取结果分类精度的影响。
本研究旨在验证ZY-1 02D国产高光谱卫星遥感数据对干旱区植被类型分类的可行性。同时1)探索结合遥感数据光谱特征和空间结构特征(纹理信息)进行植被类型分类的有效性;2)验证GNDO优化算法对高光谱波段选取的可行性;3)对比分析训练像元数量对高光谱波段选取和分类精度的影响;以期为干旱-半干旱区高光谱植被类型分类中的光谱和纹理特征选取提供一种新思路。
1 研究区与数据源
1.1 研究区概况
研究区位于青海省海西蒙古族藏族自治州都兰县宗加镇,面积50.46 km2(图1)。研究区属于高原干旱大陆性气候,平均海拔2 775 m,年平均气温4.3℃,年平均降雨量37.9~180.5 mm,蒸发量1 358~1 765 mm。地貌类型为荒漠与河流滩地。研究区南部为人类居住区,主要植被类型包括枸杞林和道路旁防风杨树林,其中枸杞林按照种植时间可分为新枸杞林(2018年后种植)和老枸杞林(2018年前种植),研究区北部为荒漠与河流滩地,植被类型主要为梭梭林和草本植物。研究区其他土地覆被类型包括裸地和建筑物。
1.2 数据源与预处理
此次数据源为2020年9月30日采集的资源1号02D数据。ZY-1 02D星重返周期为3 d,高光谱传感器(AHSI)幅宽60 km,包含可见光-近红外区间76个波段,短波红外区间90个波段,空间分辨率为30 m,多光谱传感器(VNIC)幅宽110 km,包含可见光至近红外区间8个多光谱波段(10 m分辨率)和一个全色波段(2.5 m分辨率)。
利用ENVI 5.3平台对ZY-1 02D星AHSI数据进行辐射定标、大气校正处理,获取地表反射率数据;再对AHSI数据、VNIC数据多光谱波段和全色波段进行几何校正,保证其空间位置配准,几何校正通过手动选取控制点(均匀选取36处)结合多项式法进行,校正结果平均误差为4.6 m。将VNIC数据全色波段与多光谱波段进行图像融合,获取8波段2.5 m空间分辨率数据(图1)。

注:按2018年前后种植分为老、新枸杞林。下同。
1.3 野外调查与分类样本选取
2020年8月8—10日开展野外调查,对照遥感影像,选取并验证各类型植被样本,保证研究中训练样本集与验证样本集选取的准确性,最终验证并选取枸杞林(老)样本区55处、枸杞林(新)样本区64处、草地样本区54处、梭梭林样本区66处、杨树林样本区70处、人工建筑30处、裸地32处。研究区主要植被类型图片见图2。
此次研究中使用分层随机抽样方式,选择各植被类型全部样本区的50%作为训练样本集,其余50%的样本区作为检验样本集。为了研究训练样本集中训练像元数量对各特征选取方法选取结果和植被分类精度的影响,在各植被类型训练样本数据集中,以随机采样方式分别选取30、50、100、150和200个训练像元,分别应用于光谱、纹理特征的选取和RF植被分类。
2 研究方法
此次工作在选取各植被类型检验样本集和训练样本集基础上,利用各种特征选择方法结合不同数量的训练样本集,对AHSI反射率数据进行光谱特征选取,形成基于光谱特征的分类数据集,再利用灰度共生矩阵(Gray-Level Co-occurrence Matrix,GLCM)方法,结合各植被类型训练样本,提取并选取图像纹理特征,形成结合光谱纹理特征的分类数据集,最后利用RF模型完成各分类数据集植被类型自动分类,并进行精度评价和对比分析。具体技术流程如图3所示。
2.1 光谱特征选取
GA、PSO、GWO是高光谱特征选取的常用算法,而GNDO是一种改进的特征优化算法[20],至今还未应用于高光谱数据波段选择,此次研究以ZY1-02D数据为例,旨在对比GNDO算法与传统GA、PSO、GWO算法在不同数量(30、50、100、150、200)训练像元条件下的光谱特征选取效果。考虑到计算效率,研究中GA、PSO、GWO和GNDO这4种算法中适应度函数均选择K最近邻(KNN,K-Nearest Neighbor)方法,最大迭代次数均设置为200。由于GA、PSO、GWO和GNDO算法的本质是随机性的,所以研究中分别运行20次各算法,选择各结果中重复出现15次及以上的波段作为最终波段选择结果。
2.1.1 GA算法
遗传算法(GA)是一种经典的解决优化问题的软计算技术[22],多种形式的遗传算法被提出并广泛用于解决各遥感应用问题,如图像配准[23]、图像分割与分类[24]、亚像元分类[25]和高光谱波段选择[18,26]等。此次研究,遗传算法应用于ZY1-02D高光谱数据波段选择,是将染色体作为一种波段组合,将其中每个基因座与AHSI各波段相联系并以波段号赋值[18],其优化过程主要包括“交叉”运算和“变异”运算。“交叉”运算指两个染色体可对随机选择的位置(波段)进行替换重组,以创建两个新的染色体(波段组合)。“变异”运算指随机改变染色体中某些基因座赋值,即随机选取一波段替换原波段组合中的某一波段。优化过程是以分类算法为适应度函数分代进行的,每次都会生成新的染色体。由于种群数量有限,因此只有“最佳”染色体(分类精度高的波段组合)有更多机会进行迭代、配对并产生新的染色体。优化方案由“0”和“1”组成的有限“染色体”序列表示。
2.1.2 PSO算法
粒子群优化(PSO)算法通过模拟鸟群觅食行为而发展起来的一种基于群体协作的随机搜索算法[27]。粒子群优化算法应用于ZY1-02D高光谱数据波段选择,是构造一个166维向量空间与AHSI数据166个波段对应,同时训练一个具有个粒子的种群,在此166维空间中飞行,采用二进制编码对粒子的空间飞行位置进行编码[19],其中“1”表示相应位置的波段被选中,“0”表示相应位置的波段未被选中[28],每个粒子的飞行位置代表波段选择的一个解。初始阶段,群体粒子随机分布于解空间;进化过程中,粒子基于适应度函数(分类算法),根据个体和群体历史最佳粒子位置更新速度和位置,逐渐收敛于解空间的最佳位置[29],即为波段选择最优解。
2.1.3 GWO算法
灰狼优化(GWO)算法主要通过模拟狼群中的等级制度,将整个狼群中的狼分为Alpha、Beta、Delta及Omega共4个等级,等级依次降低。算法根据狼群捕食方式分为3个阶段,即搜寻猎物阶段、围捕猎物阶段和进攻猎物阶段[30]。根据狼群社会等级制度建立数学模型,则Alpha狼在狼群中等级最高,属于统治阶级,主要负责领导捕食(寻优)过程中决策以及管理狼群等职责。选定Alpha狼为最优解,Beta狼为次优解,Delta狼更次之,Omega狼为灰狼种群的候选解。灰狼优化算法应用于ZY1-02D高光谱数据波段选择,首先从AHSI数据166个波段中随机选择(=5、10、15、20、25、30、35)个不同的波段组成一个灰狼向量,重复此操作20次,构成GWO初始化种群;其次计算种群中各灰狼向量(波段组合)的分类精度并排序,确定种群中初始Alpha、Beta、Delta及Omega狼;再对初始种群进行迭代的搜寻猎物阶段、围捕猎物阶段和进攻猎物阶段算法运算,当迭代次数达到最大值,或者满足连续两次选择同一Alpha狼,此时输出Alpha狼,即最优解(最佳波段组合)。
GWO是为解决连续优化问题而设计的。对于二进制优化问题,例如高光谱波段选取,需要GWO的二进制版本。本研究使用Emary等[31]提出的二进制灰狼优化算法。
2.1.4 GNDO算法
广义正态分布优化(GNDO)算法是一种改进的优化算法,Zhang等[20]提出了该算法并用于光伏模型参数提取。该算法应用于ZY1-02D高光谱数据波段选择,是构造一个166维向量空间与AHSI数据166个波段对应,同时构造一个×阶矩阵进行种群初始化,其中(=20)为种群中个体数量,(=166)为高光谱数据波段数。矩阵的行代表种群中的每个个体,即波段组合,个体中包含的具体波段是通过对矩阵中要素进行随机赋值(0~1),并结合阈值(>0.5)确定的。GNDO中设计的信息共享策略包括局部探索和全局探索[20]。其中局部探索是在建立广义正态分布模型的基础上进行的,以分类误差最小为适应度函数,计算初代个体的适应度(误差率),获得当前最优解(当前最佳波段组合)及最佳位置向量(在多维向量空间的位置),同时计算种群中全部个体在多维向量空间中的平均位置向量,使种群中每个个体同时向最佳位置和平均位置方向移动,实现个体位置更新(波段组合优化、更新)。全局探索与3个随机选择的个体(Random individual)有关,随机选择种群中与目标个体之外的3个个体,基于随机个体的位置对目标个体位置进行更新,起到扩大搜索空间的效果。通过局部搜索和全局搜索的迭代运行,更新种群和个体位置,获得逐渐收敛于解空间的最佳位置,即最佳波段组合。
2.2 纹理特征选取与筛选
纹理是保持一定的特征重复性的空间结构。图像纹理是反应植被冠层形态、结构的重要信息。Haralick等[32]利用灰度共生矩阵,基于图像的纹理统计信息,提出了14种纹理特征。其中均值(mean)、方差(variance)、同质性(homogeneity)、对比度(contrast)、相异度(dissimilarity)、熵(entropy)、二阶矩(second moment)和相关性(correlation)8种纹理特征常被用于植被类型自动分类[33]。本研究采用灰度共生矩阵方法,选择上述8种统计参量,对研究区VNIC数据全色波段(0.452~0.902m)提取纹理图像。同时,纹理特征提取时,移动窗口的大小十分关键,通过对比3×3、5×5、7×7、9×9、11×11、13×13六种窗口大小提取纹理图像中植被类型的可区分程度,最终选择3×3窗口进行纹理计算。
获取研究区8幅纹理图像中各植被类型训练像元的均值曲线图(图4),从曲线图中比较各植被类型训练像元均值的区别,选择区分各植被类型效果较好的纹理图像。其中,方差、二阶矩、熵纹理图像中各植被类型训练像元的均值相差较大;同质性纹理图像中杨树林训练像元均值与其他植被类型差异较大,利于区分杨树林;对比度、相异度纹理图像中枸杞林(老)训练像元均值与其他植被类型差异较大,利于区分枸杞林(老)。最终选择方差、同质性、对比度、相异度、熵、二阶矩6幅纹理图像参与研究区植被类型分类。
2.3 随机森林分类方法
随机森林(Random Forest,RF)分类算法优势在于对训练样本过拟合的适应能力强且分类时间短[21]。法国南部Languedoc地区的地中海灌木植被类型分类研究结果表明,RF算法应用于高光谱植被类型自动分类时,分类精度优于传统分类器(如决策树、线性判别分析、二次判别分析和K最近邻值),特别对区分光谱差异较小的植被类型具有优势[34]。在对相同的高光谱数据集进行植被分类时,RF算法相较支持向量机和最大似然算法运算时间更短[35]。此次研究中,为验证高光谱植被类型分类中,不同训练像元数量、不同光谱特征选择方法的图像分类精度差异,同时验证图像纹理特征的加入对植被分类精度的影响。分别对研究区AHSI 全部波段数据(ALL)、不同光谱特征选择方法(GNDO、GA、PSO、GWO)在不同训练像元数量(30、50、100、150、200)条件下获得的25组基于光谱特征的分类数据集,即:ALL30、ALL50、ALL100、ALL150、ALL200、GNDO30、GNDO50、GNDO100、GNDO150、GNDO200、GA30、GA50、GA100、GA150、GA200、PSO30、PSO50、PSO100、PSO150、PSO200、GWO30、GWO50、GWO100、GWO150、GWO200以及结合光谱与纹理特征(TEX)的5组分类数据集,即:ALL200+TEX、GNDO200+TEX、GA200+TEX、PSO200+TEX、GWO200+TEX进行RF植被分类试验。为保证对比试验的客观性,各组RF植被分类试验中选取相同决策树数目(100)。

图4 NDVI与各纹理图像中各植被类型训练像元均值特征曲线
2.4 精度评价
利用混淆矩阵方法计算总体分类精度(Overall Accuracy,OA)、生产者精度(Producer’s Accuracy,PA)、用户精度(User’s Accuracy,UA)和Kappa系数[36]等精度评价指标。将各植被类型样本区中作为训练样本外的50%的样本区作为检验样本集对各分类结果进行精度评价。检验样本集分布见图1。
3 结果与分析
3.1 光谱特征选择结果对比
图5显示,各特征选择算法在不同训练数量条件下,选取的波段数量有较大差异。由于此次光谱特征的选择是进行20次试验,并选择重复出现15次以上的波段作为各特征选择算法的光谱特征选取结果,最终选择的波段数越多表明多次试验结果的差异性越小,该特征选择算法更稳定。GNDO方法与PSO方法的波段选择数量大致随训练像元数量的增大而增大,表明随着训练像元数量增加,这两种特征选择算法的结果趋于稳定。而GWO方法与GA方法的波段选择数量与训练像元数据无明显相关性。图6为各波段选择结果的光谱区间分布情况。从各波段选取结果的光谱覆盖区间上看(图6),波段选取累计次数前三多的光谱区间为400~450 nm(38次)、700~750 nm(35次)和600~650 nm(28次),分别为蓝波段、植被红边波段和红波段。而波段选取累计次数前四少的光谱区间为800~850、1 350~1 400、1 500~ 1 500及1 800~1 850 nm,波段选取累计次数均为6次。
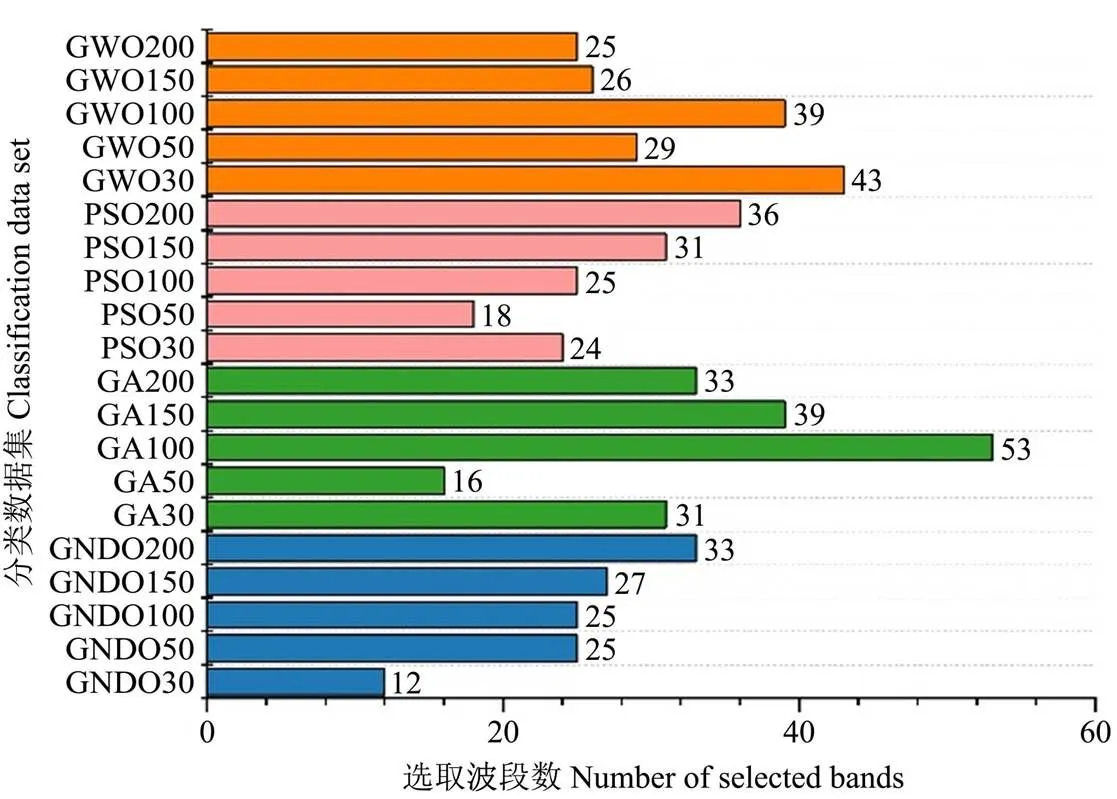
注:算法后数字代表训练像元数量。下同。

注:X轴标注1~43代表波长350~2 500 nm中以50nm为间隔的光谱区间。如1为350~400 nm,2为401~450 nm,以此类推。
3.2 植被分类结果对比
表1为各分类数据集分类精度。基于光谱特征的各分类数据中,使用200个训练像元和GNDO方法进行特征优选获取的分类数据集(GNDO200)获得最高分类精度,OA和Kappa系数分别为80.44%和0.73。随着训练像元的增加,基于光谱特征的各分类数据集总体分类精度均呈上升趋势,各特征选择方法(GNDO、GA、PSO与GWO)与原始全部波段(ALL)均在200个训练像元条件下获得最高的OA。其中,在200个训练像元条件下,PSO与GNDO方法获取的分类数据集(PSO200与GNDO200)的总体分类精度优于包含全部波段的分类数据集(OA提高1.03和2.58个百分点,Kappa系数提高0.01和0.02)。加入纹理特征的各分类数据集相较原数据集,总体分类精度均有所提升,其中GWO200+TEX获得了最高的分类精度,OA和Kappa系数分别为82.86%和0.76,ALL200+TEX总体分类精度最低,OA和Kappa系数分别为82.14%和0.75。各植被类型中,梭梭林的分类精度最高,各分类数据集PA均大于88%,UA均大于95%。
图7为ALL200、ALL200+TEX、GNDO200和GNDO200+TEX这4种分类数据集的植被分类结果图,显示出各分类数据均成功提取了研究区各类型植被的分布,主要的误分表现为研究区北部的部分裸地像元误分类为新枸杞林,以及老枸杞林和新枸杞林间的混分。GNDO2OO与ALL200的分类结果相比,老枸杞林内部误分类为新枸杞林和杨树林的现象有所减少,老枸杞林的PA提高了4.47个百分点,新枸杞林的UA提高3.80个百分点,杨树林的UA提高2.61个百分点。ALL200+TEX与ALL200的分类结果相比,杨树林的分类效果明显改善,杨树林误分类为老枸杞林的现象减少,其他植被类型误分类为杨树林的现象也有所改善,杨树林的PA和UA分别提高4.35和12.57个百分点。GNDO200+TEX与GNDO200的分类结果相比,杨树林的分类效果同样改善,杨树林的PA和UA分别提高7.82和9.63个百分点。
4 讨 论
本研究利用不同特征选择方法,在不同数量训练像元条件下进行光谱特征选取试验,并统计各光谱区间的选择频次,分析各光谱区间对于区分研究区植被类型的重要性差异。结果显示,波段选取累计次数前三多的光谱区间分别为蓝波段(400~450 nm,38次)、红边波段(700~750 nm,35次)和红波段(600~650 nm,28次)。这与稀树灌草丛生态系统高光谱植被分类研究中的波段选择结果一致[37-38],即蓝、红和红边波段光谱特征最利于区分干旱区植被类型。不同类型植被叶片色素类型和含量的差异是利用反射率光谱区分植被类型的主要依据。其中,蓝波段反射率主要受到植被叶片叶绿素吸收的影响,对于区分针叶林与阔叶林十分有效[39],可有效区分研究区中杨树和梭梭林。红波段区间,尤其是680 nm附近反射率特征与叶面叶绿素与花青素含量相关[40],被认为是区分作物类型和植被类型最重要的光谱区间[41]。此次研究中区分不同树龄枸杞树,主要依据其绿叶素、氮素等营养物质差异。前人研究证实红边区间与植被叶绿素、氮素、水分含量以及叶面积指数(Leaf Area Index,LAI)等结构特征有重要相关性[42],同时红边区域对于区分高叶绿素含量和高LAI值的植被类型十分有效[43]。

表1 各分类数据集分类精度
注:ALL代表全部波段数据;TEX代表纹理特征;OA、PA、UA分别代表总体精度、生产者精度和用户精度。下同。
Note: ALL represents all bands; TEX texture feature; OA, PA, UA represent overall accuracy, producer accuracy, and user accuracy, respectively. Same below.

图7 4种分类数据集的RF植被分类结果
在光谱特征选择方法上,本文将GNDO方法应用于高光谱特征选择,并对比了不同样本数量条件下,GA、PSO、GWO与GNDO方法的特征选择结果和分类精度。结果显示,所有光谱特征分类数据集中,GNDO200整体分类精度最高,验证了GNDO方法对于高光谱特征选择的可行性。整体上,随训练像元的增加,各特征选择结果的总体分类精度均呈上升趋势。这与Sabat-Tomala等[21]的结论一致。而不同的特征选择方法的分类精度对训练像元数量表现出不同的依赖程度。其中,GA、GWO和GNDO方法在训练像元数量达到100或更多时,总体分类精度趋于稳定,而PSO方法在训练像元数量达到50时,总体分类精度就趋于稳定。在训练像元数量较少(30个)时,GA方法的特征选择结果获得了最高的分类精度,而在训练像元数量较大(200个)时,GNDO方法的特征选择结果获得了最高的分类精度。
为验证图像纹理特征对植被分类的有效性,在对VNIC数据全色波段进行纹理特征提取和筛选的基础上,结合纹理特征与光谱特征进行图像分类发现,图像纹理特征的加入显著提升了各分类数据集分类精度,这与Fu等[11,13]的研究结论一致。Zhang等[13]发现加入纹理特征后,光谱特征相似的屋顶和道路间的混分现象显著减少。本研究结果显示,加入纹理特征后,老枸杞林和杨树林的分类精度明显提升,与其他类型植被、背景地物的混分现象明显减少,这与杨树高大的冠层形成特征图像纹理和规则种植的枸杞林形成的规则线性纹理有关。
本研究利用不同特征选取方法结合随机森林分类算法进行植被类型自动分类试验,结果显示GWO200+TEX数据集获得了最高的总体分类精度(82.86%),与George等[44]利用Hyperion数据进行喜马拉雅西部植被类型分类的总体分类精度(82.27%)接近,高于Lim等[45]利用Hyperion数据进行中国和韩国局部区域树种分类的总体分类精度(64%~81%),验证了ZY1-02D数据对干旱区植被类型分类的可行性。但研究区北部各分类结果中均有部分像元被错分为新枸杞林,这是由于新种植的枸杞树区域植被覆盖度较低,加上混合像元效应,新枸杞林像元与裸地的光谱特征接近,导致误分类,下一步工作中,将进一步研究植被覆盖度对干旱区植被类型分类的影响,同时尝试采用高光谱图像融合方法,提升高光谱数据空间分辨率,缓解混合像元效应,进一步提升分类精度。由于西北半干旱地区植被类型图像分类研究对数据时相要求严格,同时ZY1-02D数据积累有限,此次研究仅收集到一景数据进行分类试验,后续将进一步验证GNDO算法对ZY1-02D数据和其他高光谱数据的适用性和稳定性。
5 结 论
利用不同的图像光谱和纹理特征提取和筛选方法,在不同训练像元下构建各分类数据集,利用随机森林算法进行植被类型分类,验证了国产ZY1-02D高光谱卫星数据对于干旱区植被类型分类的可行性。主要结论以下:
1)ZY1-02D AHSI数据各光谱区间对于区分研究区植被类型的重要性存在差异。蓝波段(400~450 nm)、红边波段(700~750 nm)和红波段(600~650 nm)对区分研究区植被类型最敏感。
2)训练样本规模对波段选取结果和分类精度存在影响。随着训练像元的增加,各分类数据集总体分类精度均呈上升趋势,使用200个训练像元和广义正态分布优化算法(Generalized Normal Distribution Optimization, GNDO)方法进行特征优选获取的分类数据集在基于光谱特征的各分类数据集中获得了最高的总体分类精度(80.44%),表明了GNDO应用于高光谱波段选取的可行性。不同的特征选择方法的分类精度对训练像元数量表现出不同的依赖程度。其中,遗传算法、灰狼优化算法和GNDO方法在训练像元数量达到100后,总体分类精度趋于稳定,而粒子群优化算法在训练像元数量达到50后,总体分类精度就趋于稳定。
3)图像纹理特征能有效提升植被类型图像分类精度。对于各分类数据集,图像纹理特征的加入均显著提升了分类精度,其中使用200个训练像元和GWO方法进行波段优选的结果与纹理特征结合的分类数据集(GWO200+TEX)获得最高的总体分类精度(82.86%)。
[1] Dashti H, Poley A, Glenn N F, et al. Regional scale dryland vegetation classification with an integrated lidar-hyperspectral approach[J]. Remote Sensing, 2019, 11(18): 2141-2156.
[2] Kokaly R F, Despain D G, Clark R N, et al. Mapping vegetation in Yellowstone National Park using spectral feature analysis of AVIRIS data[J]. Remote Sensing of Environment, 2003, 84(3): 437-456.
[3] Lawrence R L, Wood S D, Sheley R L. Mapping invasive plants using hyperspectral imagery and Breiman Cutler classifications (Random Forest) [J]. Remote Sensing of Environment, 2006, 100(3): 356-362.
[4] 吴见,彭道黎. 基于空间信息的高光谱遥感植被分类技术[J]. 农业工程学报,2012,28(5):150-153.
Wu Jian, Peng Daoli. Vegetation classification technology of hyperspectral remote sensing based on spatial information[J].Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2012, 28(5): 150-153. (in Chinese with English abstract)
[5] 刘帅,张旭含,李笑迎,等. 基于双分支卷积网络的高光谱与多光谱图像协同土地利用分类[J]. 农业工程学报,2020,36(14):252-262.
Liu Shuai, Zhang Xuhan, Li Xiaoying, et al. Cooperative land use classification of hyperspectral and multispectral imagery based on dual branch convolutional neural network[J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2020,36(14):252-262. (in Chinese with English abstract)
[6] Pengra B W, Johnston C A, Loveland T R. Mapping an invasive plant, Phragmites australis, in coastal wetlands using the EO-1 Hyperion hyperspectral sensor[J]. Remote Sensing of Environment, 2007, 108(1): 74-81.
[7] Skowronek S, Ewald M, Isermann M, et al. Mapping an invasive bryophyte species using hyperspectral remote sensing data[J]. Biological Invasions, 2017, 19(1): 239-254.
[8] Ishii J, Washitani I. Early detection of the invasive alien plant Solidago altissima in moist tall grassland using hyperspectral imagery[J]. International Journal of Remote Sensing, 2013, 34(16): 5926-5936.
[9] 孙伟伟,任凯,肖晨超,等. 资源一号02D卫星高光谱与多光谱融合数据滨海湿地分类应用[J]. 航天器工程,2020,29(6):162-168.
Sun Weiwei, Ren Kai, Xiao Chenchao, et al. Classification of coastal wetlands based on hyperspectral and multispectral fusion data of ZY-1-02D satellite[J]. Spacecraft Engineering, 2020, 29(6): 162-168. (in Chinese with English abstract)
[10] 李毅夫,王琫瑜,高志海,等. 资源一号02D卫星影像防护林信息提取方法[J]. 航天器工程,2020,29(6):174-179.
Li Yifu, Wang Bengyu, Gao Zhihai, et al. Shelter forest in formation extraction method of ZY-1-02D satellite remote sensing images[J]. Spacecraft Engineering, 2020, 29(6): 174-179. (in Chinese with English abstract)
[11] Fu Y Y, Zhao C J, Wang J H, et al. An improved combination of spectral and spatial features for vegetation classification in hyperspectral images[J]. Remote Sensing, 2017, 9(3): 1-16.
[12] Dashti H, Glenn N F, Ustin S, et al. Empirical methods for remote sensing of nitrogen in drylands may lead to unreliable interpretation of ecosystem function[J]. IEEE Transactions on Geoscience and Remote Sensing, 2019, 57(6): 3993-4004.
[13] Zhang L F Zhang L P, Tao D C. On combining multiple features for hyperspectral remote sensing image classification[J]. IEEE Transactions on Geoscience and Remote Sensing, 2012, 50(3): 879-893.
[14] Hughes G F. On the Mean Accuracy of Statistical Pattern Recognizers[J]. IEEE Transactions on Information Theory, 1968, 14(1): 55-63.
[15] Hennessy A, Clarke K, Lewis M. Hyperspectral classification of plants: a review of waveband selection generalisability[J]. Remote Sensing, 2020, 12(1): 113-139.
[16] Wang H, Han Z Z, Xie Q Y, et al. Finite-time chaos control via nonsingular terminal sliding mode control[J]. Communications in Nonlinear Science and Numerical Simulation, 2009, 14(6): 2728-2733.
[17] 姚旭,王晓丹,张玉玺,等. 特征选择方法综述[J]. 控制与决策,2012,27(2):161-167.
Yao Xu, Wang Xiaodan, Zhang Yuxi, et al. Summary of feature selection algorithms[J]. Control and Decision, 2012, 27(2): 161-167. (in Chinese with English abstract)
[18] Vaiphasa C, Skidmore A K, Boer W F D, et al. A hyperspectral band selector for plant species discrimination[J]. ISPRS Journal of Photogrammetry & Remote Sensing, 2007, 62: 225-235.
[19] Monteiro S T, Kosugi Y. A particle swarm optimization-based approach for hyperspectral band selection[C]. IEEE Congress on Evolutionary Computation.Piscataway, NJ: IEEE, 2007: 3335-3340.
[20] Zhang Y Y, Jin Z G, Mirjalili S. Generalized normal distribution optimization and its applications in parameter extraction of photovoltaic models[J]. Energy Conversion and Management, 2020, 224: 113301-113314.
[21] Sabat-Tomala A, Raczko E, Zagajewski B. Comparison of support vector machine and random forest algorithms for invasive and expansive species classification using airborne hyperspectral data[J]. Remote Sensing, 2020, 12(3): 516-536.
[22] Yu S X, Backer S D, Scheunders P. Genetic feature selection combined with composite fuzzy nearest neighbor classifiers for hyperspectral satellite imagery[J]. Pattern Recognition Letters, 2020, 23: 183-190.
[23] Chalermwat P, El-Ghazawi T, Lemoigne J. 2-phase GA-based image registration on parallel clusters[J]. Future Generation Computer Systems, 2001, 17: 467-476.
[24] Nguyen T, Han J, Park D. Satellite image classification using convolutional learning satellite image classification using convolutional learning[C]. AIP Conference Proceedings, Melville, USA: AIP Publishing, 2013.
[25] Mertens K C, Verbeke L P C, Ducheyne E I, et al. Using genetic algorithms in sub-pixel mapping[J]. International Journal of Remote Sensing, 2003, 24(21): 4241-4247.
[26] Cogdill R P, Hurburgh C R, Rippke G R. Single-Kernel maize analysis by near-infrared hyperspectral imaging[J]. American Society of Agricultural Engineers, 2006, 47(1): 311-320.
[27] Tjiong A S J, Monteiro S T. Feature selection with PSO and Kernel methods for hyperspectral classification[C]. 2011 IEEE Congress of Evolutionary Computation (CEC). Piscataway, NJ: IEEE, 2011: 1762-1769.
[28] 王艺婷,黄世奇,刘代志,等. 基于粒子群优化算法的波段选择方法[J]. 计算机仿真,2015,32(9):417-420.
Wang Yiting, Huang Shiqi, Liu Daizhi, et al. Band selection method based on particle swarm optimization[J]. Computer Simulation, 2015, 32(9): 417-420. (in Chinese with English abstract)
[29] 刘全金,赵志敏,李颖新,等. 基于近邻信息和 PSO 算法的集成特征选取[J]. 电子学报,2016,44(4):995-1002.
Liu Quanjin, Zhao Zhimin, Li Yingxin, et al. Ensemble feature selection method based on neighborhood information and PSO algorithm[J]. Acta Electronica Sinica, 2016, 44(4): 995-1002. (in Chinese with English abstract)
[30] 王俊,冯军,张戈,等. 基于改进灰狼优化算法的医学数据特征选择应用研究[J]. 河南大学学报:自然科学版,2020,50(5):570-578.
Wang Jun, Feng Jun, Zhang Ge, et al. Application research of medical data feature selection based application research of medical data feature selection based on improved grey wolf optimization algorithm[J]. Journal of Henan University: Natural Science, 2020, 50(5): 570-578. (in Chinese with English abstract)
[31] Emary E, Zawbaa H M, Hassanien A E. Binary gray wolf optimization approaches for feature selection[J/OL]. Neurocomputing, 2016, 172: 371-381.
[32] Haralick R M, Dinstein I, Shanmugam K. Textural features for image classification[J]. IEEE Transactions on Systems, Man and Cybernetics, 1973, 3(6): 610-621.
[33] 汪雪淼,孟庆岩,赵少华,等. GF-2在城市绿地分类及景观格局度量中的应用[J]. 地球信息科学学报,2020,22(10):1971-1982.
Wang Xuemiao, Meng Qingyan, Zhao Shaohua, et al. Urban green space classification and landscape pattern measurement based on GF-2 image[J]. Journal of Geo-information Science, 2020, 22(10): 1971-1982. (in Chinese with English abstract)
[34] Sluiter R, Pebesma E J. Comparing techniques for vegetation classification using multi- and hyperspectral images and ancillary environmental data[J]. International Journal of Remote Sensing, 2010, 31(23): 6143-6161.
[35] Burai P, Deák B, Valkó O, et al. Classification of herbaceous vegetation using airborne hyperspectral imagery[J]. Remote Sensing, 2015, 7(2): 2046-2066.
[36] Congalton R G. A review of assessing the accuracy of classifications of remotely sensed data[J]. Remote Sensing of Environment, 1991, 37(1): 35-46.
[37] Cho M A, Debba P, Mathieu R, et al. Improving discrimination of savanna tree species through a multiple-endmember spectral angle mapper approach: Canopy-level analysis[J]. IEEE Transactions on Geoscience and Remote Sensing, 2010, 48(11): 4133-4142.
[38] Naidoo L, Cho M A, Mathieu R, et al. Classification of savanna tree species, in the Greater Kruger National Park region, by integrating hyperspectral and LiDAR data in a Random Forest data mining environment[J]. ISPRS Journal of Photogrammetry and Remote Sensing, 2012, 69: 167-179.
[39] Gong P, Ruilianp P, Yu B. Conifer species recognition: An exploratory analysis of in situ hyperspectral data[J]. Remote Sensing of Environment, 1997, 62(2): 189-200.
[40] Blackburn G A. Hyperspectral remote sensing of plant pigments[J]. Journal of Experimental Botany, 2007, 58(4): 855-867.
[41] Mariotto I, Thenkabail P S, Huete A, et al. Hyperspectral versus multispectral crop-productivity modeling and type discrimination for the HyspIRI mission[J]. Remote Sensing of Environment, 2013, 139: 291-305.
[42] Clark M L, Roberts D A, Clark D B. Hyperspectral discrimination of tropical rain forest tree species at leaf to crown scales[J]. Remote Sensing of Environment, 2005, 96(3/4): 375-398.
[43] Adam E, Mutanga O. Spectral discrimination of papyrus vegetation (L.) in swamp wetlands using field spectrometry[J]. ISPRS Journal of Photogrammetry and Remote Sensing, 2009, 64(6): 612-620.
[44] George R, Padalia H, Kushwaha S P S. Forest tree species discrimination in western Himalaya using EO-1 Hyperion[J]. International Journal of Applied Earth Observation and Geoinformation, 2014, 28(1): 140-149.
[45] Lim J, Kim K M, Jin R. Tree species classification using hyperion and sentinel-2 data with machine learning in South Korea and China[J]. ISPRS International Journal of Geo-Information, 2019, 8(3): 150-172.
Method for classifying vegetation types in arid areas combining spectral and textural features of ZY-1 02D
Shuai Shuang1,3, Zhang Zhi2※, Zhang Tian2, Xiao Chengzhi2, Chen Si3, Ma Zicheng2, Xie Cuirong3
(1.,(),430074,; 2.,(),430074,; 3.,430010,)
With the continuous development of hyperspectral remote sensing technology, it has been widely used in vegetation mapping. However, sparse vegetation canopy, soil background, and spectral similarity between different types of vegetation are still the main challenges for vegetation types mapping in arid areas. As a result, it is difficult to achieve reliable classification accuracy by using spectral or texture features separately. Generalized Normal Distribution Optimization (GNDO) is a new feature optimization algorithm, with advantages in quality and stability of feature extraction results, comparing to traditional optimization algorithms. But it has not yet been applied to select bands of hyperspectral data. In order to validate the feasibility of combining ZY-1 02D spectral and texture features to classify vegetation types in arid areas, to verify the effectiveness of the GNDO method for bands selection of hyperspectral data, and to explore the effects of feature selection methods and training sample numbers on the classification accuracy of vegetation mapping, different Wrapper Optimization methods, such as Genetic Algorithm (GA), Particle Swarm Optimization (PSO), Grey Wolf Optimization (GWO), and GNDO, were applied to select spectral features for vegetation mapping, taking the area around Zongjia Town, Dulan County, Qinghai Province, China as the research area, then the band selection results of these methods were analyzed. Train sample set containing 30, 50, 100, 150, and 200 pixels per class were used to select bands and to train the classifier. Different methods (ALL (without bands selection), GA, PSO, GWO, GNDO) and different sizes of the trained sample sets (30, 50, 100, 150, and 200 pixels per class) were used to obtain 25 spectral feature-based classification data sets. Simultaneously, 8 texture features (mean, variance, homogeneity, contrast, dissimilarity, entropy, second moment) were extracted using the Gray-level co-occurrence matrix (GLCM) method, and selected on basis of distinguishability for vegetation types. Texture features (TEX) were combined with spectral feature-based classification data sets. The random forest classification method was applied to classify vegetation types for the classification data sets, and the classification accuracy of classification data sets was evaluated and compared. The results show that 1) the blue region (400-450 nm), the red edge region (700-750 nm), and the red region (600-650 nm) are the most sensitive to distinguish the vegetation types in the study area; 2) the GNDO200 achieved the highest overall classification accuracy (80.44%) among the spectral feature-based classification data sets, which was better than the classification accuracy (78.86%) using all bands (ALL200); 3) with the increase of training samples, the overall classification accuracy of each classification data set showed an increasing trend, the classification accuracy of different feature selection methods showed different reliance on the number of training samples; 4) image texture features significantly improved the classification accuracy, and the GWO200+TEX dataset had the highest overall classification accuracy (82.86%). This study could verify the potential of the ZY1-02D, the new hyperspectral satellite data, for the classification of vegetation types in arid areas, and provide an idea for the selection of spectral and texture features in hyperspectral vegetation mapping.
remote sensing; algorithm;ZY1-02D; vegetation classification; feature selection; GNDO
10.11975/j.issn.1002-6819.2021.21.023
P237;TP79;S-3
A
1002-6819(2021)-21-0199-9
帅爽,张志,张天,等.结合ZY-1 02D光谱与纹理特征的干旱区植被类型遥感分类[J]. 农业工程学报,2021,37(21):199-207.doi:10.11975/j.issn.1002-6819.2021.21.023 http://www.tcsae.org
Shuai Shuang, Zhang Zhi, Zhang Tian, et al. Method for classifying vegetation types in arid areas combining spectral and textural features of ZY-1 02D[J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2021, 37(21): 199-207. (in Chinese with English abstract) doi:10.11975/j.issn.1002-6819.2021.21.023 http://www.tcsae.org
2021-06-11
2021-09-15
青海省青藏高原北部地质过程与矿产资源重点实验室专项基金(2019-kz-01);青海省科技厅创新平台建设专项项目“青海省自然资源要素与生态状况一体化遥感监测应用平台”(2019-ZJ-T04);中国地质调查局项目(DD20191011;DD20190705;DD20190511)
帅爽,博士生,研究方向为遥感地学应用。Email:21844ss@cug.edu.cn
张志,博士,教授,研究方向为遥感地学应用。Email:3slab@cug.edu.cn

