甘草PRR基因家族鉴定及表达分析
关思静, 王楠, 徐蓉蓉, 葛甜甜, 高静*,颜永刚, 张岗, 陈莹, 张明英
(1.陕西中医药大学药学院,陕西省秦岭中草药应用开发工程技术研究中心,西安712046;2.陕西中医药大学,陕西中药资源产业化省部共建协同创新中心,陕西 咸阳 712083)
原核生物中广泛存在一种信号转导机制,即双组分调节系统,该系统最关键的过程是His-ASP在HK(his-kinase)和RR(response regulator)之间的磷酸转移反应[1]。Makino等[2]在编译拟南芥所有RR时发现了一组基因,每一个都是编码ARR(authentic response regulator)类蛋白质,但是这些基因却不具有真正的RR所具有的接受磷的Asp残基,而是被Glu取代,因此将它们命名为伪答调控(pseudo response regulator,PRR)蛋白,每个PRR都包含一个N端REC的伪响应调节受体域(pseudo-receiver domain)以及C端植物特有的CCT结构域[3]。基于结构特征,在陆生植物中PRR有3个分支,包括TOC1(PRR1)、PRR7/3和PRR5/9[4]。这5个PRR基因在拟南芥生物钟系统中扮演着重要的角色,Matsushika等[5]研究证实,它们以PRR9→PRR7→PRR5→PRR3→PRR1的顺序被转录翻译。目前,PRR家族成员的分子功能尚不清楚,但其生物作用已被明确与昼夜节律相关。
昼夜节律是生活在世界上许多生物体的“时钟或振荡器”产生的,这种生物节律维持一个接近24 h的周期,与地球绕着地轴自转的周期相对应,同时受到植物体内生物钟的控制,作为一种计时机制,存在于多种生物体中,控制着生物体遗传、代谢和生理过程的时间[6],包括光合作用[7-8]、营养生长阶段的生长速度[9]、开花时间、非生物和生物胁迫反应,最具有特色的昼夜节律事件之一是花期的光周期调控[10]。目前,模式植物拟南芥生物钟分子机制已经得到了广泛的研究,其基本结构包含2个MYB相关的转录因子LHY(late elongated hypocoty)和CCA1(circadian clock-associated 1)以及中心振荡器的另一个重要组成部分,属于PRR蛋白的TOC1(timing of Cab expression)[11-12]。其中CCA1和LHY在早上表达,并抑制晚间表达的TOC1,而TOC1又抑制CCA1和LHY的转录[13-14]。除此之外,PRR家族其他成员(PRR9/7)与CCA1和LHY的启动子结合并抑制其表达;而CCA1和LHY又通过结合启动子直接促进PRR7和PRR9的表达[15-16]。
PRRs 在模式植物拟南芥中的研究已经相当清晰,在其他植物中PRRs的研究较少,多集中于农作物,如水稻中鉴定了5个与拟南芥同源的PRR基因,即OsPRR1、OsPRR37、OsPRR73、OsPRR59、OsPRR95[17];小麦TaPRR37编码PRR家族蛋白,与拟南芥APRR7和水稻OsPRR37同源[18];大麦HvPRR37编码PRR家族蛋白,与拟南芥APRR7同源[19]。甘草是常见的大宗药材,多生长在西北干旱环境下,在甘草蛋白分子研究中,已有的工作主要集中在对活性成分合成相关基因的克隆和表达分析[20-21],而对PRR蛋白在甘草中的研究却未见报道。为了揭示PRRs在不同胁迫下的潜在作用,本研究对甘草PRR基因家族(GuPRRgene family)进行了系统分析,并在全基因组水平上分析了PRR基因在干旱、盐和低磷非生物胁迫下的表达模式,阐明不同胁迫条件下PRRs基因的功能,为进一步了解植物PRRs基因的进化关系提供了信息。
1 材料与方法
1.1 试验材料
试验用乌拉尔甘草(GlycyrrhizauralensisFisch.)种子购自峰华农业有限公司,种子经消毒后,于人工气候箱(光/暗周期为12 h/12 h)中催芽1周,挑选长势一致的幼苗采用霍格兰营养液水培。选择生长1月的幼苗,分别采用15%聚乙二醇(PEG-6000)进行干旱胁迫处理、150 mmol·L-1氯化钠(NaCl)进行高盐胁处理、10 μmol·L-1磷酸二氢钾进行低磷(LowP)胁迫处理。对照(CK)仅用营养液培养,不做任何处理。3个生物学重复,在胁迫处理1周后采集对照和处理组的甘草幼苗地上部分和地下部分,-80 ℃冰箱保存备用。
1.2 试验方法
1.2.1转录组测序 委托杭州景杰生物科技有限公司利用Illumina HiSeq 2000测序平台对采集甘草样本进行高通量测序分析。将测序得到的原始图像数据文件经碱基识别转化为raw Data。使用Trimmomatic软件设置默认参数,去除Illumina平台的FASTQ序列中的接头,并根据碱基质量值对FASTQ进行修剪,得到clean reads,然后用TopHat2进行参考基因组比对分析。使用RPKM法对reads count进行均一化处理,评估基因的表达量。
1.2.2甘草中伪应答调控蛋白(PRR)的筛选与鉴定 甘草基因组数据来自Mochida等[22]已发表数据(http://ngs-data-archive.psc.riken.jp/Gur-genome/);以拟南芥PRR家族成员APRR1、APRR3、APRR5、APRR7、APRR9基因序列作为查询序列,运行TBtools工具的本地检索程序[23],筛选阈值设为1×10-5,初步获得候选甘草PRR基因家族成员;将候选基因的蛋白序列提交到NCBI,进行在线blastp比对,根据注释信息筛选出甘草PRR家族相关的基因;然后将二次比对得到的序列通过NCBI-CDD数据库和pfam数据库(https://pfam.xfam.org/)进行进一步的结构域验证,去掉结构域不完整的蛋白序列,最终鉴定出甘草PRR家族成员。
1.2.3甘草PRR家族基因的生物信息学分析 运行TBtools软件的Batch SMART程序,对甘草PRR蛋白保守结构域可视化;采用ExPASY-Prot Param(https://web.expasy.org/protparam/)分析筛选出的甘草PRR基因编码蛋白的理化性质;借助Cell-PLoc2.0(http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/)进行蛋白亚细胞定位预测;用SOMPA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)预测蛋白二级结构;同时采用SWISS-MODEL(https://swissmodel.expasy.org/)进行甘草PRR蛋白三维建模分析并用PROCHECK(https://servicesn.mbi.ucla.edu/PROCHECK/)对蛋白质三级结构检验;利用MEME(http://meme-suite.org/tools/meme)在线工具对甘草PRR蛋白进行保守基序分析;提取甘草PRR基因上游2 kb的序列信息,使用PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)预测顺式作用元件;通过获得的甘草基因序列和CDS序列,利用GSDS2.0(http://gsds.cbi.pku.edu.cn/)分析甘草PRR基因结构;采用MEGA7.0软件进行氨基酸比对和构建进化树,并用GeneDoc软件编辑比对后的序列。
1.2.4甘草PRR基因家族表达模式分析 利用转录组测序数据分析甘草PRR基因(GuPRR)的表达特征。转录组数据包含甘草干旱、高盐以及低磷胁迫处理和对照的基因表达数据,以不同实验条件下的RPKM值为表达水平,通过对数据进行对数值转换,利用TBtools的HeatMap程序做热图分析。
2 结果与分析
2.1 甘草PRR基因家族的鉴定
利用TBtools和blastp两种比对方法,从甘草基因组中鉴定出13条PRR候选蛋白序列。之后通过NCBI-CDD和pfam数据库对13条候选蛋白序列进行保守结构域验证,发现其中7条具有完整的REC、CCT结构域(图1),其余6条由于结构域不完整而去除。分析这7个甘草PRR基因编码的蛋白质理化性质和亚细胞定位(表1)发现,这些蛋白质的氨基酸大小介于553~837 aa之间;分子质量在61.930~91.683 kD之间;等电点最低为5.75(GuPRR2),最高为8.45(GuPRR4),其中等电点大于7的有4条,小于7的有3条;氨基酸不稳定系数均大于40,表明这7条甘草PRR蛋白均为不稳定蛋白质;总平均亲水性均为负值,推测这7条甘草PRR蛋白均为亲水蛋白质。亚细胞定位结果显示,这7条甘草PRR蛋白均存在于细胞核中,与Fuijiwara等[24]的研究结果吻合,进一步验证了这7个预测基因属于PRR基因家族。

表1 GuPRR基因家族理化特性

图1 甘草PRR蛋白保守结构域
2.2 甘草PRR蛋白二级及高级结构
由SOMPA预测甘草PRR蛋白二级结构,结果(表2)发现,甘草PRR蛋白二级结构由α-螺旋、β-转角、延伸链以及无规则卷曲4种成分组成,其中α-螺旋与无规则卷曲为主要组成成分,β-转角所占比例最小。随后利用SWISS-MODEL预测甘草PRR蛋白三级结构,结果如图2所示,这7个PRR蛋白三级结构可分为4种类型,GuPRR1含有的α-螺旋结构数量最少,却含有最多的延伸链;GuPRR2和GuPRR3具有相似度较高的三维结构,α-螺旋占比高于其他GuPRR蛋白;GuPRR4和GuPRR5三级结构相似度较高,GuPRR6和GuPRR7三级结构相似度较高。为了检验蛋白三级构象的合理性,采用了蛋白质结构检验工具PROCHECK对甘草的PRR蛋白三级结构进行检验,发现7个甘草PPR蛋白氨基酸残基位于最佳区和次允许区域的比例分别为98.2%、98.2%、97.8%、97.6%、96.6%、99.4%、99.4%,均超过96%,且G-factors值都大于-0.5,说明这7个PRR蛋白的三维结构是合理的。

表2 甘草PRR蛋白二级结构
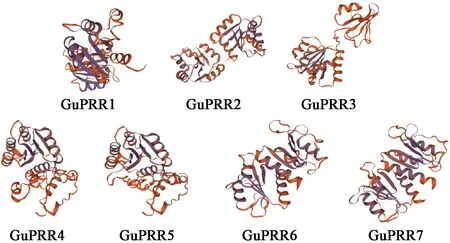
图2 甘草PRR蛋白三级结构预测
2.3 甘草PRR基因的顺式作用元件
提取甘草PRR基因上游2 kb的序列,分析其顺式作用元件,结果(图3)发现,GuPRR基因上游存在4类顺式作用元件:①光响应相关元件,如GT1-motif元件、G-box元件、Box4元件、GA-motif元件等;②植物激素响应相关元件,如响应SA代谢的TCA-element元件、响应ABA代谢的ABRE 元件、响应MeJA代谢的CGTCA-motif元件等;③植物生长发育响应相关元件,如分生组织表达相关的CTA-box、昼夜节律调节中的circadian元件等;④逆境胁迫响应相关元件,如干旱胁迫响应元件MBS、低温胁迫响应元件LTR等。进一步研究发现,7条GuPRR基因中都有光响应元件,共111个,占预测顺式作用元件42.7%,表明甘草PRR基因家族可能在光响应调节中扮演着重要角色。

图3 GuPRR基因顺式作用元件
2.4 甘草PRR蛋白系统进化关系
为了了解甘草PRR基因家族成员的进化关系,利用双子叶植物甘草、拟南芥、大豆、豌豆,以及单子叶植物水稻、小麦、高梁PRR基因家族的全蛋白序列构建进化树。结果(图4)显示,这几个物种的PRR蛋白被分为4个分支(图4),甘草的PRR蛋白分成3组。进一步分析发现,GuPRR1与APRR1相近,GuPRR2与GuPRR3与APRR5/9更加相近,而GuPRR4~7则归属于APRR3/7一组,其中GuPRR4和GuPRR5与APRR3更为相近,GuPRR6和GuPRR7与APRR7更为接近。通过对系统进化树分支分析发现,甘草与大豆、豌豆的进化距离较其他物种更短,说明甘草的PRR基因与同科的大豆、豌豆进化关系更为接近。

注:Gu—甘草;Ps—豌豆;Ga—大豆;A—拟南芥;Os—水稻;Ta—小麦;Sb—高粱。
2.5 甘草PRR家族基因结构
利用MEME在线工具预测甘草PRR蛋白的保守基序,共预测到22个motif,结果如图5所示,motif 1和motif 3作为REC结构域的保守基序、motif 2和motif 4作为CCT结构域的保守基序,这4个motif出现在所有甘草PRR蛋白中。此外,GuPRR2和GuPRR3、GuPRR4和GuPRR5、GuPRR6和GuPRR7具有相同的motif。为了进一步了解PRR基因的进化关系,对每个基因的内含子/外显子结构进行分析,结果(图6)显示,GuPRR基因的外显子数量8~12不等,内含子数量5~11不等。PRR家族基因成员的系统进化树分析表明,位于同一分支的GuPRR基因具有相似的内含子/外显子分布,如GuPRR4和GuPRR5基因都具有11个内含子和12个外显子;不同于GuPRR4和GuPRR5所在的分支,另一分支的GuPRR2和GuPRR3具有8个内含子和10个外显子,这表明不同的甘草PRR基因在进化过程中其结构与功能已经发生了分化。保守基序分析表明,预测的GuPRRs伪受体区域氨基酸序列与APRRs的氨基酸序列高度一致(图7A),且它们的中心含有独特的谷氨酸残基(E);CCT结构域序列的预测氨基酸序列也与APRRs序列一致(图7B),且REC结构域与CCT结构域中间的氨基酸序列具有很大的差异性。

图5 GuPRR基因家族保守基序

图6 GuPRR基因结构

A:伪响应调节受体域;B:CCT结构域
2.6 表达模式
图8 A所示,甘草地上部分的PRR基因在对照(CK)处理下,GuPRR1/4/5的表达量高于GuPRR2/3/6/7;盐胁迫(NaCl)处理中,仅GuPRR1上调表达,其余GuPRRs表达量下调或未检测到表达量;干旱胁迫(PEG)处理下,GuPRR1/2/3的表达量上调,GuPRR6/7/4/5表达量很低或表达量下调;低磷胁迫(LowP)处理下,仅GuPRR1表达量明显下调,其余GuPRR均上调表达。甘草根PRR基因在各处理中表达模式如8B所示,对照(CK)中,7条GuPRRs均具有较高的表达量;盐胁迫(NaCl)处理中,仅GuPRR2上调表达;干旱胁迫(PEG)与低磷胁迫(LowP)处理下,7条GuPRR基因表达量下调明显,均低于对照(CK)处理。

A:甘草地上部分;B:甘草根
3 讨论
PRRs在植物中普遍存在,并已被证明在调节植物昼夜节律中发挥作用[1,3,25]。PRRs包含有2个特殊的保守结构域,分别是N端的REC和C端的CCT,这2个保守结构域被一长段不保守的可变域分隔开。研究表明,PRRs的伪响应调节受体域来自能够磷酸化的接收域[4],这个接收域中包含1个保守的天冬氨酸-天冬氨酸-赖氨酸(DDK)基序,而在被子植物的PRRs中,第2个天冬氨酸(D)已经转化为谷氨酸(E)[1]。本研究利用生物信息学从甘草基因组中鉴定得到7个甘草PRR基因,GuPRR1、GuPRR2、GuPRR3、GuPRR4、GuPRR5、GuPRR6、GuPRR7与拟南芥和水稻的PRR基因高度同源。7个基因编码的蛋白都具有完整的REC和CCT保守结构域,预测的GuPRRs与APRRs的伪响应调节受体区域氨基酸序列一致,且在中心包含一个独特的E。进化分析将这7个甘草PRR蛋白聚为3类,与拟南芥和水稻的PRR蛋白进化一致。这些结果说明7个PRR基因属于甘草伪响应调节基因。
大部分干旱应答基因都表现出节律性表达模式,干旱引发的生理反应也受昼夜节律调节的影响[26-27]。Marcolino-Gomes等[28]采用RNA-seq和定量分析发现干旱胁迫引起大豆昼夜节律基因表达的显著变化,其中仅有PRR3在中度干旱胁迫处理下上调表达,而PRR3/7/9均表达减弱。干旱胁迫导致水稻大部分早晨生物钟基因表达振幅减弱,傍晚和夜晚生物钟基因表达增强[29]。另外,盐胁迫也会反馈调节生理时钟的振幅和周期[11],Habte等[30]研究表明,盐胁迫改变了大麦根部的时钟基因表达,使大麦对逆境胁迫的响应更为灵敏。本研究结果中,由对照处理可以看出,PRR基因在甘草根中的表达量高于地上组织,但在胁迫处理中,根中的GuPRR基因仅GuPRR2在盐胁迫处理中上调表达;而地上组织中,在不同胁迫处理下均有上调表达的GuPRR基因,如盐胁迫处理中的GuPRR1、干旱胁迫处理中的GuPRR2/3、低磷胁迫处理下的GuPRR4等基因,这表明,不同部位甘草PRR基因在胁迫响应中存在明显差异,推测地上组织中的PRR基因在胁迫响应中具有更明显的正向调控作用。
生物钟基因的转录调控可能通过这些基因启动子区域中存在的顺式作用元件发生,因此,本研究分析了PRRs启动子区域,并预测到了昼夜节律和光响应元件,包括circadian(CAAAGATATC)、G-box(ACACGTGT)等,这些结果暗示了甘草PRRs可能参与昼夜节律调控。除此之外,在启动子序列中还预测到干旱响应性元件(MBS)、低温响应性元件、无氧诱导相关元件以及与脱落酸、水杨酸、茉莉酸甲酯、生长素等激素相关的顺式反应元件。植物体内激素的合成也受昼夜节律调节。生物钟调节组分CHE(CCA1 hiking expedition)已被证明可以与水杨酸生物合成的主要基因ICS1(isochorismate synthase 1)的启动子元件结合,并调节水杨酸表达[31]。Li等[32]研究表明,水杨酸可显著提高乌拉尔甘草不定根中甘草酸、甘草次酸以及多糖等有效成分的含量。Liu等[33]通过 ChIP-seq 技术发现PRR7的靶基因在其上游区域包含脱落酸响应元件(ABRE),并调控脱落酸的表达量。研究表明,干旱胁迫下甘草根脱落酸含量显著增高,且与甘草酸的含量呈显著正相关[34]。乙烯和生长素的生物合成也被认为受到昼夜节律的调节[35-36],二者能够促进植物根的形成,进而提高植物的生物量以及抗胁迫能力。因此,可以推测甘草PRR基因可能通过调控水杨酸、脱落酸等激素水平影响其生长发育和次生代谢产物合成。
甘草是豆科多年生草本植物,除了重要的药用价值,因具有抗寒、抗旱、抗盐碱等优良特性同时也是重要的草地资源之一。当前,甘草全基因组草图序列已经公布[22],有助于进一步通过分子育种提高生物活性性状和生产力。PRR基因促进植物生长发育,同时也响应外界非生物胁迫,本研究获得的7个PRR基因,从理化性质、定位、基因结构、进化分析等方面初步鉴定属于甘草PRR基因。但PRR家族基因成员目前仅在模式植物拟南芥中研究较为广泛,其结构、功能以及表达模式仍然需要更加系统的研究,从而更全面地揭示PRR家族基因的分子机制,为今后研究甘草生长抗逆的分子机制提供科学依据。

