印度血桐与中平树基因组调查及SSR分子标记分析
李江莹 陆添权 杨俊波 田波





摘 要: 印度血桐与中平树是大戟科血桐属植物,该属植物具有多种药用价值,被广泛应用于民间医学中许多疾病的治疗,这两种植物种子中含有的神经酸也引起了研究者的高度关注。为确定适合印度血桐与中平树的全基因组测序研究策略,该研究采用二代高通量测序技术,结合生物信息学的方法首次测定了印度血桐与中平树的基因组大小、杂合率、重复率等基因组信息并初步分析了两种材料的SSR序列特征。结果表明:(1)印度血桐与中平树的基因组大小分别为986.84和946.23 M。(2)印度血桐与中平树的杂合率分别为0.75%和0.65%,重复序列比例分別为73.02%和71.5%。(3)通过对2种材料基因组序列的SSR特征分析,在印度血桐中共鉴定了4 499 185个SSR,在中平树中共鉴定了4 969 098个SSR。该研究结果为印度血桐与中平树SSR分子标记的筛选、开发以及全基因组深度测序提供了理论指导。
关键词: 印度血桐, 中平树, 神经酸, 基因组调查, SSR
中图分类号: Q943
文献标识码: A
文章编号: 1000-3142(2021)11-1897-08
Genome survey and analysis of SSR molecular markers on Macaranga indica and M. denticulata
LI Jiangying1,3, LU Tianquan1, YANG Junbo2, TIAN Bo1*
( 1. Key Laboratory of Tropical Plant Resource and Sustainable Use, Xishuangbanna Tropical Botanical Garden, Chinese Academy of Sciences, Kunming 650223, China; 2. Germplasm Bank of Wild Species, Kunming Institute of Botany, Chinese Academy of Sciences, Kunming 650204, China; 3. University of Chinese Academy of Sciences, Beijing 101408, China )
Abstract: Macaranga indica and M. denticulata belong to the genus Macaranga Thou.(Euphorbiaceae family). Plants of this genus are widely used in treatments of many diseases in Chinese folk medicine. The nervonic acid contained in the seeds of these two plants has also attracted great attention of researchers. In order to determine the strategy of the whole genome of M. indica and M. denticulata. In the present study, we first determined the genome sizes, heterozygosities, and repetitive rates of M. indica and M. denticulata through the second generation sequencing technology and bioinformatics methods, and the SSR sequence characteristics of M. indica and M. denticulata were preliminarily analyzed. The results were as follows: (1) The genome sizes of M. indica and M. denticulata were 986.84 and 946.23 M, respectively; (2) Heterozygosities and repetitive rates were 0.75% and 73.02% respectively in M. indica, and 0.65% and 71.5% in M. denticulata; (3) In addition, 4 499 185 and 4 969 098 genomic simple sequence repeat (SSR) markers in M. indica and M. denticulata were generated respectively. The results provide theoretical guidance for deep whole-genome sequencing of the two species and the screening and development of SSR molecular markers of M. indica and M. denticulata.
Key words: Macaranga indica, Macaranga denticulata, nervonic acid, genome survey, simple sequence repeat (SSR)
印度血桐(Macaranga indica)与中平树(M. denticulata)属于大戟科铁苋菜族血桐属(Macaranga Thou.)植物,二者均为叶盾状着生的高大乔木,广泛分布于我国西南地区的山谷、次生林或常绿阔叶林中。《中华本草》中记录了中平树的主要药用部位根与树皮具有退黄、清热利湿等功效,可用于治疗胃脘疼痛、黄疸型肝炎(黄建猷等,2015)。已有相关研究报道了从印度血桐中分离出了鞣花酸,异戊烯化黄酮等多种化合物,这些化合物具有如抗氧化作用、抗炎作用等多种生物活性,可发展为一种新的工业萃取源(Yang et al., 2015)。通过对印度血桐与中平树种子中所含脂肪酸的成分测定,我们发现印度血桐与中平树的种子脂肪酸组成成分中,都含有超长链单不饱和脂肪酸——神经酸,这一实验结果验证了王性炎等(2006)在多份植物样品中发现盾叶木 [注: 新版《中国植物志》已修订盾叶木(Macaranga adenantha)与印度血桐(Macaranga indica)为同一个种]是自然界已发现的植物中种子油脂中神经酸含量较高的木本植物,是目前已发现物种中较为理想的开发神经酸产品的植物资源。神经酸是大脑纤维和神经细胞的核心天然成分,与脑部神经的生物合成密切相关,具有多种重要的生物学功能,如促进大脑发育、改善记忆、延缓大脑衰老(Li et al., 2019),摄入神经酸能预防和治疗老年痴呆症、脑中风后遗症、脑萎缩、脑瘫、健忘失眠及记忆力减退等脑神经系统疾病(田德雨等,2015)。基于以上原因,神经酸的开发与应用引起了国内外专家的高度关注。因此,为满足人们对神经酸的需求,利用富含神经酸的植物开发神经酸产品已成为获取神经酸的主要途径。
目前,对印度血桐与中平树的研究主要集中在常见药用部位化学成分的药理活性及种子脂肪酸成分方面,而关于印度血桐与中平树基因组信息的研究未见报道,这给高效利用印度血桐与中平树野生资源选育植物新品种带来了极大不便,由于印度血桐与中平树均为木本植物,基因组大小尚不明确,各种因素使得血桐属植物的分子生物学研究进展缓慢,因此在对两种材料进行全基因组深度测序之前,需要先对两种材料进行低覆盖度的基因组调查,以了解材料基因组的组成特征和模式(Li et al., 2019)。深入分析DNA中的遗传信息是一项浩大的工程,其首要任务就是突破技术上的重难点(Albach et al., 2007)。植物全基因组的研究进程的迅速发展得益于新一代测序技术日益进步(施季森等,2012)。随着测序技术的逐渐成熟及测序价格的降低,基因组测序已被广泛应用到各种具有科研价值、经济价值、观赏价值的物种中。物种的基因组测序有助于我们了解各种生物体中生命现象的调控机制以及物种的群体进化、生长发育等生物学问题。目前,可对物种进行基因组大小测定的方法有流式细胞术、Feulgen分光光度法、脉冲场凝胶电泳法以及在技术不断进步革新的条件下快速发展的高通量测序技术(伍艳芳等,2014)。在大戟科植物中,麻风树、蓖麻、木薯、橡胶树等(Chan et al., 2010; Shusei et al., 2011; Simon et al., 2012; Zou & Yang, 2019)植物的基因组信息已有报道,这些已测物种的基因组可为我们研究大戟科血桐属植物的基因组信息提供参考。
本研究采用Illumina二代高通量测序技术,首次对印度血桐与中平树进行了基因组调查,并利用生物信息学方法估计了两种材料的重复率、杂合率及基于基因组调研的SSR (simple sequence repeat)特征分析,旨在为印度血桐与中平树的全基因组的测序和组装方案的制定和该属植物的进一步研究和开发利用提供依据为血桐属植物的遗传改良提供支持,也为进一步运用SSR分子标记在对两种材料的种质资源保护和遗传多样性等方面的研究提供一定参考。
1 材料与方法
1.1 材料
本实验材料印度血桐与中平树均为正常开花结果的野生植株,于2019年7月采自西双版纳傣族自治州景洪市勐龙镇勐宋村公路边,带回实验室,液氮速冻后放置于-80 ℃超低温冰箱保存,备用。
1.2 样品基因组DNA的提取、检测与测序
采用CTAB法提取印度血桐与中平树叶片基因组DNA,紫外分光光度计检测样品浓度,琼脂糖凝胶电泳检测完整性;将提取的DNA样品送至公司进行建库测序,参考其他木本植物的基因组大小及大戟科植物的C值范圍,选取1 Gb左右的基因组大小来评估印度血桐与中平树的基因组测序覆盖度。
1.3 建库信息及数据量统计
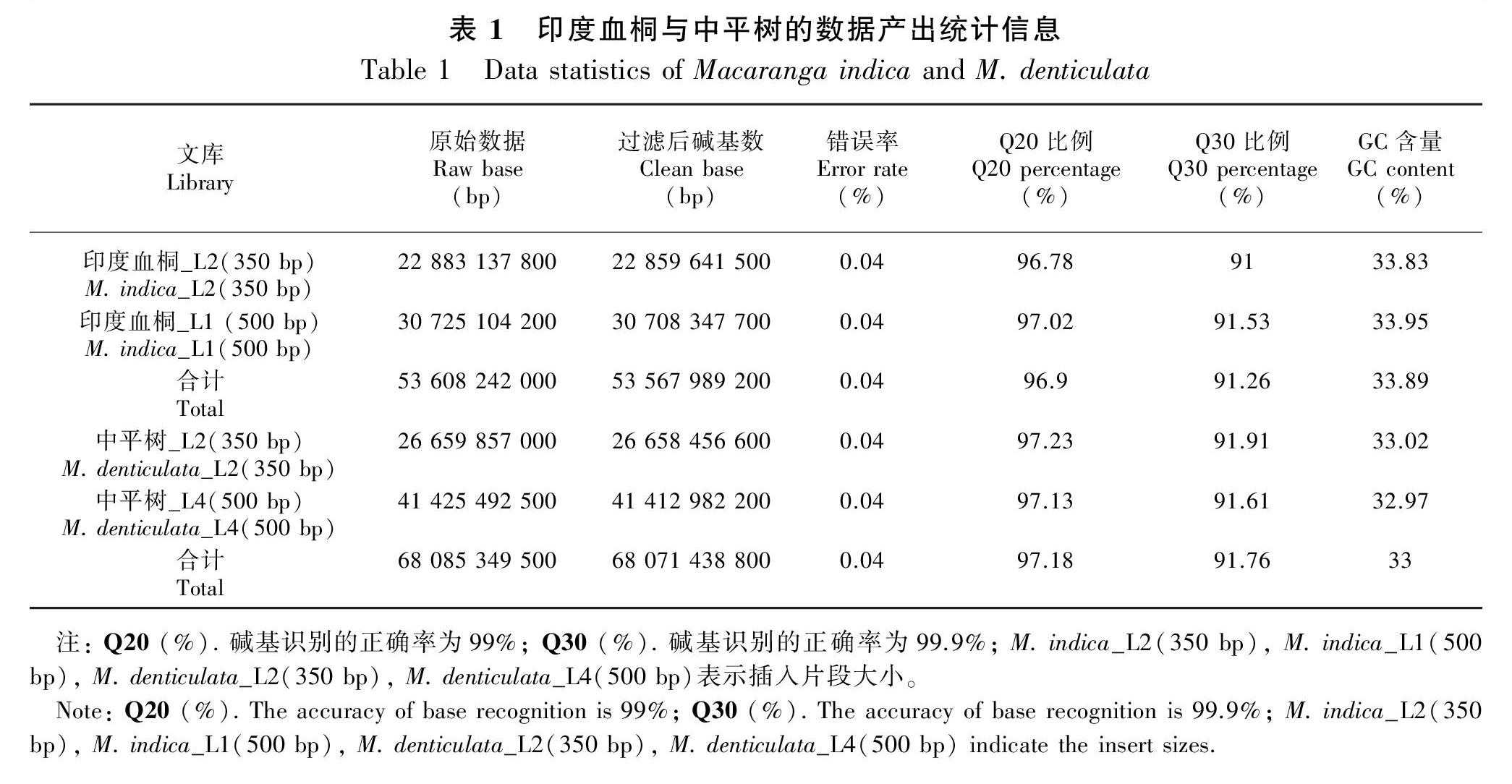
印度血桐与中平树基因组采用二代测序技术,利用WGS (全基因组鸟枪法),分别构建插入片段为350 和500 bp的DNA文库,再用Illumina HiseqTM2000平台进行双末端(Pair-End)测序,最终得到原始测序数据(raw reads),取全部原始数据对测序结果进行图像识别,去污染,去接头;统计结果包括测序read数量、数据产量、测序错误率、Q20、Q30、GC含量等。
1.4 基因组大小预测和杂合度估计
将本实验通过测序得到的序列,基于K-mer的分析方法估计印度血桐与中平树的基因组大小及杂合率,取K=17进行分析。K-mer分布图用来判断基因组的重复序列多少,如果材料的基因组重复比例较高,K-mer分布图右侧将会出现拖尾现象。mer深度分布服从泊松分布,根据曲线获得K-mer 深度期望值,用于估计基因组大小(周媛等,2019)。另外,在K-mer的分布曲线中,一般会出现一个覆盖度最高的主峰,若在主峰两侧出现另一个小峰,则说明该材料的基因组有较高的杂合度;反之,则没有。
1.5 样品污染判断
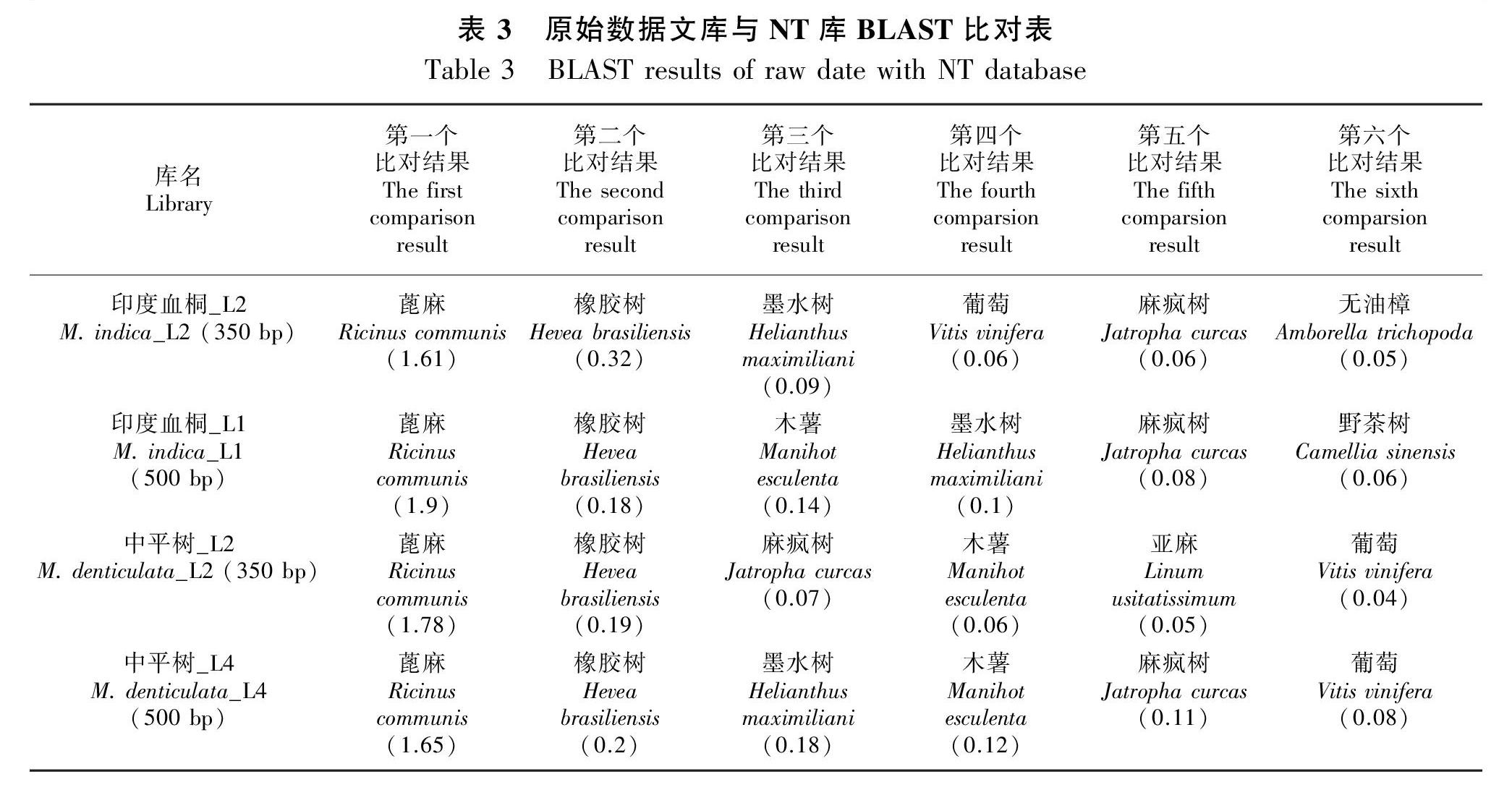
在基因组研究中,样品是否存在污染问题至关重要。若数据未被污染,可保证实验样品基因组序列的完整性,数据真实有效,结果可靠;若数据被污染,则无法获得相关信息。对过滤后的高质量数据随机抽取10 000条reads(read1和read2各5 000条)数据,通过BLAST软件比对NCBI核苷酸数据库(NT库),若比对结果是同源比对,则认为样本不存在外源污染;若比对结果出现亲缘关系较远的物种,说明样品可能存在污染(闫婧,2018)。
1.6 SSR分析
采用微卫星识别工具(microsatellite identi-fication tool, MISA) (http://pgrc.Ipk-gatersleben.de/misa/)在所有序列中搜索SSR位点,搜索参数如下:mono-10、di-6、tri-5、Tetra-5、penta-5、hexa-6。其中,复合序列中两个不同SSR之间允许的最大间隔设置为100 bp (张璟璇等,2019)。
2 结果与分析
2.1 材料的DNA提取
采用CTAB法提取印度血桐与中平树幼嫩叶片的基因组DNA。电泳图显示提取的两种材料基因组DNA质量良好(图1)。其中,印度血桐的DNA浓度为15.42 ng·μL-1,中平树的DNA浓度为10.46 ng·μL-1,可用于后续实验分析。
2.2 测序数据产量统计
利用Illumina平台对两种材料进行高通量双端测序,经过对原始测序数据的严格筛选,得到高质量的产出数据(clean data),以下统计是印度血桐与中平树4个文库的产出数据(表1)。统计结果包括测序数据数量、数据产量、错误率、Q20、Q30、GC含量等。过滤掉低质量的数据后,分别得到了53.56和68.07 Gb的印度血桐与中平树的数据用于后续分析。两种材料碱基质量正常,Q20与Q30都大于90%,测序错误率都为0.04%,印度血桐的GC含量为33.89%,中平树的GC含量为33%,结果表明原始测序质量较好,能保证后续实验分析的正常进行。
2.3 K-mer分析与基因组大小估测
采用基于K-mer的分析方法对印度血桐与中平树的53.56和68.07 Gb的数据进行分析,得到印度血桐与中平树的17-mer分布情况(图2),横坐标表示K-mer出现的总次数,纵坐标表示K-mer出现的频率(唐其等,2015),两种材料主峰之前都出现杂合峰,说明它们都有一定的杂合率。两种材料的17-mer曲线均有严重拖尾,说明它们都有很高的重复序列比例。结合表2可知,印度血桐与中平树的测序深度分别为40X和54X,印度血桐的K-mer总数为39 725 851 195,中平树的K-mer总数为51 594 983 117,根据公式基因组大小(G)的估计算法:G=K-mun/K-depth,其中K-depth表示K-mer的期望测序深度,K-mun表示K-mer的总数(闫婧,2018),由此公式可得印度血桐的大小为993.15 M,修正后的基因组大小为986.84 M;中平树的基因组大小为955.46 M,修正后的基因组大小为946.23 M;印度血桐与中平树的杂合率分别为0.75%和0.65%,印度血桐与中平树的重复率分别为73.02%和73.5%。由测序结果可知,印度血桐与中平树都属于高重复微杂合基因组。
2.4 样品污染评估——核苷酸比对结果
分别从印度血桐与中平树的350 和500 bp的序列文库中随机抽取10 000条过滤后的单端高质量reads (read1和read2各5 000条),与NT库(NCBI核苷酸数据库)进行BLAST比对,将比对结果比率最高的前六位物种展示出来(表3),比对结果显示,在印度血桐与中平树的两个文库中,比对结果比率最高的物种均为蓖麻,在印度血桐的350和500 bp文库中,蓖麻分别占比对上NT库的reads总数的1.61%和1.9%,而在中平树的350和500 bp文库中,蓖麻分别占比对上的NT库的reads总数的1.78%和1.65%,根据分类学结果可知,蓖麻属于大戟科植物,为印度血桐与中平树的近缘物种。此外,比对结果的其他物种均为植物,未发现动物或微生物等异常物种的高比率情况,因此判断样品材料无污染,可用于后续基因组调研图的正常分析。
2.5 印度血桐与中平树基因组SSR分析
利用微卫星识别工具MISA在印度血桐与中平树初步组装的所有序列中进行SSR查找,搜索结果如表4所示,在印度血桐中共搜索到4 499 185个SSR,在所含有SSR的序列中,445 117条序列包含1个以上SSR,以复合形式存在的SSR數量有492 341个;在中平树中共搜索到4 969 098个SSR,在所含有SSR的序列中,458 726条序列包含1个以上SSR,以复合形式存在的SSR序列有507 887条。分别对两种材料的不同类型的SSR核苷酸数量进行统计,在印度血桐的SSR核苷酸数量中,单、双、三、四、五和六核苷酸重复模体分别有2 800 292、1 199 707、432 509、48 890、10 498、7 289个,分别占印度血桐总重复模体的62.24%、26.66%、9.61%、1.09%、0.23%、0.16%;在中平树的SSR核苷酸数量中,单、双、三、四、五和六核苷酸核苷酸重复模体分别有3 037 613、1 321 752、522 801、63 973、11 254、11 705个,分别占中平树总重复模体的61.13%、26.60%、10.52%、1.29%、0.23%、0.24%。然后,进一步对印度血桐与中平树中每种SSR重复模体按照序列组成进行细分,分别展示出两种材料中重复类型的部分数目(表5)。
3 讨论与结论
基因组大小是比较和进化基因组学的基础,基因组的杂合率和重复率是决定基因组组装质量的关键,评估测序数据的杂合率与重复率,有助于找到合适的组装策略(Bi et al., 2019)。对印度血桐与中平树进行17-mer分析后发现,印度血桐与中平树的预估基因组大小分别为987和946 M,由测定结果来看,印度血桐与中平树的基因组大小相近,两种材料的基因组大小均比大戟科植物木薯的基因组770 M(Simon et al., 2012)、蓖麻的基因组350 M(Shusei et al., 2011)、麻疯树的基因组410 M(Chan et al., 2010),比橡胶树的基因组1.1 G(Zou & Yang, 2019)要稍小一些,这种现象可能是因为印度血桐与中平树属于大戟科血桐属植物,而木薯、蓖麻、麻风树与橡胶树分别为大戟科其他属植物,结果显示属内差异较小,而属间差异较大,这可能是由于种系发育关系较远、染色体数目不同或者自交亲和现象的出现所导致(周佳熠等,2017)。基因组大小的测定对了解物种的生长发育、起源进化等问题具有重大意义。印度血桐与中平树基因组大小测定的完成,为研究大戟科血桐属植物基因组大小变化规律提供了一定的参考依据。
判断测序数据的杂合度有利于寻找合适的基因组拼接方法,根据杂合度大小可将基因组进一步分为微杂合基因组(0.5%≤杂合率<0.8%)、高杂合基因组(杂合率≥0.8%) 以及高重复基因组(重复序列比例≥50%)(王雪等,2018)。测序结果显示,印度血桐与中平树的杂合率分别为0.75%和0.65%,重复率分别为73.03%和71.6%,两种植物的基因组都有一定的杂合率以及较高的重复率,印度血桐与中平树都属于雌雄异株的植物,这可能是导致二者含有较高杂合率的原因之一, 因此,使用WGS策略对印度血桐与中平树的基因组分析有一定的风险和难度,建议后续的研究采用二代测序(Illumina)和三代测序(PacBio)技术相结合的策略,对印度血桐与中平树基因组进行测序和组装,此外,利用Hi-C技术达到染色体水平的组装,多种方法互补,以期获得两种材料的高质量全基因组图谱。
SSR分子标记具有易操作、多态性高、成本低、数量丰富等优点。本研究基于印度血桐与中平树基因组调查数据进行SSR分析发现,印度血桐中平均2 251 bp出现1个SSR,中平树中平均2 348 bp出现1个SSR,含丰富的重复类型,结果显示两种材料的SSR分子标记具有显著的碱基偏好性,其单核苷酸重复模体中A/T含量均高于C/G含量;两种材料中双核苷酸重复模体中含量最高均为AT/AT,含量最低为CG/CG,这可能是甲基化的C残基变为T,使得两种核苷酸重复的差异较大(周佳煜等,2017)。有研究认为基因组中低级重复单元较多则表示该物种进化水平较高,而高级重复单元比例高的物种其进化时间短或变异频率低(于福来等,2019)。因此,在基因组调查测序的基础上规模化开发与筛选SSR分子标记,为进一步运用SSR标记在物种遗传图谱的构建和遗传多样性分析以及QTL定位等方面的研究提供参考。
参考文献:
ALBACH DC, LI HQ, ZHAO N, et al., 2007. Molecular systematics and phytochemistry of Rehmannia (Scrophularia
-ceae) [J]. Biochem Syst Ecol, 35(5): 293-300.
BI QX, ZHAO Y, CUI YF, et al., 2019. Genome survey sequencing and genetic background characterization of yellow horn based on next-generation sequencing [J]. Mol Biol Rep, 46(4): 4303-4312
CHAN AP, CRABTREE J, ZHAO Q, et al., 2010. Draft genome sequence of the oilseed species Ricinus communis [J]. Nat Biotechnol, 28(9): 951-956.
HUANG JY, LU WJ, TAN X, et al., 2015. Chemical constituents from Macaranga denticulata root [J]. Chin Med Mat, 38(8): 1671-1673. [黄建猷, 卢文杰, 谭晓, 等, 2015. 中平树根化学成分研究 [J]. 中药材, 38(8): 1671-1673.]
LI GQ, SONG LX, JIN CQ, et al., 2019. Genome survey and SSR analysis of Apocynum venetum [J]. Biosci Rep, 39(6): BSR20190146.doi: https://doi.org/10.1042/BSR20190146.
LI Q, CHEN J, YU XZ, et al., 2019. A mini review of nervonic acid: Source, production, and biological functions [J]. Food Chem, 301: 125286. doi: https://doi-org-443.webvpn.las.ac.cn/10.1016/j.foodchem. 2019. 125286.
SHI JS, WANG ZJ, CHEN JH, 2012. Progress on whole genome sequencing in woody plants [J]. Hereditas, 34(2): 145-156. [施季森, 王占军, 陈金慧, 2012. 木本植物全基因组测序研究进展 [J]. 遗传, 34(2): 145-156.]
SHUSEI S, HIDEKI H, SACHIKO I, et al., 2011. Sequence analysis of the genome of an oil-bearing tree, Jatropha curcas L. [J]. DNA Res, 18(1): 65-76.
SIMON P, PRADEEP R, BRIAN D, et al., 2012. The cassava genome: current progress, future directions [J]. Trop Plant Biol, 5(1): 88-94.
TANG Q, MA XJ, MO CM, et al., 2015. Genome survey analysis in Siraitia grosvenorii [J]. Guihaia, 35(6): 786-791. [唐其, 馬小军, 莫长明, 等, 2015. 罗汉果全基因组Survey分析 [J]. 广西植物, 35(6): 786-791.]
TIAN DY, WANG SA, WANG LH, et al., 2015. The biosynthesis and metabolic engineering of very long-chain monounsaturated fatty acid [J]. Biotechnol Bull, 31(12): 42-49. [田德雨, 王士安, 王立昊, 等, 2015. 超长链单不饱和脂肪酸的生物合成和代谢工程 [J]. 生物技术通报, 31(12): 42-49.]
WANG XY, FAN JS, WANG SQ, 2006. Development situation and outlook of nervonic acid plants in China [J]. Chin Oils Fats, 3: 69-71. [王性炎, 樊金栓, 王姝清, 2006. 中国含神经酸植物开发利用研究 [J]. 中国油脂, 3: 69-71.]
WANG X, ZHOU JY, SUN HG, et al., 2018. Genomic survey sequencing and estimation of genome size of Ammopiptanthus mongolicus [J]. J Plant Genet Resourc, 19(1): 143-149. [王雪, 周佳熠, 孙会改, 等, 2018. 新疆沙冬青基因组调查测序与基因组大小预测 [J]. 植物遗传资源学报, 19(1): 143-149.]
WU YF, XIAO FM, XU HN, et al., 2014. Genome survey in Cinnamomum camphora L. presl [J]. J Plant Genet Resourc, 15(1): 149-152. [伍艳芳, 肖复明, 徐海宁, 等, 2014. 樟树全基因组调查 [J]. 植物遗传资源学报, 15 (1): 149-152.]
YAN J, 2018. Genome survey of Rehmannia chingii, a rare and medicinal herb in China [D]. Xi’an: Northwest University: 17-27. [闫婧, 2018. 珍稀植物天目地黄(Rehmannia chingii)的基因组调查研究[D]. 西安: 西北大学: 17-27.]
YANG DS, PENG WB, YANG YP, et al., 2015. Cytotoxic prenylated flavonoids from Macaranga indica [J]. Fitoterapia, 103: 187-191.
YU FL, HUANG M, ZHANG YB, et al., 2019, Genome survey and characteristic analysis of SSR in Callicarpa nudiflora [J]. Chin J Chin Mat Med, 44(18): 3974-3978. [于福来, 黄梅, 张影波, 等, 2019. 裸花紫珠基因组调研及SSR特征分析 [J]. 中国中药杂志, 44(18): 3974-3978.]
ZHANG JX, TU MW, XUE S, et al., 2019. Genome survey and analysis of ssr molecular markers on traditional Chinese medicine Nauclea officinalis [J]. Mol Plant Breed, 17(23): 7829-7833. [张璟璇, 涂梦薇, 薛燊, 等, 2019. 南药胆木的基因组调查及SSR分子标记分析 [J]. 分子植物育种, 17(23): 7829-7833. ]
ZHOU JY, WANG X, GAO F, et al., 2017. Genome survey and SSR analysis of Ammopiptanthus mongolicus [J]. Genom Appl Biol, 36(10): 4334-4338. [周佳熠, 王雪, 高飞, 等, 2017. 蒙古沙冬青基因组调查及SSR分析 [J]. 基因组学与应用生物学, 36(10): 4334-4338.
ZHOU Y, ZI H, TONG J, et al., 2019. A genome survey of Rhododendron simsii and Rhododendron indicum [J]. Mol Plant Breed, 17(15): 4928-4935. [周媛, 資宏, 童俊, 等, 2019. 杜鹃花属映山红与皋月杜鹃的基因组调查 [J]. 分子植物育种, 17(15): 4928-4935.]
ZOU Z, YANG JH, 2019. Genomic analysis of Dof transcription factors in Hevea brasiliensis, a rubber-producing tree [J]. Ind Crops Products, 134: 271-283.
(责任编辑 李 莉)

