DNA条形码技术在大亚湾游泳动物卵和幼体分类鉴定中的应用*
张涛锂 李庆勇 张志敏 叶 冠 钱宏标 林海强
(1.广东大亚湾水产资源省级自然保护区管理处,广东 惠州 516081;2.惠州市渔业研究推广中心,广东 惠州516057;3.惠州市智鸿海洋科技有限公司,广东 惠州 516083)
卵和幼体是游泳动物种群补充的重要来源,其种类分布和数量变化是评估海域鱼类产卵场、亲鱼资源量、渔业资源补充量最直接有效的数据资料[1-2], 因此,游泳动物卵和幼体具有非常重要的生态地位和研究价值[3]。尽管游泳动物卵和幼体具有重要的研究价值和生态地位,但其准确鉴定依然是游泳动物、浮游生物多样性与生态学研究长期以来的瓶颈[4]。研究表明世界已鉴别的成鱼种类超过28 700 余种,而能够鉴别的仔稚鱼仅仅占成鱼种类的 1/10[5]。过去对游泳动物卵和幼体种类鉴定主要应用传统形态学方法,但是由于海洋游泳动物卵和幼体发育变化复杂,不同种间形态差异不明显,仔稚鱼生长期形态变化快,仅仅通过形态学鉴定浮游生物是不适宜的[6-8]。而且传统形态学方法过分依赖鉴定者的个人经验和观察手段, 不同鉴定者的鉴定结论时常不一致[9-11],作为种类甄别的形态特征较少,加上专业分类资料及人才的缺乏,使仅凭外部形态特征对游泳动物卵和幼体进行精确分类变得相当困难。
因此,一种准确、快速、高效、稳定的鉴定方法就显得十分必要。DNA 条形码(DNAbarcoding)技术是一种利用标准基因区域序列多态性进行物种鉴定的分子方法,条形码序列分析不依赖于形态特征,对形态特征近似,形态学分类比较困难的疑难种类的分类以及形态学分类上存有疑问种类的验证提供了新的方法和手段[12-13]。鱼类线粒体细胞色素C 氧化酶亚基I(mtCOI)基因具有序列变异水平适中,变异区域两端序列高度保守和序列长度适宜的特点,是被广泛接受的鱼类DNA 条形码标准基因[14-16]。目前鱼类DNA 条形码数据库(Barcode of Life Data System, BOLD)中已有超过17.5 万件凭证标本,代表了1.5 万种以上的鱼类种类[17],并且全球共享的基因数据库如DDBJ、ENBL、GenBank 等所收藏鱼类的各种基因及序列数量迅速增加,为鱼卵、仔稚鱼提供了大量可比对的成鱼同源性DNA 基因序列,研究者可以将DNA 条形码技术直接运用到鱼卵、仔稚鱼的鉴别中,保证了鉴定结果的准确性。
大亚湾是位于大鹏半岛东侧的典型半封闭海湾,湾内水质优良,生境类型多样,是马氏珠母贝Pinctada martensi的天然采苗场,也是鲷科等经济鱼类的重要产卵场和育幼场。然而,近几十年来,由于周边经济发展迅速,临海工业发展和涉海工程等人类活动对大亚湾带来一定的影响,同时,由于全球气候变化、环境污染和过度捕捞等问题的叠加影响,经济动物出现小型化和低值化趋势明显。因此,在大亚湾海域开展渔业资源的调查研究具有十分重要的意义。游泳动物卵和幼体不仅生命力脆弱,容易受环境变动的影响,而且是研究游泳动物补充量和资源变动的重要环节,开展大亚湾游泳动物卵和幼体的种类和数量研究,不仅可以了解环境变化对渔业资源的影响,也能反映出渔业资源变动的情况,同时对海洋生物资源的保护和管理也有一定的参考价值。本研究拟以大亚湾游泳动物卵和幼体为研究对象,采用DNA 条形码技术对采集的游泳动物进行鉴定分析,以期了解大亚湾游泳动物的产卵季节和种类分布特征,为相关种类的种群补充机制研究提供基础材料。
1 材料与方法
1.1 调查航次和站位分布
根据前期监测调查资料,分别于2020 年春季(5 月)和秋季(10 月)对大亚湾3 个游泳动物卵和幼体分布较为密集区域开展了卵和幼体定量取样调查,调查站位如图1 及表1。

表1 大亚湾游泳动物卵和幼体采样站位位置坐标Tab.1 Coordinates of sampling sites for nekton eggs and larvae in Daya Bay

图1 大亚湾游泳动物卵和幼体采样站位位置示意图Fig.1 Schematic diagram of the location of the sampling station for nekton eggs and larvae in Daya Bay
1.2 采样方法
1.2.1 网具 浅水Ⅰ型浮游生物网:网长145 cm,网口直径50 cm,网口面积0.2 m2,筛绢规格CQ14(0.505 mm)。
1.2.2 调查方法 水平调查采样(样品定性采集):将浅水Ⅰ型浮游生物网固定在船尾,拖速保持1~2 Kn,拖10 min,收集样品。垂直调查采样(样品定量采集):将浅水Ⅰ型浮游生物网放入调查站位海水中,由海底至海面垂直或倾斜拖网。落网速度为0.5 m/s,起网速度为0.5~0.8 m/s。
1.2.3 样品处理方法 样本的固定和保存:直接用70%浓度酒精固定,回实验室后分别挑出无脊椎动物、鱼卵和仔稚鱼,用100%浓度酒精保存,置于4 ℃冰箱保存。
1.2.4 数据记录 水平调查记录:网口宽,拖速,拖网时长;垂直调查记录:网口宽,入水绳长,水深。
1.3 样品处理
1.3.1 DNA 提取和扩增 无脊椎动物、鱼卵及仔稚鱼样品用双蒸水冲洗(鱼卵整个提取,仔稚鱼取尾部,以上经过形态学初步分析,按比例取出样品提取)将样品置于1.5 mL 离心管中,采用TIANGEN 公司的试剂盒(TIANamp Marine Animals DNA Kit)提取基因组DNA,提取的DNA 置于-20 ℃保存备用。Co I 引物为F(5’-TCAACCAACCACAAAGACATTGGCAC-3’),R(5’-TAGACTTCTGGGTGGCCAAAGAATCA-3’) 反应体系25 μL: ddH2O 8.5 μL,2×Taq Mix12.5 μL,模板 DNA2.0 μL,引物各1μL。扩增的程序为:94 ℃预变性2 min,94 ℃变性40 s,52 ℃退火1 min,72 ℃延伸90 s,共35 个循环;最后72 ℃延伸10 min。PCR 扩增产物通过琼脂糖凝胶(1.0%)电泳80 V,30 min;电泳完成后利用凝胶电泳成像仪观看图像。PCR 阳性产物送至上海生工生物技术有限公司进行序列测定。
1.3.2 序列分析 采用Sequencher 5.4(Gene Codes)和Clustal X2 软件将测序获得的游泳动物卵和幼体COI 基因序列进行编辑、人工校正和序列比对。通过在NCBI 中BLAST,将所得DNA 序列在Genbank 中进行相似性检索,得出物种的分类地位。同时将测序获得的COI 基因序列与BOLD系统数据库(http://www.barcodinglife.org)比对分析进行物种鉴定,在比对分析中,BOLD 系统为每个样品分配一个条形码索引号(Barcode Index Number,BIN),当样品COI 基因序列与BIN 中参考序列的相似性≥98.4%时,鉴定为种,当相似性≥95%到<98.4%之间,鉴定到属,当相似性<95%时,为未知种。BOLD 与GenBank 相互对照,进行最终的鉴定。
1.3.3 游泳动物卵和幼体统计 垂直站点进行无脊椎动物、仔稚鱼及鱼卵量计数后,进行DNA 提取、序列分析、鉴定等步骤;水平站点进行DNA提取、序列分析、鉴定等对物种多样性进行补充。
1.4 分析方法

1.4.4 Margalef 丰富度指数[18]D=(S-1)/lnN。
1.4.5 群落结构相似性分析 采用Primer5.2 对大亚湾资源调查各站点春秋两季进行聚类分析、NMDS 等进行群落结构相似性分析。检验NMDS的拟合优度采用协强系数(stress),通常认为,stress < 0.2 时,其图形具有一定的解析意义;stress < 0.1 时,具有较好的拟合度;stress < 0.05时,图形具有代表性。
2 结果与分析
2.1 种类结构
2020 年春季资源调查中,共采集游泳动物卵和幼体1 660 尾(粒),经COI 鉴定分析,共鉴定出84 种,隶属于4 门11 目48 科64 属。其中包含鱼类60 种,占物种总数71.4%,隶属8 目33 科;虾类9 种,占物种总数10.7%,隶属1 目6 科;蟹类12 种,占物种总数14.3%,隶属1 目7 科;软体动物1 种,占物种总数1.2%,隶属1 目1 科。其中鲈形目出现15 科31 种,占物种总数36.9%;十足目出现13 科21 种,占物种总数25.0%;鲱形目出现4 科12 种,占物种总数14.3%。
2020 年秋季资源调查中,共采集游泳动物卵和幼体1 580 尾(粒),经COI 鉴定分析,共鉴定出62 种,隶属于2 门11 目40 科53 属。其中包含鱼类47 种,占物种总数75.8%,隶属9目29 科;虾类10 种,占物种总数16.1%,隶属2 目7 科;蟹类4 种,占物种总数6.5%,隶属1 目4 科;虾蛄1 种,占物种总数1.61%,隶属1 目1 科。其中鲈形目出现15 科26 种,占物种总数41.9%;十足目出现10 科14 种,占物种总数22.6%;鲱形目出现2 科4 种,占物种总数6.5%;鳗鲡目出现3 科4 种,占物种总数6.5%;鲉形目出现3 科4 种,占物种总数6.5%。
2020 年春、秋两季共鉴定出4 门14 目64 科90 属120 种。
2.2 种类分布
2020 年春、秋两季共鉴定出120 种,其中A站点30 种,其中春季17 种,秋季14 种;B 站点53 种,其中春季24 种,秋季29 种;C 站点62种,其中春季49 种,秋季26 种。
2.3 丰度的时空分析
2.3.1 丰度的季节性差异 春季的平均丰度为1 359.26 尾(粒)/m3,秋季的平均丰度为821.85 尾(粒)/m3,且两个季度各站点间丰度差异不显著(P= 0.078 > 0.05)。平均丰度上春季 > 秋季,且各站点间没有显著性差异。
2.3.2 丰度的平面空间分布 春季垂直采样得到单位体积丰度区间为0 ~ 5 930 尾(粒)/m3,其中A2、A3 和D2 站点为本季度高值,分别为5 930、4 320 和3 620 尾( 粒)/m3。F2 站点、E1 站点、H3 站点、I2 站点和I3 站点为本季度低值(图2),分别为0、70、120、150、170 尾( 粒)/m3; 秋季垂直采样得到单位体积丰度区间为60 ~ 2 850尾(粒)/m3,其中C2 站点,C1 站点和D3 站点为本季度高值,分别为2 850、2 340、1 970 尾( 粒)/ m3,E1 站点、I3 站点、F3 站点、G1 站点和H1 站点为本季度低值(图3),分别为60、70、90、130、160 尾(粒)/m3。
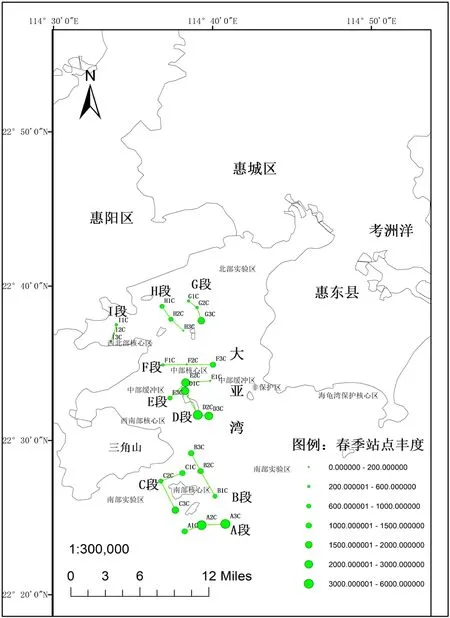
图2 2020 年春季各站位游泳动物卵和幼体调查丰度Fig.2 Survey abundance of nekton eggs and larvae at each station in the spring of 2020

图3 2020 年秋季各站位游泳动物卵和幼体调查丰度Fig.3 Survey abundance of nekton eggs and larvae at each station in the fall of 2020
2.4 优势种分析
春季优势种有4 种,最主要优势种为岛屿小公鱼Stolephorus insularis,优势度为0.061,丰度为1 540 尾(粒)/m3;秋季优势种有4 种,最主要优势种为中华管鞭虾Solenocera crassicornis,优势度为0.070,丰度为1 660 尾(粒)/m3(表2)。

表2 2020 年各站位游泳动物卵和幼体调查主要优势种Tab.2 The main dominant species in the survey of eggs and larvae of nektons at each station in 2020
2.5 群落稳定性分析
2.5.1 群落多样性分析 春季时,多样性指数变化区间为0.725~3.976,平均值为2.305,A-C 段海区多样性指数为3.227,D-F 段海区多样性指数为3.827,G-I 段海区多样性指数为4.621;秋季时,多样性指数变化区间为0.000~2.781,平均值为1.637,A-C 段海区多样性指数为3.177,D-F 段海区多样性指数为3.943,G-I 段海区多样性指数为3.061。各站点间春秋两季物种多样性差异显著(P=0.043 < 0.05),三大海区间春秋两季物种多样性差异不显著(P=0.367 >0.05)。
2.5.2 群落丰富度 春季时,丰富度指数变化区间为0.097~1.951,平均值为0.837,A-C 段海区多样性指数为1.332,D-F 段海区多样性指数为2.113,G-I 段海区多样性指数为4.161;秋季时,丰富度指数变化区间为0.000~1.093,平均值为0.405,A-C 段海区多样性指数为0.989,D-F 段海区多样性指数为2.340,G-I 段海区多样性指数为2.191。各站点间春秋两季物种多样性差异显著(P=0.014 < 0.05),三大海区间春秋两季物种多样性差异不显著(P=0.503 >0.05)。
2.5.3 群落均匀度 春季时,丰富度指数变化区间为0.064~1.000,平均值为0.809,A-C 段海区多样性指数为0.789,D-F 段海区多样性指数为0.788,G-I 段海区多样性指数为0.823;秋季时,丰富度指数变化区间为0.000 ~ 0.993,平均值为0.815,A-C 段海区多样性指数为0.834,D-F 段海区多样性指数为0.812,G-I 段海区多样性指数为0.651。各站点间春秋两季物种多样性差异不显著(P=0.927>0.05),三大海区间春秋两季物种多样性差异不显著(P=0.591>0.05)。
2.5.4 群落结构相似性分析 春季时可分为4 个聚类,D3、F3、B1 各自一个聚类,其他站点为一个聚类,(其中A1 与G3 与其他站点稍有不同而偏离),说明D3、F3、B1 所含物种群落与其他站点差别较大且各自均不相同,协强系数stress 为0.01 < 0.05,说明结果具有代表性(图4、图5);秋季时可分为6 个聚类,A1、C3、G1 和F1、D2、E2 各自一个聚类,其他站点为一个聚类,说明A1、C3、G1 和F1、D2、E2 所含物种群落与其他站点差别较大且各自均不相同,协强系数stress 为0.01 < 0.05,说明结果具有代表性(图6 ~7)。

图4 2020 年各站点游泳动物卵和幼体调查春季拟合度Fig.4 Spring fitness of nekton eggs and larvae survey at each site in 2020

图5 2020 年各站点游泳动物卵和幼体调查春季聚类树Fig.5 The spring cluster tree for the survey of eggs and larvae of nektons at each site in 2020

图6 2020 年各站点游泳动物卵和幼体调查秋季拟合度Fig.6 The fit of nekton eggs and larvae at each site in the fall in 2020
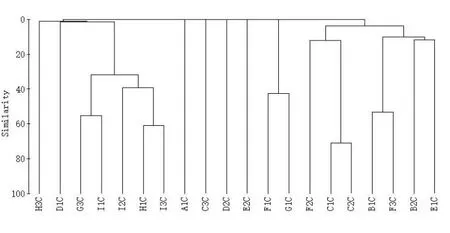
图7 2020 年各站点游泳动物卵和幼体调查秋季聚类树Fig.7 The survey of eggs and larvae of nektons at each site in 2020 cluster tree in autumn
3 结果与讨论
海洋游泳生物的卵和幼体的鉴别需要丰富的专业知识和经验,尤其是形态相似的发育初期,往往难以区分。游泳动物卵和幼体早期发育阶段时间短,形态变化复杂,物种间形态差异不明显,有记载可查的资料不超过300 种,因此形态学鉴定的局限性限制了其在诸多未知鱼类鉴定中的应用[19]。近年来发展起来的DNA 条形码技术为物种鉴定提供了一种快速、有效的方法,且易于掌握操作[20-21],并且已有鱼类鉴定的专有数据库[21]。DNA 条形码鉴定技术基因靶标以线粒体COI 基因为代表,还有基于12S rRNA、16S rRNA 和CytB基因鉴定的方法[23-25]。本研究以COI 基因为标靶,共鉴定出4 门14 目64 科90 属120 种的海洋生物,且这些种类在大亚湾历史资料中有所记载,证明了使用DNA 条形码技术做海洋渔业资源监测或其它海洋生物资源监测的可行性,也为科研基础较为薄弱的机构持续性的开展科研监测工作提供了可能性。
从2020 年调查的情况看,公洲海域(A)、小三门西面海域(B)及大三门北面海域(C)资源种类较多,分别达到了30、53 和62 种,且除B站点外,都表现出春季种类数高于秋季,且丰度上春季高于秋季,但各站点间无显著差异,结果与我们2017—2018 年开展的“蓝色家底”调查数据一致(数据未发表),同时春季种类高于秋季也从侧面验证了南海伏季休渔的必要性。
在优势种方面,此次调查按照国内外学者[26-28]公认的方法进行统计计算,结果表明春秋季优势种各有4 种,且这些物种都属于小型、低值的种类。一方面可能说明目前大亚湾海域的大型经济鱼类的保护有待进一步加强;另一方面可能是从食物丰富度的角度考虑,这些低值生物大量繁殖后,大型的、以这些低值生物为食的高值生物才会出现,以保证充足的食物[29]。在3 个海区中物种多样性差异不显著,仅表现在季节性变化,且春季多样性指数高于秋季。从我们的监测结果看,北部海域物种多样性高于南部,可能说明北部饵料较为丰富,水浅浪缓,游泳生物在此产卵繁育,这些结果与王九江[30]、林昭进[31]等在大亚湾开展调查研究的结论相似,且在游泳动物种类上也大同小异,也侧面说明了我们此次调查结果的科学性、准确性。从春季群落结构上来看,我们调查的结果说明在同一时段、不同海区群落结构基本一致,说明在大亚湾游泳动物的繁殖在同一时段,且相对来讲,亲本分布较为均匀,这也保证了种群的稳定性;秋季时,群落结构相对分散,可能是不同游泳动物繁殖季节不一样,这可能与这些生物的繁殖策略有关,主要是考虑到食物的丰富度。以上关于食物决定繁殖的推论尚无数据支撑,但理论上存在很大可能,是下阶段我们精细化研究的一个方向。
对比应用传统形态学方法鉴定游泳动物卵和幼体种类,本次采用DNA 条形码技术,避免了传统形态学专业分类人才及资料缺乏的问题,也避免了鉴定者自身具备的专业知识和经验参差不齐、主观性较强的问题,提高了鉴定效率,为掌握大亚湾水产资源状况提供了较为简便可靠的方法。虽然DNA 条形码技术在开展海洋资源调查、科研监测工作发挥出来诸多的优势,但其也存在DNA提取过程、DNA 条形码参考数据库的完整性和准确性等问题,还需要后续研究以进一步完善相关方法和技术。但总体来说,对于初涉游泳动物卵和幼体鉴定的工作人员来说,无疑是一种方便、快速的方法。

