分子动力学模拟揭示C677T诱导MTHFR及其配体FAD解离的分子机制
王若男 曹锟

摘要:亚甲基四氢叶酸还原酶(MTHFR)C677T的基因多态性会影响叶酸代谢。为了研究C677T(A177V)对于该酶构象的影响,我们采用分子动力学模拟的方法,针对MTHFR-野生型(MTHFR-WT)和A177V突变体分别进行了100 ns的模拟计算,分析了该酶整体构象差异及其FAD活性中心的变化。研究发现,与MTHFR-WT相比,A177V模拟体系无法趋于稳定,突变体a-C的均方根涨落值升高,回旋半径值增大,溶剂可及表面积增大,这些结果都表明,A177V突变会导致该酶的整体结构松散且不稳定升高;进一步的二级结构含量及FAD活性中心的分析表明,A177V突变会引起部分a螺旋含量降低、b折叠含量升高,且造成FAD结合位点残基的空间距离增大,这些结果表明A177V突变会破坏FAD结合位点微环境。本研究在原子水平揭示了A177V突变会诱导MTHFR激活剂FAD解离的关键信息。
关键词:分子动力学模拟;亚甲基四氢叶酸还原酶;黄素腺嘌呤二核苷酸;突变体;稳定性
亚甲基四氢叶酸还原酶MTHFR是机体叶酸代谢过程中的关键限速酶,它能催化5,10-亚甲基四氢叶酸向5-甲基四氢叶酸转换,随后会进入甲基传递通路,为DNA、蛋白质甲基化提供甲基,为同型半胱氨酸提供甲基形成甲硫氨酸。叶酸是人体体内重要的水溶性维生素B族营养素,是对具有蝶酰谷氨酸分子结构化合物的总称[1-2]。叶酸在细胞分裂、蛋白质合成等途径中具有重要作用,叶酸摄入不足或者代谢障碍,可能造成人体同型半胱氨酸积累、血红蛋白生成减少进而导致贫血,甚至可能导致神经管畸形、智力低下、心脑血管等疾病[3]。
MTHFR C677T具有基因多态性,这直接决定了酶活性以及叶酸利用率。根据文献报道,与MTHFR-WT相比,C677T基因突变会造成该酶的熔解温度下降6℃且酶活性仅有26%,从而引起叶酸、甲基代谢紊乱,所以该酶参与了人类重要疾病的发生发展[4]。本研究项目将通过分子动力学模拟的手段揭示C677T对于该酶构象的影响,并阐明其诱导FAD配体解离的分子机制。
1材料与方法
1.1分子模拟的条件
本研究中大肠杆菌MTHFR的结构(PDB:1b5t)来自于RCSB蛋白质数据库(https://www.rcsb.org/)。在使用Gromacs2020.4软件进行分子模拟计算之前,需要先除去MTHFR结构中的结晶水和杂质离子,此后需要通过PymoL软件将Ala-177突变成Val-177,即A177V。针对MTHFR和A177V设置两个独立的体系,采用SPC水模型、Gromos43a1力场进行总时长为100 ns模拟[5-6]。将MTHFR和A177V的晶体构象放置在立方周期盒中并作为起始构象,将蛋白与盒子边界的最小距离设定为1.0 nm,模拟系统的周期性边界条件适用于X/Y/Z三个方向。在溶胶中加入0.15 mol/L NaCl盐溶质以便于中和蛋白体系的电荷。接下来,采用蛙跳算法计算每个原子的运动,用粒子网格法计算PME静电相互作用。我们利用最速下降能量法进行400步能量最小化,并对MTHFR和A177V的模拟体系进行了50 ps的位置约束仿真。采用随机初始速度作为正式动力学模拟的初速度。利用PyMOL、VMD以及Origin8.5软件生成数据结果。
1.2模拟结果的分析
我们利用VMD-1.9.1软件观察两个不同模拟体系的轨迹中蛋白构象变化[7],采用gmx rms、gmx rmsf和gmx gyrate工具分别计算了蛋白的RMSD变化、每个氨基酸残基的a-C的均方根涨落RMSF以及回旋半径[8]。并采用gmx distance分别测量了MTHFR和A177V中的典型FAD结合位点残基的平均距离。并用PyMOL绘制结构图,用Origin8.5软件绘制曲线图。
2 结果与分析
2.1 MTHFR-WT与A177V的整体构象变化
大肠杆菌MTHFR酶的三维构象主要呈现桶状结构,由296个氨基酸残基组成,其分子量约为33.1 kDa。其中,Ala-177与人类MTHFR中的Ala222相对应,位于桶底附近,远离FAD。大多数a-螺旋位于桶的外侧周围,主要作用是向内形成一个疏水囊;而桶的内部是辅酶因子FAD的主要活性区域,由7个b-折叠形成疏水区域,这对于稳定蛋白的整体构象至关重要。
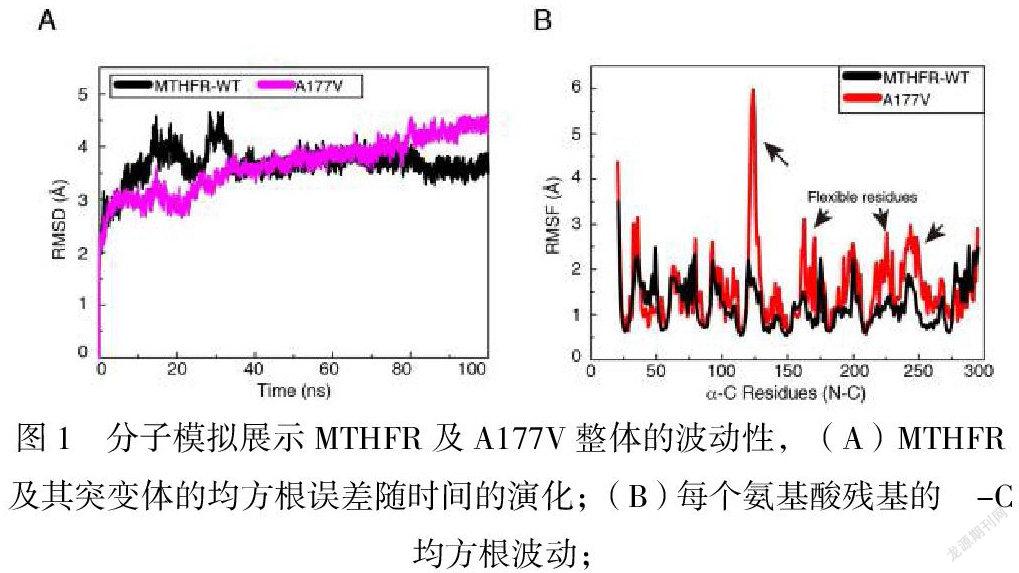
均方根误差(root mean square deviation,RMSD)是衡量体系是否达到平衡状态的重要依据。MTHFR-WT在70 ns即达到平衡狀态,对应的平均RMSD值最低约为3.6 Å(图1A);而A177V突变体蛋白的RMSD值在100 ns的总模拟时间内无法达到平衡状态,其RMSD最大值高达4.3 Å以上,说明Ala-177残基被突变成Val-177会导致蛋白结构不稳定。
通过对比MTHFR-WT与A177V中每个a-C原子相对于其平均位置的涨落(RMSF)值的变化,能够直观反应出Ala-177突变引起的残基柔性差异。通常情况下,RMSF值越低则该残基的灵活性越小,我们将MTHFR-WT的RMSF值定义为正常值;与正常值相比,A177V突变体的多数残基的RMSF值增大,这表明Ala-177的突变已经对蛋白的碳骨架的波动性造成较大影响(图1B)。因此,这些结果都表明A177V的突变会导致多数残基的波动性增大,甚至造成蛋白构象不稳定性,这不利于蛋白发挥酶活性。
2.2 蛋白的回旋半径及其构象的溶剂可及表面积分析
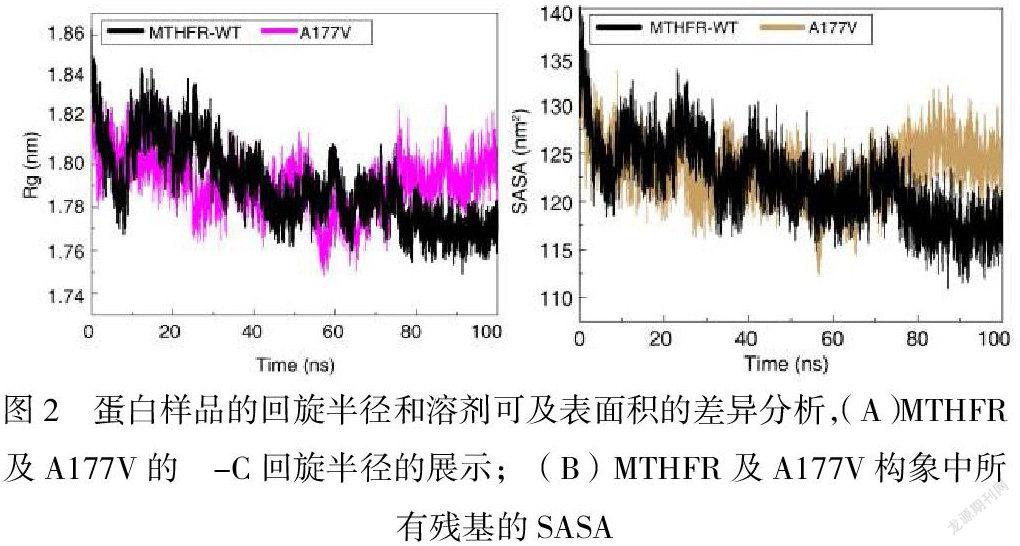
接下来,我们统计了MTHFR-WT及其突变体的回旋半径(Rg)随模拟时长的变化情况,蛋白的Rg值越大表示蛋白构象越灵活,该值越小说明构象越趋近于刚性。在MTHFR-WT和A177V的体系中,蛋白样品a-C的Rg值分别为1.77 nm和1.81 nm(图2A)。接下来,我们采用gmx sasa分别计算了蛋白整体的溶剂可及表面积SASA随着时间变化情况,平衡状态下的MTHFR-WT所对应的SASA值降低至117.5 nm2,与之相比A177V的SASA值则升高至125.1 nm2(图2B)。这些结果说明,回旋半径与溶剂可及表面积的数据趋势与RMSD及RMSF结果保持一致,即Val-177突变将导致该酶的空间构象由致密变得更加松散,这不利于蛋白构象保持稳定。该结论从原子水平解释了A177V突变体的熔解温度比野生型下降了6℃这一科学问题。
2.3對比MTHFR-WT与A177V的二级结构变化以及FAD的结合能力
为了探究Ala-177被突变成Val-177所引起的二级结构变化差异,我们采用do_dssp插件解析了蛋白的二级结构含量。A177V模拟体系的二级结构改变情况与MTHFR-WT正常体系形成鲜明对比,MTHFR-WT平均有122个残基参与a螺旋的形成,19个形成b-转角(图3A);然而,在A177V突变体中有125个残基形成a螺旋,17个形成b-转角(图3B)。这些数据表明Ala-177被突变成Val-177可能会导致一部分氨基酸残基由b-转角转化为a螺旋,从而改变了二级结构含量占比。由于MTHFR酶的b-转角具有稳定蛋白骨架的作用,因此b-转角含量的降低可能会破坏其结构稳定性,甚至降低该酶的催化活性,这个结果与文献报道的A177V酶活性仅有26%相一致。
由于FAD是MTHFR酶的底物辅酶因子,接下来我们分析了MTHFR-WT和A177V分别与底物FAD分子的结合位点,经过分析,我们确定了大肠杆菌MTHFR-WT的FAD结合位点残基为Trp-60、His-88、Arg-118、Gly-119、Asp-120、Ala-132、Trp-152、His-156、Asp-165、Asn-168、Arg-171和Lys-172(图3C),由此可见FAD的结合方式极为严苛。然而,上述诸多证据已经揭示A177V突变体的氨基酸残基波动性较大、整体构象趋于松散状态,因此这些结合位点残基之间的空间距离也随之增大(图3D),具体表现在Trp-60和His-88与其他残基之间的距离增大为12.2 Å和15.9 Å,而其余结合位点的空间位置则被压缩,不利于FAD底物分子的结合,这可能是由于氨基酸残基的灵活性较大、局部二级结构被转换导致的结果。总之,这些数据从原子水平证实A177V突变体不利于结合辅酶因子FAD,故无法充分发挥其催化活性。
3 讨论
据文献报道,Brian D.Guenther等人已证实MTHFR-WT的熔解温度大约为53.1 °C,而A177V的熔解温度仅有46.0 °C,并且A177V的酶活性仅为野生型的26%[9-10],然而这一科学问题并未从原子水平角度被更好的解释。因此,本研究通过分子动力学模拟的手段,探究A177V突变体所造成的蛋白构象变化,具有一定的生物学意义。在这项研究中,我们采用分子动力学模拟的手段分析了MTHFR-WT和A177V突变体蛋白的原子空间构象差异,从RMSD、RMSF、Rg、SASA、二级结构改变及FAD结合能力等方面证实,A177V突变体会引起氨基酸残基的波动性增大、碳骨架的不稳定性升高、蛋白空间构象由致密变得松散等结论,并导致底物分子FAD的活性区域被破坏,最终造成该酶的熔解温度降低以及酶活性降低。在临床病例中,MTHFR的大多数突变属于C677T突变,即形成A177V突变体,这将诱导MTHFR酶无法正常结合FAD配体分子。总之,本研究在原子水平揭示了C677T对于该酶构象的影响,并阐明其诱导FAD配体解离的分子机制。
参考文献
[1]AYGUN B,AK DY.vitamin b12 and folic acid levels after iron therapy in iron-deficiency anemia[J].Cukurova Anestezi ve Cerrahi Bilimler Dergisi,2020,3(3):261-267.
[2]A,EGJ VERMEULEN,et al.Effect of homocysteine-lowering treatment with folic acid plus vitamin B6 on progression of subclinical atherosclerosis:a randomised,placebo-controlled trial.Lancet,2019,3(92):517-522.
[3]HILDEBRAND LA,DUMAS B,MILROD C,et al.Folate Deficiency in an Urban Safety Net Population[J].Blood,2020,136(Supplement 1):46-46.
[4]MD MARTINIS,SIRUFO MM,NOCELLI C,et al.Hyperhomocysteinemia is Associated with Inflammation,Bone Resorption,Vitamin B12 and Folate Deficiency and MTHFR C677T Polymorphism in Postmenopausal Women with Decreased Bone Mineral Density[J].International Journal of Environmental Research and Public Health,2020,17(12):4260-4266.
[5]CAO K,ZHANG J,MIAO XY,et al.Evolution and molecular mechanism of PitAs in iron transport of Streptococcus species[J].Journal of Inorganic Biochemistry,2018:113-123.
[6]VITALINI F,F NOÉ,KELLER B G.Molecular dynamics simulations data of the twenty encoded amino acids in different force fields[J].Data in Brief,2016,7(C):582-590.
[7]VERMAAS JV,HARDY DJ,STONE JE,et al.TopoGromacs:Automated Topology Conversion from CHARMM to Gromacs within VMD[J].Journal of Chemical Information and Modeling,2016,56(6):1112-1116.
[8]CHEN J,III C.Can molecular dynamics simulations provide high-resolution refinement of protein structure?[J].Proteins:Structure,Function,and Bioinformatics,2007,67(4):922-930.
[9]GUENTHER B D,SHEPPARD C A,TRAN P,et al.The structure and properties of methylenetetrahydrofolate reductase from Escherichia coli suggest how folate ameliorates human hyperhomocy steinemia.[J].Nat Struct Biol,1999,6(4):359-365.
[10]FROESE DS,KOPEC J,REMBEZA E,et al.Structural basis for the regulation of human 5,10-methylenetetrahydrofolate reductase by phosphorylation and S-adenosylmethionine inhibition[J].Nature Communications,2018,9(1):2261.
基金項目:国家自然科学基金青年项目(82002104);广东省基础与应用基础研究基金项目区域联合基金-青年基金项目(2019A1515 110659);广东省医学科学技术研究基金项目(A2020381);2019年度广东医科大学博士学位人员科研启动基金(B2019018);2020年度广东医科大学科研基金自然科学类面上培育项目(GDMUM2020016)。
*通讯作者:曹锟(1989-),男,助理研究员,博士研究生,研究方向:医学生物化学。
作者简介:王若男(1991-),女,助理实验师,硕士研究生,研究方向:食品科学和生物化学。

