汾酒发酵室新旧土壤酵母群落结构对比研究
耿添霈 蔚慧欣 甄 攀 王军燕
(山西杏花村汾酒厂股份有限公司,山西汾阳 032200)
汾酒酿造历史悠久,以绵甜清香的风味著称,是清香型白酒的典型代表。汾酒的生产采用豌豆和大麦制曲,高粱酿酒,清蒸二次清工艺,经贮存勾兑而成。但是,传统汾酒开放式的生产方式导致其酒的品质和产量与整个酿造过程中微生物群落结构变化有着十分紧密的联系。汾酒发酵过程中发酵酒醅与发酵室地缸之间的土壤(俗称“缸花”)有着直切的接触,汾酒在每年停产季节会将发酵室地缸之间的旧土换为新土,因此,对比研究汾酒发酵室新土与旧土酵母群落结构,不仅有助于提高人们对汾酒酿造机理的认识,而且还为进一步研究汾酒微生物发酵规律,稳定质量,提高产量提供理论支撑。
本试验利用可培养微生物分离方法对汾酒发酵室中的新旧土壤中酵母菌进行分离培养,并进行26S rDNA 序列分析,分析对比汾酒发酵室新旧土壤酵母群落结构差异,初步建立了汾酒发酵室新旧土壤酵母群落结构对比研究的方法以及为汾酒发酵室更换土壤提供理论依据。
近年来,以核酸为研究对象的分子生物学技术以其简单、准确等优点逐渐应用到酵母菌的分类鉴定中。在rRNA 中,26SrRNA 是核糖体RNA 的一个亚基,而26S rDNA 是编码该亚基的基因,26S rDNA 在细胞内有数以百计的拷贝,增加了扩增的可能性。而26S rDNA 的D1/D2 区域位于大亚基的5' 端,序列长度在600 左右,目前这一区域序列多应用于酵母菌的分类研究中。
1 材料与方法
1.1 样品来源
选取汾酒热季停产阶段2 个发酵室地缸间地表未更换的土壤各3 份,另选取1 份准备更换的新土壤作为对照组。
1.2 主要试剂
Marker DL2000、2×Taq PCR MasterMix、乙二胺四乙酸钠盐(EDTA-Na2)、三羟甲基氨基甲烷(Tris)、NaOH、Gelred(索莱宝)、酵母浸粉、葡萄糖、蛋白胨、琼脂、氯霉素、乙醇、乙酸、乳酸等。
1.3 主要仪器设备
ML204 电子天平,梅特勒-托利多集团;Milli Q Plus 超级纯水仪,伯乐生命医学产品有限公司;MAC-350P 高压灭菌锅,三洋电气公司;WP750 中型机械微波炉,广东格兰仕电器制造有限公司;ZHJH-C1109C 超净工作台,上海智城分析仪器制造有限公司;移液枪,艾本德股份公司;LT-36VLC8 人工气候培养箱,PERCIVAL 科技公司;5417R 高速低温离心机,艾本德股份公司;BIO-PARK 琼脂糖水平电泳槽及电泳仪电源,伯乐生命医学产品有限公司;PTC-220 PCR 仪,MJ-Research 公司;UVP Bioimaging System 凝胶电泳成像分析系统,美国UVP 公司;Centrifuge 5415D离心机,艾本德股份公司。
1.4 方法
1.4.1 培养基的配制
参考关凯乐等培养基配制的方法,略作修改。YPD 培养基的配制:酵母浸粉10 g/L、葡萄糖20 g/L、蛋白胨20 g/L、琼脂20 g/L、氯霉素200 mg/L、水1 L。115 ℃条件下灭菌30 min,在超净工作台无菌条件下加1 mL 乙酸,2 mL 乳酸。
1.4.2 样品处理及微生物的分离培养、计数、鉴定
取新旧土壤样品20 g 于180 mL 无菌水中,充分震荡混匀,形成10-1的菌悬液,吸取1 mL 于玻璃试管中,作为原液。在超净工作台内进行10 倍梯度稀释,依次稀释3 个梯度,吸取100 μL 稀释液于倒好培养基平皿中,每个稀释度做3 个平行,倒置于恒温培养箱,在30 ℃条件下培养。
接种后,每天定时观察,待菌落长出,选择稀释度合适,菌落数量合适的平板,先拍照计数,再随机选取具有形态差异的单菌落进行二次划线得到微生物的纯化条带。
1.4.3 基因组DNA 的提取
参考吴飞等的方法,所得DNA 溶解于TE 缓冲液中,放于-20 ℃保存备用。
1.4.4 26S rDNA 的PCR 扩增及测序
以酵母菌总DNA 为模板,采用真菌通用引物:引物1(NL1):5’-GCATATCAATAAGCGGAGGAAAAG-3’,引物2(NL4):5’-GGTCCGTGTTTCAAGACGG-3’。PCR 反应体系12 μL,DNA 模板2 μL,TaqDNA 聚合酶0.5 μL,无菌超纯水9 μL,PCR 反应条件:95 ℃预变性5 min,95 ℃变性1 min,50 ℃退火1 min,72 ℃延伸1 min50 s,共进35 个循环,最后72 ℃延伸10 min。经PCR 反应扩增出26S rDNA 产物,PCR 产物用1%琼脂糖凝胶电泳检测后,送上海迈谱股份有限公司进行纯化测序,将得到的序列与NCBI 进行BLAST 比对,得到的同源序列和测定序列在Clustal X 软件包中进行分析,形成一个多重序列匹配排列阵。导入MEGA4.0 软件,采用邻位相连(Neighbor-jioning)法构建系统发育树。
2 结果与分析
2.1 新旧土壤菌株分离比较
由此表1 可知,旧缸花中酵母菌数量显著高于新缸花中酵母菌数量,通过梯度稀释、平板分离培养计数的方法,前者分离得到了7975 株,而后者只分离到了765 株。

表1 新旧土壤菌株分离计数比较
2.2 26S rDNA 序列的同源性分析及系统发育树的构建
经过酵母菌的菌落形态及显微镜特征观察,从中挑选17 株有表征差异的代表性酵母菌株,提取其DNA,利用NL1 与NL4 引物扩增目标片段后,将测序结果序列在NCBI 中用BLAST 进行同源性分析并构建系统发育树,见下页图1。
从图1 中可以看出,汾酒缸花中的酵母菌有Pichia、Wickerhamomyces、Saccharomycopsis、Candida 与Meyerozyma 这5 个属。
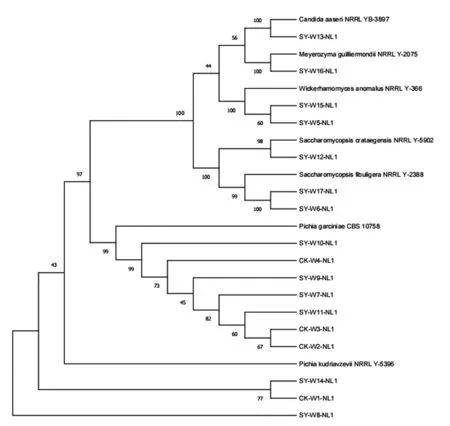
图1 系统发育树
在系统发育树上,W1、W8、W14 菌株属于Pichia kudriavzevii;菌株W2、W3、W4、W7、W9、W10、W11 属于Pichia manshurica;W5 和W15 属于Wickerhamomyces anomalus;W6 和W17 属 于Saccharomycopsis fibuligera;W12 属于Saccharomycopsis malanga;W13 属于Candida aaseri;W16 菌株属于Meyerozyma guilliermondii。其中菌株Saccharomycopsis malanga 首次在清香型白酒酿造的环境中被发现。其中绝大部分的酵母菌都在旧缸花中发现,而新缸花中只分离到一株P.kudriavzevii 与两株P.manshurica。
3 小 结
整体来讲,汾酒旧缸花中酵母菌的遗传多样性高于新缸花,且优势菌群与清香型大曲酵母菌结构基本一致,这些菌株可能来自入缸发酵前的酒醅。值得关注的是,此次分菌并未发现酿酒酵母,这可能与土壤的微环境不适宜其生长有关。通过比较新旧缸花菌群差异,可以促进对厂区的酿造环境微生物多样性的了解,为深入了解更换旧缸花是否对酒质产生影响提供客观依据。

