食源性致病菌污染估计中删失数据分析的研究进展
孙天妹,刘阳泰,王 翔,董晓璐,刘 弘,李红梅,董庆利,*
(1.上海理工大学医疗器械与食品学院,上海 200093;2.上海市疾病预防控制中心,上海 200336)
食品安全已成为国际性公共卫生问题,根据世界卫生组织2019年的报告,全球每年至少有6亿 人患食源性疾病,由此引发的死亡人数高达42 万,所致疾病负担累计为3 300万 人年[1]。2017年全国各省份通过国家突发公共卫生事件报告管理信息系统报告的食物中毒事件的统计结果表明,细菌性食物中毒事件的报告数量和中毒人数最多,分别占食物中毒事件总数和中毒总人数的31.61%(110/348)和57.60%(4 256/7 389),所涉及的食源性细菌主要有沙门氏菌、单核细胞增生李斯特菌(以下简称“单增李斯特菌”)和致泻性大肠杆菌等[2]。因此,对食品中的致病菌进行风险评估对保障食品安全、降低食源性疾病暴发率具有重要意义。
微生物定量风险评估(quantitative microbial risk assessment,QMRA)作为有效工具之一,通过量化的数值描述致病菌对人群健康产生不良作用的风险,为监管者提供科学的风险管理依据[3]。QMRA分为危害识别、危害特征描述、暴露评估和风险特征描述4 个模块[4],其中包含大量的不确定性因素,可能影响最终评估结果的准确性。暴露评估作为风险评估的核心内容,主要依据目标致病菌在目标食品中的污染量,结合居民食品消费量,估计致病菌暴露于消费者的可能性水平[5-6]。因此,食品中致病菌的污染量信息是定量风险评估中不确定性的重要构成因素之一[7]。
致病菌在食品中的污染情况主要依靠食品采样和微生物学检验获悉。最大可能数法和平板计数法是微生物定量检测中较常用的两种方法[8]。除此之外,荧光定量聚合酶链式反应(quantitative polymerase chain reaction,qPCR)、变性梯度凝交电泳和宏基因组测序等分子生物学技术作为快速检测方法,在致病菌的定量检测中也得到了初步应用[9]。在实际情况中,由于检测方法的限制,当致病菌污染水平低于或高于相对应的检测阈值时,因无法完全定量食品中存在的致病菌而产生大量删失数据(censored data),造成食源性致病菌污染检测信息的不完整。在对食源性致病菌的污染检测数据进行浓度估计时,若不考虑删失数据的存在,其结果往往会产生较大的偏差,提升风险评估结果的不确定性。与此同时,致病菌在食品中的污染水平较低,其检测数据具有一定的变异性。因此,有效处理致病菌污染检测中出现的删失数据有利于保证食源性致病菌污染水平估计的精准化,提高风险评估结果的准确性。
近年来,删失数据分析主要用于应对临床医学、生命科学及环境科学等领域的生存类数据,而在食源性致病菌污染水平估计方面尚无完整的删失数据分析体系,缺乏不同处理方法对估计结果影响的认知。故本文围绕食源性致病菌污染检测数据中删失数据的类别、常用分析方法以及不同删失数据情况下污染水平估计的精准化展开综述和讨论,以期为风险评估人员和风险管理人员提供理论参考,进一步加强食品安全的风险控制。
1 食源性致病菌污染检测中删失数据的分类
食源性致病菌污染检测的结果一般以不可检测(non-detect,ND)、阳性或某一分析单元中的浓度表示,其中ND值即为删失数据。统计学上将删失数据定义为“未量化的观测值”,易对统计数据的整体描述产生干扰。在食品微生物学领域,根据食源性致病菌的检测特点,将其删失数据主要分为以下3大类:左删失数据(left-censored data)、右删失数据(right-censored data)以及区间删失数据(interval-censored data)[10]。
1.1 左删失数据
食源性致病菌的定性检测和定量检测方法均有定性检测限(limit of detection,LOD)或定量检测限(limit of quantification,LOQ)。当检测样品中致病菌的浓度过低,低于所用检测方法的LOD或LOQ时,此类ND值为左删失数据[11]。对左删失数据进行处理分析,可降低部分样品中存在致病菌污染而检测结果为阴性的数据对食源性致病菌整体浓度估计产生的偏差。
1.2 右删失数据
食源性致病菌定量检测方法除了存在一个较低的LOQ外,往往还有一个最大的检测限值与之相对应。当检测样品中的致病菌浓度过高,超出该检测方法的最大检测阈值时,通常会以多不可数的结果进行报告,此类ND值为右删失数据[12]。致病菌在食品中的污染水平较低,因此在实际检测中,出现右删失数据的情况较少。
1.3 区间删失数据
在食源性致病菌检测中,同一样品可能会经多种方法进行检测与鉴定。由于检测方法的不同,其检测限、敏感性和特异性也有所不同[13],因此样品在检测过程中往往会出现在某一检测方法下不可检测,而在另一检测方法下产生阳性数据的现象。如某一样品同时进行定性检测和定量检测,该样品在定性检测时结果为阳性,在定量检测时结果呈现ND值(即阴性)。在食源性致病菌的污染检测中,具有此类特征的数据被称为区间删失数据[14]。对区间删失数据进行有效地统计分析可避免因检测方法的差异而产生的假阳性或假阴性数据对食品中致病菌浓度水平的低估或高估。
2 食源性致病菌污染检测中删失数据的分析方法
在食源性致病菌的污染检测数据中,删失部分无法提供准确的检测浓度。通过选择适当的统计学方法来分析删失数据,可从包含ND值的污染检测数据中提取最大数量的可靠信息,从而提高食源性致病菌污染浓度估计的准确性。图1整理了医学、生命科学及环境科学等领域用于删失数据处理分析的相关统计学方法,主要包括替代法、多重填补(multiple imputation,MI)法、参数估计法、非参数估计法、假设检验比较法及回归分析法这6大类,其中一些方法可引入食源性致病菌污染检测删失数据的分析中[15-17]。根据文献调研,较常用于食源性致病菌污染检测删失数据定量估计的方法主要有替代法、参数估计法、非参数估计法和MI法,表1对这4 类常用分析方法的特点和局限性进行了逐一比较。由于假设检验比较法多用于比较可检测值的数据集和包含ND值数据集之间的差异显著性,而回归分析法需满足数据的独立性假设,在处理具有相互依赖关系的数据集时具有一定难度,故这两种方法在食源性致病菌的污染水平估计中使用频率较低。
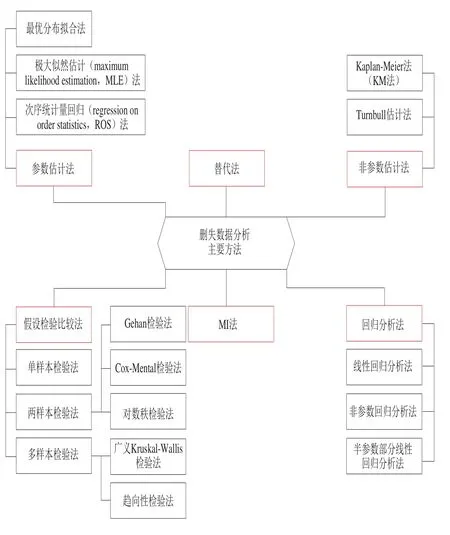
图1 删失数据分析的主要方法Fig.1 Methods commonly used for censored data analysis

表1 食源性致病菌删失数据分析中4 类常用方法的特点和局限性比较Table 1 Comparison of the advantages and disadvantages of four commonly used methods for the analysis of foodborne pathogens censored data
2.1 替代法
早期研究通过借鉴分析化学检测中ND值的处理,在食品微生物学中采用删除法来处理检测中出现的ND值,即在分析过程中将ND值去除,只保留可检测值用于分析。由于食源性致病菌污染水平对ND值较为敏感,通过该方法估计的结果可能存在较大偏差,故该方法不适宜用于处理食源性致病菌的污染检测删失数据。由此,在随后的食源性致病菌污染检测数据分析中采用替代法来处理ND值,较为常见的是将ND值作“0”值处理。然而,检测中ND值的产生,一方面可能由于检测样品未受微生物污染,为真实的“0”值;另一方面可能由于样品中存在目标致病菌,因受方法限制而未成功检出[22]。由于后一种情况的存在,将ND值作“0”值处理并不恰当。Shoari等[18]通过蒙特卡洛方法模拟环境样中的污染数据,将低于LOD的ND值用LOD的不同形式(如LOD、LOD/2或LOD/等常数)来替代,结果表明替代法的性能随数据集中删失比例的降低而提高。由于替代法是用单一值替代数据集中所有的ND值,因此当ND值在整个数据集中占比较大时,使用该方法会使食源性致病菌整体的浓度估计产生较大偏差[23-24]。
2.2 参数估计法
参数估计法需要对数据进行特定的分布假设,主要分为最优分布拟合法、MLE法和ROS法[19]。最优分布拟合法是运用合理的统计分布模型来描述食品中致病菌的频率分布,从而可近似估计致病菌的污染水平[25]。国际生命科学协会在已发表的理论研究中提出,在选择恰当的统计分布模型时可根据以下5 个特征,分别是非负、允许“0”值、离散型、可递减(或近似)泊松分布以及致病菌数量较高时,应近似对数正态分布[26]。表2列举了不同种类的统计分布模型分别具备的特征以及描述致病菌在不同食品基质中污染情况的应用。
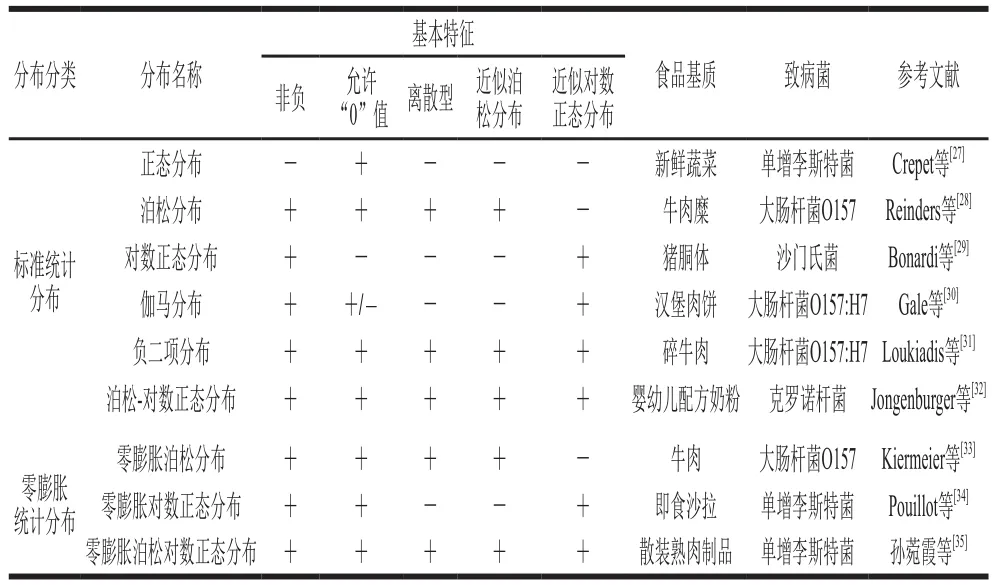
表2 不同种类的统计分布模型所具备的特征以及描述致病菌在食品基质中污染情况的应用Table 2 Characteristics of different types of statistical distribution models and their applications for describing the contamination of pathogens in food matrix
当食源性致病菌污染检测数据中ND值比例较小时,可选取恰当的标准统计分布模型进行近似拟合。若食源性致病菌污染检测数据中存在大量删失数据,导致数据集呈零膨胀(zero-inflated)现象时,零膨胀模型更适宜。Kiermeier等[33]发现零膨胀泊松分布可很好地拟合牛肉中大肠杆菌O157的检测数据。孙菀霞等[35]在散装熟肉制品单增李斯特菌左删失数据的处理分析中发现,零膨胀对数正态分布与零膨胀泊松对数正态分布的拟合度优于标准统计分布。然而,在运用零膨胀模型时,若对模型本身产生的“0”值和致病菌检测中产生的“0”值处理不当,可使拟合得到的致病菌浓度产生偏差[36]。
MLE法是假定数据集中的删失部分和未删失部分都遵循相同的分布(如对数正态分布等),再通过似然函数进行参数估计,使数据与假定的分布实现较高程度的吻合。当数据特征接近于假定的分布且模型拟合良好时,使用MLE法可有效处理删失数据。而ROS法是仅对数据集中的未删失部分进行某一特定的分布假设,建立次序统计量回归方程,并对删失数据进行填补[37]。MLE法和ROS法在应用中也存在一定的缺陷性,如这两种方法易受到异常值的影响,且对假定的分布选择较为敏感。由于研究者们习惯假定数据服从于对数正态分布,因此在估算平均值和标准差时易产生反向转换偏差。为了解决这些问题,Kroll[38]和Helsel[39]等分别提出了稳健MLE法和稳健次序统计量回归法,从而可以较好地避免数据转换中平均值和标准差的异常估计。部分研究者认为MLE法是处理删失数据的“黄金标准”,当数据集中包含至少30~50 个可观测值时,MLE法具有最佳性能;而当数据集高度偏斜且删失程度较高时,MLE法可能并不适用[40]。加拿大卫生部在《关于复杂人类健康的详细化学定量风险评估指南》中建议,MLE法适用于大样本量的数据处理,而ROS法适用于一般样本量的数据处理[41]。
2.3 非参数估计法
由于删失数据的存在,研究人员常常无法得到足够多的信息来判断数据资料服从何种分布假设。非参数估计法无需计算参数,也无需假定数据的分布,不受样本数据从属总体分布的限制,因此在删失数据处理分析中应用较多[20]。KM法作为一种非参数法,基于一个累积分布函数,对包含随机删失数据的观测值直接估计累积概率。KM法最初用于右删失数据的处理,后经Turnbull[42]推广到左删失数据分布函数的非参数拟合中,即形成Turnbull估计法。Helsel[43]在分析处理环境水样检测中毒性当量浓度的左删失数据时比较了替代法和KM法,结果表明使用KM法进行浓度估计效果要优于替代法,且KM法适用于数据精度较低的情况。同时,也有研究表明当数据呈严重删失或呈高度偏态时,使用非参数的KM法进行处理效果更优[44]。然而,当数据集的样本量较大时,选择非参数估计法进行删失数据的处理具有一定的局限性。
2.4 多重填补法
MI法基于贝叶斯理论,在替代法(即用一个单一值替代每个ND值)的基础上作出改进,从观测值的后验概率分布中随机抽取一系列(m个)合理的填充值替代每个ND值,通过创建若干个完整数据集再对每一完整数据集分析后综合得到结果[21]。MI法需要所有参与分析的变量服从联合概率分布。2014年,美国国家环境保护局提出用MI法来处理删失数据将更为严谨[45]。Lin等[46]在一个艾滋病临床试验的案例研究中将多变量纵向数据中的ND值进行多重填补,结果表明在删失比例较高的纵向数据处理中,MI法优于其他删失数据分析方法。Canales等[47]比较了替代法、服从对数正态分布假设的MLE法、KM法、基于对数正态分布估计参数的MI法和基于均匀分布估计参数的MI法这5 种方法在低、中和高3 种删失程度的左删失数据处理中的应用情况,结果表明两种MI法的数据处理效果优于其余3 种方法。同时,该作者又提出这两种MI法是通过使用R语言中自带的程序包实现的,未考虑分布误选的因素。因此,在删失数据的处理中,有必要在MI法中纳入更多合理的分布模型以提高数据拟合的准确性。MI法虽在删失数据的无偏估计中占有优势,但其在反复填补ND值时往往会占用较大内存,比较耗时。
综上,在选择合理的删失数据分析方法时,除依据均方根误差或偏差等统计学评价指标的比较外,更应侧重于对所需拟合分析的食源性致病菌污染检测数据特征进行判断,如数据集样本量的大小、数据集内删失数据的类别或数据集的删失比例等。因此,将删失数据分析方法应用于食源性致病菌的污染水平估计时,需考虑食源性致病菌污染检测数据删失情况的不同。
3 数据删失情况下的食源性致病菌污染水平估计
食品中致病菌污染水平的确定取决于对致病菌污染检测数据的准确估计。由于检测方法的限制,食源性致病菌的污染检测数据集中往往会包含一定比例的左删失数据、右删失数据或区间删失数据。基于不同类别的删失数据特征,综合前述的食源性致病菌删失数据常用分析方法,可应用于不同评估要求食源性致病菌污染水平的估计。
3.1 左删失或右删失数据下的致病菌污染定量估计
食品中致病菌的污染水平通常较低,在定量检测数据中左删失数据(<LOQ)的占比要远远高于右删失数据,且致病菌检测中出现的右删失数据可通过连续稀释到适宜梯度进行浓度计数,因此针对于食源性致病菌污染检测数据中左删失数据处理分析的研究居多。基于食品采样及致病菌检测所得到的定量数据,对其左删失数据进行处理分析,将对风险的预测具有显著影响。Duarte等[48]通过区分微生物检测中的真实“0”值和人为“0”值(即小于LOQ的左删失数据),开发一种新的模型进行微生物浓度分布及患病率的估计,结果表明正确处理检测中得到的“0”值是准确表征微生物总体污染水平的关键之一,且微生物浓度分布和患病率的估计具有较高的相关性。Duque等[49]从法国4 个不同屠宰场中进行鸡胴体样本采集并开展空肠弯曲杆菌的检测,将包含左删失数据和未包含左删失数据的两个数据集进行比较,并通过正态分布、对数正态分布和伽马分布3 个模型进行数据拟合,结果表明正态分布拟合效果最优。由正态分布拟合到的弯曲杆菌浓度平均值为2.74 (lg(CFU/g)),且不确定性范围缩小至1(lg(CFU/g)),进而较精准地定量了鸡胴体中空肠弯曲杆菌的污染水平。Cantoni等[50]在对饮用水和地下水中微量污染物检测中的左删失数据分析处理时,首先选择对数正态分布、正态分布、Weibull分布和伽马分布等模型进行拟合,再通过MLE法进行参数估计,可更精确地进行水质分析(如水源中污染物随时间的变化趋势和对污染物的处理效率),从而更有利于评价人类健康风险。因此,在食源性致病菌污染检测数据的定量估计中,左删失数据扮演着重要的角色,根据数据集特征的多样性来选取恰当的统计分析方法是保证致病菌准确定量估计的关键。
3.2 区间删失数据下的致病菌污染定量估计
在食源性致病菌的监测工作中,传统定量检测方法实验周期长,需耗费大量人力物力,因此多用定性方法检测致病菌,与此同时造成致病菌定量数据的大量缺失。如何将致病菌的定性数据有效转化为定量数据,是目前开展QMRA的难点之一。
定性检测方法和定量检测方法具有不同的检测限、敏感性和特异性,这使得在定性数据和定量数据的比较中可能会存在一部分区间删失数据。对区间删失数据进行正确的处理分析有助于提高食源性致病菌定性数据到定量数据转化过程中的精确性。Jarvis[51]基于食品中致病菌服从泊松分布的假设,提出了一种基于定性检测数据来确定一批样品中致病菌平均污染浓度的有效方程。目前,该方程已广泛用于只有定性检测数据的食品样品中致病菌的定量估计。Andritsos等[52]对猪肉糜中单增李斯特菌首先基于PALCAM、ALOA和RAPID’L.mono 3 种不同的选择性培养基进行传统定性检测,得到阳性率分别为16%、19%和26%,再经分子生物学方法鉴定,对多种检测方法下的数据进行真实阳性、真实阴性、假阳性和假阴性等多组数据集的整理,并基于不同检测方法敏感性和特异性的计算,得出猪肉糜中单增李斯特菌的真实污染率为22%。随后假设猪肉糜中单增李斯特菌服从泊松分布,并构建真实污染率和敏感性的关系式,进而估计出猪肉糜中单增李斯特菌的浓度为14~17 CFU/kg,实现定性数据到定量数据的转化。随后,Sun Wanxia等[53]在此基础上,假设熟肉制品中的单增李斯特菌服从零膨胀分布(即零膨胀泊松分布或零膨胀泊松对数正态分布),并基于贝叶斯方法对熟肉制品中单增李斯特菌浓度进行估计,构建了一种基于定性数据的定量转化概率模型。
目前,传统致病菌检测方法向基于分子生物学技术等快速检测方法转变的趋势越来越明显。这些快检方法所得数据将生成定量数据,最终需要转化为致病菌的浓度。根据检测方法的不同,其检测限具有较大差异,因此在区间删失数据处理模型的选取方面应考虑该模型是否允许不同检测限的输入。Kato等[54]提出了一种基于截断对数分布的贝叶斯随机模型,可允许不同样本不同LOD的输入,并将此模型用于环境水样检测中指示菌和致病菌在基因水平上浓度比的估计。Poma等[55]使用qPCR对水样中的诺如病毒和肠炎病毒进行定量检测,并将低于方法LOD的删失数据使用样本检测限值(the sample limit of detection,SLOD)的不同形式进行替代,即SLOD、SLOD/2、SLODa(average SLOD)、SLODm(median SLOD),以保证每个样本检测值的唯一性。基于此方法将水样中诺如病毒和肠炎病毒检测的qPCR定量数据转化为污染水平,并结合不同的暴露场景,开展进一步的定量风险评估。随着检测方法的更新及组学技术的发展,食源性致病菌定量风险评估不应局限于传统定量检测下的致病菌定量数据,充分利用新检测技术下的数据,确保多种数据信息在致病菌定量估计中具有同等的有效性,是当下开展新一代定量风险评估的突破口之一[56-58]。
准确估计食品中致病菌的污染水平是降低风险评估不确定性的前提条件。综上所述,选择恰当的方法处理删失数据可一定程度上弥补检测信息的不足,避免食源性致病菌污染水平的有偏估计,减少风险评估过程中不确定性的产生。同时,不可忽视食源性致病菌污染检测数据的变异性因素对最终结果的影响。变异性是由特定群体在时间、空间或个体上呈现的风险异质性产生,为固有属性[59]。如何在食源性致病菌污染检测删失数据处理的基础上纳入其检测数据的变异性分析仍是目前实现食源性致病菌污染定量估计精准化的瓶颈之一。致病菌在不同的食品基质中具有不同的空间分布特征,在固体及粉末状食品中多呈集群(cluster)分布[60]。若某批次内的食品中致病菌呈集群分布,由于采样的随机性,通过检测可得的致病菌定量数据往往会呈现出较大的变异性,主要体现在致病菌污染的季节变异性、批次间变异性、批次内变异性和菌株变异性等。为更好地描述变异性,一方面食源性致病菌的风险监测计划应更具体且全面,需充分考虑采样时间、采样点、样品类别、检测方法、采样批次/样本/分析单元等信息的有效收集。Mikkela等[61]通过构建采样批次-样本-分析单元的分层结构,并提出了一个基于贝叶斯理论的时序-污染率-浓度模型,对呈高度异质性的鸡肉中弯曲杆菌的检测数据进行处理分析,以精准估计鸡肉中弯曲杆菌的定量水平用于进一步的风险评估。另一方面,需要在相关模型的构建及软件程序应用的更新方面推进研究。1992年,Frey[62]提出二维蒙特卡洛模拟(two-dimensional Monte-Carlo simulation,2D-MC)方法,可用来估计由于参数不确定性引起的风险估计的不确定性。由于2D-MC可通过对风险评估中的变异性因素和不确定性因素分别抽样来反映两者的分布情况,并能在输出结果中分别估计变异性和不确定性,故该方法在QMRA领域已被广泛应用。Pouillot等[63]开发了用于建立和研究2D-MC的R语言工具包,可直接将QMRA中的变异性和不确定性因素分开估计,风险评估人员可利用工具包首先对变异性进行分析估计,后在此估计基础上再评估不确定性,为后续风险评估研究的进一步开展提供了便利。因此,在开展食源性致病菌的定量风险评估时,评估人员应谨慎分析致病菌污染检测数据的变异性及污染信息不足导致的不确定性,确保评估结果的准确性,进一步加强风险监测、风险评估和风险管理的互动交流。
4 结 语
食源性致病菌的污染检测数据中往往包含大量的删失数据。根据数据集的删失特点,选择恰当的分析方法进行数据处理,利用转化后的食源性致病菌浓度可较准确地估计人群患病风险。因此,删失数据的分析研究在食源性致病菌定量风险评估中具有实际意义。在此基础上,评估人员还需关注食源性致病菌污染检测数据的变异性,实现对致病菌污染水平估计的精准化,以进一步推进我国食源性致病菌污染定量的建模工作。综合国内外有关微生物检测中删失数据处理分析的研究现状及我国食源性致病菌污染检测数据的定量估计所存在的问题,提出如下建议:1)当前用于删失数据分析的相关模型在数据特征及处理条件方面均存在或多或少的局限性,应在统计学领域尝试更多有用模型的组合,如在MLE法的分布假设时考虑结合最优分布模型的选择,进一步优化方法使其更符合数据条件,从而降低食源性致病菌风险评估的不确定性;2)应进一步推进食源性致病菌的风险监测工作,以不同监测批次为纵向检测目标,以同一监测批次下致病菌的定性数据、定量数据和分子分型数据等作为横向检测目标,进行同一批次下多横向监测数据和同一横向检测目标下多纵向监测数据的收集,实现致病菌风险监测指标多维化,挖掘不同食品基质中致病菌污染数据的变异性因素,实现监测大数据的有效整合;3)随着当前食品安全检测技术的不断推进,培养组学、宏基因组和宏转录组等新技术下的多种检测信息应被有效利用,对致病菌新型检测污染数据进行定量转化后用于暴露评估中是下一代定量风险评估工作的核心内容,可利用快检方法的优势提高风险预警能力。

