甘薯全基因组WRKY转录因子的基因鉴定与逆境胁迫表达分析
毕楚韵,黄小芳,王和寿,陈其俊,胡韵卓,黄碧芳,许 明,杨志坚,林世强,,陈选阳,1d
(1福建农林大学a作物生物技术福建省高校重点实验室,b农学院,c生命科学学院,d教育部作物遗传育种与综合利用重点实验室,福建 福州 350002;2 宁德市农业农村局,福建 宁德 352100;3 福建省种子总站,福建 福州 350003)
在植物生长发育过程中,植物免疫系统通过转录因子调控基因表达来抵御病虫害和非生物胁迫[1]。转录因子通常以家族的形式存在,植物的重要转录因子家族有WRKY、MYB、HLH、NAC等,其中WRKY转录因子是高等植物中最为常见的转录因子家族之一,在拟南芥(Arabidopsisthaliana)中有72个[2]、水稻(Oryzasativa)中有102个[3]、枣树(ZiziphusjujubaMill.)中有54个[4]。在结构方面,WRKY转录因子具有高度保守的序列,其蛋白N端一般包含WRKYGQK七肽序列构成的WRKY结构域,随后是C2H2(CX4-5CX22-23HXH)或C2HC(CX7CX23HXC)组成的锌指结构[5]。在功能方面,WRKY转录因子参与植物响应生物胁迫和非生物胁迫,例如拟南芥AtWRKY33调控植物对抗葡萄孢菌(Botrytiscinerea)的激素合成[6],过表达水稻OsWRKY6能使水稻(Oryzasativa)对病原物产生更强的抗性[7]。有研究发现,当WRKY转录因子增强植物对生物胁迫的耐受性时,同时会一定程度上降低对另一种胁迫的抗性[8]。在拟南芥中过量表达杨树(Populustrichocarpa)PtrWRKY73可增强植株对丁香假单胞菌(Pseudomonassyringae)的抗性,但对葡萄孢菌(Botrytiscinerea)的抗性则降低[9]。在非生物胁迫下,WRKY转录因子通过参与复杂的信号转导过程调控植物的抗性,如在高温高湿条件下,甜椒(Capsicumannuum)CaWRKY6可通过激活CaWRKY40以增强其耐受性[10]。此外,WRKY转录因子在植物生长发育,例如种子萌发和休眠[11]、开花期[12]、叶片衰老[13]等过程中也发挥着重要作用。
甘薯是重要的粮食作物,也是淀粉加工原料和能源作物,最早被克隆研究的WRKY转录因子来自甘薯的SPF1[14]。研究表明,SPF1有调控蔗糖生成的潜在功能,生物信息学分析发现其N端具有叶绿体定位功能,可能参与细胞核和叶绿体基因的表达调控[15]。然而到目前为止,有关甘薯WRKY基因结构和功能方面的研究报道仍然较少。本研究应用生物信息学方法挖掘甘薯全基因组WRKY转录因子,并对其序列、保守结构域、潜在复制关系等进行分析,利用SRA数据库中甘薯在生物胁迫(蔓割病菌侵染)和非生物胁迫(贮藏期低温)条件下的转录组数据,分析WRKY基因的差异表达,为甘薯WRKY基因功能与调控的分子机制研究以及WRKY基因的应用提供参考。
1 材料与方法
1.1 甘薯全基因组基因注释
从https://www.ncbi.nlm.nih.gov/genome/?term=Ipomoea+batatas下载甘薯全基因组序列[16]。使用拟南芥、水稻和线虫的HMM模型分别对甘薯染色体蛋白质编码区进行预测[17]。从结果文件中随机选取500组制作针对甘薯的HMM模型,以此模型预测得到甘薯全基因组蛋白序列[18]。
1.2 WRKY的基因预测与鉴定
从Pfam网站[19]下载WRKY(PF03106)的HMM模型,运行Hmmsearch检索甘薯全基因组蛋白序列,选取E值≤4.2×10-11的序列构建针对甘薯的HMM模型[20]。以此特异的HMM模型预测得到124个候选WRKY蛋白序列,上传至Conserved Domains Tool[21],筛选得到90个WRKY蛋白候选序列。对这些序列使用InterProScan[22]确认WRKY结构域,得到89个WRKY蛋白候选序列。核对每个蛋白序列后,得到56个结构域完整的WRKY蛋白。应用AUGUSTUS[23]对其余33个WRKY结构域不完整的基因进行序列预测和校对;同时下载Lin等[16]的甘薯SRA数据,将基因序列与转录组测序得到的read序列进行比对,从而修补基因的WRKY结构域序列,得到14个完整的WRKY蛋白,两组相加共得到70个WRKY蛋白。用拥有完整WRKY结构域的70个WRKY蛋白在甘薯全基因组蛋白库运行Blastp,检索更多的WRKY候选蛋白序列,再次对序列的WRKY结构域进行确定和序列修补,最终得到82个甘薯WRKY蛋白,根据其相应基因在染色体上的位置,以IbWRKY1~IbWRKY82进行编号并建立甘薯IbWRKY蛋白数据库。
1.3 WRKY基因序列潜在复制关系的分析
对甘薯所有WRKY基因序列进行Blastn比对,筛选出序列相似度大于75%并且两条序列比对上的序列长度大于较长序列长度75%的基因对,提取基因在染色体上的位置以及基因对的潜在复制关系,使用Circos[24]绘图。
1.4 WRKY蛋白的亚家族分类
根据参考文献[2,5,25-26]的分类依据,下载拟南芥WRKY蛋白序列(http://www.arabidopsis.org/)与甘薯WRKY蛋白序列进行比对分析,分类成各亚家族。
1.5 WRKY蛋白保守结构域序列的分析
应用Clustal Omega软件[27]对WRKY蛋白序列进行多序列比对后,使用Jalview软件[28-29]分析并绘图。
1.6 WRKY蛋白亚家族系统进化树的构建
利用软件MEGA X[30],使用ML(maximum likelihood)方法,并添加拟南芥WRKY蛋白序列后,对甘薯WRKY蛋白序列的各亚家族构建系统进化树。
1.7 WRKY蛋白保守基序分析
使用MEME[21]对82个甘薯WRKY蛋白进行保守基序预测,保守性基序的数目限制为10,分别命名为基序1~基序10。
1.8 WRKY基因的抗逆表达模式分析
从SRA数据库分别下载蔓割病菌[16]和低温胁迫下的甘薯转录组数据[32],将82个甘薯WRKY基因序列与甘薯转录组进行比对,提取基因在不同处理组和对照组中的表达信息。应用DESeq2对数据进行标准化处理,数据筛选的Padj设为小于0.05,只保留以2为底差异表达倍数的对数(log2FoldChange)值大于1或者小于-1的数据,使用Pheatmap绘制热图。
1.9 甘薯SPF1相似IbWRKY蛋白检索
从NCBI下载SPF1的氨基酸序列(GenBank ID:BAA06278.1)。本地运行Blastp软件,检索1.2节中建立的IbWRKY蛋白数据库,搜索与SPF1相似的蛋白,并使用EMBOSS needle软件进行序列比对和分析。
2 结果与分析
2.1 甘薯WRKY转录因子的鉴定、染色体定位和潜在复制关系
经鉴定分析,在甘薯全基因组中得到82个WRKY基因,对其进行染色体定位,发现其在15条染色体上分布不均匀(图1),其中8号染色体和15号染色体上各仅有1个,12号染色体和14号染色体上最多,均为11个。基因在染色体上的分布可分为2种形式:单个存在和2个及以上在同一区域形成基因簇。甘薯WRKY基因在多条染色体上存在基因簇,拥有最多基因簇的是12号染色体,有3个基因簇,其中2个基因簇包含3个基因,另1个基因簇包含2个基因。

染色体之间和染色体内的潜在复制基因连线分别为蓝色和橘色Inter-chromosomal and intra-chromosomal potential duplication relationships are represented by blue and orange lines,respectively图1 甘薯82个WRKY基因在15条染色体上的定位及其潜在复制关系Fig.1 Analysis of chromosomal localization and potential duplication relationship of 82 Ipomoea batatas WRKY genes in 15 chromosomes
根据Gu等[33]确定的基因潜在复制关系的分析方法,发现有25对基因存在潜在复制关系,包括10对染色体间潜在复制基因和15对染色体内潜在复制基因,分布于1、7、8、9、11、12和14号染色体上。11号染色体与12号染色体之间的潜在复制基因对最多,有4对,但仅涉及4个基因,而且它们之间的位置非常接近。染色体内的潜在复制通常发生在较为相近的基因之间,例如1号染色体上的3对潜在复制基因,分别为IbWRKY5与IbWRKY6(距离338 810 bp)、IbWRKY6与IbWRKY4(距离405 185 bp)、IbWRKY4与IbWRKY5(距离64 233 bp);但也有染色体内潜在复制基因之间的距离较远,例如9号染色体上的IbWRKY40与IbWRKY37之间的距离为5 317 598 bp。
2.2 甘薯WRKY亚家族分类与保守结构域的序列分析
按照Eulgem等[2,5,25-26]的WRKY蛋白分类方法,引入拟南芥WRKY蛋白进行比对后,可将82个甘薯WRKY蛋白分为Ⅰ、Ⅱ、Ⅲ 3大类,其中Ⅱ类蛋白还可进一步分为Ⅱ-a、Ⅱ-b、Ⅱ-c、Ⅱ-d和Ⅱ-e等5个亚类。不同类的蛋白数量不同,Ⅰ类蛋白18个,Ⅱ-a亚类4个,Ⅱ-b亚类14个,Ⅱ-c亚类20个,Ⅱ-d亚类8个,Ⅱ-e亚类13个,Ⅲ类5个。
为了比较和分析甘薯WRKY蛋白的保守结构域,截取82个WRKY蛋白中长度约65个氨基酸残基的结构域进行多序列比对。结果显示绝大多数Ⅰ类WRKY转录因子拥有2个WRKY结构域:Ⅰ-N和Ⅰ-C(分别位于蛋白N端和C端),WRKY七肽高度一致,而锌指结构的保守性相对较弱(图2)。此外,在Ⅰ类WRKY转录因子中有少数缺失保守结构域,例如IbWRKY59和IbWRKY1缺失N端的WRKY结构域,IbWRKY65和IbWRKY42缺失C端WRKY结构域,IbWRKY45和IbWRKY25的C端WRKY结构域缺失锌指结构域。Ⅲ类WRKY转录因子只有1个WRKY结构域,WRKYGQK高度保守,而锌指结构保守性较弱(图2)。

依照Clustal X的配色原则[27],蓝色为疏水性氨基酸,红色为正电荷氨基酸,紫色为负电荷氨基酸,绿色为极性氨基酸,粉色为半胱氨酸,橙色为甘氨酸,黄绿色为脯氨酸,蓝绿色为芳香族氨基酸,无配色为非保守性氨基酸。蛋白名斜杠后的数字为截取的氨基酸范围。图3同The amino acids are colored according to the guidelines of Clustal X in reference[27].Blue indicates hydrophilic amino acids,red indicates positively charged amino acids,purple indicates negatively charged amino acids,green indicates polar amino acids,pink indicates cysteines,orange indicates glycine,yellow-green indicatesprolines,and blue-green indicates aromatic amino acids.The non-conservative amino acids are uncolored.The number behind forward slash represents the range of selected amino acids.The same for Fig.3.图2 甘薯WRKY转录因子Ⅰ和Ⅲ类WRKY的保守结构域分析Fig.2 Conservative domains of subfamily-Ⅰ and subfamily-Ⅲ WRKY transcription factors in Ipomoea batatas
在82个甘薯WRKY转录因子中,Ⅱ类的数量最多,占72%。Ⅱ类转录因子拥有1个保守的WRKY结构域,根据其结构域的序列差异,分为5个亚类[5]。在截取的长度约为65个氨基酸残基的结构域中,Ⅱ-a、Ⅱ-b、Ⅱ-d和Ⅱ-e这4个亚类的保守结构域较为保守,Ⅱ-c亚类的氨基酸序列结构域则存在较多变异(图3)。

图3 甘薯WRKY转录因子Ⅱ类WRKY的保守结构域分析Fig.3 Conservative domains of subfamily-Ⅱ WRKY transcription factors in Ipomoea batatas
甘薯WRKY蛋白中的WRKY结构域和锌指结构域大部分比较保守,但是在一些WRKY蛋白中存在不同程度的氨基酸序列变异(表1),其中Ⅱ-c亚类中8个WRKY蛋白存在WRKY结构域序列的氨基酸变异,其WRKYGQK变异为WRKYGKK;锌指结构域的变异多见于Ⅱ类WRKY转录因子,其中Ⅱ-b亚类中最多,变异类别包括锌指结构中Cys到His之间的氨基酸残基数目的增加或减少,例如氨基酸残基减少的变异形式,由CX5CX23HXH变异为CX5CX16HXH。还有两个Cys之间的氨基酸残基的数量变异,如属于Ⅱ-d亚类的蛋白由CX5CX25HXH变异为CX6CX25XXX的形式。此外还有一些WRKY蛋白缺失WRKY七肽结构域或锌指结构,这类缺失除了这些WRKY蛋白本身缺失相应结构域的原因之外,也不排除由于基因组质量偏低造成[34]。

表1 甘薯WRKY转录因子保守结构域的变异情况Table 1 Variation of conservative domain in Ipomoea batatas WRKY transcription factors
2.3 甘薯WRKY转录因子的系统进化分析
为了对甘薯WRKY蛋白进行更为准确的分类,引入拟南芥AtWRKY蛋白参与系统进化树的构建。由于WRKY转录因子Ⅰ类亚家族拥有2个WRKY保守结构域,将蛋白N端的WRKY结构域和C端的WRKY结构域分别独立构建系统进化树,若存在缺失某个WRKY结构域的情况,则该WRKY蛋白只保留另一端的WRKY序列参与进化树构建。甘薯和拟南芥的Ⅰ类WRKY转录因子保守域进化树如图4所示。

蛋白名称+C/N代表该蛋白的C/N端WRKY结构域。节点处的数字代表换算成百分数的自助抽样自展值(例如最右上方的72代表在1 000次的自助抽样建树中,IbWRKY62C和IbWRKY44C大约720次为邻位,四舍五入为72%)。图5和图6同The protein name+C/N indicates the WRKY domain in the C/N-terminal domain of the protein.The number at the node denotes the bootstrap value in percentage (for example,the 72 at the top right means there are about 720 out of times,rounding to 72%,that the IbWRKY62C and IbWRKY44C are the nearest neighbors during the bootstrap sampling for phylogenetic tree construction).The same for Fig.5 and Fig.6
由图4可知,Ⅰ类WRKY家族蛋白的N端与C端WRKY保守结构域序列在进化上大部分距离较远,大部分蛋白呈现出按物种少量聚集的现象,即包含5~7个节点的甘薯WRKY分支与拟南芥WRKY分支相邻。
由图5可以看出,Ⅱ-a、Ⅱ-b和Ⅱ-c这3个亚类蛋白属于同一分支,进化关系较近,而Ⅱ-e亚类WRKY蛋白与其他4类蛋白的进化关系较远。由图5还可以看出,甘薯和拟南芥WRKY蛋白在几个亚类内的位置一致,大部分分支中的拟南芥WRKY蛋白和甘薯WRKY蛋白互相交错排列。
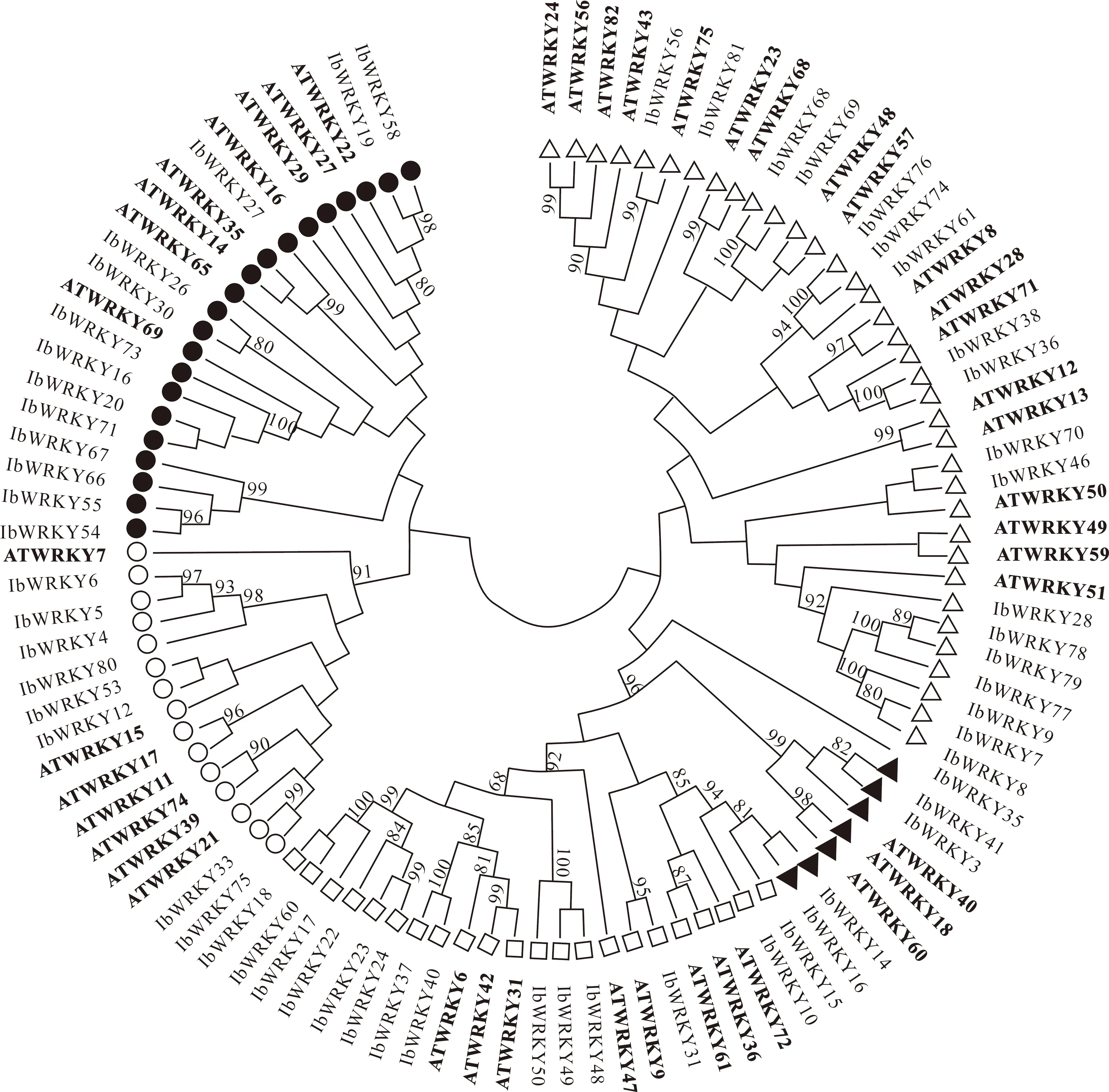
用实心圆、空心圆、空心正方形、实心三角形、空心三角形Ⅱ类甘薯与拟南芥WRKY家族蛋白分别标记Ⅱ-e、Ⅱ-d、Ⅱ-b、Ⅱ-a和Ⅱ-c亚类。加粗字体标记甘薯WRKY家族蛋白For subfamily-Ⅱ WRKY proteins,the solid circle,hollow circle,hollow square,solid triangle,hollow triangle indicate Ⅱ-e,Ⅱ-d,Ⅱ-b,Ⅱ-a and Ⅱ-c subgroups,respectively.The blod font indicates proteins in I.batatas WRKY family
Ⅱ-c亚类的WRKY蛋白数量最多,与Ⅱ-a亚类的蛋白从同一分支进化而来,进化关系较近,但Ⅱ-c亚类的WRKY蛋白与Ⅱ-e亚类的进化关系最远。另外还存在一些只有拟南芥WRKY或甘薯WRKY的分支,例如Ⅱ-b亚类的IbWRKY18所在的进化树分支只包含甘薯WRKY转录因子。Ⅱ-a亚类的WRKY蛋白数量最少,且不统一在一个进化树分支内,IbWRKY16和IbWRKY14的亲缘关系与Ⅱ-b亚类较近。
甘薯WRKY家族有5个蛋白属于Ⅲ类,相比拟南芥(13个)较少。从进化树(图6)上看,甘薯和拟南芥WRKY蛋白整体上分为2个进化树分支,均有甘薯WRKY分布在内,其中IbWRKY21和IbWRKY29亲缘关系较近。

图6 甘薯和拟南芥的Ⅲ类WRKY转录因子进化树Fig.6 Phylogenetic tree of subfamily-Ⅲ WRKY transcription factors of I. batatas and A. thaliana
2.4 甘薯WRKY转录因子保守基序分析
应用MEME对甘薯82个WRKY蛋白进行基序分析,每亚类内按照蛋白序列长度排序,结果见表2和图7。由图7结果可以看出,每个亚类内甘薯WRKY家族蛋白的基序比较一致,而亚类间则存在一定的差异性。82个甘薯WRKY蛋白中有70个蛋白内部存在基序1-基序4-基序2的基序串,占全部蛋白数量的85.4%。Ⅰ类WRKY蛋白的N端大多数都有基序7和基序3,其中基序3为Ⅰ类WRKY家族特有的基序。基序7除了存在于I类蛋白的N端WRKY结构域中,也存在于一些蛋白的C端结构域,如IbWRKY43和IbWRKY51等。Ⅱ-a亚类WRKY蛋白的基序与Ⅱ-b亚类较为相似。Ⅱ-b亚类WRKY蛋白的典型基序串为基序1-基序4-基序2-基序6。Ⅱ-c亚类的蛋白均具有典型的WRKY结构域,个别蛋白还含有基序5;Ⅱ-d亚类蛋白均有基序9(除IbWRKY53外)。Ⅱ-e亚类蛋白的保守基序与Ⅱ-d亚类相似。Ⅲ类WRKY蛋白的基序数量较少,而Ⅰ类和Ⅱ-b亚类WRKY蛋白的基序种类较为丰富。由图7和表2可知,10个基序的长度为8~57个氨基酸,其中2个属于WRKY保守结构域,分别为基序1和基序7;2个属于锌指结构,分别为基序2和基序3。基序7-基序3形式的基序串只出现在Ⅰ类WRKY蛋白中,是N端WRKY结构域的保守基序;基序4是最短的基序,存在于WRKY七肽结构域与锌指结构域之间。

表2 甘薯WRKY转录因子的保守基序长度及其氨基酸序列Table 2 Length and amino acid sequence of motifs for the WRKY transcription factors in I. batatas

左侧罗马数字为甘薯WRKY转录因子所属的亚类,最下方横轴为蛋白序列长度轴Roman numbers in the left side represent the subgroups of I. batatas WRKY transcription factors and the lowest horizontal axis indicates length of protein sequences
2.5 甘薯WRKY转录因子在蔓割病菌侵染下的差异表达分析
笔者之前用蔓割病强致病菌株F07与非致病菌株F04对甘薯高抗品种JS57(金山57)与高感品种XZH(新种花)进行侵染,以茎段作为材料进行转录组测序[16]。本研究利用该转录组数据,经过基因表达差异显著性分析,筛选到19个存在显著差异的甘薯WRKY基因(图8)。由图8可知,存在差异表达的WRKY基因类型丰富,分别为Ⅰ类3个、Ⅱ-a亚类2个、Ⅱ-b亚类4个、Ⅱ-c亚类7个、Ⅱ-e亚类2个、Ⅲ类1个。其中Ⅱ-c亚类的WRKY基因最多,Ⅱ-a亚类的IbWRKY14和IbWRKY16在进化树上属于同一分支,在XZH_F07中均为下调表达。Ⅱ-e亚类的IbWRKY67和IbWRKY66进化关系近,但在XZH_F07中分别为下调和上调表达。
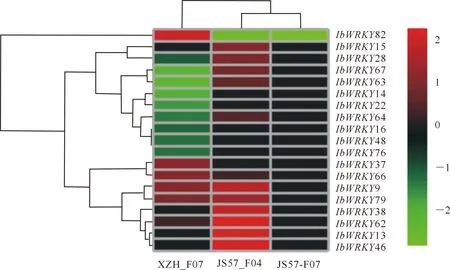
XZH_F07代表甘薯品种XZH受F07侵染的试验组,JS57_F04代表甘薯品种JS57受F04侵染的试验组,JS57_F07代表甘薯品种JS57受F07侵染的试验组。热图左侧部分为Pheatmap软件根据各基因在不同处理中的以2为底差异表达倍数的对数值以欧式距离建立的聚类关系,热图上部为不同处理中包含的所有基因以2为底差异表达倍数的对数值以欧式距离建立的聚类关系,右侧色卡代表相应的以2为底差异表达倍数的对数值。图9同The XZH_F07 indicates the test group of I.batatas cultivar XZH infected by F07.The JS57_F04 indicates the test group of I. batatas cultivar JS57 infected by F04.The JS57_F07 indicates the test group of I. batatas cultivar JS57 infected by F07.The left part of the heat map shows the clustering relationship among the different genes with regard to the log2FoldChanges based on the Euclidean distance according to the Pheatmap.The top part of the heat map shows the clustering relationship among the different treatments in terms of the comprised genes with regard to the log2FoldChanges based on the Euclidean distance.The color card in the right side indicates the corresponding log2FoldChange values.The lines in the left and up sides indicate the data clustering and thecolor card in the right side indicates the corresponding log2 FoldChange values.The same for Fig.9图8 甘薯WRKY基因在甘薯蔓割病菌胁迫下的差异性表达Fig.8 Differential expression of WRKY genes in I. batatas under the stress of Fusarium oxysporum f. sp.batatas
对转录组数据进行分析表明,高感品种XZH在强致病菌株F07侵染后,许多WRKY基因出现了较为明显的上调或下调变化;而高抗品种JS57在F07侵染条件下,只有IbWRKY82基因表达显著下调(以2为底差异表达倍数的对数为-2.83)。JS57_F04中大部分存在差异表达的WRKY基因为上调。在XZH_F07中下调表达的基因在JS57_F04中为上调表达,如IbWRKY67、IbWRKY63和IbWRKY64;或差异未达显著性,如IbWRKY76。IbWRKY82在JS57受到非致病菌株F04侵染后下调表达(以2为底差异表达倍数的对数为-2.63),但在XZH_F07试验组内为上调表达(以2为底差异表达倍数的对数为2.05)。
2.6 甘薯WRKY转录因子在块根储藏期低温条件下的差异表达分析
低温储存是延长甘薯货架期的重要方式,但在较低温度条件下,会导致源于热带的甘薯受到冷害。Ji等[32]对低温敏感甘薯品种Xushu 15-4和对低温耐受性较好的Xushu 15-1,分别设置4 ℃条件贮存2周或6周,进行甘薯块根储藏期低温试验,分别测得2个品种的转录组数据。以该转录组数据为基础,分析甘薯在低温储藏过程中WRKY基因的差异表达情况,结果如图9所示。由图9可知,82个甘薯WRKY基因有34个存在表达差异。其中Ⅰ类WRKY基因最多,达到13个;其余分别为Ⅱ-a亚类1个,Ⅱ-b亚类3个,Ⅱ-c亚类8个,Ⅱ-d亚类4个,Ⅱ-e亚类3个,Ⅲ类2个。4个处理中均存在WRKY基因表达上调或下调的情况,以2为底差异表达倍数的对数值为-6.42~8.60;下调表达的WRKY基因数量(18个)略多于上调表达的基因数(16个)。

Xushu 15-1_6w代表甘薯品种Xushu 15-1冷藏6周的试验组,Xushu 15-4_2w代表甘薯品种Xushu 15-4冷藏2周的试验组,Xushu 15-1_2w代表甘薯品种Xushu 15-1冷藏2周的试验组,Xushu 15-4_6w代表甘薯品种Xushu 15-4冷藏6周的试验组The Xushu 15-1_6w indicates the test group of I. batatas cultivar Xushu 15-1 stored at low temperature for 6 weeks.The Xushu 15-4_2w indicates the test group of I. batatas cultivar Xushu 15-4 stored at low temperature for 2 weeks.The Xushu 15-1_2w indicates the test group of I. batatas cultivar Xushu 15-1 stored at low temperature for 2 weeks.The Xushu 15-4_6w indicates the test group of I. batatas cultivar Xushu 15-4 stored at low temperature for 6 weeks图9 甘薯WRKY基因在甘薯低温胁迫下的差异性表达Fig.9 Differential expression of WRKY genes in I. batatas under low temperature stress
不同甘薯WRKY基因在冷藏2周、6周和整个冷藏过程中的差异表达情况不同,仅在冷藏2周、6周和冷藏全期存在差异表达的WRKY基因数分别有7,5和21个。有些WRKY基因在2个甘薯品种块根中冷藏全期存在表达差异,可以观察到在6周时,Xushu 15-4块根中WRKY基因差异表达的幅度相比冷藏2周时更低,而甘薯Xushu 15-1仍保持着较高的差异水平。有些基因只在Xushu 15-1块根中存在差异表达,而在Xushu 15-2中冷藏全期均不存在差异表达,如IbWRKY33、IbWRKY63和IbWRKY64等。由此可见,不同低温敏感度的甘薯品系间存在表达差异的WRKY基因,其可能参与甘薯对低温胁迫的响应。
2.7 甘薯SPF1蛋白与IbWRKY蛋白的比较
1994年,Ishiguro等[14]从甘薯高系14(Kokei no.14)中克隆出第1个WRKY基因,取名为SPF1。本研究从NCBI下载SPF1的氨基酸序列,在建立的WRKY蛋白数据库内检索,结果得到4个相似蛋白,分别为IbWRKY32、IbWRKY44、IbWRKY63和IbWRKY64,这4个蛋白的氨基酸序列与SPF1的氨基酸序列一致度分别为85.4%,52.0%,50.5%和38.4%。IbWRKY32与SPF1的一致度最高,位于第7号染色体上的第9 504 956~9 514 219 位核苷酸处。SPF1与IbWRKY32的氨基酸个数分别为549和582个,在SPF1蛋白的第29~549区域内,IbWRKY32与SPF1的氨基酸序列完全一致(一致度为100%),占SPF1蛋白序列全长的95%(图10)。不同之处在于,IbWRKY32的N端氨基酸比SPF1少而C端氨基酸比SPF1多,这种差异可能与甘薯品种差异或基因组测序质量有关。

灰色标记为WRKY保守结构域Conservative WRKY domains were highlighted with gray color图10 甘薯IbWRKY32与SPF1的氨基酸序列比对Fig.10 Alignment of amino acid sequences of I. batatas IbWRKY32 and SPF1
3 讨 论
WRKY转录因子家族作为重要的一类转录因子,参与植物生长发育过程并响应生物胁迫以及非生物胁迫。甘薯是我国重要的粮食作物和淀粉加工来源,其基因组序列已测定但基因尚未注释,因而无法直接研究甘薯WRKY转录因子。本研究使用隐马尔可夫模型,在甘薯全基因组中预测和鉴定甘薯WRKY转录因子基因并进行一系列分析,包括染色体定位、基因潜在复制关系、保守结构域、进化树构建以及保守基序分析等,同时结合Lin等[16]和Ji等[32]的甘薯RNA转录组数据分析,得到差异表达的WRKY基因,获得可能参与抗蔓割病机制和抗低温胁迫机制的WRKY基因。
本研究从甘薯全基因组筛选并鉴定到82个甘薯WRKY转录因子家族成员,分为Ⅰ、Ⅱ、Ⅲ 3个大类,其中Ⅱ类转录因子分为5个亚类。从蛋白数量上看,Ⅱ-c亚类的蛋白最多,占全部蛋白的24.4%;其次是Ⅰ类,占全部蛋白的22.0%。在其他植物的WRKY转录因子分类中,也是Ⅰ类和Ⅱ-c亚类的数量较多,例如草莓(Fragaria×ananassaDuch.)的WRKY蛋白数量为47[35],其中Ⅰ类和Ⅱ-c亚类的数量均为10;拟南芥的WRKY蛋白数量为72[2],其中Ⅰ类和Ⅱ-c亚类的数量分别为13和19。甘薯的WRKY转录因子中,发生WRKYGKK变异的8个蛋白都在Ⅱ-c亚类,与苹果[36](MalusdomesticaBorkh.)的一致。从系统进化树的位置看(图5),这8个蛋白中除了IbWRKY46外,其余7个蛋白均为进化树的同一分支,其中IbWRKY78、IbWRKY77和IbWRKY79这3个基因在甘薯第14号染色体内存在潜在复制基因对(图1),基序的种类和排列顺序也一致(图7);推测甘薯的部分WRKY基因在进化过程中发生了变异和复制,造成WRKY结构域七肽变异也高度相似。有研究结果表明,转录因子家族的进化和扩张得益于这样的基因复制事件[37]。
甘薯蔓割病作为一种真菌病害,多在苗期发病,病株蒂部常发生腐烂,严重影响甘薯的生长和产量。JS57(金山57)是蔓割病高抗品种,在蔓割病菌株F04和F07的侵染下,JS57_F07的试验结果显示,甘薯JS57的WRKY基因发生差异表达的数量很少,相比之下JS57_F04受影响程度较大,大部分差异表达基因呈现上调表达。甘薯起源于热带地区[32],适应性广,在我国种植面积大,在较冷的北方地区也有广泛种植。因此,探索影响甘薯抗低温机制相关的WRKY基因具有重要的实践价值。在2个耐冷性不同的甘薯品种的低温贮藏试验中,甘薯WRKY基因在低温贮藏2周和6周时的表达存在差异,表明甘薯块根在低温条件下贮藏前期与后期的抗低温胁迫机制可能存在差异。
SPF1来自于甘薯品种高系14,是WRKY转录因子家族报道的第一个基因[14]。在82个WRKY转录因子中进行检索后发现,IbWRKY32与SPF1的蛋白序列一致度达到85.4%,WRKY保守结构域完全一致,定位于甘薯的第7条染色体上。根据进化树(图4)发现,IbWRKY32与AtWRKY33的N端WRKY保守结构域进化关系较近,AtWRKY33在过量表达的情况下可以增强拟南芥对NaCl胁迫的耐受性[38]。在甘薯块根贮藏期的冷胁迫下,IbWRKY32在耐冷性不同的2个甘薯品种中均上调表达,以2为底差异表达倍数的对数值在Xushu 15-1_2w、Xushu 15-1_6w、Xushu 15-4_2w和Xushu 15-4_6w试验组中分别为1.42,2.78,1.00和2.00,可看出基因在冷胁迫6周时上调表达的幅度增大,表明IbWRKY32可能参与增强甘薯块根耐冷过程。
本研究应用隐马尔可夫方法从甘薯全基因组中鉴定得到82个WRKY基因,并分析其染色体定位、蛋白保守结构域、系统进化以及蔓割病胁迫与低温胁迫条件下的差异表达,为甘薯WRKY转录因子的机制研究及其实践应用奠定了基础。

