液相芯片技术在花生品种纯度鉴定中的应用
郝明国 张 君 陈光辉 杨松楠 陈亮宇 袁 美 梁嘉宁韩 笑*
(1.吉林农业大学农学院,吉林 长春 130118;2.山东省花生研究所,山东 青岛 266100)
花生是世界范围内广泛栽培和利用的油料和经济作物,是重要的植物油脂和蛋白质来源。我国拥有全世界最大的花生加工产业,具有举足轻重的地位。随着中国花生产业的不断壮大,花生的消费需求逐年递增,为适应国际和国内市场需求的变化,中国花生育种无论在育种技术还是育种目标方面都面临着新的挑战[1-3]。
目前,我国的花生育种工作正处于对相关性状遗传机理研究和产量、抗性、品质等育种目标的提升阶段[4]。优良的花生品种对产量和品质的改善均居重要地位。在国家标准GB4407.2-2008(现行)中,种子纯度是用来判定种子优劣的主要指标之一。做好种子纯度检验工作是维护种子品质、减少制种过程遗传漂变、减缓品种经济性状衰退、确保作物增产增收的重要前提及保障。
种子纯度的鉴定方法由最初传统的田间鉴定法逐步发展成利用蛋白质多态性的蛋白质电泳鉴定法,但是该技术稳定性和准确性仍不能满足更深入的研究。由于种子纯度鉴定具有重要性和迫切性的特点,纯度鉴定新方法在不断突破和更新,随着分子生物学实验技术的发展,如PCR(Polymerase Chain Reaction)技术的出现,品种鉴定进入了DNA 分子水平。以RFLP(Restricted Fragment Length Polymorphism)、RAPD(Rampid Amplification Polymorphism DNA)、AFLP(Amplified Fragment Length Polymorphism)、SSR(Simple Sequence Repeat)、SNP(Single Nucleotide Polymorphism)等为代表的分子标记技术成功应用于花生品种鉴定[5-9]。作为第三代分子标记,SNP标记被认为是最有前景的分子标记,具有高密度、高遗传稳定性和易实现计算机大规模、自动化分析等特点[10]。高通量测序是大量获得SNP的主要方式,目前主流的二代测序技术正逐步过渡为三代测序技术。全基因组重测序法是检测SNP最直接、最准确的方法,被认为是“金标准”[11]。基因芯片和靶向测序(分为多重PCR 和液相芯片两条技术路线)等技术作为检测SNP的不同发展方向各有优势,也为花生品种鉴定带来了新机遇。
液相芯片(液相杂交捕获测序),是利用碱基互补配对原理,将设计好的核酸探针与目标区域结合,富集目标区域DNA,在主流测序平台进行高通量测序的一项技术。该技术可应用于不同植物,进行种质资源鉴定、遗传多样性分析、全基因组关联分析(Genome-wide Association Study,GWAS)、功能基因/QTL(Quantitative trait locus)定位、分子设计育种等相关研究[12-18]。目前尚未见液相芯片技术应用于花生品种纯度鉴定方面的报道。本研究使用花生10K 液相芯片技术结合基于Illumina测序平台的高通量测序,获得目标区域SNP,使用R语言、PLINK、RMEGA-X等软件分析SNP位点信息,分析了100份东北王花生品种的纯度,并与传统鉴定方法进行比较,以期为花生育种者提供新的品种鉴定思路和研究方法。
1 材料与方法
1.1 供试材料
东北王、豫花9327等花生品种由吉林农业大学提供,种植于长春市农业农村部大豆区域技术创新中心。
1.2 DNA 提取与花生10K 液相芯片检测
田间取100株花生样品幼叶,使用植物基因组DNA提取试剂盒(DP305,北京天根生化有限公司)提取叶片DNA。超微量紫外分光光度计(QUAWELL Q5000)结合1%琼脂糖电泳验证DNA品质。花生10K液相芯片和高通量测序由北京伊鲁生物科技有限公司(下文简称公司)完成。
1.3 基因分型数据与SNP分布图绘制
公司交付压缩后大小约95 Gb的压缩clean data,430 Mb 的vcf(variant call format)文件以及SNP数据(大小4.74 Mb,格式为Microsoft Excel)。累计100万个SNP位点信息绝大多数以二等位基因形式表示。为了获得SNP 在花生参考基因组(Tifrunner.gnm1.KYV3)及染色体上的分布情况,用R(v4.0.4)加载R包Cmplot,绘制SNP分布图。
1.4 基因分型结果的验证
随机选择7 个SNP 位点,在PeanutBase网站(http://www.peanutbase.org)下载位点前后各300 bp的序列信息。用primer premier5.0设计7对引物(编号H1~H7,表1),委托吉林省库美生物科技有限公司进行引物合成。以东北王和豫花9327的DNA 为模板,进行PCR 扩增,PCR扩增产物用1%琼脂糖电泳检测。挑选扩增效果好的两对引物,随机选择16 份花生样本进行PCR,PCR产物测序委托库美公司完成。

表1 引物信息Table 1 Primer information
对PCR产物测序结果,使用Chromas(v2.6.5)查看测序品质。所选取序列依次导入MEGA-X(v10.2.2)进行多序列比对找到SNP位点,与公司交付的SNP数据对比,以验证SNP位点的准确性。
1.5 SNP品质控制及数据处理
SNP 数据需要品质控制(Quality Control,QC)才能后续分析。本研究使用PLINK(v1.90)进行品质控制操作。PLINK 是一个免费、开源的全基因组关联分析工具集,版本1.9以上可用于非模式生物的基因组数据分析[19]。品质控制后的SNP数据便可借助PLINK 进行数据格式转换、主成分分析、计算IBS(Identical By State)并通过R 包进行多维标度(MDS)可视化、亲缘关系(Identity By Descent,IBD)检测等相关操作。
将PLINK转换的vcf格式文件使用vcf2phylipmaster(v2.4)转换成phy格式文件。然后用MEGAX将phy格式文件转换成meg格式文件,用MEGAX打开meg格式文件,进行聚类分析[20]。
1.6 表型鉴定
田间调查100株花生的主茎高、侧枝长、分枝数等性状。收获后进行考种,包括单株荚果质量、荚果网纹深浅、籽仁形状及种皮颜色等性状。花生品质包括油酸、亚油酸、蛋白质和含油量等指标使用傅里叶变换红外光谱仪测定(仪器型号:MPA;测试地点为山东省花生研究所)。
2 结果与分析
2.1 基因分型结果
基于SNP数据绘制SNP在花生染色体上的分布图(图1),结果显示分布在各组染色体上的SNP数量平均约为500个,表现为染色体两端密集,中间稀疏的特点。SNP 标记数量众多,在目标区域呈现高密度广泛平均分布的特点,是一种理想的分子标记。

图1 SNP在染色体上的数量及分布情况(滑动窗口大小4 Mb)Fig.1 The number of SNPs on chromosome within 4 Mb window size
2.2 基因分型结果准确性验证
图2可知,在7对引物中,引物H2、H3效果较好。使用H2、H3对16份样本进行PCR扩增产物测序,得到理想的测序峰图(图3)。MEGA-X多序列比对的部分结果显示,设计验证的SNP位点在无星号(*)位置上。将此处碱基序列和公司交付的SNP数据进行比较,两者完全一致,证明了液相芯片技术基因分型结果的高准确性(图4)。
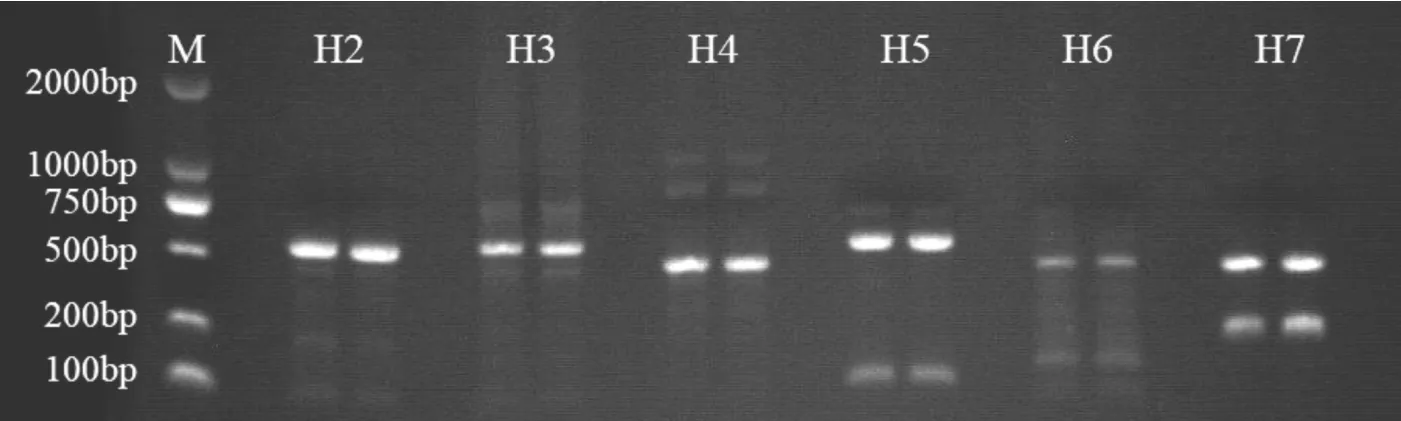
图2 引物筛选Fig.2 Screening of primers
图3 PCR 产物的测序品质Fig.3 Sequence quality of PCR products
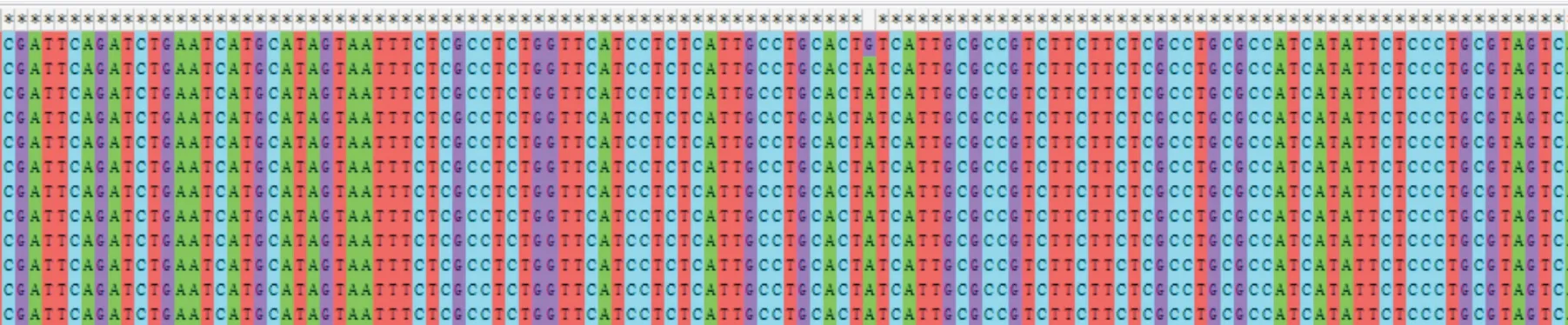
图4 多序列对比部分结果Fig.4 Alignment of multiple sequences
2.3 SNP品质控制及纯度分析
PLINK 品质控制过程中删除了8个样本和2366个SNP数据,剩余7634个SNP和92个样本通过过滤器和品质控制,数据用于进一步分析。使用PLINK进行主成分分析(Principal components analysis,以下简称PCA),分析结果用R包scatterplot3d(v0.3-41)绘图展示(图5)。从三维视图来看,91份东北王样品很容易与豫花9327区分。育种者可通过样本在三维空间的距离直观地判定品种纯度。
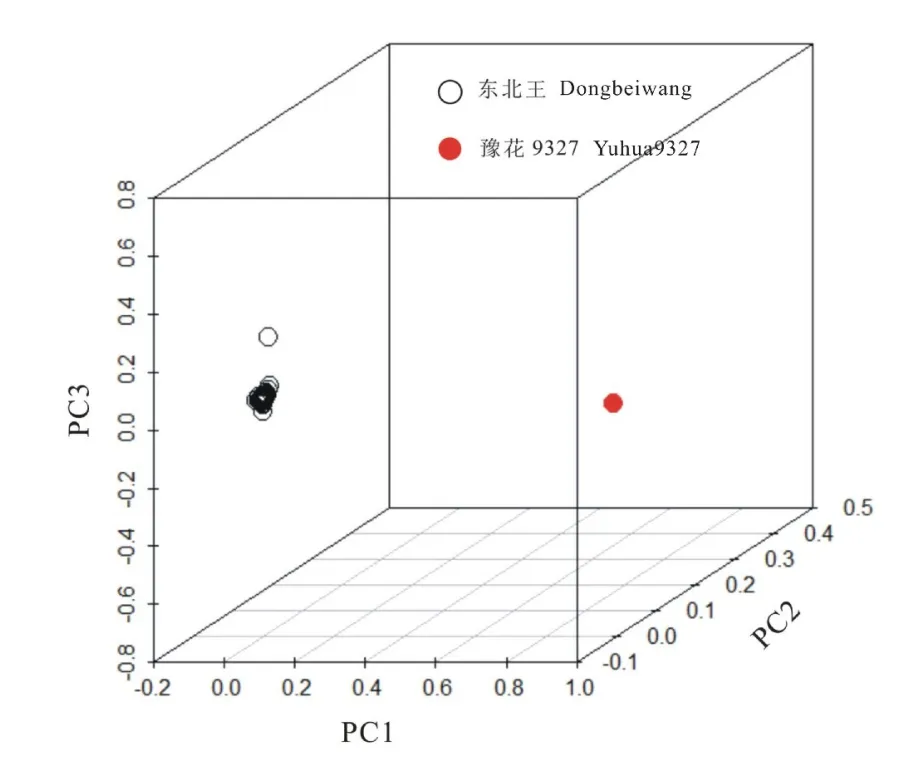
图5 主成分分析Fig.5 Principal component analysis
多维标度法(Multidimensional Scaling,MDS)是一种经典的数据降维方法。它要求原始空间中样本之间的距离在低维空间中保持,即当仅能获得样本之间的相似性矩阵时,如何由此来重构它们的欧几里德坐标。使用PLINK 计算IBS距离矩阵,通过R语言包实现MDS可视化。这种方法计算样本中任何一对样本之间共享等位基因的全基因组平均比例,从而为每个样本生成遗传变异的定量指数(成分),以探索样本在基因层面上的相似程度[21]。多维标度法对SNP数据分析显示,91份东北王样品和豫花9327分布在二维平面的两个不同位置,东北王样品的纯度为98.9%,说明液体芯片技术可以高效用于品种纯度鉴定(图6)。PCA 和MDS两种方法的算法不同,但其本质都是通过数据降维,实现数据可视化。遗传学中描述等位基因的同源关系时,会有状态同源(IBS)和血缘同源(IBD)两个概念[22]。使用PLINK 进行样本间的亲缘关系(IBD)估计,将任何1个样本与其他91个样本进行比较,根据排列组合公式,共有C(92,2)=4186种组合。用记事本打开PLINK 生成的*.genome格式文件,除去表头外,共有4186行,查看RT(Relationship type inferred from .fam/.ped file)列,根据RT列的数值(表2)可判断个体间的亲缘关系。若样本包含亲缘关系,可使用Z值(Z0=P(IBD=0),Z1=P(IBD=1))可视化这些亲子关系。由图7可看出,91份东北王两两之间的关系为“其他”;91份东北王和豫花9327之间的关系为“不相关个体”。92份样本之间不含亲子代关系。
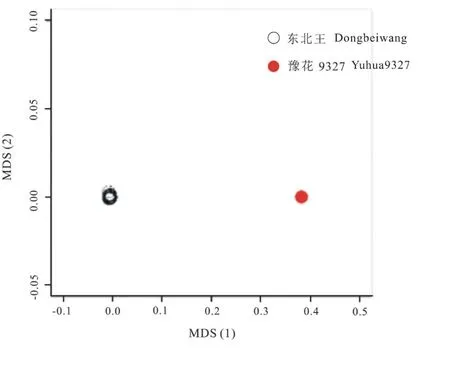
图6 多维标度法分析Fig.6 Multidimensional scaling analysis

表2 RT 列取值含义Table 2 Meaning of RT column value
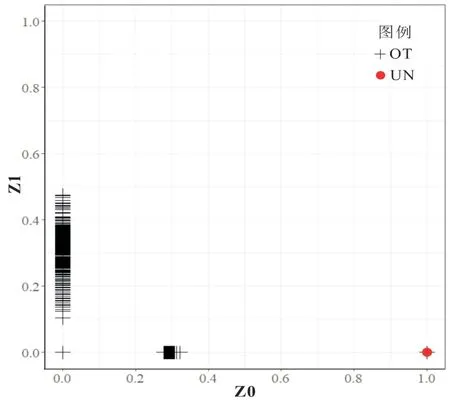
图7 Z值可视化图Fig.7 Visualizationof Z value
2.4 基于聚类分析的品种纯度鉴定
基于7634个SNP 标记,对92份花生样本进行聚类分析。由图8可知,92份样本被分为2类。类型Ⅰ包含91份,彼此之间标尺为0,表明这91份样本为同一个花生品种;类型Ⅱ包含1份(编号100),与类型Ⅰ之间标尺为1.60,表明两者为不同品种。聚类分析可把东北王和豫花9327两个品种完全分开,这与基于降维分析法(主成分分析法和多维标度法)的结果完全一致。

图8 92份花生样品的聚类分析图Fig.8 Cluster analysis of 92 peanut individuals
2.5 基于表型的纯度鉴定
东北王和豫花9327两个花生品种在植株形态上差异不大(图9)。主茎高、分枝数等植物学性状无明显差异。单株荚果质量、荚果网纹深浅、种子油酸、亚油酸、蛋白质含量等性状有差异(数据未列出),但差异不显著,判断标准存在一定主观因素且难以界定。东北王荚果为串珠形,籽仁种皮深红色,而豫花9327荚果为普通形,种皮浅红色,因此荚果形状和种皮颜色是区分这两个品种的鉴别性状。据此二性状,可将东北王和1株豫花9327区分开来,表型鉴定结果与SNP 数据分析结果完全一致。

图9 两个花生品种的植株和荚果性状比较Fig.9 Traits comparison on plant and pod between two peanut cultivars
3 结论与讨论
本研究通过10K 花生液相芯片技术结合高通量测序技术共分析100份东北王花生样品,获得目标区域内1万个SNP位点,使用计算机软件进行大规模、自动化分析,并将分析结果可视化,三种分析方法(PCA、MDS和聚类分析)均成功地将1份豫花9327与91份东北王两个品种完全分开,与表型鉴定结果完全一致。与使用少数几个SSR 标记分析相比,上万个SNP位点(图1)对鉴定结果准确性的提高是显而易见的。SNP 标记的处理分析主要依靠计算机,可以把育种者从SSR 繁复的操作步骤解放出来,节省人力成本和时间成本。液相芯片技术虽然达不到全基因组重测序99%的基因组覆盖度,但测序成本可以降低80%甚至更高。
目前花生10K 液相芯片的使用价格是170元/样本,使用成本相对高昂。对于花生育种者来说,液相芯片用来鉴定品种纯度未来可期。当下,液相芯片鉴定杂交种纯度是一个比较好的过渡选择。获得亲本SNP数据后,从中挑选1~2个在父母本具有二态性的SNP(如本研究中东北王和豫花9327之间有3518个二态性SNP),通过PCR产物直接测序法[9]进行杂交种的鉴定。除了首次使用液相芯片成本较高,后续每年使用PCR 产物测序法的成本约32元/样本。这些亲本SNP数据除了应用于杂种F1纯度鉴定,还可用于构建花生种质资源的标准DNA指纹库[24],为新品种保护和亲本选配提供理论依据。

