MIXTA/MIXTA-like基因特征及其对植物表皮细胞分化的调控
周芳伟,吴怀通,尹佟明
(教育部林木遗传与生物技术重点实验室,江苏省林木遗传与高效培育重点实验室,南京林业大学林学院,南方现代林业协同创新中心,江苏 南京 210037)
MYB (V-myb avian myeloblastosis viral oncogene homolog)转录因子是植物中最大的转录因子家族之一,调控表皮毛发育、根毛密度、花和种子发育,参与次生代谢以及对生物和非生物胁迫反应[1-2],与bHLH转录因子和WD40蛋白结合形成MBW转录激活复合物启动下游基因的表达。根据保守结构域的重复次数,MYB 家族分为4个亚家族,其中R2R3 MYB是最大的亚家族[3-4],也是目前功能研究最为广泛的一类MYB转录因子,通常在N端包含2个不完全的MYB重复序列,在C端包含转录激活或抑制域[5]。R2R3 MYB转录因子在拟南芥(Arabidopsisthaliana)中有125个基因成员,依据C端序列差异分为25个亚组,每个亚组有较为独立的特殊功能,例如第6亚组基因参与花青素的生物合成、第22亚组成员在非生物胁迫响应过程发挥重要作用、第21亚组成员广泛参与腋芽分生组织调节。其中,MIXTA/MIXTA-like基因属于第9亚组[3,5-7]。
MIXTA/MIXTA-like基因主要参与表皮细胞分化,第1个MIXTA基因是从金鱼草(Antirrhinummajus)突变体中鉴定出来的,命名为AmMIXTA,是金鱼草花瓣表皮细胞分化为锥形细胞的关键调控因子[8]。后续还鉴定出3个与MIXTA基因功能相似的同源基因分别是AmMYBML1(MYBMIXTA-like1)、AmMYBML2和AmMYBML3[9-10],都在金鱼草花瓣表皮细胞分化过程中发挥重要作用,它们的功能相似或相同但并不冗余[10]。近几年,越来越多的研究报道了MIXTA/MIXTA-like基因在不同植物中参与调控表皮细胞锥形化,同时对角质层及表皮蜡质合成、表皮毛发育也起到调控作用,这类基因被证实是参与植物表皮细胞分化的主要调控因子。
植物表皮细胞分化后主要形成锥形化细胞、角质层以及多种形态的表皮毛等植物表皮附着物。锥形化细胞是表皮细胞分化形成的一种高于扁平细胞的凸起状锥形细胞[8,11-12],其中花瓣表皮细胞的锥形化在自然界中广泛存在[13],且不同植物之间的细胞大小,圆锥形的角度和高度也存在显著差异。圆锥形细胞与花瓣的颜色、对光的捕获和反射、气味的产生、润湿性以及传粉媒介在花表面的附着力有关[13-15]。
许多陆地植物组织器官的表面有角质层,是抵抗生物和非生物胁迫的第1道屏障[16-17]。植物角质层的保护功能取决于其组成成分的物理化学性质。植物角质层通常由2个主要成分组成:角质支架以及围绕角质支架的表皮蜡[18]。角质层蜡质是减少非气孔失水的主要屏障之一,对植物耐旱性发挥重要作用[17-18]。目前在拟南芥、番茄(Solanumlycopersicum)、洋桔梗(Eustomagrandiflorum)和地钱(Marchantiapolymorpha)等多个植物种中均发现MIXTA/MIXTA-like基因对角质层及表皮蜡质合成具有调控作用。
表皮毛是植物表皮细胞凸起、分化后附着在植物表面的一种特殊结构。表皮毛的存在加强了对表皮的保护作用,可以减少食草动物和取食昆虫的侵害[19-21],还可以减少蒸腾,有利于植物生长[22-23]。在拟南芥、番茄、黄花蒿(Artemisiaannua)、黄瓜(Cucumissativus)及杨树(Populus)等多个植物种中均发现MIXTA/MIXTA-like基因参与表皮毛的发育调控[20-23]。种毛是一种特殊的表皮毛,是附属在植物种子表面的毛状纤维,起到辅助种子扩散和传播的作用。如棉花(Gossypiumhirsuta)的棉纤维发育起始于胚珠表皮细胞,由单细胞分化而来。目前棉纤维发育已得到较为深入的研究[24-25],棉纤维细胞的起始分化期决定了有多少胚珠表皮细胞发育成棉纤维[26-27],因此阐明调控棉纤维起始分化的关键基因GhMML(GossypiumhirsuteMYBMIXTA-Like)[28],对提高棉花产量有重要意义,也为其他植物的种毛发育研究提供参考[29-31],特别是对杨树、柳树(Salixspp.)和法国梧桐(Platanusorientalis)等的飞絮治理有重要意义。
研究发现,MIXTA/MIXTA-like基因在调控表皮细胞分化过程中发挥重要作用[32],不同植物中的MIXTA/MIXTA-like基因数目不同,但MIXTA/MIXTA-like基因在结构和表达模式上有较高的相似性,因此,MIXTA/MIXTA-like基因是鉴定表皮细胞分化潜在遗传机制的重要候选基因。了解MIXTA/MIXTA-like基因的特征及其对植物表皮细胞分化调控的研究进展,对于阐明木本植物表皮细胞分化的分子调控,尤其是对加速林木飞絮污染防治基础研究,具有重要意义。
1 MIXTA/MIXTA-like基因的序列特征
MIXTA/MIXTA-like基因属于MYB转录因子家族中的一类序列和功能保守的亚家族基因,有典型的MYB转录因子序列特征。MYB转录因子通常在N端含有高度保守的MYB结构域,该结构域是一段51~52个氨基酸的肽段,包含4个不完整的氨基酸重复序列(R),每个重复序列在三维空间中构成 3 个α-螺旋,第2个和第3个螺旋以3个色氨酸残基为疏水核心构成“螺旋—转角—螺旋(HTH)”结构,MYB 转录因子通过该结构与 DNA 结合[33-34]。MYB结构域根据碱基数目和色氨酸保守性上存在的差异分为R1、R2和R3 这3种类型,根据结构域含有的R重复片段数将MYB超家族成员分为 1R-MYB (MYB-related proteins)、2R-MYB(R2R3)、3R-MYB(R1R2R3)和 4R-MYB(R1R2R2R1/2-MYB) 4个亚家族,R2R3 MYB亚家族是植物中最丰富的类型[3,33]。在拟南芥中已发现大约125个R2R3 MYB成员,这些成员在系统发育树上被聚成25个亚组[34]。
R2R3 MYB转录因子第9亚组(subgroup 9,SBG9)的基因除N端2个高度保守的R2R3 MYB结构域外,在C端还存在1个高度保守的第9亚组特征基序(SBG9基序)[3],这种高度保守的SBG9基序一般由10~22个氨基酸组成(序列通常为AQWESAR**AE*RL*RES)[8,31,34]。通过对SBG9基因序列分析,发现SBG9基因C端保守性较低,出现较高频率的插入/缺失突变,以及同义和非同义替换[31]。在种子植物中,SBG9基因序列C端存在2个差异明显的基序,进一步将含SBG9基序的基因分成含SBG9-A基序和含SBG9-B基序这两类基因(图1)。含SBG9-A基序的基因在真双子叶植物基因组复制事件中又产生了2个明显分支,与最早鉴定出来的金鱼草中的AmMIXTA基因聚在同一分支的命名为MIXTA,另一分支命名为MIXTA-like。含SBG9-B基序的基因在真双子叶植物基因组复制事件中也产生了2个分支,分别称为MYB17和MYB17-like(图1)。
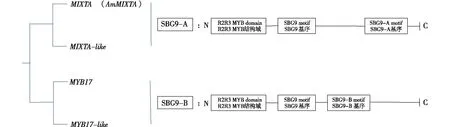
图1 R2R3 MYB SBG9基因的分类及功能域结构示意图
2 MIXTA/MIXTA-like基因的表达特征
真核生物基因组大且结构复杂[35],基因表达调控是一个复杂的生物学过程。解析MIXTA/MIXTA-like基因的生物学功能,需要首先了解MIXTA/MIXTA-like基因在不同植物中及在不同组织器官中的时空表达模式。研究发现,MIXTA/MIXTA-like基因在不同植物中的表达有一定的组织特异性,主要在花和叶片中高水平表达。例如:首先在金鱼草中鉴定出来的AmMIXTA在花瓣中优势表达[36],其同源基因AmMYBML2基因在花瓣发育中表达相对较晚,明显晚于AmMIXTA的表达高峰,在刚刚开放的花冠中表达量最大。AmMYBML2在叶片中同样也有表达,尤其是在扩展期和成熟期叶片中[36]表达量更高。同样,拟南芥的AtMYB16和AtMYB106也具有相似的表达模式,他们在花尤其是花瓣中表达量最高,但这2种基因在叶片中也有表达,尤其是在较幼嫩的莲座叶中优势表达[34,37-38]。矮牵牛(Petuniahybrida)的MIXTA-like基因PhMYB1也在花中高表达,并且表达量与花发育的不同阶段有关[36]。猴面花(Mimulushybridus)中鉴定的MlMYBML7基因的表达仅限于花组织,且在花冠发育的9~10 mm阶段达到顶峰[39]。洋桔梗的EgMIXTA1和黄花蒿的AaMIXTA1都在花芽中有较高的表达[40]。番茄的SlMIXTA-like表达谱显示其在花瓣中显著高表达[41-42]。此外,也有一些MIXTA基因在子房中优势表达,例如黄瓜的CsMYB6在黄瓜果刺发生过程中主要表达于子房表皮[43-44]。同时也存在一些MIXTA基因在胚珠发育过程中特异表达,例如棉花的10个GhMML基因,他们主要在棉纤维起始发育过程的胚珠表皮细胞中优势表达,其中一些GhMML基因(GhMML3、GhMML4、GhMML7、GhMML8、GhMML9、GhMML10)在无絮突变体(SL-7-1、MD17、XZ142f1)中表达下调[17]。
综上,MIXTA/MIXTA-like基因在不同植物中,表达部位可能不完全一致,主要在花和叶片中高水平表达,也有在胚珠和子房中优势表达。造成这个差异的原因,可能是植物在进化过程中,不同植物的组织器官发生特化,MIXTA/MIXTA-like基因家族成员也发生了不同程度的扩张或收缩。
3 MIXTA/MIXTA-like基因对表皮细胞分化的调控作用
3.1 MIXTA/MIXTA-like基因对表皮细胞锥形化的调控作用
虫媒花植物中,花的颜色和构造是影响授粉昆虫吸引的关键因素。花瓣颜色由花色素成分和含量、环境以及花瓣表皮细胞的特性决定。花瓣在吸引授粉昆虫的过程中不断进化,表皮细胞形状发生改变进化出圆锥形表皮细胞,可以增强花瓣表面的颜色强度和亮度[8,22-23]。MIXTA/MIXTA-like编码的R2R3 MYB转录因子已经在多个物种中被鉴定出可以促进扁平细胞分化形成锥形表皮细胞。在金鱼草中首先鉴定的AmMIXTA被证实对花瓣锥形表皮细胞形成起积极调控作用。随后又在金鱼草中鉴定出3个MIXTA-like同源基因(AmMYBML1、AmMYBML2和AmMYBML3)也在花瓣中表达,表明在同一物种的同一组织中会有多个MIXTA基因发挥作用。AmMYBML1主要对金鱼草腹瓣的锥形表皮细胞形成起调控作用[45]。AmMYBML2在花冠中表达量最高,与矮牵牛的PhMYB1和拟南芥的AtMYB16的亲缘关系最为密切,它们具有相似的生物学功能,促进细胞单向扩张,决定花瓣表皮细胞伸展生长的程度[36]。唐松草属(Thalictrum)的MIXTA-like转录因子TtMYBML2具有与AmMYBML2和PhMYB1相似的功能,参与调控花瓣表皮细胞锥形化,在烟草(Nicotianatabacum)过表达可诱导心皮表皮细胞向外生长,并显著增加花瓣正面表皮圆锥细胞的高度[46]。
MIXTA/MIXTA-like基因除了参与花瓣中锥形表皮细胞的形成,在猴面花中还鉴定出一种编码R2R3 MYB的MIXTA-like转录因子MlMYBML7,通过调节花瓣腹侧表皮细胞锥形化,使其在花瓣腹侧形成 2 个毛茸茸的形状类似于脊的结构,称之为“花蜜向导”[47]。MlMYBML7控制猴面花中的花蜜向导形成,同时还促进类胡萝卜素色素沉积,花蜜向导对紫外线反射的响应较强烈,类胡萝卜素使花瓣色彩鲜艳,花心部位颜色较深,从而形成鲜明的颜色对比以吸引昆虫授粉[39]。此外,在番茄中还发现SlMIXTA-like沉默植株番茄果实表皮锥形细胞发育受影响,同时采后失水并且对病原体的抵抗力发生了变化[41]。说明MIXTA/MIXTA-like基因对果实中表皮细胞锥形化也起到调控作用。
3.2 MIXTA/MIXTA-like基因对角质层及表面蜡质的调控作用
植物角质层一般由植物表皮细胞合成和分泌,在植物与环境的界面形成疏水层[48-49],限制非蒸腾作用,防止水分流失[50-51]。此外,角质层还可以作为物理屏障,抵御紫外线辐射、病原体攻击和机械损伤[27,29]。拟南芥被作为研究遗传学和分子生物学的一种模式植物,具有典型的角质层结构及组分,在拟南芥中首先发现了参与角质层生物合成以及蜡沉积的AP2/EREBP 超家族ERF亚家族的WAX INDUCER1/SHINE1(WIN1/SHN1)转录因子[52-53],过表达WIN1/SHN1可以增加角质和蜡质的产生[54]。近期的研究在维管植物角质层形成的生化和遗传基础方面取得了相当大的进展[29-30,55], 揭示了维管植物中调节角质层生物合成的遗传网络,其中MYB转录因子起着核心作用。
拟南芥中调节表皮细胞发育的编码R2R3 MYB的MIXTA-like转录因子MYB16和MYB106通过直接激活角质层生物合成基因WIN1/SHN1来控制拟南芥中的角质层形成[51,56]。MYB106和MYB16的敲除导致表皮缺陷,角质和蜡质减少。相反,MYB106过表达可诱导角质和蜡质合成,并提高WIN1/SHN1的表达[57]。WIN1/SHN1表达被MYB106-VP16(在MYB106编码序列的C端添加VP16 转录激活域序列)诱导并被MYB106-SRDX(在MYB106编码序列的C端添加SRDX 转录抑制域序列)抑制,表明MIXTA-like蛋白和WIN1/SHN1协同调节角质的生物合成和蜡积累[58]。番茄的MIXTA-like基因SlMIXTA-like对果实角质层和锥形表皮细胞的形成均有积极的调节作用,并与角质层聚合物的形成、角质层的组装和表皮细胞的模式化有关[41]。地钱的MIXTA-like基因MpSBG9也是角质层生物合成的关键调节因子,其优先调节角质形成的同源基因的表达,而不是蜡的生物合成基因[59]。还有一些MIXTA-like基因在角质层发育过程中主要对表皮蜡起重要调控作用,洋桔梗EgMIXTA1基因过表达的植物在叶表面有更多的蜡晶沉积,并且角质层蜡的总量也明显增加。表明它参与角质蜡的生物合成[40]。综上,MIXTA/MIXTA-like基因参与了不同植物的角质层的生物合成,在不同物种间的功能具有保守性。尤其是在植物进化早期的物种苔藓中,也发现了MIXTA/MIXTA-like基因参与了角质层的合成调控,这意味着陆地植物特有的MIXTA-MYB谱系在角质层的早期起源和进化中起着重要作用。
3.3 MIXTA/MIXTA-like基因对表皮毛发育的调控作用
植物表皮毛是由表皮细胞分化而来的一种特殊的单细胞或多细胞结构[60],分为有分泌功能的腺毛和不具分泌功能的非腺毛[61]。在植物表面起保护作用,如提供机械屏障保护植物免受动物和昆虫侵害,有的植物腺毛还能够产生、分泌或储存具有重要价值的次生代谢产物[40,62]。拟南芥的茎、叶片和萼片上的表皮毛是单细胞凸起分化形成的毛状结构,已在拟南芥中发现多个基因调节表皮毛的发育[3,63-64]。例如,编码R2R3MYB的控制拟南芥表皮毛形成的关键正调控基因GL1是最早克隆出来的[65-67],其突变体出现茎叶无毛的表型。同样编码R2R3 MYB的拟南芥MIXTA-like基因AtMYB106/NOK是拟南芥是表皮毛分枝的负调节因子[38],nok突变体中表皮毛分支明显增多并具有玻璃状表型外观[68],而在nok突变体中过表达AtMYB106基因,减少了表皮毛分支的形成[69]。进一步研究发现,TCP15也参与调控拟南芥表皮毛分支的形成,TCP15与MYB106的启动子在体内结合,调控MYB106基因的表达[70]。在其他植物中也发现了一些与拟南芥MIXTA-like基因亲缘关系较近的基因调控表皮毛的发育。例如黄瓜的MIXTA-like转录因子CsMYB6负调控果刺形成,时空表达分析表明,CsMYB6在黄瓜果刺起始过程中的子房表皮中高水平表达。在黄瓜中过表达CsMYB6,转基因植株中果刺数量和大小明显减少[43]。
腺毛是具有分泌功能的植物表皮毛,主要作用是分泌和储存次生代谢产物,是由多细胞分化形成的毛状结构。研究发现,MIXTA/MIXTA-like基因对腺毛的发育也有一定的调控作用。番茄和黄花蒿是研究腺毛发育的理想材料[62,71]。番茄表皮毛主要受 R2R3 MYB 转录因子、HD-Zip Ⅳ蛋白、C2H2锌指蛋白以及螺旋—环—螺旋结构的 bHLH转录因子调控。其中番茄中的MIXTA-like基因SlMX1编码R2R3 MYB转录因子,过表达后转基因植株的腺毛数量远高于野生植株,并且叶片厚度增加。SlMX1基因表达下调,则叶片的腺毛数量减少,表明SlMX1是番茄腺毛发育的正调节因子[72-73]。黄花蒿是抗疟药青蒿素的主要来源,青蒿素作为植物的次级代谢产物,主要在腺毛中合成和储存,因此腺毛密度直接关系到黄花蒿中有效成分青蒿素的含量。HD-ZIP IV转录因子AaHD1和MIXTA-like蛋白AaMIXTA1在促进黄花蒿腺毛发生中起重要作用。AaHD1是黄花蒿腺毛形成的正调控因子[74]。AaMIXTA1主要在黄花蒿腺毛的基底细胞中表达。AaMIXTA1的过表达和抑制分别导致转基因植株中腺毛数量和青蒿素含量的增加和减少。表明AaMIXTA1是腺毛发育的正调控因子,促进腺毛的发育而不会对腺毛的形态结构产生任何异常影响[72]。研究还发现一种新的HD-ZIPIV/MIXTA复合物可以促进黄花蒿腺毛的发育。HD-ZIP IV转录因子AaHD8与MIXTA-like转录因子AaMIXTA1相互作用形成一种复合物,提高了调控黄花蒿腺毛起始发育的关键基因AaHD1基因的转录活性,促进了腺毛的形成[74]。
种毛是促进植物种子靠风力传播的一种表皮毛,长期以来,杨树、柳树和法国梧桐等的飞絮污染是造林和城市绿化中的重大困扰。这些植物的飞絮是在种子发育过程中由胎座的表皮细胞发育而来[75]。种毛发育研究是飞絮治理和开展不飞絮精准育种的前提。研究MIXTA/MIXTA-like基因对种毛发育的调控有重要的理论意义和应用价值。棉纤维是已知自然界中最长的单细胞分化的种毛,有着重要的经济价值[76]。对棉纤维的起始发育研究相对比较深入,研究发现,MIXTA-like转录因子在棉纤维发育过程中发挥了重要作用。根据MIXTA序列特征,在棉花基因组中一共鉴定出10个GhMML基因[17]。其中,与拟南芥AtMYB06同源性最高的GhMYB25(GhMML7)是棉纤维发育的关键调节因子,GhMYB25沉默纤维起始分化细胞减少,伸长速度减缓,出现“短纤维”表型[77]。而AtMYB106负调控拟南芥毛状体分支,表明棉纤维是不同于毛状体的特殊种毛[34]。研究者利用正向遗传学的图位克隆方法在棉纤维突变体中克隆并验证了GhMYB25-like(GhMML3)以及其串联重复基因GhMML4,分别调控棉花短绒与长绒的起始发育[78-81]。在无絮突变体以及GhMML3-RNAi沉默植株中,包括GhMML4在内的5个GhMML(GhMML4、GhMML7、GhMML8、GhMML9、GhMML10)基因表达量均被下调,表明GhMML3是其他GhMML基因的上游调节因子[17]。
研究表明,MIXTA/MIXTA-like基因在不同组织器官中的表达主要与表皮细胞的分化相关(表1)。植物的表皮层是表皮细胞分化形成的功能多样、结构复杂的组织。在植物不断进化过程中,对于不同物种,表皮细胞分化方式不同,MIXTA/MIXTA-like基因也随之衍生出不同的调控方式。表1列出了不同植物中MIXTA/MIXTA-like基因家族成员在表皮细胞分化过程中的代表性功能研究。如:首次在金鱼草中鉴定出来的AmMIXTA及其同源基因AmMYBML1,参与调节花瓣表皮细胞锥形化;拟南芥的MYB106功能不仅体现在对表皮毛分支起调控作用,还体现在对其角质层及表皮蜡质形成的调控上。

表1 MIXTA/MIXTA-like基因家族成员在不同植物中的功能研究
综合分析MIXTA/MIXTA-like基因在不同物种中的功能发现,其作用部位和作用方式在不同植物种中有所不同,但最终调控目的都集中在表皮细胞分化上(表1)。如:金鱼草、矮牵牛和唐松草的MIXTA/MIXTA-like基因参与调控表皮细胞凸起形成锥形表皮细胞;拟南芥、番茄、洋桔梗和地钱的MIXTA/MIXTA-like基因对植物表皮细胞合成和分泌的角质层及表皮蜡质起调控作用;拟南芥、黄瓜、黄花蒿以及棉花的MIXTA/MIXTA-like基因对表皮细胞分化形成的表皮毛起调控作用,以上结果表明MIXTA/MIXTA-like基因在不同植物种表皮细胞分化过程中功能是高度保守的。但随着植物在进化过程中对环境的适应以及MIXTA/MIXTA-like基因在不同物种基因组中扩张和收缩不同,MIXTA/MIXTA-like基因在植物不同组织和器官中功能分化存在差异,因此MIXTA/MIXTA-like基因一些潜在功能也有待进一步挖掘。
4 展 望
植物表皮细胞是植物与外界环境接触的最外层细胞,其结构特征与功能密切相关。表皮毛由表皮细胞分化而来,可以防止食草动物和取食昆虫的侵害,也可以降低蒸腾速率。角质层及表皮蜡质形成了非气孔失水的最外层屏障,在应对环境胁迫中发挥重要作用。锥形表皮细胞可以直接影响花瓣的颜色,吸引授粉昆虫。棉纤维是附属在种皮上的特殊的表皮细胞,是天然纤维的重要来源。杨树、法国梧桐等的飞絮可以极大提高种子的扩散能力,但同时会带来严重的环境污染。综合表皮细胞分化的重要功能,对表皮细胞分化分子机制研究有重要理论意义和应用价值。研究发现,R2R3 MYB、bHLH 和 HD-ZIP IV亚家族均有成员参与表皮细胞分化,其中编码R2R3 MYB 蛋白的MIXTA/MIXTA-like基因被证明广泛参与表皮细胞分化调控,但由于不同植物中基因功能的分化,MIXTA/MIXTA-like基因一些潜在的生物学功能还有待挖掘,其调控网络也需要进一步研究,并且由于木本植物组培体系不完善、育种周期长等特点,目前关于MIXTA/MIXTA-like基因在木本植物中参与表皮细胞分化的报道较少。随着生物信息学和分子生物学的快速发展,将极大推进对MIXTA/MIXTA-like在表皮细胞分化过程中的作用机制进行更全面、更深入的研究。同时随着基因编辑技术在林木中的广泛应用,可以实现对林木中与表皮毛相关的重要性状的精准改良,例如在杨树中对MIXTA/MIXTA-like基因进行基因编辑,提高无絮杨树新品种创制的可能性。
参考文献(reference):
[1]KHOSLA A, PAPER J M, BOEHLER A P, et al. HD-Zip proteins GL2 and HDG11 have redundant functions inArabidopsistrichomes, and GL2 activates a positive feedback loop via MYB23[J]. Plant Cell, 2014, 26 (5): 2184-2200. DOI: 10.1105/tpc.113.120360.
[3]DUBOS C, STRACKE R, GROTEWOLD E, et al. MYB transcription factors inArabidopsis[J]. Trends Plant Sci, 2010, 15(10): 573-581. DOI: 10.1016/j.tplants.2010.06.005.
[4]FELLERA,MACHEMER K,BRAUN E L,et al.Evolutionary and comparative analysis of MYB and bHLH plant transcription factors[J].Plant J,2011,66(1):94-116.DOI:10.1111/j.1365-313x.2010.04459.x.
[5]MARTIN C,PAZ-ARES J.MYB transcription factors in plants[J].Trends Genet,1997,13(2):67-73.DOI:10.1016/S0168-9525(96)10049-4.
[6]李宗艳,李名扬.调控植物类黄酮生物合成的转录因子研究进展[J].南京林业大学学报(自然科学版),2011,35(5):129-134.LI Z Y,LI M Y.Advance in transcriptional factors regulating flavonoid biosynthesis[J].J Nanjing For Univ(Nat Sci Ed),2011,35(5):129-134.DOI: 10.3969/j.issn.1000-2006.2011.05.029.
[7]JUNGBLUT P R,SCHAIBLE U E,MOLLENKOPF H J,et al.Comparative proteome analysis ofMycobacteriumtuberculosisandMycobacteriumbovisBCG strains:towards functional genomics of microbial pathogens[J].Mol Microbiol,1999,33(6):1103-1117.DOI:10.1046/j.1365-2958.1999.01549.x.
[8]NODA K,GLOVER B J,LINSTEAD P,et al.Flower colour intensity depends on specialized cell shape controlled by a Myb-related transcription factor[J].Nature,1994,369(6482):661-664.DOI:10.1038/369661a0.
[9]MARTIN C,BHATT K,BAUMANN K,et al.The mechanics of cell fate determination in petals[J].Philos Trans Royal Soc Lond Ser B Biol Sci,2002,357(1422):809-813.DOI:10.1098/rstb.2002.1089.
[10]JAFFÉ F W,TATTERSALL A,GLOVER B J.A truncated MYB transcription factor fromAntirrhinummajusregulates epidermal cell outgrowth[J].J Exp Bot,2007,58(6):1515-1524.DOI:10.1093/jxb/erm020.
[11]KAY Q. More than eye can see: the unexpected complexity of petal structure[J]. Plants Today, 1988, 87: 109-114. DOI: 10.1111/j.1749-6632.2002.tb07570.x.
[12]KAY Q,DAOUD H S,STIRTON C H.Pigment distribution,light reflection and cell structure in petals[J].Bot J Linn Soc,1981,83(1):57-83.DOI:10.1111/j.1095-8339.1981.tb00129.x.
[13]WHITNEY H M, CHITTKA L, BRUCE T J, et al. Conical epidermal cells allow bees to grip lflowers and increase foraging effificiency[J]. Curr Biol, 2009, 19: 948-953. DOI: 10.1016/j.cub.2009.04.051.
[14]NEINHUIS C,BARTHLOTT W.Characterization and distribution of water-repellent,self-cleaning plant surfaces[J].Ann Bot,1997,79(6):667-677.DOI:10.1006/anbo.1997.0400.
[15]SCHREIBER L. Annual plant reviews Volume 23: Biology of the plant cuticle[M]. Oxford: Blackwell Publishing Ltd, 2007.
[16]SHEPHERD T,WYNNE GRIFFITHS D.The effects of stress on plant cuticular waxes[J].New Phytol,2006,171(3):469-499.DOI:10.1111/j.1469-8137.2006.01826.
[17]YEATS T H,ROSE J K.The formation and function of plant cuticles[J].Plant Physiol,2013,163(1):5-20.DOI:10.1104/pp.113.222737.
[18]FICH E A,SEGERSON N A,ROSE J K.The plant polyester cutin:biosynthesis,structure,and biological roles[J].Annu Rev Plant Biol,2016,67:207-233.DOI:10.1146/annurev-arplant-043015-111929.
[19]SCHILMILLER A L,LAST R L,PICHERSKY E.Harnessing plant trichome biochemistry for the production of useful compounds[J].Plant J,2008,54(4):702-711.DOI:10.1111/j.1365-313x.2008.03432.x.
[20]NEAL J J,STEFFENS J C,TINGEY W M.Glandular trichomes ofSolatiumberthaultiiand resistance to the Colorado potato beetle[J].Entomol Exp et Appl,1989,51(2):133-140.DOI:10.1111/j.1570-7458.1989.tb01223.x.
[21]BODNARYK R P.Physical and chemical defences of pods and seeds of white mustard(SinapisalbaL.) against tarnished plant bugs,Lyguslineolaris(Palisot De Beauvois)(Heteroptera:Miridae)[J].Can J Plant Sci,1996,76(1):33-36.DOI:10.4141/cjps96-006.
[22]CHOINSKI J S,WISE R R.Leaf growth development in relation to gas exchange inQuercusmarilandicaMuenchh[J].J Plant Physiol,1999,154(3):302-309.DOI:10.1016/S0176-1617(99)80172-2.
[23]PÉREZ-ESTRADA L B,CANO-SANTANA Z,OYAMA K.Variation in leaf trichomes of Wigandia arens:environmental factors and physiological consequences[J].Tree Physiol,2000,20(9):629-632.DOI:10.1093/treephys/20.9.629.
[24]PATERSON A H,WENDEL J F,GUNDLACH H,et al.Repeated polyploidization ofGossypiumgenomes and the evolution of spinnable cotton fibres[J].Nature,2012,492(7429):423-427.DOI:10.1038/nature11798.
[25]ZHANG TZ,HU Y,JIANG W K,et al.Sequencing of allotetraploid cotton(GossypiumhirsutumL.acc.TM-1) provides a resource for fiber improvement[J].Nat Biotechnol,2015,33(5):531-537.DOI:10.1038/nbt.3207.
[26]HAIGLER C H,BETANCUR L,STIFF M R,et al.Cotton fiber:a powerful single-cell model for cell wall and cellulose research[J].Front Plant Sci,2012,3:104.DOI:10.3389/fpls.2012.00104.
[27]TURLEY R B, KLOTH R H. Identification of a third fuzzless seed locus in upland cotton(GossypiumhirsutumL.)[J]. J Hered, 2002, 93(5): 359-364. DOI: 10.1093/jhered/93.5.359.
[28]TIAN Y, DU J J, WU H T, et al. The transcription factor MML4_D12 regulates fiber development through interplay with the WD40-repeat protein WDR in cotton[J]. J Exp Bot, 2020, 71(12): 3499-3511. DOI: 10.1093/jxb/eraa104.
[29]STEWART JM.Fiber initiation on the cotton ovule(Gossypiumhirsutum)[J].Am J Bot,1975,62(7):723-730.DOI:10.1002/j.1537-2197.1975.tb14105.x.
[30]WU YR,MACHADO A C,WHITE R G,et al.Expression profiling identifies genes expressed early during lint fibre initiation in cotton[J].Plant Cell Physiol,2006,47(1):107-127.DOI:10.1093/pcp/pci228.
[31]BROCKINGTON S F,ALVAREZ-FERNANDEZ R,LANDIS J B,et al.Evolutionary analysis of theMIXTAgene family highlights potential targets for the study of cellular differentiation[J].Mol Biol Evol,2013,30(3):526-540.DOI:10.1093/molbev/mss260.
[32]OGATA K,MORIKAWA S,NAKAMURA H,et al.Solution structure of a specific DNA complex of the Myb DNA-binding domain with cooperative recognition helices[J].Cell,1994,79(4):639-648.DOI:10.1016/0092-8674(94)90549-5.
[33]MILLARD P S,KRAGELUND B B,BUROW M.R2R3 MYB transcription factors-functions outside the DNA-binding domain[J].Trends Plant Sci,2019,24(10):934-946.DOI:10.1016/j.tplants.2019.07.003.
[34]STRACKE R,WERBER M,WEISSHAAR B.The R2R3-MYB gene family inArabidopsisthaliana[J].Curr Opin Plant Biol,2001,4(5):447-456.DOI:10.1016/s1369-5266(00)00199-0.
[35]JAENISCH R, BIRD A. Epigenetic regulation of gene expression: how the genome integrates intrinsic and environmental signals[J]. Nat Genet, 2003, 33: 245-254. DOI: 10.1038/ng1089.
[36]BAUMANN K,PEREZ-RODRIGUEZ M,BRADLEY D,et al.Control of cell and petal morphogenesis by R2R3 MYB transcription factors[J].Development,2007,134(9):1691-1701.DOI:10.1242/dev.02836.
[37]KRANZ H D,DENEKAMP M,GRECO R,et al.Towards functional characterisation of the members of theR2R3-MYBgene family fromArabidopsisthaliana[J].Plant J,1998,16(2):263-276.DOI:10.1046/j.1365-313x.1998.00278.x.
[38]JAKOBY M J,FALKENHAN D,MADER M T,et al.Transcriptional profiling of matureArabidopsistrichomesreveals that NOECK encodes the MIXTA-like transcriptional regulator MYB106[J].Plant Physiol,2008,148(3):1583-1602.DOI:10.1104/pp.108.126979.
[39]YUAN Y W,SAGAWA J M,DI STILIO V S,et al.Bulk segregant analysis of an induced floral mutant identifies a MIXTA-like R2R3 MYB controlling nectar guide formation inMimuluslewisii[J].Genetics,2013,194(2):523-528.DOI:10.1534/genetics.113.151225.
[40]WANG L,XUE W,LI X,et al.EgMIXTA1,a MYB-type transcription factor,promotes cuticular wax formation inEustomagrandiflorumleaves[J].Front Plant Sci,2020,11:524947.DOI:10.3389/fpls.2020.524947.
[41]LASHBROOKE J,ADATO A,LOTAN O,et al.The tomato MIXTA-like transcription factor coordinates fruit epidermis conical cell development and cuticular lipid biosynthesis and assembly[J].Plant Physiol,2015,169(4):2553-2571.DOI:10.1104/pp.15.01145.
[42]XU J S,VAN HERWIJNEN Z O,DRGER D B,et al.SlMYC1 regulates type VI glandular trichome formation and terpene biosynthesis in tomato glandular cells[J].Plant Cell,2018,30(12):2988-3005.DOI:10.1105/tpc.18.00571.
[43]ZHAO L,ZHU H,ZHANG K,et al.The MIXTA-LIKE transcription factor CsMYB6 regulates fruit spine and tubercule formation in cucumber[J].Plant Sci,2020,300:110636.DOI:10.1016/j.plantsci.2020.110636.
[44]YANG S,CAI Y L,LIU X W,et al.A CsMYB6-CsTRY module regulates fruit trichome initiation in cucumber[J].J Exp Bot,2018,69(8):1887-1902.DOI:10.1093/jxb/ery047.
[45]PEREZ-RODRIGUEZ M,JAFFE F W,BUTELLI E,et al.Deve-lopment of three different cell types is associated with the activity of a specific MYB transcription factor in the ventral petal ofAntirrhinummajusflowers[J].Development,2005,132(2):359-370.DOI:10.1242/dev.01584.
[46]DI STILIO V S,MARTIN C,SCHULFER A F,et al.An ortholog ofMIXTA-like2 controls epidermal cell shape in flowers ofThalictrum[J].New Phytol,2009,183(3):718-728.DOI:10.1111/j.1469-8137.2009.02945.x.
[47]LEONARD A S,BRENT J,PAPAJ D R,et al.Floral nectar guide patterns discourage nectar robbing by bumble bees[J].PLoS One,2013,8(2):e55914.DOI:10.1371/journal.pone.0055914.
[48]RAVEN J A.The evolution of vascular land plants in relation to supracellular transport processes[J].Adv Bot Res,1977,5:153-219.DOI:10.1016/S0065-2296(08)60361-4.
[49]KOLATTUKUDY P E.Biopolyester membranes of plants:cutin and suberin[J].Science,1980,208(4447):990-1000.DOI:10.1126/science.208.4447.990.
[50]KOLATTUKUDY P E.Polyesters in higher plants[J].Adv Biochem Eng,2001,71:1-49.DOI:10.1007/3-540-40021-4_1.
[51]NAWRATH C.Unraveling the complex network of cuticular structure and function[J].Curr Opin Plant Biol,2006,9(3):281-287.DOI:10.1016/j.pbi.2006.03.001.
[52]AHARONI A,DIXIT S,JETTER R,et al.The SHINE clade of AP2 domain transcription factors activates wax biosynthesis,alters cuticle properties,and confers drought tolerance when overexpressed inArabidopsis[J].Plant Cell,2004,16(9):2463-2480.DOI:10.1105/tpc.104.022897.
[53]BROUN P,POINDEXTER P,OSBORNE E,et al.WIN1,a transcriptional activator of epidermal wax accumulation inArabidopsis[J].PNAS,2004,101(13):4706-4711.DOI:10.1073/pnas.0305574101.
[54]KANNANGARA R,BRANIGAN C,LIU Y,et al.The transcription factor WIN1/SHN1 regulatesCutinbiosynthesisinArabidopsisthaliana[J].Plant Cell,2007,19(4):1278-1294.DOI:10.1105/tpc.106.047076.
[56]TAKETA S,AMANO S,TSUJINO Y,et al.Barley grain with adhering hulls is controlled by an ERF family transcription factor gene regulating a lipid biosynthesis pathway[J].PNAS,2008,105(10):4062-4067.DOI:10.1073/pnas.0711034105.
[57]OSHIMA Y, SHIKATA M, KOYAMA T, et al. MIXTA-like transcription factors and WAX INDUCER1/SHINE1 coordinately re-gulate cuticle development inArabidopsisandToreniafournieri[J]. Plant Cell, 2013, 25(5): 1609-1624. DOI: 10.1105/tpc.113.110783.
[58]YAN T, LI L, XIE L, CHEN M,et al. A novel HD-ZIP IV/MIXTA complex promotes glandular trichome initiation and cuticle development inArtemisiaannua[J]. New Phytol, 2018, 218(2): 567-578. DOI: 10.1111/nph.15005.
[59]XU B, TAYLOR L, PUCKER B,et al. The land plant-specific MIXTA-MYB lineage is implicated in the early evolution of the plant cuticle and the colonization of land[J]. New Phytol, 2021, 229(4): 2324-2338. DOI: 10.1111/nph.16997.
[60]MARTIN C, GLOVERB J. Functional aspects of cell patterning in aerial epidermis[J]. Curr Opin Plant Biol, 2007, 10: 70-82. DOI: 10(1).1016/j.pbi.2006.11.004.
[61]Advances in botanical research-incorporating advances in plant pathology[M].Amsterdam:Elsevier,2005.DOI:10.1016/s0065-2296(04)x4200-2.
[62]TISSIER A.Glandular trichomes:What comes after expressed sequence tags?[J].Plant J,2012,70(1):51-68.DOI:10.1111/j.1365-313x.2012.04913.x.
[63]LARKIN J C, BROWN M L, SCHIEFELBEIN J. How do cells know what they want to be when they grow up? lessons from epidermal patterning inArabidopsis[J]. Annu Rev Plant Biol, 2003, 54(1): 403-430. DOI: 10.1146/annurev.arplant.54.031902.134823.
[64]SERNA L, MARTIN C. Trichomes: different regulatory networks lead to convergent structures[J]. Trends Plant Sci, 2006, 11(6): 274-280. DOI: 10.1016/j.tplants.2006.04.008.
[65]OPPENHEIMER D G, HERMAN P L, SIVAKUMARAN S, et al. A myb gene required for leaf trichome differentiation inArabidopsisis expressed in stipules[J]. Cell, 1991, 67(3): 483-493. DOI: 10.1016/0092-8674(91)90523-2.
[66]BLOOMER R H, JUENGER T E, SYMONDS V V. Natural varia-tion in GL1 and its effects on trichome density inArabidopsisthaliana[J]. Mol Ecol, 2012, 21(14): 3501-3515. DOI: 10.1111/j.1365-294x.2012.05630.x.
[67]ZHAO M, MOROHASHI K, HATLESTAD G, et al. The TTG1-bHLH-MYB complex controls trichome cell fate and patterning through direct targeting of regulatory loci[J]. Development, 2008, 135(11): 1991-1999. DOI: 10.1242/dev.016873.
[68]FOLKERS U,BERGER J,HÜLSKAMP M.Cell morphogenesis of trichomes inArabidopsis:differential control of primary and se-condary branching by branch initiation regulators and cell growth[J].Dev(Camb Engl),1997,124(19):3779-3786.
[69]GILDING E K,MARKS M D.Analysis of purified glabra3-shapeshifter trichomes reveals a role for NOECK in regulating early trichome morphogenic events[J].Plant J,2010,64(2):304-317.DOI:10.1111/j.1365-313x.2010.04329.x.
[70]CAMOIRANO A, ARCE A L, ARIEL F D, et al. Class I TCP transcription factors regulate trichome branching and cuticle development inArabidopsis[J]. J Exp Bot, 2020, 71(18): 5438-5453. DOI: 10.1093/jxb/eraa257.
[71]SHI P,FU X,SHEN Q,et al.The roles of AaMIXTA1 in regulating the initiation of glandular trichomes and cuticle biosynthesis inArtemisiaannua[J].New Phytol,2018,217(1):261-276.DOI:10.1111/nph.14789.
[72]EWAS M,GAO Y Q,WANG S C,et al.Manipulation of SlMXl for enhanced carotenoids accumulation and drought resistance in tomato[J].Sci Bull,2016,61(18):1413-1418.DOI:10.1007/s11434-016-1108-9.
[73]EWAS M,GAO Y Q,ALI F,et al.RNA-seq reveals mechanisms of SlMX1 for enhanced carotenoids and terpenoids accumulation along with stress resistance in tomato[J].Sci Bull,2017,62(7):476-485.DOI:10.1016/j.scib.2017.03.018.
[74]YAN T,CHEN M,SHEN Q,et al.HOMEODOMAIN PROTEIN 1 is required for jasmonate-mediated glandular trichome initiation inArtemisiaannua[J].New Phytol,2017,213(3):1145-1155.DOI:10.1111/nph.14205.
[75]LI J X,XIA X F,XU S J,et al.Development,structure and evolutionary significance of seed appendages inSalixmatsudana(Salicaceae)[J].PLoS One,2018,13(9):e0203061.DOI:10.1371/journal.pone.0203061.
[76]樊汝汶, 吴琼美. 响叶杨(PopulusadenopodaMaxim)种子发育的胚胎学观察[J]. 南京林学报, 1982, 25(3): 116-128. FAN R W, WU Q M. The embryological observation of the seed development ofPopulusadenopoda[J]. J Nanjing For Univ, 1982, 25(3): 116-128. DOI: 10.3969/j.jssn.1000-2006.1982.03.010.
[77]MACHADO A,WU Y,YANG Y,et al.The MYB transcription factor GhMYB25 regulates early fibre and trichome development[J].Plant J,2009,59(1):52-62.DOI:10.1111/j.1365-313x.2009.03847.x.
[78]WAN Q,GUAN X,YANG N,et al.Small interfering RNAs from bidirectional transcripts of GhMML3_A12 regulate cotton fiber development[J].New Phytol,2016,210(4):1298-1310.DOI:10.1111/nph.13860.
[79]WALFORD S A,WU Y,LLEWELLYN D J,et al.GhMYB25-like:a key factor in early cotton fibre development[J].Plant J,2011,65(5):785-797.DOI:10.1111/j.1365-313x.2010.04464.x.
[80]ZHU QH,YUAN Y,STILLER W,et al.Genetic dissection of the fuzzless seed trait inGossypiumbarbadense[J].J Exp Bot,2018,69(5):997-1009.DOI:10.1093/jxb/erx459.
[81]WU H,TIAN Y,WAN Q,et al.Genetics and evolution ofMIXTAgenes regulating cotton lint fiber development[J].New Phytol,2018,217(2):883-895.DOI:10.1111/nph.14844.
[82]PASTORE J J,LIMPUANGTHIP A,YAMAGUCHI N,et al.Late meristem IDENTITY2 acts together with LEAFY to activate APETALA1[J].Development,2011,138(15):3189-3198.DOI:10.1242/dev.063073.
[83]TAN J,WALFORD S A,DENNIS E S,et al.Trichomes control flower bud shape by linking together young petals[J].Nat Plants,2016,2:16093.DOI:10.1038/nplants.2016.93.
[84]WENG L,TIAN Z,FENG X,et al.Petal development inLotusjaponicus[J].J Integr Plant Biol,2011,53(10):770-782.DOI:10.1111/j.1744-7909.2011.01072.x.
[85]SCOVILLE A G,BARNETT L L,BODBYL-ROELS S,et al.Differential regulation of a MYB transcription factor is correlated with transgenerational epigenetic inheritance of trichome density inMimulusguttatus[J].New Phytol,2011,191(1):251-263.DOI:10.1111/j.1469-8137.2011.03656.x.
[86]PLETT J M,WILKINS O,CAMPBELL M M,et al.Endogenous overexpression ofPopulusMYB186 increases trichome density,improves insect pest resistance,and impacts plant growth[J].Plant J,2010,64(3):419-432.DOI:10.1111/j.1365-313x.2010.04343.x.

