深度学习——生物医学研究和临床诊断的新助手
李心怡 贾婉楠 吴健复旦大学上海医学院,上海000;复旦大学基础医学院,上海000;复旦大学附属中山医院消化科,上海000
0 引言
20世纪50年代,计算机刚刚出现,就有人提出“人工智能”的概念。机器学习是实现人工智能的一种方法,是计算机通过各种算法对数据进行分析,不断训练用以预测或决策现实生活的问题。而深度学习(deep learning)是机器学习的一种技术,能整合庞大的数据集,学习由输入值到输出值之间复杂的函数关系。各种神经网络组成深度学习模型,在生物研究和生物医学中有诸多应用,例如预测遗传变异在具体发病机制中的细胞学过程,筛选调节治疗相关蛋白质活性的小分子以及医学影像的判读。深度学习广阔的应用前景也给研究者们带来了巨大挑战,在技术层面上,深度学习模型的性能需要不断提高;在应用层面上,深度学习需要获得医生、监管部门和其他相关机构的信任,才能成为辅助决策的良好助手。本文对深度学习的基本神经网络、在生物医学和研究中的应用、其面临的挑战及应用前景进行综述。
1 深度学习的神经网络
深度学习的名称来源于深度神经网络(deep neural networks,DNN),框架基础为人工神经网络(artificial neural networks,ANN)。本文首先介绍几种基本的神经网络,以期有助于加深对深度学习的理解。
1.1 ANN
20世纪60年代,科学家发现猫在视物时,视皮层的不同神经元能被激活,证明眼睛与视皮层的神经元之间联系密切,可逐级处理视觉信息[1-2]。此后,通过抽象和模拟人脑神经元,建立了多层复杂模型,以层为单位将提取到的颜色、形状、轮廓等各种特征进行整合处理,这一数据运算模型被称为ANN。
1.2 DNN
DNN可看作由输入层、隐藏层和输出层组成的变换数学函数,有多个层次,前一层的输出作为后一层的输入来运算,其模式如图1A所示。深度学习的另一个重要特性是可训练性,常用的训练方式是反向传播[3]。在设计一个神经网络时,人们不能确保初始设定的权重能使误差最小,所以常需反复调整权重。数据依次通过输入层、隐藏层和输出层,正向传播得到预测值;继而将预测值与真实值进行比较;若误差不能达到目标,则通过不断调整权重减小两者的误差。调整权重的过程呈现反向传播的特点,“反向”是指从输出值到输入值的计算流程,类似于生物体内反馈调节原理。
1.3 循环神经网络
在不同的深度学习构架中,循环神经网络(recurrent neural networks,RNN)在处理序列输入的方面占优势。RNN的特点是记忆功能,即在输入新数据时能读取储存的旧数据,其模式如图1B所示。与其他构架相似,RNN也可用反向传播来训练;其在RNA序列剪接、语音识别和机器翻译等方面应用广泛[4]。
1.4 卷积神经网络
卷积神经网络(convolutional neural networks,CNN)在深度学习中运用广泛,主要用于图像识别和分类,在DNN中加入卷积层,其实就是加入了特征学习的部分。每一卷积层可提取输入的不同特征,称之为不同感受野[5-6],类似于人脑在处理视觉信息时对不同特征的提取。卷积层提取特征是个由低级到高级的递进过程,首个卷积层的滤波器提取初级特征,其输出信号可作为下一卷积层的输入,如此逐层推进,达到识别人脸或图像等复杂特征之功效。卷积计算过程的模式如图1C所示。
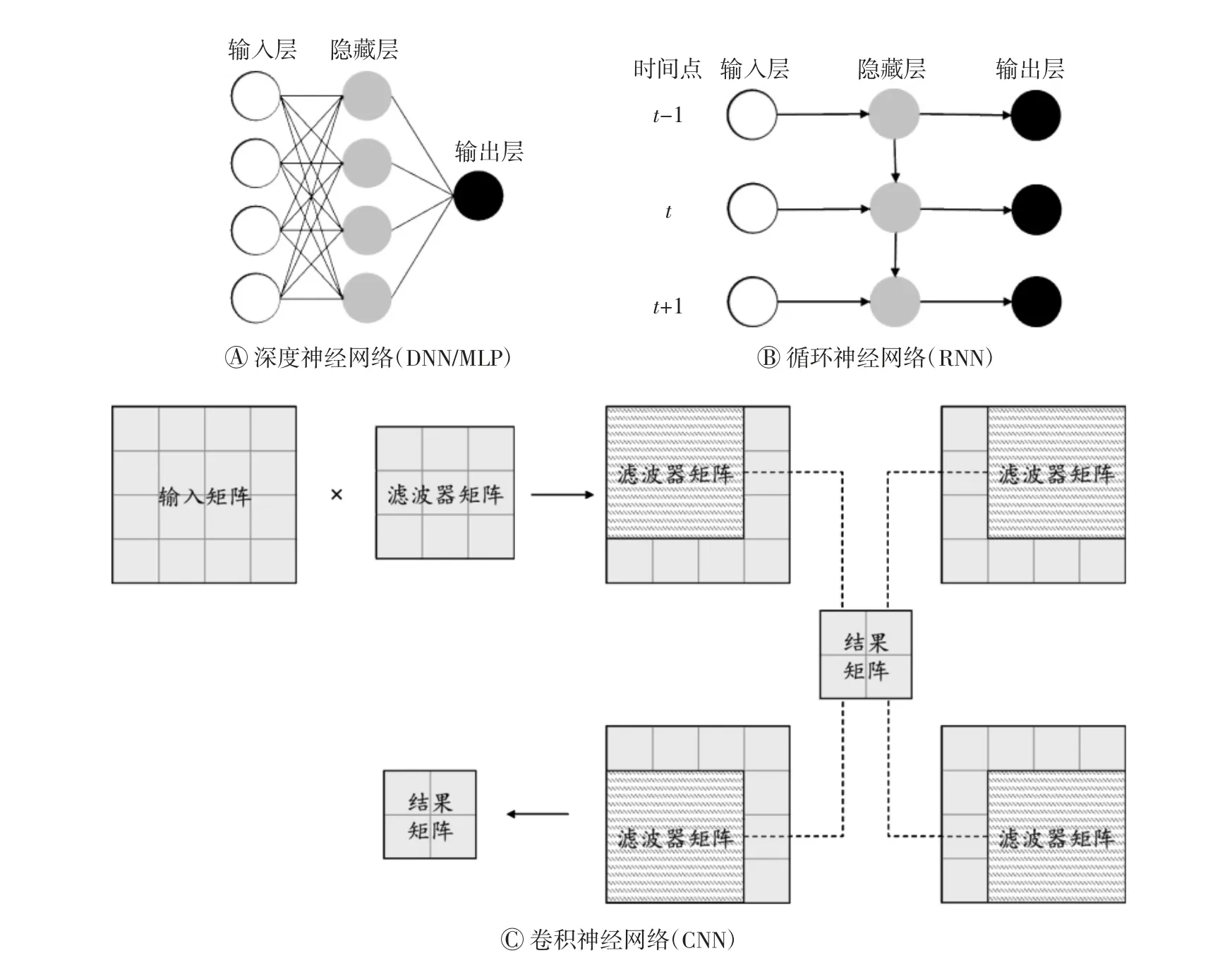
图1 深度学习中不同神经网络的基本原理及流程
2 深度学习在生物医学影像中的应用
长期以来,计算机视觉识别广泛运用于生物医学影像的自动分析。研究聚焦于人工智能对图像的处理,如分类、分割、配准、目标探测和追踪等。这些新技术已应用于医学成像、诊断、病理学、高内涵筛选、分子成像等领域。
利用深度学习可分析X线、MRI、CT扫描图片、脑电图等,其具体应用主要分为3类:医学诊断、生物学图像分析和数据建模。要将千变万化的影像学信息转变成能被计算机识别、加工的数据,并给出相应的诊断或建议,关键在于如何标注大批量数据,并监控标注数据的质量,这有赖于受过专门培训专业人员的素养和效率。临床上对同一张影像常无标准答案;对“需由几个医生分若干阶段进行标注”的问题缺乏共认标准。这使得图像辅助识别更为困难。
2.1 医学诊断
深度学习模型通过对医疗数据进行分析和建模,能推断出异常病变或发病风险,作为临床诊治的一种辅助手段,在一定程度上可提高诊断效率和准确度,减轻医生的工作负担。
Son等[7]利用深度学习模型筛选异常的眼底视网膜图像,测试10万多张图像的超过30万个读数和2个外部数据集,内容包括出血、硬性渗出物、黄斑裂孔、脉络膜萎缩或瘢痕等12个指标。将深度学习模型的灵敏度和特异性与眼科专家的检查结果对比后发现,深度学习模型能对黄斑中心视网膜眼底图像进行分类,推动了视网膜眼底图像自动筛查系统的临床应用。
胶囊内镜在小肠检查中应用广泛,但其拍摄到的视频长达8~10 h,对医生来说非常费时。Ding等[8]开发了一种基于CNN的算法,帮助医生分类和评估小肠胶囊内镜图像。检测结果表明,该模型发现视频异常的灵敏度为99.88%,对病灶的灵敏度为99.90%;而医生常规读片对病例的灵敏度为74.57%,对病灶的灵敏度为76.89%。每位患者的常规读片时间平均为96.6 min,而该模型缩短为5.9 min。因此,CNN算法对小肠胶囊内镜图像分析的敏感性可超过医生,同时也提高了效率。
Hsieh等[9]建立了一个分析中小血管病变MRI图像的辅助诊断系统。该系统采用CNN分析脑血管闭塞程度,并将结果输出到标记图像中。标记内容包括小血管的坐标位置、阻塞范围、面积大小,以及是否可能导致卒中,最后对患者所有的MRI图像进行合成,清晰显示脑内小血管的三维结构,帮助临床医生快速判断患者卒中的可能性和严重程度。
目前美国食品药品监督管理局已批准深度学习系统用于心脏分割、肿瘤追踪和视网膜病变检测。随着系统输出结果在相关领域内可信度的提高,科学家将研发出更多新系统,助推更高级别的诊断决策。图2列出了人工智能适用于辅助诊断的疾患及病变类型。
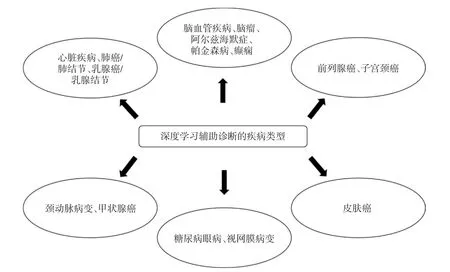
图2 深度学习在医学影像诊断中的具体应用
2.2 生物学影像分析
除了医学图像分析,一些基础研究的图像也需要进行高通量手段处理。
细胞分裂速率和各分裂阶段耗时的差异是区分正常和肿瘤细胞的重要指标[10]。肿瘤细胞在分裂时会出现有丝分裂构型的差错,所以细胞有丝分裂及其机制对开发抗肿瘤药物意义重大,但人工处理有丝分裂构型难以实现。Mao等[11]利用时域显微图像解决了有丝分裂中定位和分期的难题,该法对有丝分裂事件的定位准确率达99.2%,回收率达98.0%。
外周血显微检查是临床常规检查。识别不同类型白细胞需要技术人员通过光镜观察,利用专业知识和经验[12],耗时且带主观判断因素;利用基于CNN的分类方法能自动识别上述不同类别的白细胞。Wang等[13]利用含14 700幅标注图像数据集训练一个检测模型,并利用包含11类外周血白细胞的测试集对模型进行了评估,检测准确度达90%,每幅图像平均耗时仅53 ms,而人工读片时间平均需约20 min。
2.3 影像数据建模
应对精准医疗的需求,临床亟需从宏观影像信息中获取遗传变异、表观遗传学差异等微观因素,并应用于预测预后、指导诊断和治疗过程。目前深度学习技术已用于统计方法建模,完善医学影像体系,辅助临床决策。
Larson等[14]开发了一种基于深度学习的骨龄X光片评估模型,并与放射科专家和自动化模型比较评估结果。研究者共采集了14 036张手部X光片,训练了一个包含50层、每秒浮点运算次数为3.8×109的深度残差网络。在200例测试数据集中,模型的估计值与专家预测结果的平均差为0岁,平均绝对误差为0.50岁,均方根差为0.63岁。故认为该深度学习模型能准确估计骨龄,且性能与专家预测水平相当。
确保活产是辅助生殖技术的最终目标,产妇年龄是影响活产率的主要因素。为了实现在无创前提下按照年龄预测患者的活产概率,Miyagi等[15]建立了识别囊胚电子显微镜图像的分类模型,每个年龄段分类模型的设计原理均以CNN为基础,评估结果显示该模型的敏感性和特异性均优于常规胚胎检测方法。
3 深度学习在组学数据分析中的应用
随着分子生物学的飞速发展,高通量基因测序技术不断进步,全基因组测序也越来越易于完成,测序产生大量组学数据等待分析。无论是微生物种属比较和新种属鉴定、核酸的复制、转录和表达,还是疾病分类和药物作用预测,每项突破均依赖于组学数据的新发现。深度学习模型对处理以大数据为特征的组学信息极为有助,采用合适模型,经过训练,可大大加快处理速度。
3.1 鉴定基因表达信息
通过比较正常组织与病灶(如肿瘤组织)的基因表达信息,得到两者间差异,根据基因组学对疾病进行诊断或分类。以肿瘤为例,高维度的复杂信息是数据分析的巨大挑战。Fakoor等[16]在深度学习中运用主要成分分析将数据进行降维且保留其主要特征,使上述问题得以圆满解决;另外,由于某些类型肿瘤的数据量较小,他们用未分类的数据代替分类数据,使小数据量肿瘤类型在诊断时也可应用其他类型肿瘤学习得到特征性数据。Yu等[17]发现在疾病分类中,单隐藏层或双隐藏层的DNN更适用于RNA-seq和高通量代谢组学的表型分类,而CNN的分析结果不尽人意,这可能是由于后者在空间结构上不擅长分析组学数据矩阵。
测试全基因组表达谱的费用较高,一个替代方案是先得到L1000——即1 000个精心挑选基因的表达谱,然后据此推断出剩余基因的表达谱。Chen等[18]提出了D-GEX深度学习方法,以多层前馈神经网络来完成上述推断,与先前使用的线性回归相比,该法误差更小,性能更好。在对该法的性能评估时,研究人员发现当该神经网络的隐藏层数增多,且每层隐藏单位相应增多,推断结果的整体误差就越小,可见深层的构架要比浅层构架更具代表性。
深度学习在根据转录组数据预测药物-靶标的相互作用方面具有独特作用。药物-靶标作用对新药研发具有重大意义,可从疾病或药物的角度来研究药物定位,并预测药物的不良反应[19]。Xie等[20]建立了药物相关的基因表达数据库,对含有2 000个隐藏单位的DNN模型进行训练,并对数据进行约200倍的降维,使得DNN模型可鉴定患者样本间的差异,以此来预测药物-靶标作用的差异。
3.2 预测基因表达过程的调控
深度学习还可推测基因的复制、转录、剪接等过程。Liu等[21]提出了DNN与隐马尔科夫模型(hidden Markov model,HMM)的DNN-HMM混合模型,其利用DNA序列来识别新突变的复制结构域。增强子位于DNA非编码区,与转录过程相关,是调控基因表达的重要元件。Liu团队[22]开发了PELDA的深度学习算法框架,将前述的DNN-HMM作为其中的一个单元,在识别复制结构域的同时识别增强子,预测增强子调控基因转录能力;DNN-HMM需要在单一细胞类型中训练,而PELDA可在多种细胞或组织类型中训练,更为灵活;PELDA还可扩展到其他功能元件或识别功能域,有助于研究启动子、增强子或抑制因子对转录过程的影响。
预测选择性剪接对研究基因产物具有重要意义。Zhang等[23]依据深度学习建立了RNA-seq转录剪接分析模型——DARTS,其中DNN部分通过外显子的序列特征和基因调控特征来预测选择性剪接。DARTS已应用于研究上皮-间质转化过程中的选择性剪接,对分析胚胎发育和癌症转移意义重大。传统RNA-seq分析选择性剪接需要高序列覆盖,而该模型可预测低表达基因的选择性剪接,有效改进了传统方法。
在选择性剪接中发挥重要作用的还有RNA结合蛋白(RNA binding protein,RBP)。由于与RBP结合的单链RNA空间构型变化多端,需要多个结合结构域的组装才能完成正确结合,因此深度学习模型对RBP的预测比对转录因子更为困难。Alipanahi等[24]开发的DeepBind工具通过卷积层、纠正层、池化层和神经网络层4个阶段,以体外参数进行训练,能较准确地鉴定体内的相关结合序列。另外,DeepBind还能识别与结合位点产生或消失相关的突变序列,分析其对蛋白质结合位点的影响,在精准医疗领域应用前景广阔。
4 深度学习在蛋白质结构及相互作用分析方面的应用
X射线晶体学、核磁共振光谱学和电子显微术等实验方法可用于确定蛋白质结构,但技术要求高、成本高、耗时长以及人工需求大等因素限制了这些技术的普遍开展,也为深度学习在该领域提供了用武之地。深度学习不仅可用于识别、预测蛋白质的二级、三级结构,还可分析蛋白质-药物结合和蛋白质-蛋白质相互作用,以揭示疾病发病机制、研发药物与疫苗以及寻找合适的药物靶点。
4.1 预测蛋白质的空间结构
蛋白质二级结构是三级结构的基础,传统的观点是将其分为3部分——α螺旋、β折叠和无规则卷曲,称为Q3;但现已将Q3扩展为Q8。Q8蛋白质二级结构预测较Q3更为复杂。
Zhang等[25]使用一个卷积-双向RNN对上述内容进行了预测。与二维CNN不同,这里使用的一维CNN适用于序列分析,模拟相邻氨基酸的相互作用;双向RNN由两部分组成,分别为从序列起始端正向分析和从序列末端逆向分析的RNN。得益于RNN能记忆过去输入值的特点,双向RNN可模拟远距离氨基酸的相互作用。结合相邻氨基酸作用和远距离氨基酸作用,该模型能预测蛋白质的二级结构。DN-Fold模型[26]具有相似的应用,可预测样本和数据库中模板蛋白质是否具有相同的折叠结构,以此鉴定蛋白质的三级结构是否相似;该模型在大数据集和不同倍数识别水平的应用中性能良好。
4.2 分析蛋白质之间的结合作用
蛋白质-蛋白质相互作用与疾病的发病机制关系密切。为了研究戈谢病中酶功能丧失的分子机制,Romero等[27]应用深度学习模型(其中卷积层能捕获低维潜在空间内大规模构象运动),比较了活性与非活性状态葡糖脑苷脂酶(glucocerebrosidase,GCase)-鞘脂激活蛋白C(saposin C,SPAC)复合物的构象改变,从原子层面研究突变GCase在戈谢病的机制。该研究结果发现GCase与SPAC的相互作用能稳定酶底物的结合位点,而突变GCase无法与SPAC进行正常的相互作用,从而引起功能障碍和疾病发生。
深度学习有助于寻找与主要组织相容性复合体Ⅰ(major histocompatibility complexⅠ,MHC-Ⅰ)类分子具有高亲和力的肽链。具有二级结构肽链折叠形成的超二级空间结构称为结构模体(structural motif),它是MHC与肽链结合的重要结构,但仅仅通过核酸序列无法精确定位模体。Xiao等[28]开发了模体激活定位网络,从MHC-Ⅰ-肽复合物中提取模体,然后在模体中替换氨基酸,在保持模体功能结构不变的基础上改变其亲和力,从而得到高亲和力MHC分子结合肽,用于疫苗的开发。
综上所述,深度学习在医学影像、组学数据和蛋白质空间结构等生物医学数据分析中应用广泛,并不断改进优化。为便于查询,现将已知深度学习工具的开发年份、主要任务和所用模型归纳于表1。
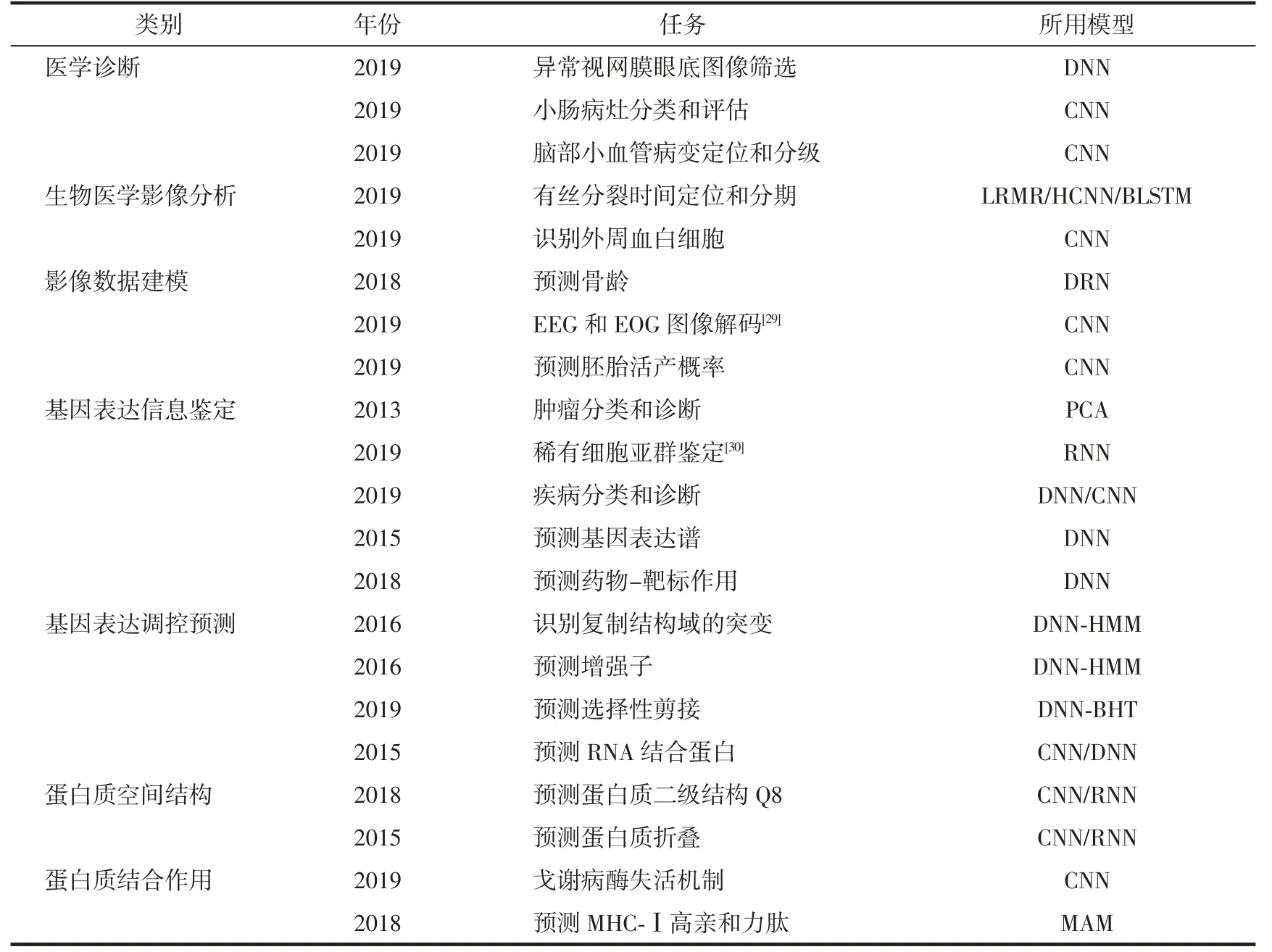
表1 深度学习在生物医学数据分析中的应用
5 深度学习的局限性
深度学习将生物医学信息分析引入了一个新时代,随着各种技术的开发和应用,人们也发现了一些具体问题,有待深入探索。
训练数据的质与量是模型可靠与否的核心。深度学习依赖于大量的标注数据,影像标注数据的质量决定了深度学习模型训练的结果。目前,国内影像标注的质量尚未达到统一规范标准,数据质量参差不齐且种类繁多,这必定会影响模型的准确性和普适性。在组学研究领域,虽然很容易获取大量基因组数据,但在个别研究中,如罕见病,仍不能满足深度学习模型训练所需要的大样本量。
深度学习也存在模型本身造成的限制。由于许多组学数据有其特殊的多层级结构,传统深度学习模型难以整合不同组学数据层,因此无法对这类结构进行处理,限制了其应用。在代谢组学研究中,由于深度学习模型缺乏解释性,所以有时不能识别对特定结果起主要作用的影响因子。在蛋白质结构预测方面,现主要依赖于目标蛋白质与已知蛋白质序列和结构的相似性;但在实际折叠过程中,并不确定两者之间的联系,这导致现有预测方法往往不能得出准确结果。要解决这个问题,不仅要增加训练集的数据库,还要使深度学习模型能利用完整的输入数据,包含整个蛋白质序列。
6 展望
深度学习能从数据中寻找深层次、具有鉴别性的特征,已应用于医学影像分析等多个领域,并取得了突破性进展。随着云计算和多图形处理器高性能并行计算技术的发展,深度学习能在海量医学图像数据中训练;同时,医学图像数据库和分割数据集的公开均能用来验证深度学习算法的精确性。深度学习在医学影像分析领域的研究以技术为主,采用的评价标准主要涉及计算机领域。为更具专业性和可信度,需要更多从事基础和临床医学专家的参与,按照不同专业考察指标对相关技术进行评估,来展示这一技术在特定专业应用的前景。
针对深度学习的直接训练需依赖大量数据库,目前主要采用的方法是微调网络,即通过预训练的网络,将大数据量的特征迁移到小数据量的目标任务上,使得目标任务对于数据量的要求大大减小,这成为发展趋势。另外,鉴于医学影像的数据库相对较少,无监督网络也成为一个重要手段。近来Campanella等[31]利用4万余张未标注的数字化病理切片,以弱监督方式训练模型。训练后模型推断准确率高于98.3%,表明读取癌症切片的特异性很高。这是直接训练未标注生物数据而成功建立深度学习模型的一个实例。
深度学习能分析几乎所有类型的组学数据,虽然还存在局限性,比如需要专家逐一解释复杂表型的因果关系,但可以预见,深度学习参与的精准健康管理模式——通过移动设备实时监控基因突变、基因转录表达和疾病的早期发生发展将成为可能。
目前,可从商业渠道获得许多深度学习的组织框架。科研人员即使无计算机专业背景,也能根据组织框架来设计模型,比从头建立神经网络更为便捷。人们在不断发掘深度学习的优势,基于现有基础,不仅需要扩展处理数据的类型,更需要提高预测的准确性和运行速度,以满足生物医学及临床实践服务的需求。
利益冲突所有作者均声明不存在利益冲突

