头颈部鳞癌中酪蛋白激酶1γ2的表达及临床相关性研究
杨震 黄春英 江河 姚金光右江民族医学院口腔医学院,百色533000;右江民族医学院附属医院口腔科,百色533000
0 引 言
头颈部鳞癌(head and neck squamouscell carcinoma,HNSC)是包括口腔癌、咽癌、喉癌等在内的头颈部肿瘤的总称,其总发病率较高,列所有癌症的第6位,5年总生存率仅约50%[1]。对HNSC发生机制和预后的研究将有助于临床上更好地对其进行防治。
酪蛋白激酶1(casein kinase1,CK1)家族由7个已知的同工型(α、β、γ1、γ2、γ3、δ和ε)组成,普遍存在于真核生物中,其进化保守,并作用于囊泡运输、DNA损伤修复、核糖体生物合成和细胞分裂分化等多个细胞功能[2-3]。CK1家族常被认为是一类潜在的致癌基因,尤其是CK1α、CK1δ和CK1ε,可能通过p53和mdm2磷酸化影响β-连环蛋白稳定性,抑制细胞凋亡以及调节微管稳定性和中心体特异性功能而具有致癌作用[3]。
酪蛋白激酶1γ2(casein kinase 1 gamma 2,CSNK1G2)是CK1家族的一种同工型,有研究结果表明CSNK1G2能调节Wnt及Hedgehog信号通路,同时抑制转化生长因子β的功能,进而促进上皮细胞的增殖,有利于肿瘤生长[4]。Li等[5]的研究结果证实CSNK1G2可磷酸化胰岛素受体底物4,通过HSC70相互作用蛋白的羧基末端引发泛素/溶酶体途径促进胰岛素受体底物4的多聚泛素化和降解,从而影响骨肉瘤细胞系生长。也有文献报道,黏液性乳腺癌等其他恶性肿瘤中亦存在CSNK1G2的变异[6-7]。然而关于CSNK1G2在HNSC发生中的作用机制尚不完全清楚,本研究将应用基于癌症基因组图谱(The Cancer Genome Atlas,TCGA)的UALCAN和Linked Omics数据库网站[8-9]、蛋白质组学数据库STRING[10]结合临床病例标本分析HNSC中CSNK1G2的表达情况及其临床相关性。
1 资料与方法
1.1 临床资料
选择2019年1月至2020年8月于右江民族医学院附属医院口腔科就诊被确诊为口腔鳞状细胞癌并接受手术治疗的患者21例。其中男性17例,女性4例,年龄(62.5±13.7)岁。收集手术切除的新鲜原发肿瘤组织标本及对应的癌旁组织标本各21例,标本取出后即冻存于液氮中。本研究符合2013年修订的《赫尔辛基宣言》的要求,所有患者均签署知情同意书。
1.2 主要材料与仪器
CSNK1G2基因引物序列、内参基因甘油醛-3-磷酸脱氢酶引物序列[生工生物工程(上海)股份有限公司],一步法反转录PCR试剂盒[翌圣生物科技(上海)有限公司]。PB100匀浆机(英国Prima公司),LightCycler96 PCR仪(瑞士Roche公司)。
1.3 方法
1.3.1 UALCAN数据分析
登录UALCAN网站,点击“analysis”,输入基因“CSNK1G2”,选择癌症类型“head and neck squamous cell carcinoma”,点击“Explore”后下载获取不同分类的基因表达信息。
1.3.2 LinkedOmics数据分析
(一)CSNK1G2拷贝数突变和甲基化对CSNK1G2 mRNA表达量的影响
登录LinkedOmics网站,选择研究癌症群组为“TCGA_HNSC”,数据库为“SCNA”,研究基因为“CSNK1G2”,目的数据库为“RNAseq”,选择统计学方法“Spearman Correlation test”,提交并下载有关CSNK1G2 mRNA表达量的分析结果。将研究数据库改为“Methylation”,重复上述操作获得CSNK1G2甲基化与CSNK1G2 mRNA表达量的关系。
(二)CSNK1G2拷贝数突变、甲基化和mRNA表达量的生存分析
登录LinkedOmics网站,选择研究癌症群组为“TCGA_HNSC”,数据库分别为“SCNA”、“Methylation”、“RNAseq”,研究基因为“CSNK1G2”,目的数据库为“clinical”,选择统计学方法“Non-parametric T Test”,提交并下载“COX回归检验”生存分析结果。
(三)CSNK1G2表达相关的基因富集分析和相关蛋白筛选
登录LinkedOmics网站,选择研究癌症群组为“TCGA_HNSC”,数据库为“RNAseq”,研究基因为“CSNK1G2”,目的数据库分别为“RNAseq”和“RPPA”,选择统计学方法“Spearman Correlation test”,提交后获取相关数据。使用该网站内置工具通过京都基因与基因组百科全书(KEGG)数据库注释进行基因富集分析并选择Affinity Propagation方法聚类后得到的结果。以P<0.05为差异具有统计学意义。
1.3.3 STRING蛋白互作网络分析
登录STRING网站,选择“Multiple proteins”,输入上述过程中筛选得到的前10个最显著相关的蛋白名称,选择人类蛋白数据库,提交搜索,下载结果。
1.3.4 实时荧光定量PCR实验
将收集的HNSC肿瘤及癌旁组织标本取出,解冻,匀浆机匀浆,使用Trizol法提取标本总RNA。引物序列包括:CSNK1G2基因正向引物序列5′-CGAGGTGCTCTGCGAGAACTTC-3′,反向引物序列5′-GAAGCCACTGCGGTCGAAGAG-3′;内参基因甘油醛-3-磷酸脱氢酶正向引物序列5′-CAGGAGGCATTGCTGATGAT-3′,反向引物序列5′-GAAGGCTGGGGCTCATTT-3′。使用一步法反转录PCR试剂盒配置反应体系进行实时荧光定量PCR(r eal-time fluorescence quantitative PCR,RT-qPC R)。
1.4 统计学方法
采用SPSS19.0统计学软件处理数据,符合正态分布的计量资料以均值±标准差(Mean±SD)表示,临床病例标本的RT-qPCR结果采用配对t检验,以P<0.05为差异具有统计学意义。通过UALCAN数据库分析不同分期分型HNSC癌组织中的CSNK1G2 mRNA表达差异,数据比较采用Kruskal-Wallis检验,以P<0.05为差异具有统计学意义。通过LinkedOmics数据库分析CSNK1G2拷贝数突变和甲基化与CSNK1G2 mRNA表达的相关性(Spearman相关性分析)、3者的生存分析(非参数t检验)以及与CSNK1G2表达相关的基因和蛋白(Spearman相关性分析),以P<0.05为差异具有统计学意义。
2 结果
2.1 不同分期分型HNSC组织标本中的CSNK1G2 mRNA表达量差异
使用UALCAN对TCGA数据库中有关HNSC的转录组测序数据进行分析,结果显示HNSC癌组织中的CSNK1G2 mRNA表达量明显高于正常组织(P<0.001,图1A);随后的RT-qPCR实验结果再次证实了原发肿瘤中的CSNK1G2 mRNA表达量高于癌旁组织(P=0.011,图1B)。深入分析数据发现,不同病理分期、不同年龄阶段和不同淋巴结转移分期(N分期)患者其原发肿瘤中的CSNK1G2 mRNA表达量差异均无统计学意义(均P>0.05,图1C~1E);男性患者的CSNK1G2 mRNA表达量高于女性患者(P<0.001,图1F)。而在肿瘤分化程度上CSNK1G2的mRNA表达量具有以下趋势:G1分期(高分化)原发肿瘤中的CSNK1G2 mRNA表达量相对其他分期降低(均P<0.05),G2分期(中分化)较G3分期(低分化)CSNK1G2 mRNA表达量降低(P<0.05),但G2分期与G4分期(未分化)、G3分期与G4分期间CSNK1G2 mRNA表达量差异均无统计学意义(均P>0.05,图1G)。此外,HPV阳性的HNSC患者较HPV阴性患者有着更高的CSNK1G2 mRNA表达量(P=0.001,图1H)。

图1 不同分期分型HNSC组织标本中的CSNK1G2 mRNA表达量差异
2.2 CSNK1G2拷贝数突变和甲基化对CSNK1G2 mRNA表达量的影响
运用LinkedOmics进行相关性分析,结果发现CSNK1G2拷贝数突变与其mRNA表达量呈显著正相关(P<0.001,图2A),而CSNK1G2甲基化程度与其mRNA表达量呈负相关(P<0.001,图2B)。
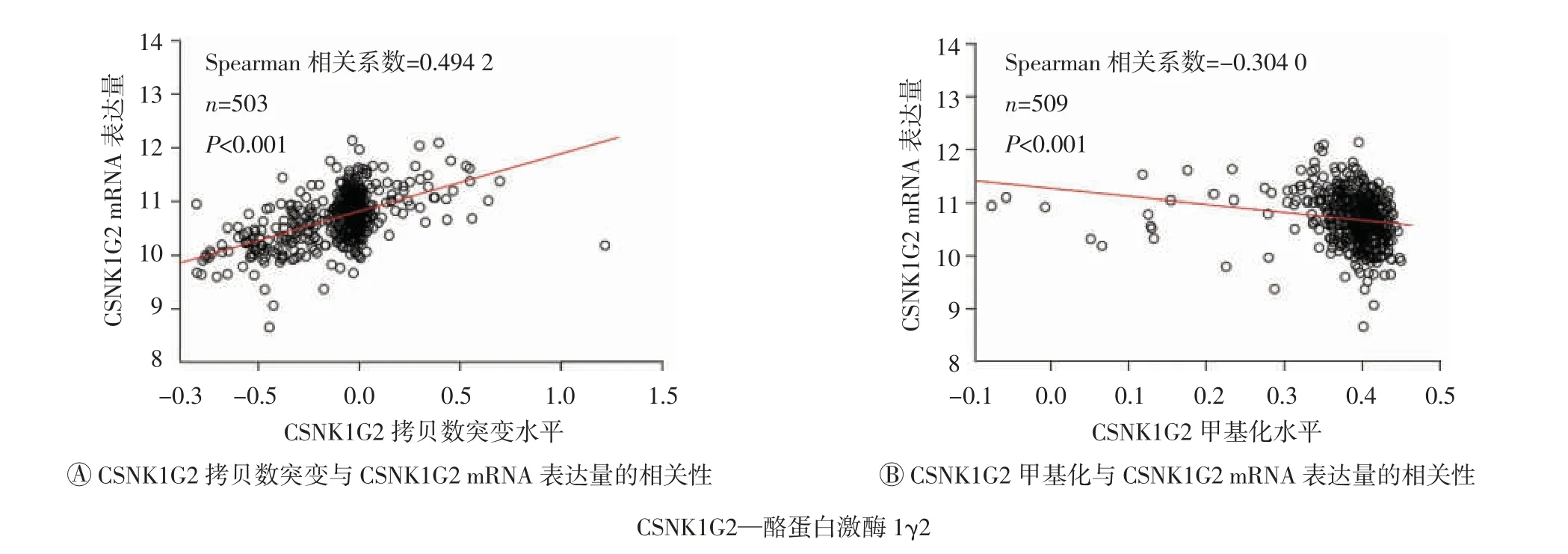
图2 CSNK1G2拷贝数突变和甲基化对CSNK1G2 mRNA表达量的影响
2.3 CSNK1G2拷贝数突变、甲基化和mRNA表达量的生存分析
LinkedOmics生存分析结果显示:CSNK1G2 mRNA高表达预示更好的生存率(P=0.033,图3A),而CSNK1G2基因甲基化的患者与基因野生型患者相比其生存率差异无统计学意义(P=0.458,图3B);CSNK1G2拷贝数突变的患者其生存率较野生型患者高(P=0.015,图3C)。
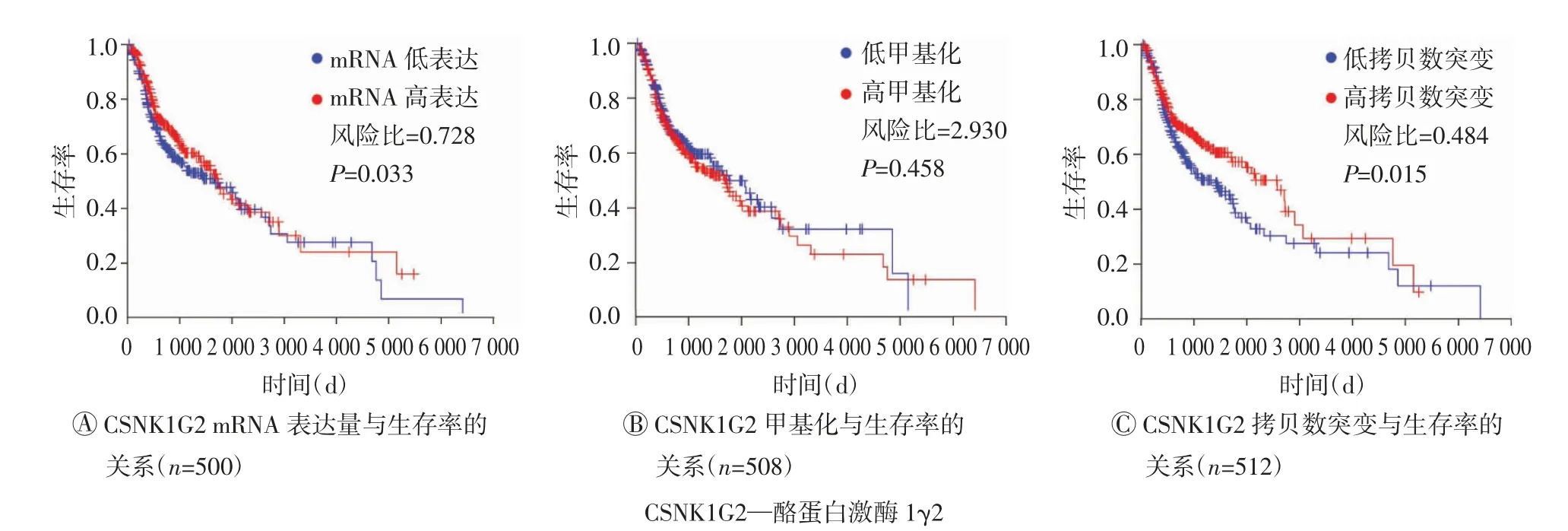
图3 CSNK1G2 mRNA表达量、拷贝数突变和甲基化的生存分析
2.4 CSNK1G2表达相关基因的基因富集分析和蛋白互作网络分析
利用LinkedOmics数据库分析出与CSNK1G2表达呈明显相关的基因(图4和5、表1),基因富集分析结果显示相关的协同基因主要作用于DNA复制等方面(图6)。同时,筛选出与CSNK1G2表达显著相关的蛋白,并对最显著相关的前10个蛋白运用STRING数据库进行蛋白互作网络分析,结果显示TP53、CHEK1和CHEK2可能在CSNK1G2介导的生理病理过程中发挥着关键作用(图7、表2)。
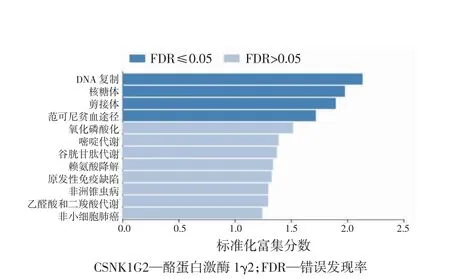
图6 与CSNK1G2表达正相关的基因富集注释
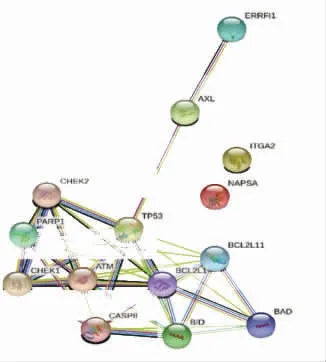
图7 与酪蛋白激酶1γ2表达相关的蛋白互作网络分析
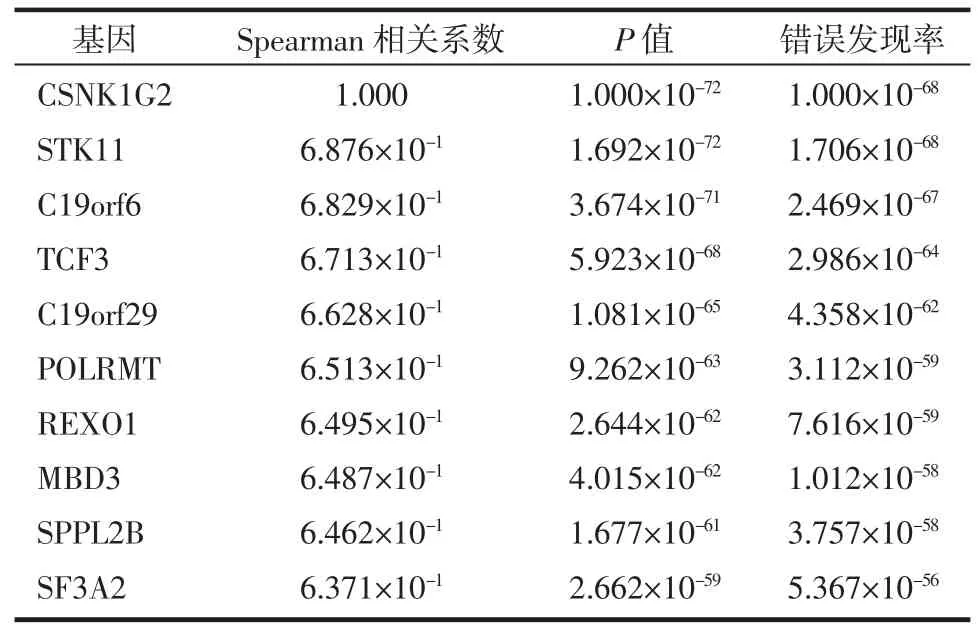
表1 CSNK1G2表达正相关基因

表2 与CSNK1G2表达显著相关的蛋白
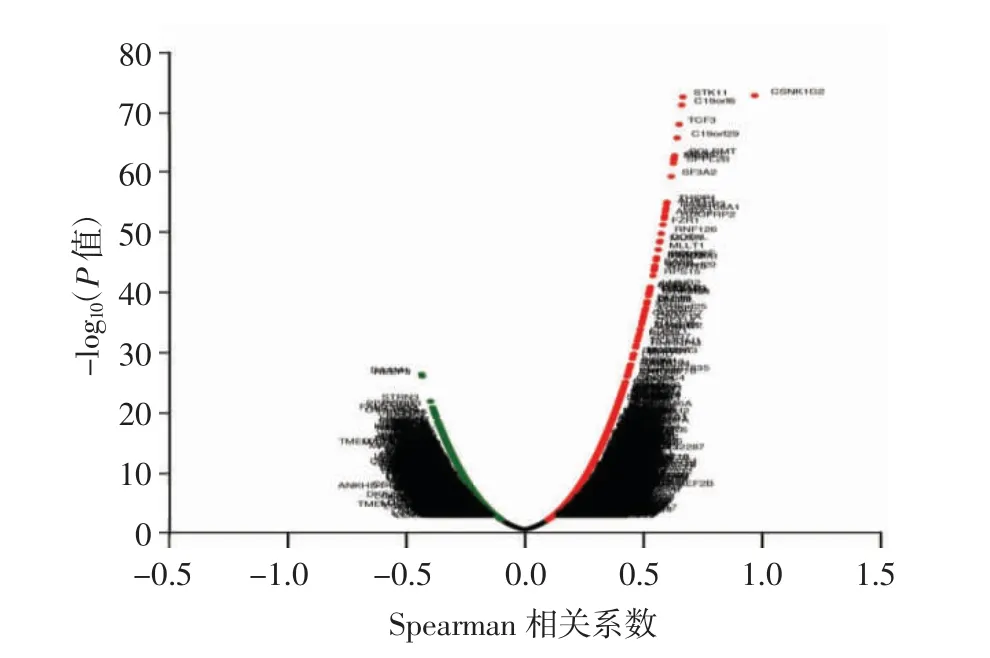
图4 与CSNK1G2表达显著相关的基因
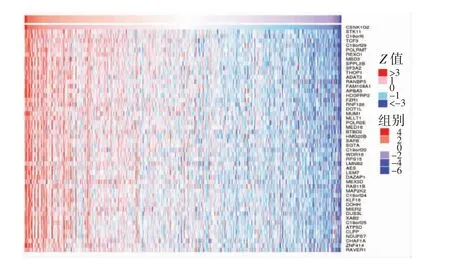
图5 与CSNK1G2表达正相关的基因
3 讨论与结论
本研究结果发现,HNSC肿瘤中的CSNK1G2 mRNA表达量较正常组织明显上调,且原发肿瘤的分化程度越高CSNK1G2 mRNA表达量亦越高,这可能预示着CSNK1G2基因对于肿瘤生长与增殖极为关键。同时笔者注意到男性HNSC患者原发肿瘤中的CSNK1G2 mRNA表达量高于女性,这与口腔癌、口咽癌等癌种中男性发病率高于女性的趋势相一致[11]。作为HNSC中口咽癌、鼻咽癌等的一项重要危险性因素,HPV感染也与CSNK1G2表达存在明显的正相关[11-12]。本研究结果还发现,CSNK1G2 mRNA表达量在不同淋巴结转移分期、不同病理分期和不同年龄阶段均无差异。这些结果在一定程度上表明CSNK1G2在HNSC中表达上调可能是HNSC发生的重要危险因素,但其具体机制尚不清楚。
DNA甲基化和体细胞拷贝数突变常常是癌症发生的重要原因[13-14]。在本研究中,CSNK1G2 mRNA表达量与CSNK1G2拷贝数突变存在正相关,而与CSNK1G2的甲基化程度呈显著负相关;同时相关的生存分析结果显示,更高的CSNK1G2 mRNA表达量和拷贝数突变预示着更好的生存率,而甲基化程度的差异与生存率无关。由此笔者认为高水平的CSNK1G2拷贝数突变导致CSNK1G2 mRNA表达升高的同时促进了癌症的发生。
进一步探寻CSNK1G2促进HNSC发生的分子机制发现,CSNK1G2相关的共表达基因主要存在于DNA复制等环节。相关蛋白的蛋白互作网络分析结果显示,癌基因TP53、CHEK1和CHEK2在与CSNK1G2相关的蛋白相互作用中处于核心位置。TP53是HNSC中最常见的突变基因,其通过调节参与细胞周期停滞、凋亡、衰老、DNA修复和新陈代谢等细胞功能的多种下游靶基因的转录来发挥肿瘤抑制功能,并与HNSC患者的短期生存率显著相关[15]。CHEK1和CHEK2作为重要的细胞周期蛋白在肿瘤发生中亦具有重要作用[16-17]。上述研究结果提示TP53、CHEK1、CHEK2与CSNK1G2可能存在协同作用共同促进了HNSC的发生。
综上所述,CSNK1G2可能通过与TP53、CHEK1、CHEK2的协同作用促进HNSC的发生,但对于肿瘤的转移和扩散无影响,同时CSNK1G2的高表达预示着较好的临床生存率。笔者推测CSNK1G2在HNSC的高表达导致肿瘤增长迅速,这使患者早期就诊成为可能,并因此具有更佳的生存预后,但相关机制仍需探索明确。未来将通过对相关基因和蛋白的进一步研究进行更深入的探讨。
利益冲突所有作者均声明不存在利益冲突

