基于字符卷积神经网络的生物医学变异实体识别方法
宋雅文,杨志豪,罗 凌,王 磊,张 音,林鸿飞,王 健
(1. 大连理工大学 计算机科学与技术学院,辽宁 大连 116024;2. 军事医学科学院,北京 100850)
0 引言
随着生物医学研究的发展,领域专家和学者们为了记录研究成果和促进领域交流,发表了大量的生物医学文献,从而促进了生物医学文本挖掘技术的发展。其中在复杂疾病研究领域中,为了分析和解释复杂疾病的序列变异,从文献中挖掘变异信息成为一项重要的研究内容。
序列变异是指机体内的基因水平的物质(通常是DNA)发生改变造成的变异。从文献中挖掘变异信息一般指挖掘文本信息中的序列变异,序列变异的文本提及(text mention)通常称为变异实体,变异实体识别是变异信息抽取的研究重点之一。本文提到的变异实体主要包含DNA变异、蛋白质变异和单核苷酸多态性SNP三类,如图1所示,标注部分c.356G>A属于DNA变异,rs13267属于单核苷酸多态性SNP,C119Y属于蛋白质变异。

图1 tmVar数据集变异实体示例
目前变异命名实体识别主要采用基于规则、基于机器学习和基于深度学习的方法。传统基于规则的方法依赖于大量手工设计的正则表达式,其规则的生成也需要领域知识的支撑。如MutationFinder[1]可以识别出文本中的核苷酸或氨基酸变异,该类型变异的命名形式单一固定,仅用简单规则就可达到很好的识别效果。在基于传统机器学习方法中,基于条件随机场(conditional random filed, CRF)的方法成为了目前主流。如VTag[2]使用CRF方法识别文本中描述的基因组畸变类型、基因组位置和基因组状态变化信息;tmVar[3]采用基于CRF的综合模型提取蛋白质、DNA和RNA水平描述的广泛序列变异。CRF模型虽然可以和基于规则的方法相结合,但其性能在很大程度上依赖于复杂的特征工程。
近年来,随着深度学习研究的不断深入,通用新闻领域提出几种相似的应用于命名实体识别任务的神经网络结构并取得了不错的结果[4-7]。其中,结合条件随机场的双向长短期记忆网络(BiLSTM-CRF)模型表现较为突出。在生物医学领域,Habibi等人[8]将BiLSTM-CRF模型应用到了生物医学语料上(包含药物、疾病、物种、基因蛋白和细胞实体识别五大类语料),其中药物和疾病语料上的实验结果都处于当今先进水平,优于传统的CRF基线系统。Zhu等人[9]对比了BiLSTM-CRF模型和CNN-CRF模型在生物医学领域实体识别上的效果,发现两种模型存在差距,但并不明显。Matos等人[10]将BiLSTM-CRF模型应用到变异实体识别任务上,并比较了以字符和单词分别作为模型输入单元的影响,结果表明以字符为输入单元的模型效果更好,但由于没有根据变异实体名的特点进行针对性优化,导致实验结果没有超越基于丰富特征的CRF模型。
针对现有方法存在的问题,首先本文提出一种基于字符卷积神经网络的变异实体识别方法。针对变异实体名的特点,本文利用一个多窗口大小的卷积神经网络学习字符级词表示,以充分利用变异实体名的内部字符信息,还能在一定程度上缓解未登录词问题。其次,本文对比了基于词和基于字符的不同神经网络模型(CNN-CRF和BiLSTM-CRF模型)在变异实体识别任务上的效果。结果表明,与基于词的BiLSTM-CRF模型相比,基于字符的CNN-CRF模型效果更好、效率更高。最后,本文方法在MutationFinder[1]和tmVar[3]两个语料上均取得了目前的最好结果,模型简单高效且不依赖于复杂的特征工程。
一般实体识别任务被看作序列标注问题,因此本文也采用序列标注最常用的标注机制IOB(inside,outside, beginning)机制[11]。
1 变异实体识别模型
本文提出了一种基于字符卷积神经网络的变异实体识别方法,模型CharCNN-CNN-CRF整体框架如图2所示。模型主要分为基于字符卷积神经网络的词表示层CharCNN、编码器CNN层和解码器CRF层三部分。

图2 CharCNN-CNN-CRF模型整体框架
1.1 基于字符卷积神经网络的词表示层
词表示层模型CharCNN如图3所示。模型将目标词的初始化字符向量送入卷积神经网络[12](convolutional neural network,CNN)和最大池化层,抽取词的字符特征,输出目标词的字符级词表示。字符向量选择随机初始化,且在模型的整个网络结构中都是可训的。字符特征抽取过程如式(1)所示。
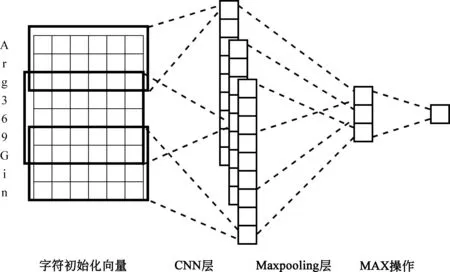
图3 抽取字符特征的卷积神经网络示例窗口大小3,4,5

(1)
受N-Gram思想启发,本文选择多窗口大小的CNN学习字符级词表示,定义窗口大小为k(k∈(1,2,3,4,5)),每个窗口大小的卷积核数均为64。经过各自的最大池化层后,我们尝试了5种不同窗口大小的特征合并方式,定义为f,分别为最大化(Max)、平均化(Avg)、拼接(Con)和相加(Add)操作。合并过程定义如式(2)所示。
1.2 编码器CNN层
一般认为CNN通过卷积操作提取特定文本粒度下特定窗口大小中的信息,因此更擅长提取重要的局部特征,导致其无法考虑长距离的依赖信息和词序信息,但实践中可以通过栈式叠加多层来缓解。在变异实体识别任务中,变异实体的识别对于长距离信息和词序信息没有太大的依赖性,更加依赖于实体本身的字符组合信息,我们选择如图2所示的多层CNN的栈式叠加作为文本编码器。
以CharCNN抽取的字符级词表示作为输入,文本编码器先用CNN层捕获文本中目标词和它相邻词的关联信息,再利用多层CNN抽取句子不同语言粒度的局部上下文特征,最后将CNN层输出到两层全连接网络,将该词标注作为每一种标签的最终分值。
1.3 解码器CRF层
本文将变异实体识别任务当作是序列标注问题。与分类问题相比,序列标注问题中当前的预测标签不仅与当前的输入特征相关,还与之前的预测标签相关,即预测标签序列之间是有强相互依赖关系的。例如,在IOB标注机制中,B表示一个实体的开始部分,I表示该实体其余部分,O表示非实体部分。所以在合理的标签序列中I标签不应该出现在O标签之后。为了考虑预测标签之间的依赖性,本文使用CRF模型来进行解码,从训练数据中获得约束性的规则,保证预测的标签是合法的。
CRF模型是一种对数线性模型。给定一个句子,CRF模型将为句子中每一个词预测一个标签。定义输入句子的单词序列x=(x1,…,xn),预测标签序列y=(y1,…,yn),Y(x)是所有可能的标注序列集合,Pi,yi是CNN编码器输出的第i个词标注为标签yi的预测分值。定义一个标签转移矩阵Tyi,yi+1,其中Ti,j表示从标签i转化为标签j的分数。模型最终的得分函数s(x,y)如式(3)所示。
使用归一化指数函数计算生成给定输入序列x的标签y的条件概率分布是P(y|x),如式(4)所示。然后通过最大化对数似然概率来训练模型参数,如式(5)所示,使用Adam优化器来优化参数,选择得分最高的标签序列作为标签预测序列。最后通过维特比算法[13]寻找最佳标签序列,如式(6)所示。
其中,Y(X)表示所有可能的标签序列集合,y*是得到的最佳标签序列。
2 实验分析
2.1 实验设置
我们选用MutationFinder[1]数据集和tmVar[3]数据集作为实验数据集。tmVar数据集中标注的变异实体主要包含DNA变异、蛋白质变异和单核苷酸多态性SNP三类。MutationFinder数据集中标注的变异实体均为核苷酸或氨基酸变异(属于蛋白质变异类别)。数据集统计信息如表1所示。模型的超参数设置如表2所示。此外,从两个数据集的训练集中分别抽取10%的样本作为开发集,用于超参数的选择,并通过在开发集上的早停策略[14]选择模型训练迭代次数。

表1 数据集统计信息

表2 模型的超参数设置
训练Word2Vec的数据包括tmVar数据集文摘、MutationFinder数据集文摘和从PubMed上爬取的一部分生物医学文摘,共计2GB。所有文摘在训练之前用nltk工具包[15]进行标准分词。
变异实体识别中,当实体左右边界和实体类别均正确时为预测正确,否则均为预测不正确。我们使用查准率(precision,P)、召回率(recall,R)和F值(F-score)对模型在测试集上的预测结果进行评价。
2.2 字符级词表示实验
为了探索适合变异实体的词表示方法,基于CNN-CRF模型,本文设计了一组词表示实验。实验比较了抽取字符特征的CNN和BiLSTM模型,对比了CNN层中合并字符特征的不同方式,如表3所示。结果显示窗口大小 固 定为3的CNN层模型的F值是86.91%,比BiLSTM的F值85.01%高了近两个百分点。可能原因是绝大多数变异实体名中的字符组成遵循一定的行业标准,有一定的结构特性(图4),所以其实体名对字符组成这种局部信息有依赖性。虽然CNN和BiLSTM都能学习上下文信息,但BiLSTM的优势主要是捕获长距离依赖信息,而文本中每个词的长度有限,词内部字符之间的上下文联系紧密,不至于依赖词外部长距离的字符信息,故BiLSTM的优势在词表示方面无法体现,因此词表示模型选用卷积神经网络效果更好,且更轻便。
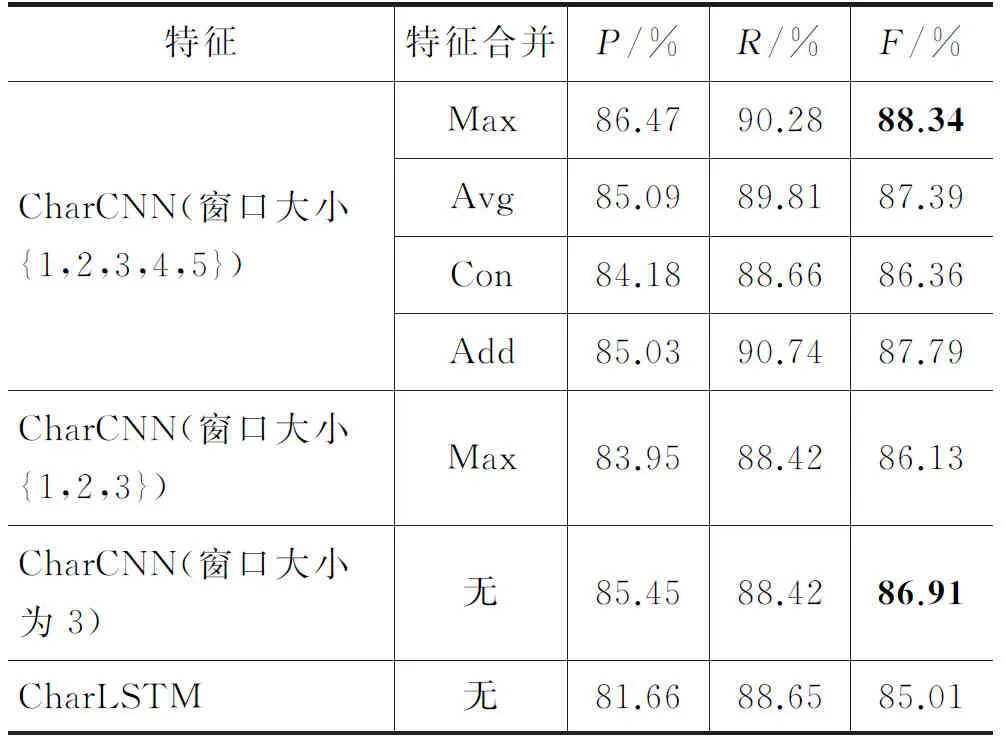
表3 字符级词表示实验结果

图4 样本1灰色高亮为蛋白质变异实体,样本2灰色高亮为DNA变异实体
进一步探索如何更好地抽取字符特征,受N-gram思想的启发,输入先各自通过窗口大小分别为1,2,3,4,5的卷积神经网络,再经过各自的最大池化层,最后以不同的方式合并这些特征,生成字符级别的词表示。我们将抽取字符级词表示的模型命名为CharCNN。
模型CharCNN的F值为88.34%,F值和窗口大小仅为3的模型相比高约1.4%。这说明CharCNN获得了比常见卷积层窗口大小固定的模型更好的词表示,原因可能是窗口大小不同的卷积层能捕捉到多层次、多角度的字符信息,词表示中包含了更加丰富、对预测更有用的字符特征。
合并方式中最大化操作(Max)的结果F值为88.34%,优于其他三种方式。相加操作仅次于最大化操作,F值为87.79%,而最常用的拼接操作F值最低,是86.36%。可能的原因是Max合并可以保留对实体识别最关键、最显著和更有利的字符特征信息,为预测阶段奠定更好的基础。
此外实验结果中召回率均高于准确率,通过可视化测试集预测结果发现,预测结果中存在一定量的样本,如图5所示,样本中某些词被模型错误预测为实体,其词结构与数据集中DNA变异和蛋白质变异实体的结构极其相近,导致了模型的误判。因此,我们认为这种现象的存在是降低模型预测准确率的原因之一。

图5 错误预测示例
2.3 字符和词特征在不同神经网络上的对比实验
常见的命名实体识别任务多以词特征为主体特征,而将字符特征作为辅助特征,以便更好地刻画单词的属性。对于神经网络模型BiLSTM,由于其能学习到句子长距离依赖信息的优势而在命名实体识别任务中得到广泛的应用。
为了探索不同特征和不同神经网络对模型性能的影响,我们在tmVar数据集上设计了一组对比实验,比较了基于字符特征和基于字符特征与词特征组合的CNN-CRF模型。对比了基于字符特征的CNN-CRF模型和BiLSTM-CRF模型,实验结果如表4所示。
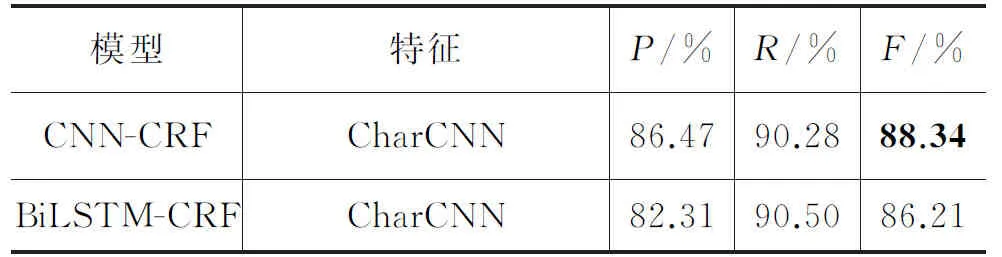
表4 tmVar数据集上字符特征和词特征在不同神经网络上的对比实验结果

续表
对比CNN-CRF模型和BiLSTM-CRF模型,当特征仅有字符特征时,模型编码器选用CNN的F值是88.34%,选用BiLSTM的F值是86.21%,两者相差近两个百分点,且CNN-CRF模型的结果较高。可能的原因是当判断目标词是否属于某个变异实体的一部分时,词序信息或者该词与句子中其他词的长距离依赖信息的联系不够大,更多地是依赖于变异实体名中的局部上下文、字符的组合信息和单词的前缀、后缀等信息。因此当选用字符特征时,CNN既能捕获词的局部上下文信息,又能从词表示中获取字符信息,而且效率相对较高,导致BiLSTM能捕获长距离依赖信息的优势无法体现。
基于词特征的模型中BiLSTM-CRF模型的F值比CNN-CRF模型高,可能的原因是当无法利用字符信息时,模型只能通过上下文内容进行预测。此时,BiLSTM和CNN相比更有优势。
当仅用词特征时, CNN-CRF模型和BiLSTM-CRF模型的F值与各自基于字符特征的模型相比均大幅下降,分别是61.53%和67.15%。可能的原因是基于词特征的模型仅仅包含词级别的信息,无法获取词的字符信息,因此实体识别过程只能依赖于仅有的词信息。这类模型需要事先选择一个词汇量固定的词汇库,不属于词汇库的词汇被映射为未知词。在生物医学文本中,变异实体分词结果与普通词汇相比词频偏低,因而当用Word2Vec训练词嵌入时,这类词会被过滤而成为输入文本中的未登录词(out of vocabulary, OOV)。Labeau等人[16]提出了一种基于字符或者词组件方式构建词表示方法,相较于传统语言模型词嵌入初始化的词表示可以有效解决未登录词问题,其结论与本文实验结果一致。
综合各方面因素,我们选择卷积神经网络作为模型的编码器。本文提出的卷积神经网络抽取字符级别词表示模型虽然属于词级别模型,但具有不受词汇表限制、能充分利用字符信息等优点,能缓解基于词特征模型存在的未登录词问题。此外,构建字符特征需要的词汇库很小,计算成本低。
2.4 主流方法对比实验
本文提出的模型是CharCNN-CNN-CRF,在MutationFinder数据集上的对比方法包括基于规则的MutationFinder方法、基于CRF模型的tmVar方法。在tmVar数据集上的对比方法有tmVar方法、Matos[10]提出的基于深度学习的方法和基于规则的方法。基于规则的方法复现了tmVar提供的正则表达式,实验结果如表5所示。
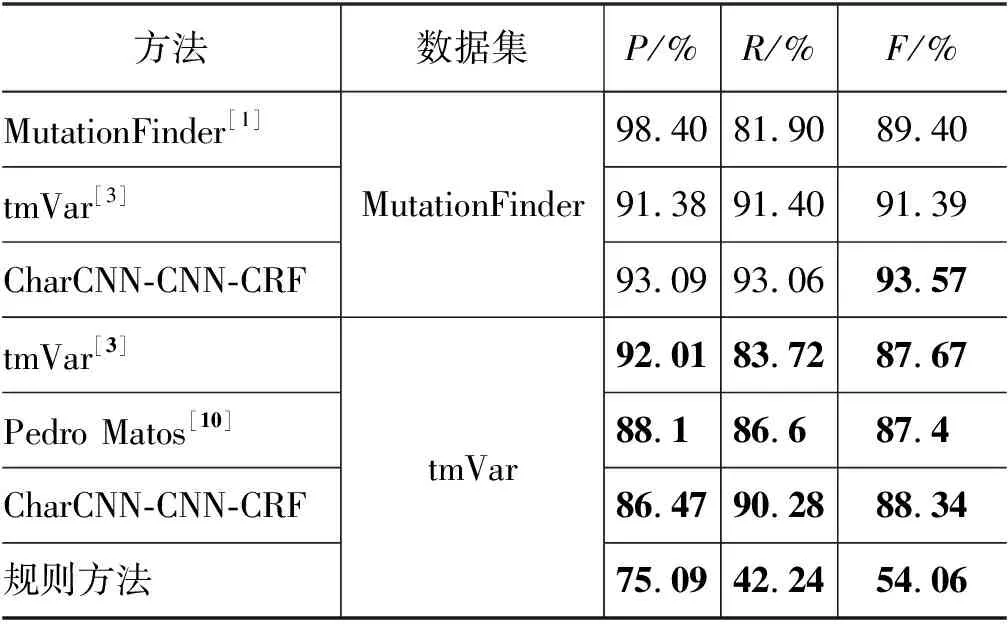
表5 主流方法对比实验结果
对于MutationFinder数据集,本文方法CharCNN-CNN-CRF的F值达到了93.57%,相比于MutationFinder方法有所提高,可能原因是该数据集中的变异实体形式单一,模型中的字符级词表示层能充分学习变异实体名的字符特征,生成更具优势的词表示,从而获得较好的实体识别能力。
对于tmVar数据集,CharCNN-CNN-CRF在没有后处理的情况下,与tmVar的基于综合CRF模型F值87.67%相比有所提升。原因可能是语料中除遵循HGVS命名规则的结构化变异实体名外,不可避免地存在一些结构信息不明显、需要上下文信息才能识别出来的变异实体,如图6所示。本文方法不仅能通过字符表示层学习实体名的字符等内部细节信息,还能通过编码器编码文本内容,互补地学习到变异实体名外部的上下文信息,从而从多方面提高模型的实体识别的能力。

图6 不遵循HGVS标准的正样本示例
Matos[10]等人在tmVar语料上比较了词级和字符级BiLSTM-CRF模型,其实验结果显示字符级模型的效果更好,F值为87.4%,不过仍低于本文实验结果。可能的原因是字符级模型是预测文本序列中每一个字符的标签,没有考虑词的边界和词的上下文等词级别的细节信息,所以即使最终结果优于预测文本序列中每一个词标签的词级模型,但仍有局限性。
CharCNN-CNN-CRF虽然也属于词级模型,但CharCNN层的输出是字符级的词表示,文本编码器通过栈式叠加多层CNN能够学到词的上下文信息,因此该模型既包含词的字符信息,又包含必要的词信息,为标签预测过程提供更多的关键信息,不仅可以识别出结构化变异实体名,还能识别出不遵循标准的变异实体名。
CharCNN-CNN-CRF模型在两个不同语料上均取得最好的结果,表明该模型具有较好的泛化能力。
3 结论
本文提出了一种用于生物医学变异命名实体识别的基于字符卷积神经网络的CNN-CRF模型,实验结果表明,该模型仅使用字符特征和简单的CNN-CRF模型就能在tmVar和MutationFinder语料上取得最好的结果。我们还进一步发现:
(1) 在变异命名实体识别任务上,单独的字符特征比常见实体识别模型中单独的词特征或拼接词特征与字符特征的效果更好,还能缓解未登录词问题。
(2) 基于N-gram思想的字符级卷积神经网络通过编码实体内字符信息,抽取出对变异命名实体识别更有利的字符特征,从而获得更好的词表示。
(3) 使用CNN作为模型的文本编码器比BiLSTM效率更高。
(4) CRF层的使用能极大地降低预测中非法标签序列出现的概率。
目前的方法仅用字符级的词表示,虽包含一定的上下文信息,但未能找到适用于变异实体的直接结合词特征的方法,因此不能充分挖掘和利用上下文词信息,所以如何在词级别上提高实验性能是未来研究的重点。

