基于DNA条形码对温州洞头海域游泳生物的鉴定
吴晓雯 王铁杆 刘颖 张鹏
(浙江省海洋水产养殖研究所,温州市海洋生物遗传育种重点实验室,浙江温州 325005)
海洋游泳动物是海洋生态系统和海洋生物资源的重要组成部分,包括海洋鱼类、头足类、甲壳类等,可以直接给人类提供高营养价值的食物,具有重要的经济利用价值。对海洋游泳生物进行调查鉴定是海洋生物资源监测、海洋生物多样性研究的基础工作,也是海洋资源开发利用和海洋保护区建设重要的参考依据。长期以来,都是根据生物的外部形态来鉴定物种。通常鱼类在早期的形态特征差异不够明显或者相类似,很难根据其早期阶段的形态特征进行精确分类[1]。由于物种表型具有可塑性,其形态特征会受到生物或者非生物因素的干扰[2];生物在不同的生长发育阶段或者两性异形也会影响鉴定的准确性[3]。此外,形态学鉴定还受到分类者经验和能力的影响,需要大量的专业知识积累。因此,准确地鉴定物种显得尤为重要。
随着生物分子技术的快速发展,DNA条形码分析成为物种系统演化和分类鉴定的1种新的手段[4],是专业人员可以运用的1种有效鉴定工具[5]。Hebert 等[6]明确提出了DNA 条形码(DNA barcoding) 的概念,即通过使用短的、标准化的基因片段来进行物种鉴定,以提高真核生物识别效率,也使得DNA 序列辅助分类方法得到一定程度的统一。DNA条形码技术的快速发展,在物种鉴定方面表现出较高的优越性,尤其是DNA条形码数据库的构建,为种类鉴定奠定了基础。目前,线粒体细胞色素C氧化酶亚基Ⅰ(COI) 基因被公认为标准DNA条形码[7-8],可以有效鉴定鱼类[9-12]、头足类[13]、甲壳类[14]和多毛类[15]。
洞头海域处于浙江省南部,该海域受浙闽沿岸流和台湾暖流的低、高盐水系交汇的影响,水文条件适宜,海域开阔,海洋生物资源丰富。虽然已有该海域游泳动物的物种组成和群落结构季节性变化的研究报道[16-17],但鲜有关于DNA条形码数据收集的报道。为了监测该地区的游泳生物资源,本研究通过测定和比对温州海域常见游泳生物的线粒体COI基因的DNA 条形码序列,分析该基因在温州海域游泳生物鉴定中的可行性,并为建立温州海域游泳生物DNA 条形码库提供方法和基础数据,研究结果可为该地区渔业监测、海洋生物资源保护、海洋保护区的建立提供参考依据。
1 材料和方法
1.1 样品采集与鉴定
2020年4月在浙江省洞头海域进行张网调查(张网网口长10 m、宽7 m、高5 m, 80目),生物学测定按照《海洋调查规范 第6 部分:海洋生物调查》(GB/T 12763.6—2007)进行。将每个调查站位的渔获物全部取样装入样品袋,做好渔捞记录和样品袋编号记录后,冰鲜保存,带回实验室进行分析和鉴定,对主要品种进行生物学测定。称量用电子天平精确度为0.001 g。游泳动物中软体动物和节肢动物种类名称及分类地位参考《中国海洋生物名录》[18],鱼类参考《中国海洋鱼类》[19]《浙江海洋鱼类》[20]等文献资料。
1.2 试验方法
取渔获物的肌肉组织,用双蒸水洗去杂物,再用滤纸吸干,采用海洋动物组织DNA提取试剂盒(北京全式金生物技术有限公司)提取基因组DNA,4 ℃保存备用。利用通用引物对目的基因进行扩增,引物详见表1。
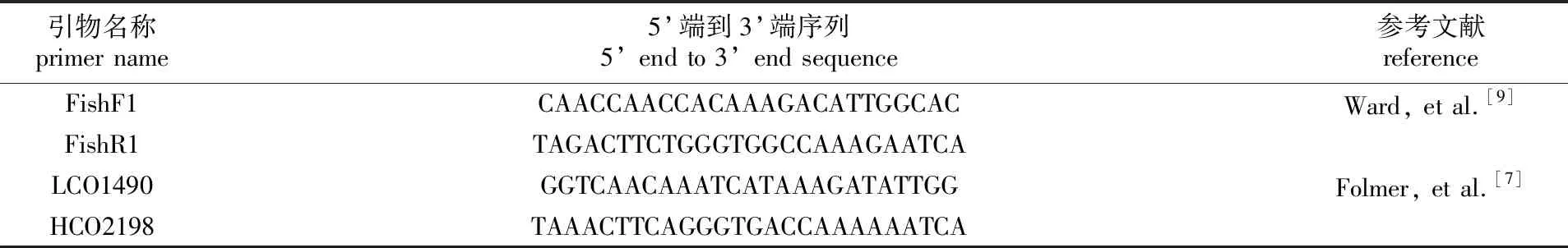
表1 引物序列信息
PCR反应体系为25 μL:上下游引物各1 μL(10 μM),DNA模板2 μL,金牌PCR Mix(green,北京擎科生物技术有限公司)21 μL。PCR反应程序为:98 ℃预变性2 min;98 ℃变性10 s,53 ℃退火15 s,72 ℃延伸15 s,35个循环;72 ℃延伸5 min。以上试验均设置阴性对照。取2 μL扩增产物进行1%琼脂糖凝胶电泳检测,将单一目的条带的PCR产物送到杭州擎科生物技术有限公司进行纯化和双向测定,以确保序列的可靠性。
1.3 数据处理
对获得的全部序列进行人工比对并进行人工矫正,截取有效准确片段进行后续分析,将所有序列在NCBI数据库中进行blast序列相似性比对,采用两两序列间遗传相似度>98%的为同一物种、92%~98%为同一属、85%~91%为同一科的标准对洞头海域的游泳生物进行种类鉴定[21-22]。然后通过MEGA 6.0[23]对序列进行排列,计算所获序列的长度、GC含量、多态位点、简约信息位点等参数。结合数据库中与样品DNA 条形码相似度最高的序列,利用邻接法(neighbor-joining) 构建系统发育树。邻接法分支树的可信度采用Bootstrap检验,经1 000次自检获得分支树节点的支持率。
2 结果和分析
2.1 基因序列
本研究共获得29条序列,经过比对,平均长度为666 bp,其中鱼类序列共有19条,无脊椎动物10条,所有序列没有插入、缺失碱基。保守位点316个,变异位点350个,单变异位点13个,简约信息位点337个。T、C、A和G含量分别为31.8%、24.9%、25.1%和18.1%。
在脊椎动物(鱼类)19条序列中,保守位点379个,变异位点287个,其中单变异位点30个,简约信息位点257个。T、C、A和G含量分别为30.2%、27.1%、24.3%和18.4%。
在无脊椎动物(头足类、甲壳类和多毛类)10条序列中,保守位点346个,变异位点320个,其中单变异位点41个,简约信息位点279个。T、C、A和G含量分别为34.8%、20.8%、26.9%和17.5%。
2.2 序列同源性分析
将所获得的29条有效序列在GenBank数据库中进行blast比对,比对结果见表2。其中鱼类19种,头足类2种,甲壳类5种,多毛类3种。共有15条序列相似度为100%,12条序列相似度为99.0%,1条序列相似度为98.8%,大于98%的序列共有28条,占所有序列的96.5%。只有长吻沙蚕(Glycerachirori)相似度为86.19%。将所有序列上传到GenBank数据库,获得相应的登录号(见表2)。
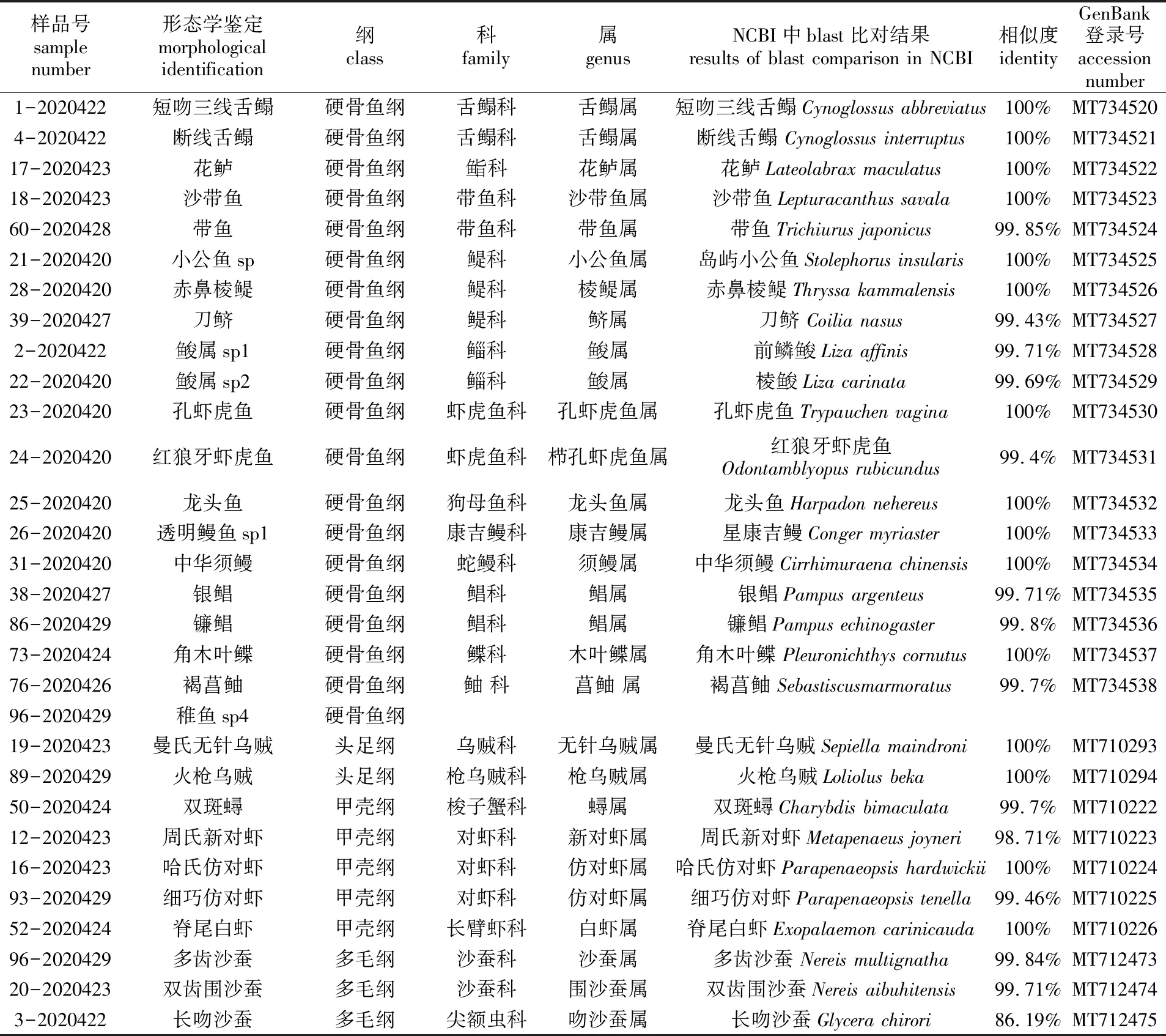
表2 样品信息
2.3 遗传距离分析
K2P双参数模型为生物条形码协会(Consortium for the Barcode of Life,CBOL) 推荐的遗传距离计算方法[24]。本研究采用K2P 双参数模型,结合GenBank 数据库检索到的29条序列,统计试验中样品的种内、种间和科间的遗传距离,结果见表3。根据K2P 模型计算各分类阶元的遗传距离,结果显示,硬骨鱼纲的种内遗传距离为0.11%,种间遗传距离为24.87%;甲壳类的种内遗传距离为0.24%,种间遗传距离为23.99%;多毛类的种内遗传距离为0.24%,种间遗传距离为29.47%。
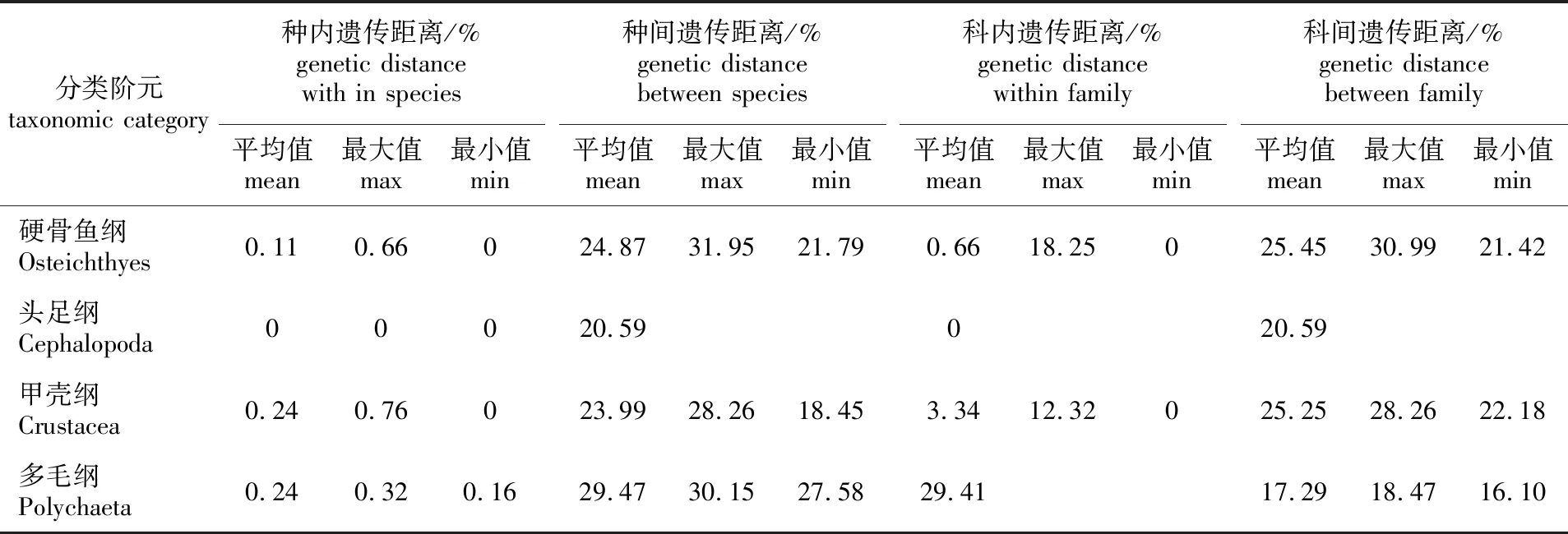
表3 各个分类级别遗传距离
2.4 系统进化分析
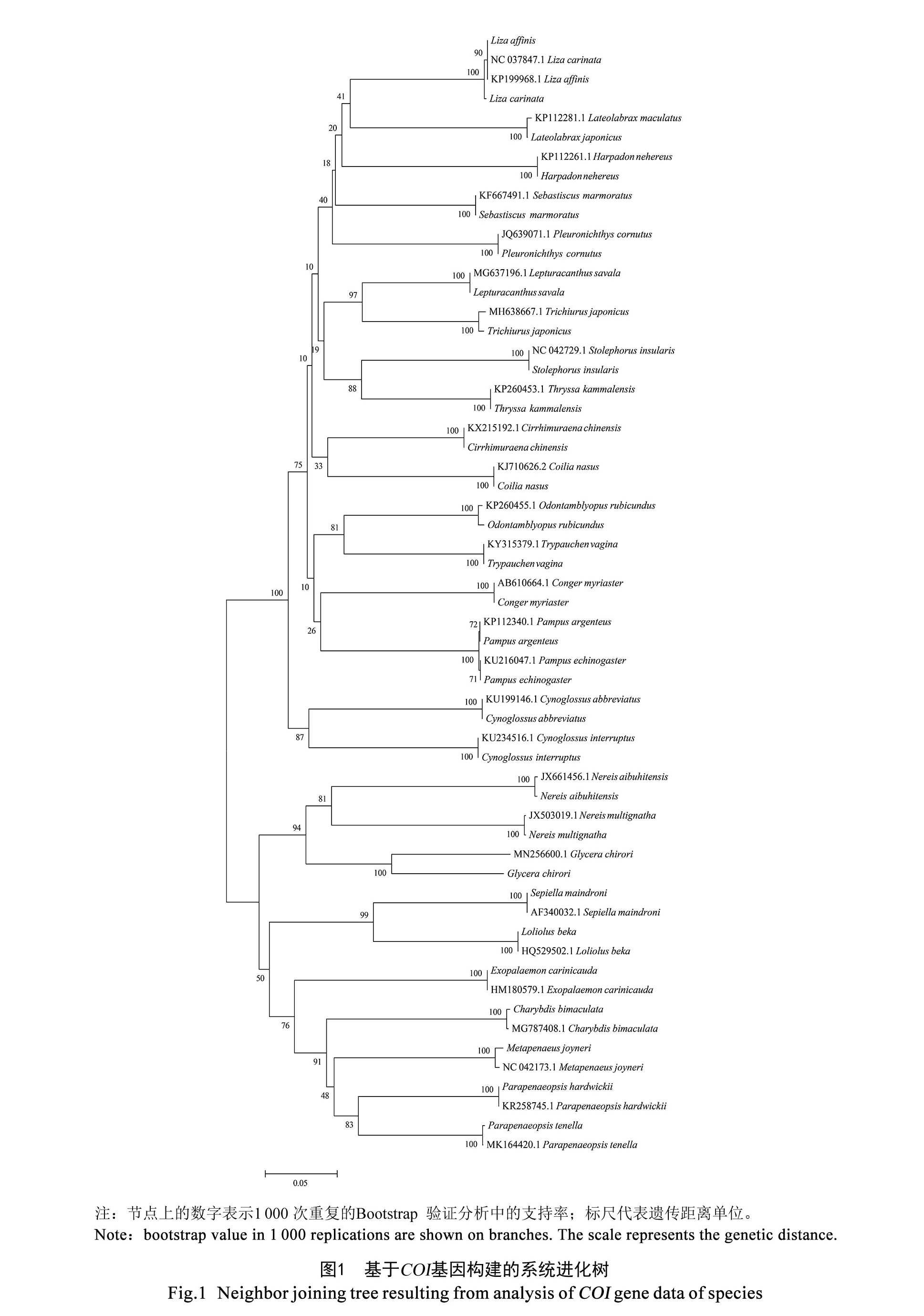
本研究构建的NJ 树由58条序列构成,在种的水平上,所有序列均与GenBank中的序列聚合,与形态学鉴定结果一致。所有种类能够聚为独立分支,同种物种均能聚合在一起,同一物种的序列聚合后,再与同一属的样品序列聚合,同属和同科的样品序列均聚合在一起,同形态分类一致。在科以上阶元中,NJ树与形态学鉴定结果不一致,各目未能完全聚合在一起,存在一定的交叉(见图1)。
3 讨论
传统的形态分类物种鉴定过度依赖于鉴定者的个人能力和经验,分类单元的表型可塑性也会加大错误识别的概率[1]。DNA条形码方法已被证明是1种有效的物种识别工具,尤其是对于损坏、不完整或由几个形态学上不同阶段组成的标本,可以使物种鉴定过程实现信息化和标准化[25]。DNA 条形码技术能够准确地鉴定形态相似种、隐存种以及处于不同生活史阶段的物种,为海洋生态调查、物种遗传进化以及环境资源保护提供数据和支持。但是,DNA条形码也有局限性,如在某些情况下,近缘物种序列相似度较大,可能会导致DNA条形码无法识别而失败。因此,DNA条形码可以作为鉴定物种的辅助工具,但不能替代形态分类学分析[26]。本研究中,通过DNA条形码鉴定,所鉴定的小公鱼sp与岛屿小公鱼(Stolephorusinsularis)的相似度为100%。但是根据《台湾鱼类志》[27]记载,岛屿小公鱼(Stolephorusinsularis)分布于印度-太平洋热带海域,包括我国台湾南、北、东北及西部近海,在我国东海未有记载。本次利用DNA条形码鉴定出的岛屿小公鱼(Stolephorusinsularis)值得探讨,遗憾的是由于样本个体过小,无法对其进行形态学的深入比较。出现该结果的原因可能有以下2个方面:一是因待测样品同时与GenBank数据库中其他物种序列相似度相同,即种间界限不明显,DNA片段难以将物种间的遗传距离拉开,因而无法做出判定,需要借助其他序列辅助来综合判定。利用DNA 条形码技术虽然可以快速鉴别物种,但这项技术还需要结合传统的形态鉴定方法才能获得准确的种类序列,从而为DNA 条形码鉴别工作累积有效的资料;另一方面,GenBank 是1个开放的共享数据库,数据资料虽然丰富且庞大,但是其中的基因序列质量良莠不齐,上传者在进行形态鉴定时也可能存在偏差,导致比对的序列同源性存在偏差。为了避免此情况,在此基础上应该使用其他的条形码进行辅助鉴定。
Hebert等[5]最早提出DNA条形码的定义,以COI基因部分DNA序列作为物种分类指标,种内遗传距离一定要小于种间距离,种间遗传距离在2%以上,且是种内遗传距离的10 倍以上。本次研究中,硬骨鱼纲的种内遗传距离为0.11%,种间遗传距离为24.87%,种间遗传距离约为种内遗传距离的226倍;甲壳类的种内遗传距离为0.24%,种间遗传距离为23.99%,种间遗传距离约为种内遗传距离的100倍;多毛类的种内遗传距离为0.24%,种间遗传距离为29.47%,种间遗传距离约为种内遗传距离的122倍。以上结果表明以COI基因进行游泳生物鉴定是可行的。在构建的系统进化树中,所有物种都能聚为独立分支,且以属、科作为分类单元均能在进化树上成功聚类,说明DNA条形码技术能成功应用于游泳生物的鉴定。
本研究遵循:相似度>98%的序列为同一种类,90%~98%为同一属,80%~89%为同一科的标准对洞头海域游泳生物进行种类判定[1]。所获物种获得的COI基因的DNA片段与相应基因的标准DNA条形码进行序列同源性比对,得到的比对结果相较于传统的形态学鉴定更为精准。本研究根据DNA 条形码的鉴定结果,共鉴定出了28种游泳生物,其中鱼类19种,头足类2种,甲壳类5种,多毛类1种,序列比对的相似度均大于98%,条形码鉴定和形态学判别结果互相吻合,因而可精准判别样品所属的物种,与预期结果一致。结果表明,该方法稳定、精准、易于操作,可应用于物种鉴定,值得推广。
本研究中还存在一些不足的地方需要改进。例如有样品的基因组DNA提取失败,导致目的基因扩增失败,无法进行后续实验,因此未能获得该物种的序列。该样品在捕获时未能及时进行冰冻保存,在鉴定时出现反复冻溶的情况,可能因此导致基因组DNA降解严重。此外,为了避免肠道内其他生物对实验的影响,一般只取背部肌肉来提取基因组DNA,由于一些稚鱼个体较小,其肌肉组织非常少,提取过程中稍有操作不当,即导致样品基因组DNA提取失败,未能获得有效的基因组DNA。在今后的研究中需对操作进行优化,以提高实验的检出率。
本研究基于COI基因的DNA条形码分析能够识别洞头海域的的大部分游泳生物,其鉴定结果与形态鉴定结果相吻合。DNA条形码技术已成功应用于识别其他地理区域中的海洋鱼类[1,28]。本研究建立了一个较可靠的洞头海域的游泳生物DNA条形码参考文库,这将有助于更好地进行该地区的渔业监测、海洋生物资源保护和管理。
4 结论
基于COI基因的DNA 条形码技术将随机选取的29个样品中的28个样品准确鉴定到种,表明COI基因序列适用于温州海域游泳生物物种的分类鉴定。在形态学分类资料缺乏或样品形态特征缺失的情况下,DNA 条形码技术可以作为游泳生物鉴定的一种便捷有效的方式,同时可以用来对游泳生物形态学分类系统进行补充和修订。
由于COI基因的突变速率相对于线粒体DNA 控制区低,在鉴定分化程度较低的物种(如圆属)时,利用COI序列无法准确鉴定到种。为了更好地发挥DNA 条形码技术在物种鉴定中的作用,DNA 条形码数据库在数量和种类上亟需完善,以使物种鉴定更为精确。

